基于TCGA和ICGC数据库构建肾透明细胞癌的免疫相关预后模型*
2022-03-05梁德宏王艳张全颖陈燃谢朝阳罗海清李祥勇
梁德宏,王艳,张全颖,陈燃,谢朝阳,罗海清,李祥勇
(1.广东医科大学生物化学与分子生物学教研室/广东省教育厅医学生物活性分子研究重点实验室,广东湛江524023;2.广东医科大学医学技术学院,广东东莞523808;3.广东医科大学附属医院肿瘤医院,广东湛江524001)
肾透明细胞癌(kidney clear cell carcinoma,KIRC)起源于肾小管上皮,是肾细胞癌的主要组织病理亚型,具有更高的肿瘤复发率和转移率[1]。早期KIRC可通过手术或消融策略成功治愈,但晚期、转移性KIRC患者的总体预后仍然较差[2]。随着免疫治疗的不断发展,其应用于KIRC的临床收益获得持续提升,但大多数 KIRC患者仍没有获得持久的疗效收益,甚至出现免疫相关不良事件,这主要归因于肿瘤免疫微环境(tumor microenvironment, TME)的异质性和复杂性[3-4]。研究表明,具有强免疫原性的KIRC,其预后模型主要基于病理分期等常规临床指标,为最大化免疫治疗的临床获益,有必要进一步探索KIRC免疫相关生物标志物和TME的特征[5]。因此,在本研究中,笔者拟通过生物信息学的方法筛选出免疫相关的差异表达基因,以此构建和验证KIRC免疫相关预后模型,分析TME特征,以期为KIRC预后管理和免疫治疗决策的制定提供重要证据。
1 材料与方法
1.1数据获取 基于UCSC Xena平台( https://xenabrowser.net/datapages/),下载源自癌症基因组图谱(The Cancer Genome Atlas, TCGA)的KIRC队列(593例样本)和国际癌症基因组联盟(International Cancer Genome Consortium,ICGC)的KIRC队列(158例样本)的mRNA表达数据以及对应患者的临床信息,包括人口统计学指标(年龄、性别等)、预后数据(生存时间、死亡状态等)和病理参数等。其中,TCGA的KIRC数据集作为目标数据集,ICGC的KIRC数据集作为验证数据集。
1.2筛选免疫相关的差异表达基因 基于R包limma,对目标数据集进行差异表达基因(differentially expressed genes,DEGs)分析,筛选标准为矫正后P值小于0.05和差异倍数|Fold Change|大于1.5,即被视为DEGs。将得到的DEGs与从免疫学数据库和分析平台(Immunology Database and Analysis Portal,ImmPort)数据库(https://immport.niaid.nih.gov/)获取的参考免疫相关基因集取交集,得到免疫相关的DEGs,用于后续分析。
1.3构建免疫相关预后风险模型 基于R包Survival,对免疫相关DEGs与患者总生存时间(overall survival,OS)进行单因子回归分析,筛选显著关联(P<0.05)的基因作为候选预后因子。接着,通过LASSO cox回归分析,进一步筛选出重要的预后相关基因及其回归系数,以此建立KIRC预后风险评估模型(风险分数=∑差异基因的回归系数λi×归一化处理后的基因表达量βi),并利用R包survivalROC绘制ROC曲线来评估预后模型的性能。以真阳性率与假阳性率之间的差值最大处作为最佳风险评分临界值,将肿瘤样本分为高风险组和低风险组,通过R包SUR-Miner 绘制两组的生存曲线。
1.4肿瘤微环境的评估分析 采用CIBERSORT算法分析22种肿瘤浸润免疫细胞在高风险组和低风险组的比例,以此评估和比较肿瘤免疫浸润细胞类型的构成和组间差异(通过非配对t检验进行)[6]。
1.5构建和验证列线图 基于R包rms,结合临床信息(年龄、性别和肿瘤分期),对预后风险评分模型进行列线图模型构建,以此分析其在不同患者临床特征的预后价值。临床信息、风险评分与OS之间的关系通过森林图形式展示,其中,一致性指数(C-index)表明列线图的预测准确性。
1.6统计学分析 采用R软件对数据进行处理和统计分析,R包survivalROC绘制ROC曲线并评估预后模型的性能,Wilcoxon检验用于检验两组间的差异,生存分析曲线的组间差异采用Log-Rank检验。以P<0.05为差异具有统计学意义。
2 结果
2.1筛选关联预后的免疫相关DEGs 纳入TCGA的KIRC队列为目标数据集,共计593个样本(表1),对肿瘤组织样本和癌旁组织样本的mRNA表达谱进行分析,共发现694个DEGs,其中326个基因表达上调(图1A)。将所得DEGs与ImmPort数据库的参考免疫基因集(1 509个)取交集后,筛选出57个免疫相关的DEGs(图1B),进一步通过单因子回归和LASSO cox回归分析,得到11个与OS显著相关的免疫相关DEGs(P<0.05,表2),以及各个基因对应的回归系数(图1C)。NOS1、ANGPTL3和AVPR1B基因高表达与良好的预后显著相关,而其他8个基因则相反。根据多因素回归分析模型,Akaik信息标准(AIC)为705.42,C指数为0.71。值得注意的是,仅ANGPTL3与OS显著相关(P=0.035),其风险率为0.89(95%CI:0.80~099),见图1D。
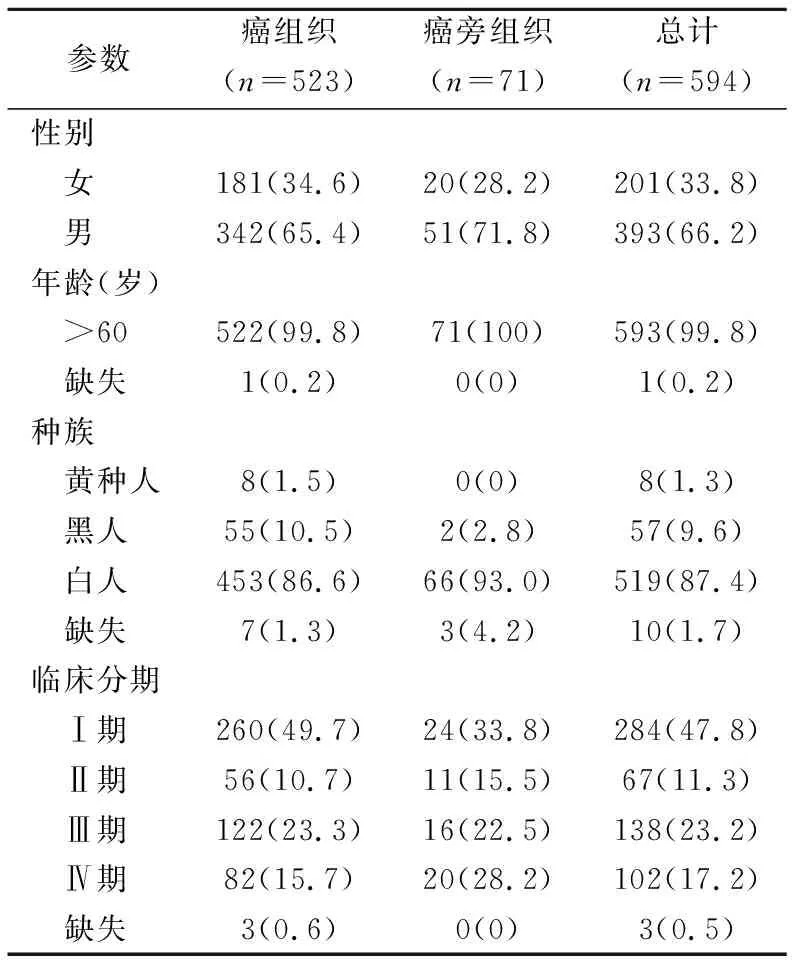
表1 TCGA KIRC数据集的样本信息[n(%)]
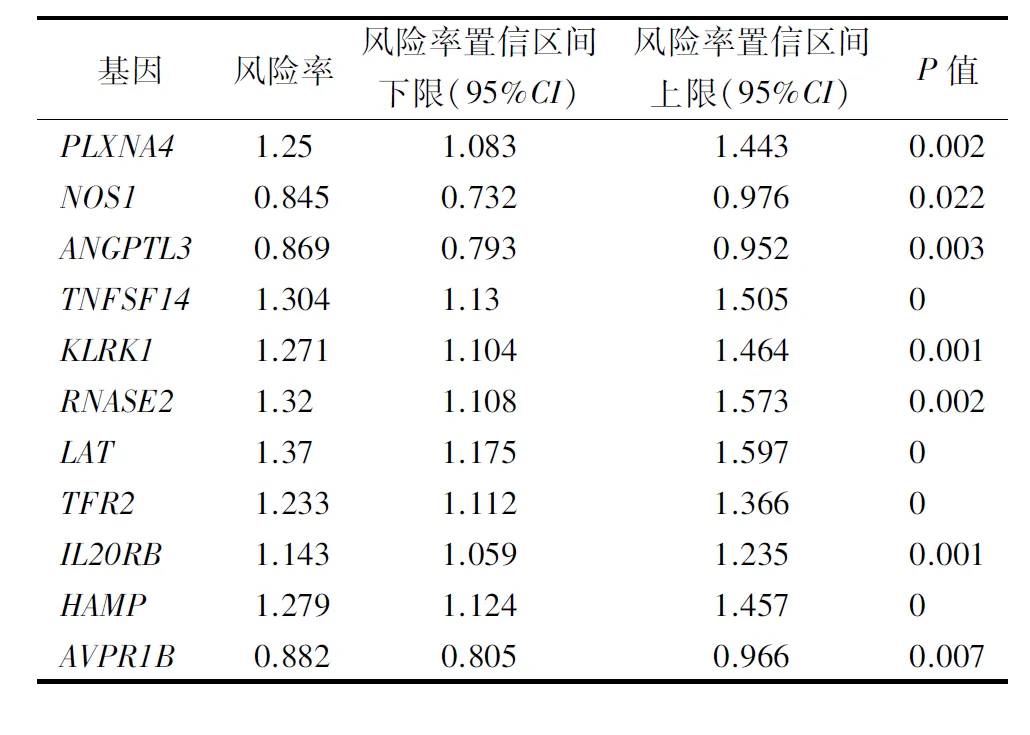
表2 与KIRC预后相关的基因列表(单因素回归分析)
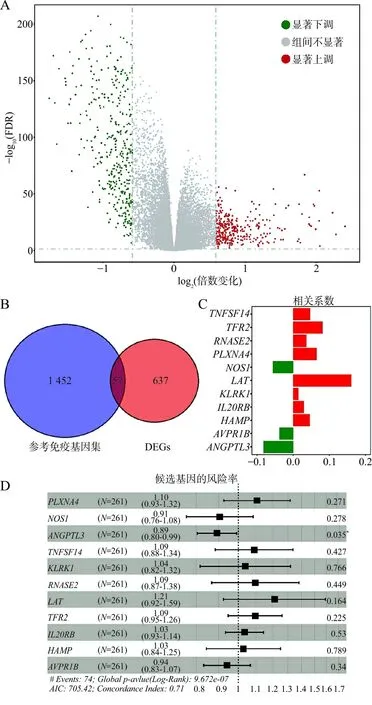
注:A,TCGA KIRC数据集的差异基因分析,横坐标为差异倍数的对数值(以log2为底),纵坐标为校正后P值的对数值(以 log10为底)的相反数;B,差异表达基因(DEGs)和ImmPort参考免疫基因集的交集分析;C,基因的回归系数柱状图;纵坐标为基因,横坐标为回归系数;D,多因素回归分析的森林图;纵坐标为风险因子(基因),横坐标为风险因子的风险率。
2.2免疫相关预后模型的搭建 基于11个显著与OS相关的免疫相关DEGs及其回归系数构建的免疫相关预后风险模型,将风险得分的阈值设为-0.02,以此将所有KIRC样本分为高风险组和低风险组(图2A)。结合预后数据分析,发现高风险组的死亡比例显著高于低风险组,提示风险评分对评估肾透明细胞癌患者生存状况具有一定的应用价值(图2B)。值得注意的是,相比高风险组,低风险组具有更高的总生存时间(P<0.001),表现出NOS1、ANGPTL3和AVPR1B表达量上调,而其他8个基因表达量下调(图2C~D)。
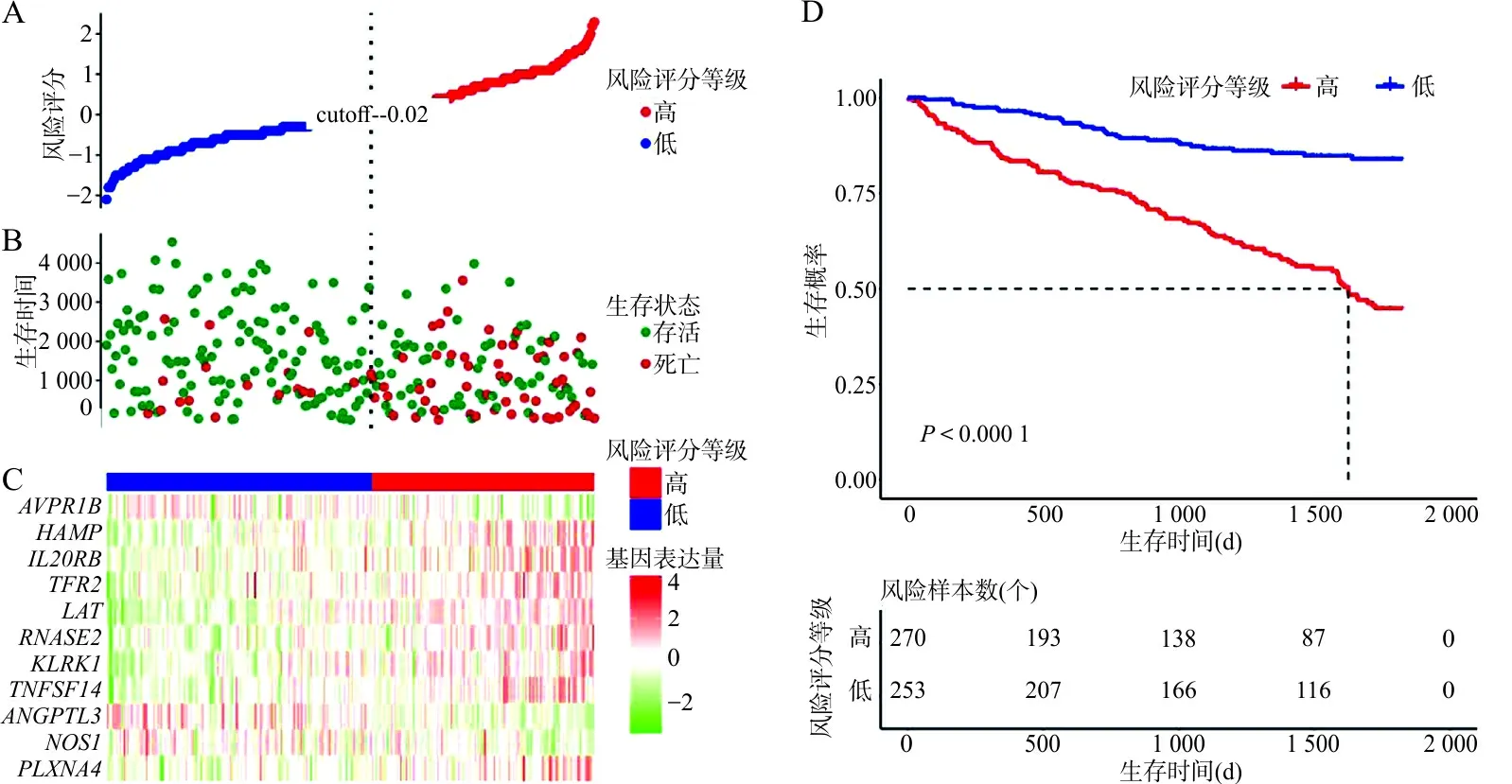
注:A,高风险组和低风险组的风险曲线,纵坐标代表风险评分,横坐标代表风险评分等级;B,生存状况分布,纵坐标代表生存时间(d),横坐标代表风险评分等级;C,11个免疫相关DEGs在高风险组和低风险组的比较;D,总生存时间在高风险组和低风险组的比较。
2.3免疫相关预后模型的性能评估和验证 绘制用于评估免疫相关预后风险模型性能的ROC曲线,发现该模型在评估3年、4年、5年生存概率的AUCROC分别为0.669、0.707和0.750(图3A),且风险评分在KIRC不同肿瘤分期中表现出显著的差异,倾向于临床分期越高,则风险评分越高(图3B)。同时,基于ICGC的KIRC队列数据集,选择其中具有mRNA表达数据和预后信息的肿瘤样本共158例,以此作为预后模型的验证集,其中男性100例,女性58例,年龄大于60岁共101例,小于60岁共57例。结果显示,在验证集中,该免疫相关预后风险模型在评估3年、4年、5年生存概率的AUCROC分别为0.628、0.653和0.677(图3C),高风险评分与较短生存期显著相关(P<0.000 1),见图3D。

注:A,基于目标数据集搭建预后模型用于预测3/4/5年生存率的ROC曲线;B,目标数据集中不同临床分期的肿瘤样本的风险评分比较;C,预后模型在验证集中表现出来的预测3/4/5年生存率的ROC曲线;D,基于预后模型给验证集样本计算的风险评分的生存分析曲线。
经过多因素回归模型分析,发现年龄(≥60岁)、肿瘤分期(Ⅲ和Ⅳ期)和基于免疫相关预后模型得到的风险评分均与OS显著相关,风险评分与肿瘤Ⅲ期的风险率相当,且可通过列线图估算3年、4年和5年的生存概率(图4A~B)。通过绘制校准曲线验证列线图的性能,可以看出预测曲线接近理想曲线(图4C),表明构建的列线图模型性能良好,其预测准确性较先前的免疫相关预后模型得到进一步提高(C指数:0.78 vs 0.71)。值得注意的是,风险评分对生存时间的风险率(1.93,95%CI:1.59~2.3)比任一单个免疫相关DEGs都高。
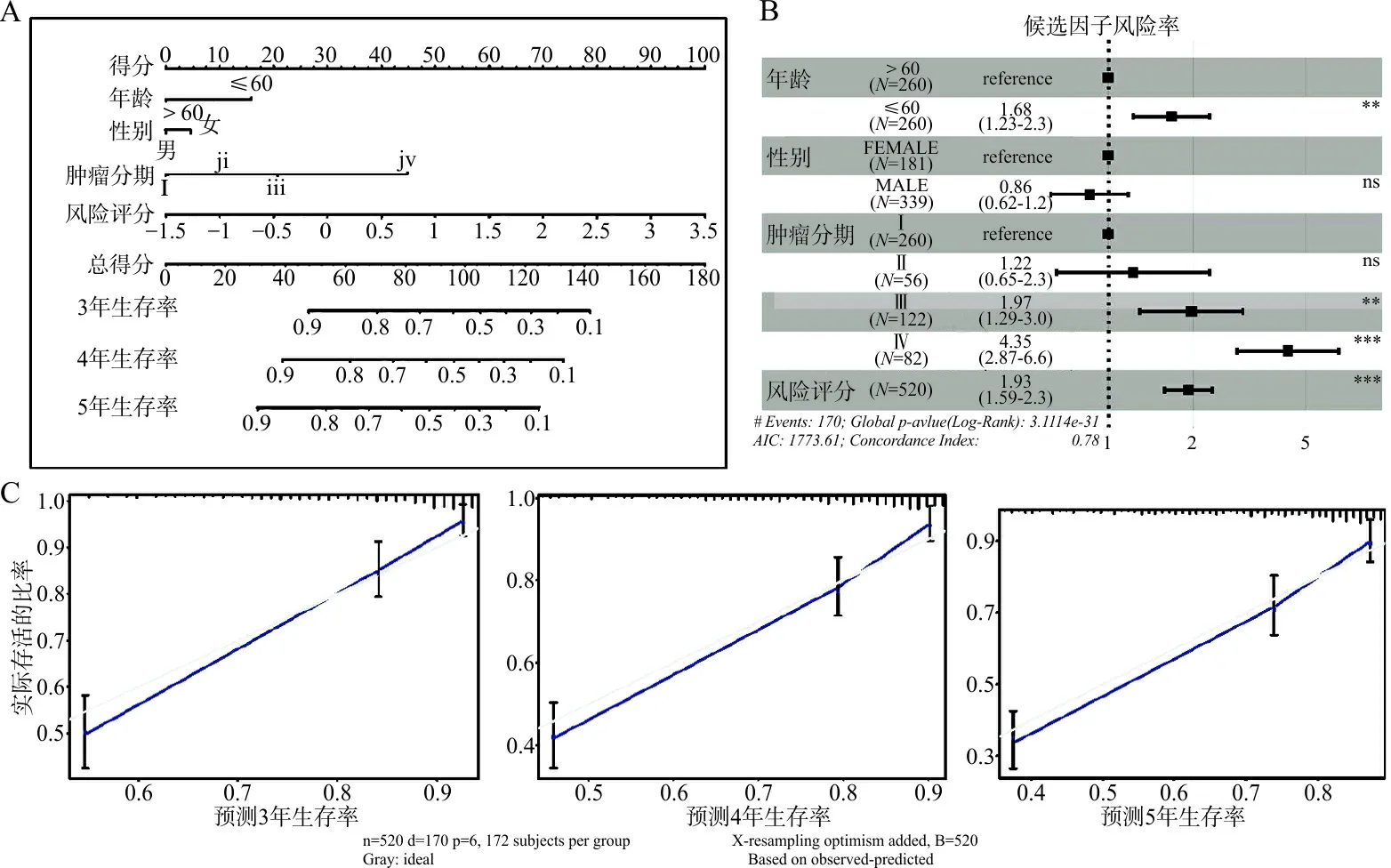
注:A,基于目标数据集,将患者年龄、性别、病理分期、风险评分分别作为变量预测患者3年、4年和5年生存率的列线图;B,多因素回归分析的森林图;C,分别针对3年、4年和5年的预后模型绘制的校准曲线,灰色直线代表理想状态,蓝色折线代表实际状态。
2.4探索KIRC肿瘤免疫微环境 基于mRNA表达谱,通过CIBERSORT算法来估算每例KIRC样本中22种免疫细胞的比例,结果显示,12种免疫细胞类型在高风险组和低风险组间的占比差异具有统计学意义(P<0.05),其中,浆细胞(plasma cell)、调节T细胞(regulatory T cell,Tregs)、单核细胞(Monocyte)等6种细胞的组间差异最为显著(P<0.000 1,图5A)。值得注意的是,在高风险组中表现为更高浸润程度的调节T细胞、M0型巨噬细胞(macrophages M0)、活化的记忆CD4+T细胞(T cells CD4 memory activated)与较差的预后相关,同时,在高风险组中浸润程度更低的休眠树突状细胞(dendritic cells resting)则与较好的预后相关(图5B~E)。
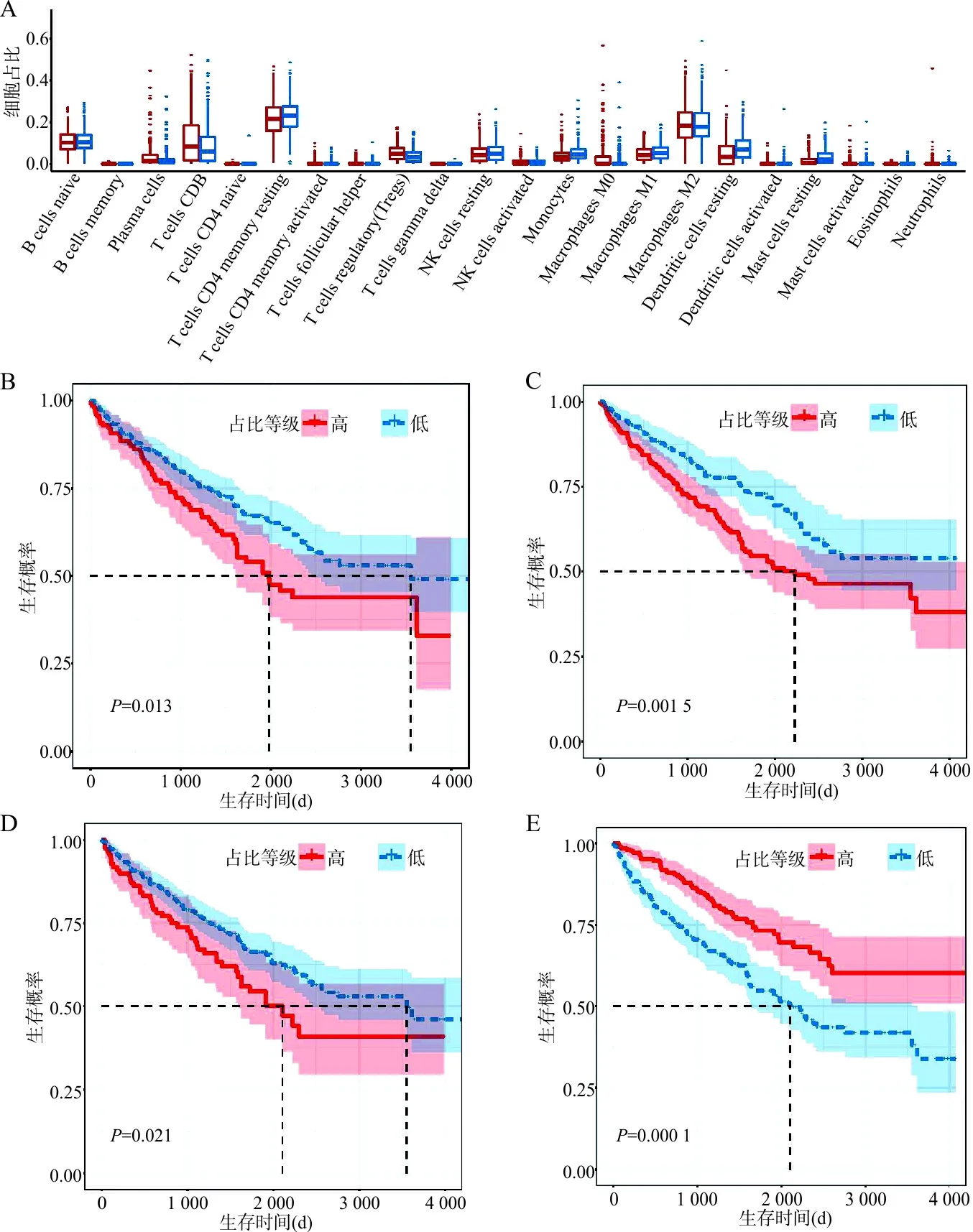
注:A,高风险组(蓝色)和低风险组(红色)免疫细胞浸润程度的比较,纵坐标为细胞比例,横坐标为免疫细胞类型;B,M0型巨噬细胞浸润程度高低在生存时间上的比较分析;C,调节T细胞浸润程度高低在生存时间上的比较分析;D,活化的记忆CD4+T细胞浸润程度高低在生存时间上的比较分析;E,休眠树突状细胞浸润程度高低在生存时间上的比较分析。
此外,进一步对高风险组和低风险组间免疫检查点及其配体的表达水平进行分析,结果表明,相比于低风险组,高风险组中PD-1、CTLA-4和LAG3的表达水平显著上调(P<0.000 1),而PD-L1显著下降(图6A~D)。
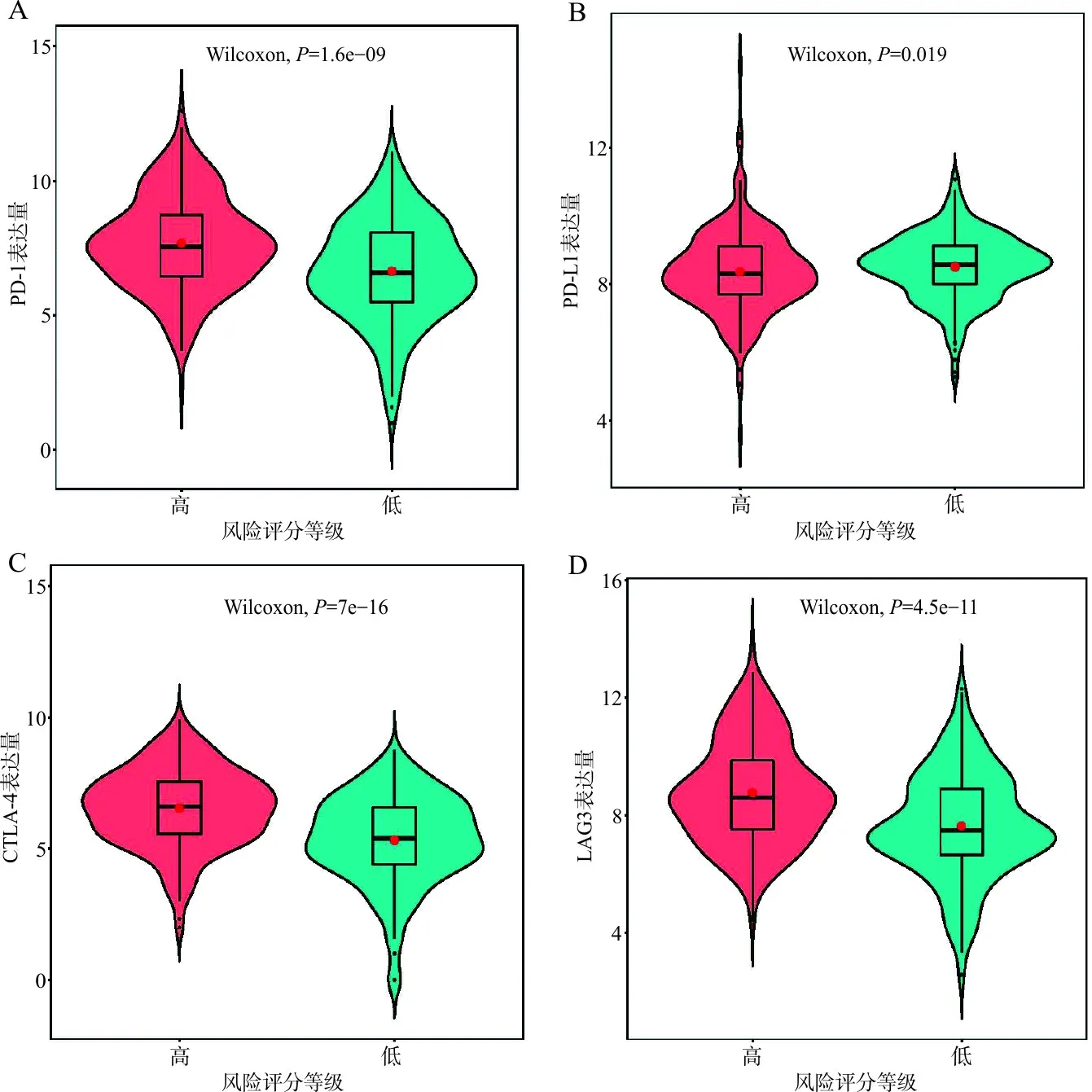
注:A,PD-1的表达量与不同风险评分等级的比较分析;B,PD-L1的表达量与不同风险评分等级的比较分析:C,CTLA-4的表达量与不同风险评分等级的比较分析;D,LAG3的表达量与不同风险评分等级的比较分析。
3 讨论
为探索免疫相关分子标志物在KIRC预后中的预测价值,笔者从TCGA的KIRC数据集中筛选出11个显著关联KIRC患者总生存时间的免疫相关DEGs。其中包括鲜有被报道的ANGPTL3,在本研究中无论单因素还是多因素回归分析结果,均表现出与KIRC预后显著的关联性,且其高表达被证实可抑制肿瘤的增殖和转移,进而关联更好的预后[7-8]。此外,单因素回归分析结果显示,NOS1和AVPR1B基因的高表达均与良好的预后相关,但在多因素回归分析中不存在相关性,表明这2个基因对KIRC预后的预测价值仍需进一步探索。
鉴于单个免疫相关DEGs对预后结果的影响程度有限,笔者以这11个免疫相关DEGs构建了预后模型,并通过ICGC的KIRC数据集进行验证。结果显示,基于该模型得到的风险评分越高,肿瘤分期等级越高,生存时间越短,且风险评分在评估3年、4年、5年生存概率上也表现出一定的预测性能。同时,风险评分对KIRC预后的影响程度相比于单个免疫相关DEGs更强(风险率:1.93 vs 1.37),且与肿瘤分期Ⅲ期相当(风险率:1.93 vs 1.97),这进一步支持该模型的临床应用潜力。
鉴于KIRC中TME异质性和复杂性可影响免疫治疗的临床获益,故而笔者进一步分析了风险评分与TME的关联。结果表明,组间显著差异的免疫细胞类型高达12种,其中,浆细胞、调节T细胞和M0型巨噬细胞在高风险组中表现出更高的浸润程度,且均与较差的预后相关。其中,调节T细胞被认为是KIRC中有效抗肿瘤免疫的主要障碍,可通过负调节肿瘤内部或外部的免疫和非免疫细胞,限制抗肿瘤免疫反应,进而不利于KIRC患者的预后结果[9]。同时,相比低风险组,在高风险组中,浸润程度更高的活化的记忆CD4+T细胞和M0型巨噬细胞与较差的预后相关,而浸润程度较低的休眠树突状细胞,则与较好的预后相关,这与先前的研究结论相一致[10]。此外,笔者还分析了免疫检查点及其配体的表达情况,发现高风险组中PD-1、CTLA-4和LAG3显著上调,而PD-L1显著下调。以上结果表明与风险评分相关联的调节T细胞、休眠树突状细胞、记忆CD4+T细胞、M0型巨噬细胞和免疫检查点可能是KIRC的独立预后因素,有望为预后管理和免疫治疗提供证据。
本研究存在以下局限性,一是研究数据均源自TCGA数据库,样本来源以白人为主,该结果是否适用于黄种人尚需验证;二是研究数据的来源人群以60岁以上的男性为主,或许仅限这小部分人群可得到结果复现和应用;三是该模型的应用价值还需要进一步的临床研究验证。
综上所述,本研究聚焦于KIRC患者群体,基于11个关联OS的免疫相关DEGs构建和验证了1个性能良好的KIRC免疫相关预后风险模型,并在此基础上,探索不同风险评分下的TME特征,有望为KIRC患者的临床风险分层和免疫治疗决策提供支持。
