基于临床表型的致病基因排序方法的模拟分析及验证
2022-03-05许熠腾靳光付
许熠腾,靳光付
(1.南京医科大学公共卫生学院,南京 211166;2.博圣医学检验实验室,杭州 310000)
外显子测序(exome sequencing,ES)是目前遗传检测领域最常用方法之一,该方法只需对全基因组序列中1%~2%的区域进行测序,就可以检测出85%的基因致病性突变[1]。ES测序结果相对于传统的核型分析、染色体微阵列分析或一代测序等方法在提供更大数据量的同时,也增加了数据判读与临床表型准确关联的难度。为了更好地利用临床表型进行辅助诊断,遗传学者以及信息学者共同开发出了基于语义相似性框架——人类表型本体(human phenotype ontology, HPO)[2-4]的致病基因筛选方法,其将患者的临床特征与HPO数据库中收录的标准化疾病表型进行匹配,通过计算信息量(information content,IC)为2个表型之间的相似性进行打分,继而为候选基因与给定表型谱之间的匹配程度进行排序,以此辅助诊断未知原因的遗传缺陷。在被广泛认可的基于信息量的方法——Resnik[5]中,特异性较高的表型间相似性应高于特异性较低表型间相似性的特征未得到体现,其他基于Resnik的改进计算方法(如Lin[4],JC[5],Rel[6]等)在既往研究中的排序准确率低于Resnik。Gong等[7]的方法则直接定义1个临床观测到的表型对疑似疾病的贡献值,最后累加所有贡献值并计算临床观察到的表型集与相关疾病(或基因)的关联。该方法不计算表型间的关联性,直接排除了所有不能精准匹配上的疑似表型所关联的疾病,因此在临床应用中可能导致敏感性降低。本研究开发了一种基于拓扑结构的度量方法,该方法与原有的几种疾病—基因关联方法相比具有较高的敏感性。报道如下。
1 材料与方法
1.1测试数据来源 采用常规方法[7-9]生成与41种畸形综合征相关的3 075例模拟病患作为测试数据。并通过本地的ES测序数据以及相关患者表型集作为真实的测试数据集进行验证。
1.1.1模拟患者数据集 HPO表型与疾病(基因)关联数据库下载自其网站(https://hpo.jax.org//)。41种畸形综合征以及其相对应的HPO表型发生频率列表下载自Genecards[10]网站(https://www.genecards.org//)。
为了给每种畸形综合征生成25位模拟患者,首先对每个疾病关联表型生成1个介于 0 和 100 之间的随机整数,如果该数字小于100例患者的相对发生率(频率*100),则保留相应的表型;生成的随机数小于相对发生率的表型则剔除该名模拟患者的表型集。最终生成的初始数据集1共包含1 025例模拟患者以及其对应的模拟表型集的数据集1(无噪声数据,无不准确数据)。随之,将与潜在疾病无关的表型(数量为初始数据集中各样本的模拟表型数量的一半)添加到初始表型集数据中来生成具有“噪声”的模拟患者数据集2(有噪声数据,无不准确数据)。最后,通过将数据集1中的每个表型随机替换成其的先祖表型以生成具有“不准确性”的数据集,并加入数据集2中生成的噪声表型,来获取同时具有不准确性和噪声表型组的数据集3(有噪声数据,有不准确数据),也是模拟数据集中最贴近临床诊断环境的模拟患者数据集。
1.1.2真实患者数据集 选取385例经临床医师诊断的南京地区患者样本作为后续的测试数据,数据来自博圣医学检验室的ES测序数据,采样时间为2021年9月至2022年3月。所有样本均经过临床医师诊断并确诊相关疾病。其中产前样本134例,超声及影像学检验报告异常后送检;产后样本中男性136例,女性患者115例,年龄为0~54岁,中位年龄6岁,经临床医师诊断疑似患有遗传性疾病后送检。各送检单位在检测前均签署知情同意书。测序数据根据美国医学遗传学和基因组学学会(the American College of Medical Genetics and Genomics, ACMG)指南[11],构建质量和人群频率阈值、致病性规则,过滤掉低质量、高频率和良性变异,得到可用于医学遗传分析的变异结果。患者所患疾病包括骨骼系统疾病、泌尿系统疾病和眼科疾病等单基因疾病。
1.2方法
1.2.1表型间相似性计算方法 HPO提供人类疾病中用于描述表型异常的标准词汇,以及其所对应疾病数据,来源于分类学方法对人类孟德尔遗传数据库(Online Mendelian Inheritance in Man, OMIM)[12]中的表型进行的文本挖掘和归纳。HPO中的表型依据相互之间的关系被组织为有向无环图,以描述表型特征及相互之间的联系。见图1。
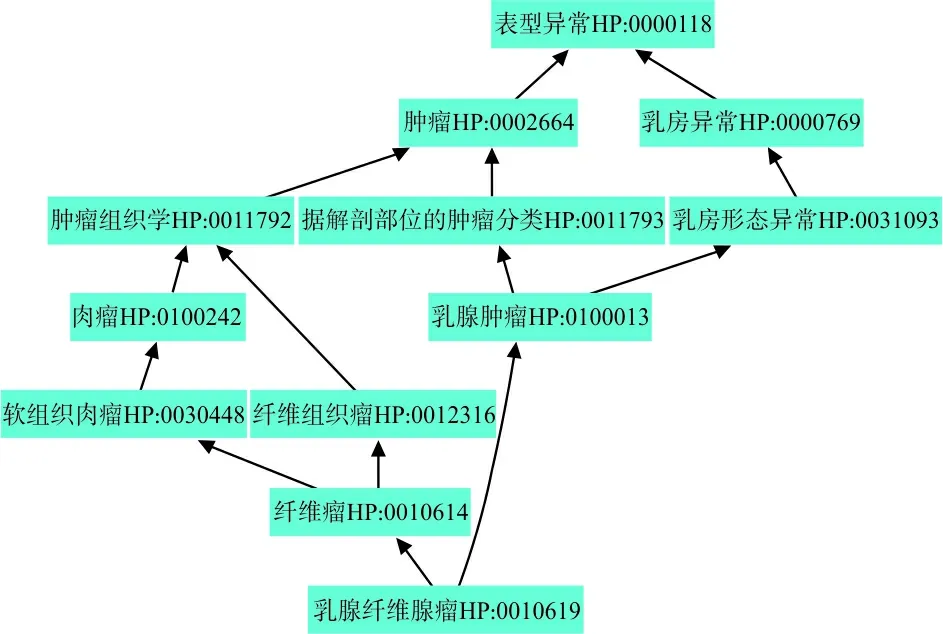
注:表型之间的关系由分类学中的概念is_a(is ancestor)定义,即箭头所指的表型为箭头另一端表型的先祖表型。父表型比子表型更具有通用性,而子表型比父表型更具有特异性[13]。
本研究提出的新方法Depth对任意表型之间相似性度量值计算公式如下:
在上述公式中,root为HP:0000001,ta、tb为任意2个HPO表型,dist(ta,tb)表示ta,tb2个表型到其任意共同先祖的最短距离之和的最小值,也即是将HPO的有向无环图转换为无向图后,ta,tb2个表型之间的最短距离。
1.2.2表型集之间相似性计算方法 本方法选择使用效果相对较好的单边搜索算法获取临床观察到的患者表型集与相关疾病(基因)所对应的表型谱之间的相似性,公式:
simone-sided(Q→D)=avg[∑t1∈Qmaxt2∈Dsim(t1,t2)]
(2)
其中Q是要查询的HPO表型集(观察到的患者表型),D是注释到给定疾病的HPO表型集,sim(ta,tb) 即为公式1中求得ta,tb的相似性。
1.3统计学分析 使用Python的random包生成模拟测试数据。通过Matplotlib绘制折线图进行图画化的性能分析。Depth和其他已有方法的性能比对则通过对患者表型集与每种候选疾病之间的相似性度量值排序,统计患者对应疾病排名在每种方法的候选疾病列表中前二十位以内的患者数量,该数字越大则对应方法的敏感性越高。在难以直接通过图表直观确定方法的优劣时,采用Python包scipy进行Wilcoxon秩和单边检验以验证不同方法之间的性能优劣。
2 结果
2.1模拟患者的检验结果 本研究将Depth和其他5种已有方法分别计算患者表型集与每种候选疾病之间的相似性,然后根据它们的相似性度量值对所有候选疾病进行降序排列。真实疾病的排名越靠前,算法的性能就越好。在每个模拟数据集中,统计患者对应疾病排名在每种方法的候选疾病列表中前二十位以内的患者数量,该数字越大则对应方法的敏感性越高。3个模拟数据集中的比较结果见表1及图2。
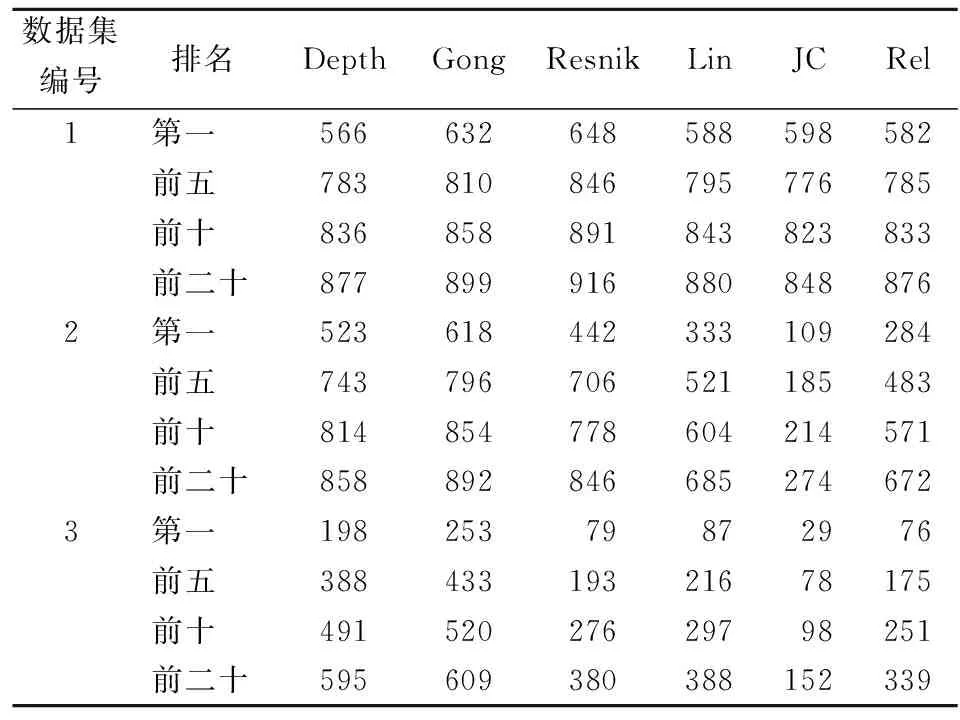
表1 6种方法在3个模拟数据集中的排序结果汇总
在数据集1中,Depth的敏感性低于Resnik和Gong的方法,与剩余3种方法相似。见图2A。在数据集2中,Depth的敏感性仅次于Gong,高于其他4种方法。见图2B。在同时包含噪声数据以及不准确数据的数据集3中,Depth在模拟患者数据集中展示出了与Gong类似的性能,Gong的方法对应疾病排名在候选列表的第一、前五、前十、前二十位的百分比分别为24.68%、42.24%、50.73%、59.41%,Depth对应疾病排名在候选列表的第一、前五、前十、前二十位的百分比分别为19.31%、37.85%、47.90%、58.04%,而其他几种方法中最常使用的Resnik对应疾病排名在候选列表的第一、前五、前十、前二十位的百分比分别为7.71%、18.83%、26.92%、37.07%,新方法与包括Resnik在内的其他4种方法排序的敏感性相比均有了较大的提升。见图2C。
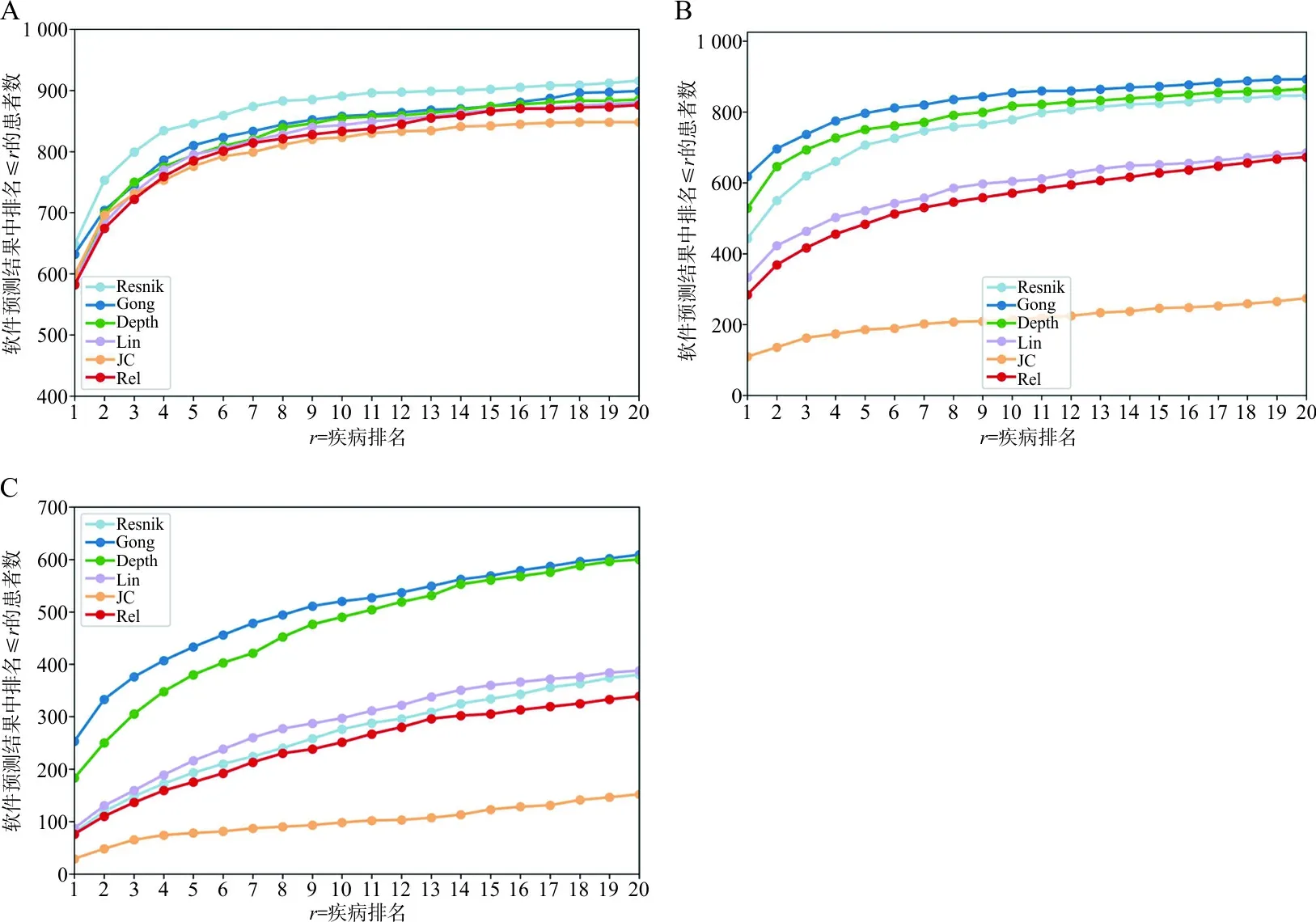
注:A,B,C分别为数据集1,2,3;横轴为模拟病例所对应的真实疾病在6种方法中的排序秩次,纵轴为排序结果小于对应秩次的患者数。
2.2385例本地数据结果 本地测序数据集上的比较结果见表2及图3。Depth对应致病基因排名在候选列表的第一、前五、前十、前二十位的百分比分别为41.56%、51.43%、58.18%、64.68%,而这些数据在Gong和Resnik方法生成的排序结果中则分别为37.40%、51.43%、56.88%、64.94%以及35.58%、45.94%、53.52%、60%。分别将排名前二十位的患者数量进行单边Wilcoxon秩和检验,结果表明Depth的排序敏感性优于Resnik方法和Gong方法(P<0.001)。
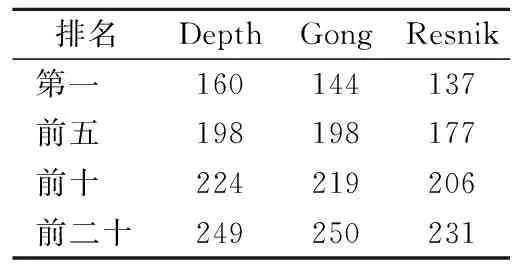
表2 3种方法在真实患者数据集上的排序结果汇总
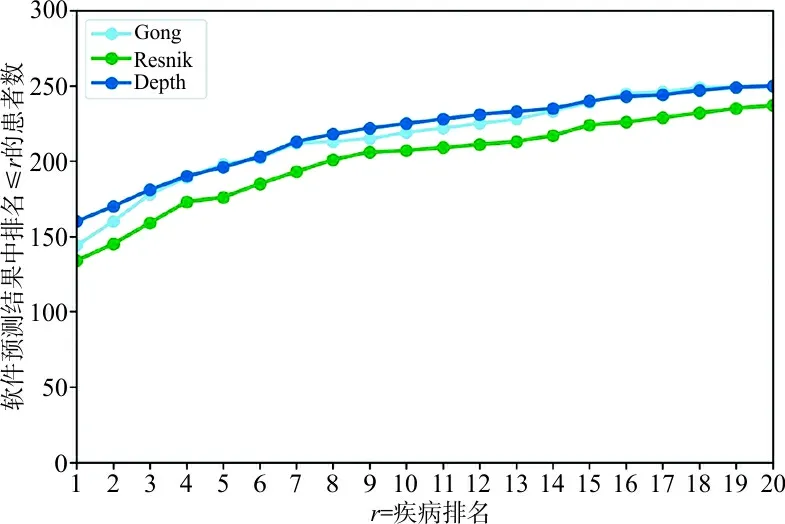
注:横轴为本地数据集上3种方法的排序秩次,纵轴为排序结果小于对应秩次的患者数。
3 讨论
近年来,HPO已被广泛应用到各种表型相关的领域中。Phenomizer的Web应用程序提供基于HPO的本体相似性搜索,以帮助临床医生识别罕见病病因[6]。PheNominal是1个嵌入在电子健康记录(electronic health record,EHR)工作流程中的即时网络应用程序,用于从非标准化文本中捕获标准化的表型数据[14]。AMELIE解析了PubMed提供的2 900万篇摘要以及数十万篇全文文章,从文献中挖掘与表型相关的疾病信息[15]。此外,有数种方法将基于HPO表型信息与基因突变的基因型数据进行结合,以进行基因变异致病性的优先级排序,这类方法包括PHIVE/Exomiser、PhenoVar和Xrare等。
本方法提出的基于HPO拓扑信息的新表型相似性度量方法,可用于识别致病基因并加速疾病诊断。依据2个表型与DAG根节点的总距离设计了正则化项,使得在表型间拓扑距离相同时,相似度计量值跟随表型与根节点的距离增加而减小,既满足了分类学中相似性度量的三大原则,又兼顾了HPO中由上而下特异性不断提高的特点。另一方面,本方法的计量值计算仅依据拓扑信息,不依赖注释到每个表型的疾病信息来计算IC值,因此在计算尚未有相关联疾病或致病基因被发现的表型间相似性时,计算出的相似性度量值比依据IC值类方法更准确。
模拟患者检验结果显示,本方法在最接近临床的数据集3(有噪声数据且有不准确数据)中的敏感性仅次于Gong的方法,高于其他4种方法;而在385例本地数据测试中,本方法的敏感性经Wilcoxon秩和检验高于其他方法。本方法的局限性有以下几个方面:(1)虽然使用了真实患者的ES测序结果作为测试数据,但样本量仍不够多,有待于更多实际临床应用的验证;(2)完全基于HPO所生成的有向无环图进行计算,因此方法的排序准确性高度依赖该图中表型分类的精确性;(3)未能结合更多的信息(蛋白质通路分析等)进行综合性判断,相似性计算结果仍依赖于表型信息。综上所述,本研究提出的新方法具有较高敏感性,增加了临床中各类先天性疾病识别的准确性,有望提高临床医生的工作效率,具有较高的临床应用潜力。
