1株利用乳酸的猪源丁酸梭菌的分离鉴定及基因组序列分析
2022-03-03胡晓冰林标声王振伟郑黎静
胡晓冰,林标声,王振伟,郑黎静
(1.黄河水利职业技术学院环境工程学院,开封 475004;2.龙岩学院生命科学学院,龙岩 364012;3.龙岩学院,预防兽医学与生物技术福建省高等学校重点实验室,龙岩 364012)
丁酸梭菌(Clostridiumbutyricum),又名丁酸菌、酪酸梭菌、丁酸梭状芽孢杆菌,为革兰氏阳性厌氧杆菌[1]。丁酸梭菌是人和动物体内肠道正常益生菌群,广泛存在于腐乳、酸奶、奶酪等食品及一些酒窖土壤中[2]。丁酸梭菌主要代谢产物是丁酸,而丁酸是促进肠道上皮组织细胞再生和修复的重要物质[3]。大量研究表明,丁酸梭菌对肠道常见致病菌幽门螺杆菌、沙门氏菌和大肠杆菌等均有良好的抑制增殖作用[4];而且丁酸梭菌在肠道中还能分泌淀粉酶、纤维素酶、糖苷酶、蛋白酶等,生产促进机体消化吸收的葡萄糖、麦芽糖等营养物质,以及部分寡糖,为其他益生菌提供营养物质[5]。因而,食用丁酸梭菌可以维持肠道优势益生菌群,促进微生态平衡。近年来,丁酸梭菌被制成颗粒、胶囊、菌液剂等保健食品,在食品领域的研究逐渐受到重视,得到较为广泛的应用[6-7]。在养殖业中,丁酸梭菌作为饲料添加剂,可以促进机体健康,提高动物生产性能和畜产品品质[8-9]。此外,制备乳酸菌-丁酸梭菌联合益生菌发酵饲料可以改善单独使用乳酸菌发酵饲料引起的畜禽肠道乳酸过多的不良反应[10]。因此,本研究以乳酸为唯一碳源和能源制备筛选培养基,从使用乳酸菌发酵饲料的健康仔猪肠道内分离能转化乳酸为丁酸的丁酸梭菌临床株,对其菌群进行16S rDNA鉴定及Illumina Miseq二代测序,揭示其种属亲缘关系和基因组相关信息,以期为深入研究该菌株的遗传多样性及丁酸梭菌益生菌制剂的推广应用提供相关背景信息。
1 材料与方法
1.1 材料
1.1.1 样品 4%乳酸菌发酵饲料饲喂的未接受任何抗生素的仔猪肠道内容物,采自福建省漳平市顺佳农牧发展有限责任公司生猪养殖场。
1.1.2 培养基 乳酸分解培养基(LADM培养基):酪蛋白10 g,酵母膏2.5 g,磷酸二氢钾0.45 g,磷酸氢二钾 0.45 g,半胱氨酸1 g,NaCl 0.9 g,氯化钙 0.09 g,硫酸镁0.09 g,生物素10 μg,血红素10 mg,钴胺素10 μg,吡哆胺150 μg,叶酸50 μg,对氨基苯甲酸30 μg,核黄素0.5 μg,硫胺素0.5 μg,刃天青1 mg,添加5 mL乳酸(0.6%左右,66 mmol)作为碳源,蒸馏水定容至1 000 mL,pH 6.0~6.5,固体培养基另添加琼脂20 g,121 ℃灭菌20 min。
梭菌增殖培养基(RCM培养基):葡萄糖5 g,牛肉膏10 g,胰蛋白胨10 g,酵母膏3 g,可溶性淀粉1 g,三水合乙酸钠3 g,氯化钠5 g,刃天青0.3 g,半胱氨酸盐酸盐0.5 g,蒸馏水定容至1 000 mL,pH 6.5~7.0,固体培养基另添加琼脂20 g,121 ℃灭菌20 min。
1.1.3 主要试剂与仪器 细菌基因组DNA提取试剂盒(DP302)购自天根生化科技(北京)有限公司;测序仪(3730XL)和PCR仪(2720 thermal cycler)购自应用生物系统公司;板式离心机(5810R)购自Eppendorf公司;凝胶成像装置(JY04S-3C)和电泳仪(JY300C Power Supply)均购自北京君意东方电泳设备有限公司;高效液相色谱仪(LC-16)购自岛津公司;厌氧手套箱(YQX-11)购自上海海向仪器设备厂。
1.2 方法
1.2.1 菌株的筛选 初筛:取采集的样品10 g,加入50 mL LADM液体培养基中,在30 ℃下震荡厌氧(培养基中加入0.1%的硫乙醇酸钠厌氧还原剂)富集培养5 d,然后按5%比例接入含LADM的厌氧管继续培养,定期监测乳酸的消耗,当乳酸可快速降解时,按10倍稀释法稀释菌液,在厌氧箱内均匀涂布于LADM固体培养基平板上,在37 ℃下厌氧培养3~5 d,挑选单一菌落,划线接种于LADM固体培养基上进行菌株纯化,直至获得单一菌株克隆后,接种于斜面厌氧管保存。
复筛:将初筛出的菌株接种到装有10 mL RCM液体培养基的厌氧管中,80 ℃下热敷10 min,用水冷却后置于37 ℃恒温培养箱中厌氧富集培养4 d,定期监测乳酸的消耗,挑选有大量气泡生成的培养液,进一步在RCM 固体培养基中进行富集、传代,筛选利用乳酸快、丁酸转化率高的为目标菌株,进行下一步研究。
1.2.2 形态鉴定 将获得的目标菌株进行平板菌落观察、革兰氏染色及芽孢染色观察,具体方法参照《微生物学实验》[11]。
1.2.3 16S rDNA序列分析 按试剂盒说明书提取纯化细菌的基因组DNA,采用16S rDNA通用引物(27F:5′-AGAGTTTGATCMTGGCTCAGC-3′;1492R:5′-GGTTACCTTGTTACGACTT-3′)进行PCR扩增。PCR反应总体系为50 μL:DNA模板1 μL,2×TsingKE Master Mix 25 μL,上、下游引物(10 μmol/L)各1 μL,ddH2O 22 μL。PCR反应程序:94 ℃预变性10 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸90 s,共30个循环;72 ℃延伸10 min。PCR扩增产物经1.0%琼脂糖凝胶电泳后回收纯化,送北京奥维森基因科技有限公司进行测序。测得的16S rDNA序列在NCBI数据库进行序列比对,下载相关菌株的序列信息,采用Mega 6.0软件构建系统发育进化树。
1.2.4 乳酸转化特性试验 将所筛选的菌株制备成菌悬液(108CFU/mL),按5%比例接种到LADM液体培养基中,37 ℃厌氧培养5 d,定期取样测定菌株生长D600 nm值、乳酸消耗量和丁酸生成量,分析所筛选菌株生长情况与乳酸转化的特性。乳酸、丁酸含量使用高效液相色谱仪测定[7]:色谱柱HypersilC 184.6 mm×150 mm,5 μm;移动相2.5 mmol/L磷酸二氢铵,pH 2.65,流速1.0 mL/min;检测波长210 nm。
1.2.5 菌株的重测序及分析 采用高通量测序技术(next-generation sequencing technology,NGS)对所筛选菌株进行重测序及分析,具体流程如下:提取所筛选的菌株基因组DNA,样品的DNA纯度、完整性检测合格后,构建DNA小片段文库;再经质量检测合格后,按照文库有效浓度及期望数据量的需求对文库进行汇集(pooling),最后根据测序读长,在测序仪上机测序。所测得的有效测序数据比对到参考基因组(BioProject登录号:PRJNA478481)中,获得单核苷酸多态性(single nucleotide polymorphism,SNP)位点、插入缺失(insertion/deletion,InDel)、结构变异(structure variation,SV)、拷贝数变异(copy number variation,CNV)等遗传变异信息并进行比对分析[12]。
1.2.6 菌株的二代基因组测序及功能分析 在北京奥维森基因科技有限公司Illumina MiSeq测序平台通过二代基因组测序获得所筛选菌株的Raw reads数据,经Trimmomatic(v0.36)软件进行数据的质控,获得Clean reads,再经SPAdes v3.13.0软件拼接、组装,获得菌株的基因组基本数据[13]。通过基因预测分析菌株基因组的编码基因及非编码DNA结构、串联重复序列、CRISPR结构组件等,得到菌株的基因组组分。再将预测得到的蛋白质编码基因与各个标准功能数据库进行比对注释,并进行特定功能数据库注释、比较基因组等高级分析[14]。
2 结 果
2.1 菌株的分离鉴定
本研究通过LADM培养基初筛、RCM培养基复筛,筛选到1株菌株。所筛选菌株菌落呈奶油色,圆形,边缘整齐、稍突,表面湿润光滑,不透明,有酸臭味(图1A)。 显微镜下,其基本形态为革兰氏阳性杆菌、梭状,次端生芽孢、卵圆,(0.6~1.1) μm×(4.0~7.0) μm(图1B)。将此分离菌株命名为LY33。 LY33菌株的16S rDNA的PCR产物电泳结果见图2,其PCR产物大小为1 400 bp左右,与预期结果一致。所得序列经NCBI BLAST比对发现,LY33菌株与丁酸梭菌(C.butyricumTK2(登录号:KJ558432))一致性达到99.8%(图3),属于丁酸梭菌。

A,平板菌落形态;B,显微镜下的菌体形态(1 000×)

图2 LY33菌株16S rDNA PCR电泳图

图3 基于16S rDNA基因序列的系统进化树
2.2 菌株的乳酸转化性能分析
LY33菌株菌体生长、乳酸消耗及丁酸生成如图4所示,乳酸消耗及丁酸生成过程基本与LY33菌株菌体生长同步,LY33菌株利用乳酸生产丁酸的能力较强。当菌体在对数生长期生长较快时(2~6 d),LY33菌株对乳酸的转化速度较快,丁酸的生成量较大,在第6天可将66 mmol的乳酸基本转化完全,生成17 mmol左右的丁酸。

图4 LY33菌株菌体生长、利用乳酸生产丁酸的曲线图
2.3 菌株重测序结果分析
2.3.1 与参考基因组的比对结果 LY33菌株经高通量测序技术上机测序,获得原始碱基1 693 386 900 bp、有效率99.77%、GC含量29.83%。使用BWA程序把有效测序数据与参考基因组比对,经SAMtools对结果进行排序,匹配率为90.13%、测序深度为1×的最小覆盖范围为89.33%,测序深度为4×的最小覆盖范围为89.26%。LY33菌株的测序深度的分布及累积分布如图5所示,LY33菌株测序所得数据能较好的覆盖参考基因组。

图5 LY33菌株测序深度及累计分布图
2.3.2 基因组的变异分析 采用ANNOVAR软件对LY33菌株进行遗传变异信息的注释,结果表明与参考基因组相比,LY33菌株全基因组杂合比率为0.022‰,SNP位点变异总数21 590个(占整个基因组0.4666%),InDel突变总和为594个(占0.0128%,其中包括插入突变总和299个、缺失突变总和295个),SV注释的变异总数103个(占0.0003%,其中包括缺失76个、倒置12个、染色体内部迁移8个、染色体间的迁移7个),不存在CNV注释的变异。将所获得的SNP、InDel、SV和CNV遗传变异信息与参考基因组上对应的位置信息分析表明,突变基因主要为假设蛋白(hypothetical protein)、淀粉结合蛋白(starch-binding protein)、生物素合成酶BioB(biotin synthase BioB)、生物素转运体BioY(biotin transporter BioY)、酰基-sn-甘油-3-磷酸酰基转移酶(acyl-sn-glycerol-3-phosphate acyltransferase)、碳水化合物激酶(carbohydrate kinase)、1-脱氧-D-木酮糖-5-磷酸合酶(1-deoxy-D-xylulose-5-phosphate synthase)、甲基化趋化蛋白(methyl-accepting chemotaxis protein)、蔗糖-6-磷酸水解酶(sucrose-6-phosphate hydrolase)、含有蛋白的转运体-底物结合域(transporter substrate-binding domain-containing protein)等与菌株生长、能量代谢调节、维生素合成(特别是生物素)有关的基因,分布于2条染色体多个序列上。
2.4 菌株二代基因组测序及功能注释分析
2.4.1 菌株基因组基本信息 通过Illumina MiSeq测序平台获得了LY33菌株高质量基因组序列,获得Raw reads 5 644 623个,使用SPAdes v3.13.0软件对测序菌株质控后的测序数据进行组装,最终获得菌株基因组大小4 627 127 bp,GC含量28.60%,使用Prokka1.13.7软件对细菌基因组进行注释预测,LY33菌株预测基因总长度3 892 563 bp,预测基因数量4 171个,编码区总长度占全基因组84.12%。将测序原始数据上传至NCBI数据库,获得BioProject登录号:PRJNA761144。
2.4.2 菌株编码基因蛋白的功能注释 将LY33菌株所预测的基因蛋白编码序列输入NR、SwissProt、GO、KEGG、COG、InterproScan、CAZyme数据库中进行比对(Diamond,Evalue≤1e-5),各数据库中注释到的基因个数(非冗余)分别为:4 166、2 683、2 491、2 086、3 060、3 691和175个。
其中GO数据库注释得到三大分类统计中基因种类和数量最多的是生物过程(biological process),其次是分子功能(molecular function),细胞组分(cellular component),注释到的基因种类和数量最少(图6)。 KEGG注释中,数量最多的代谢通路类别分别是:碳水化合物代谢(carbohydrate metabolism)、全部和全局观览图(global and overview maps)、膜运输(membrane transport)和氨基酸代谢(amino acid metabolism)(图7)。COG数据库注释中数量最多的是碳水化合物运输和代谢(carbohydrate transport and metabolism)(图8)。表明LY33菌株的基因功能与细胞代谢密切相关,并参与了多种物质的代谢途径转化。此外,CARD数据库中共有109个抗性基因得到注释,其中基因个数最多的前13个抗性基因见图9,基因个数最多的抗性基因前3位是ARO:3000535(39个)、ARO:3002987(19个)和ARO:3003728(14个),分别与抗大环内酯类药物、抗杆菌肽、VanI糖肽抗性基因有关。

图6 LY33菌株的GO功能注释

图7 LY33菌株的KEGG通路分析

图8 LY33菌株的COG功能分类
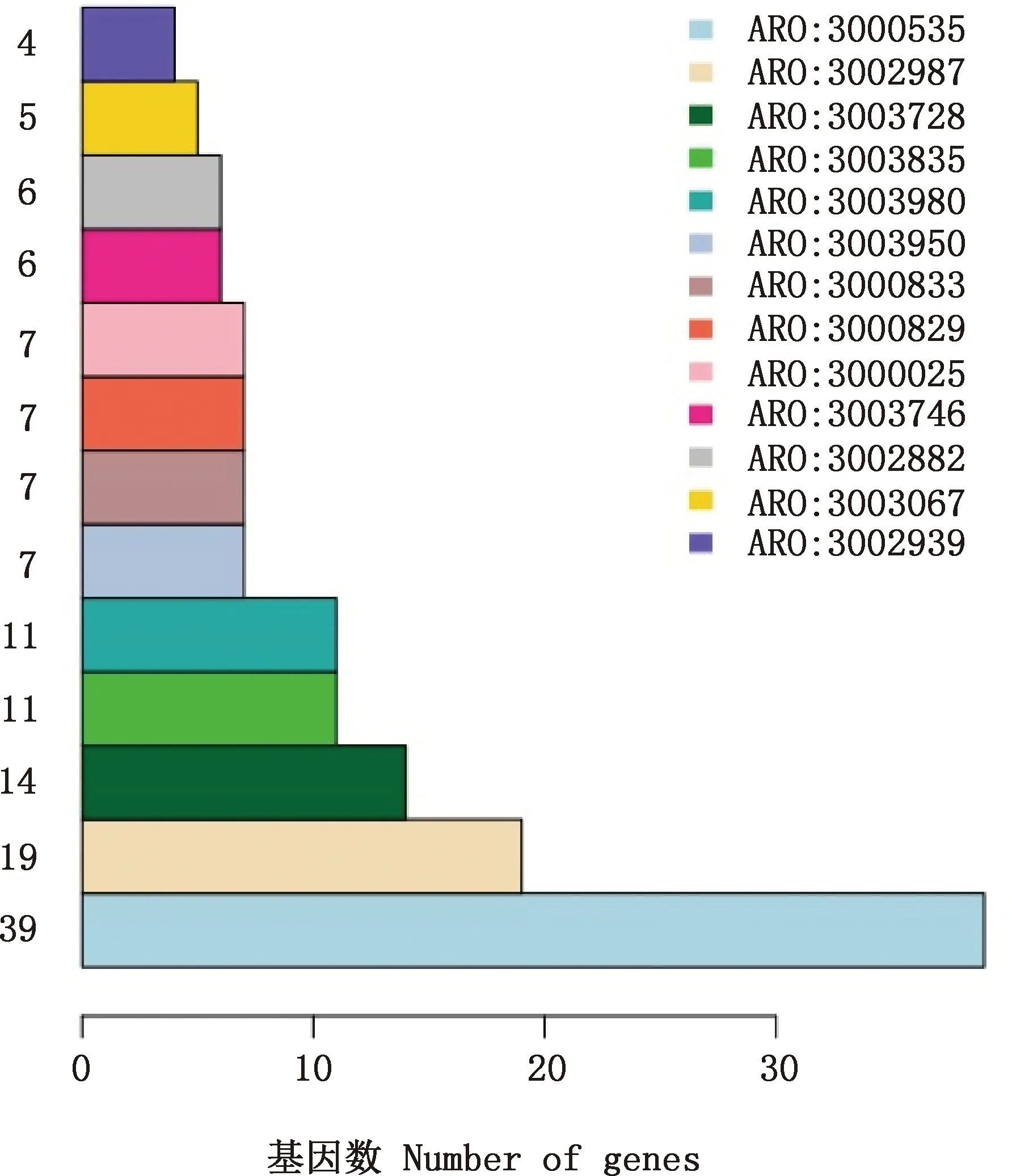
图9 LY33菌株的前13个抗性基因的CARD数据库注释个数
3 讨 论
除了饲喂乳酸菌等益生菌发酵饲料能在动物体内积累乳酸外,动物和人消化道内有许多微生物如嗜酸性乳酸杆菌(Lactobacillusacidophilus)、粪链球菌(Streptococcusfaecalis)、屎肠球菌(Enterococcusfaecium)等也都能产生乳酸[15]。乳酸对酸化饲料、降低畜禽胃内容物pH、杀灭肠道有害微生物,改善肠道菌群结构、刺激消化酶分泌等均具有良好的功效[16],但过多的乳酸容易导致畜禽短肠综合征、溃疡性结肠炎等疾病[10]。而丁酸在维持肠道生理平衡、保持上皮细胞完整性,降低肠炎和糖尿病等方面发挥重要作用[17]。因此肠道内能够代谢乳酸、产生丁酸的丁酸梭菌及其发酵制剂的研究越来越引起了研究者的重视,对饲用益生素的进一步发展有较好的实际意义[18-19]。
本研究对所分离的丁酸梭菌进行重测序,结果表明,LY33菌株基因组特别是编码基因整体变异较小,假设蛋白、淀粉结合蛋白、生物素合成酶BioB、生物素转运体BioY、酰基-sn-甘油-3-磷酸酰基转移酶、碳水化合物激酶等与菌体生长、能量代谢,维生素合成(特别是生物素)的功能基因变异增强。经查阅测序的基因描述(gene description)文件,LY33菌株基因组中确实含有丰富的维生素合成基因,如硫胺素合成酶(thiamine phosphate synthase)、核黄素合酶(riboflavin synthase)、双功能叶酸聚谷氨酸合酶(bifunctional folylpolyglutamate synthase)、6-羧基四氢叶黄素合酶(6-carboxytetrahydropterin synthase)、生物素-(乙酰辅酶A羧化酶)连接酶(biotin--(acetyl-CoA-carboxylase) ligase)、生物素依赖性羧基转移酶家族蛋白(biotin-dependent carboxyltransferase family protein)等,进一步支持了丁酸梭菌具有合成B族维生素、淀粉酶等功能的研究报道[20-21],在动物肠道内应用具有保健作用。此外,LY33菌株框架图测序及其编码基因蛋白的功能注释表明,LY33菌株生长过程中涉及了各种的能量、膜转运、氨基酸代谢的通路,推测与其产生淀粉酶、维生素有关,且对大环内酯类、多肽类抗生素具有较强的抗性,可通过抗生素表型试验做进一步的验证。
随着生物技术的不断发展,高通量测序技术已成为微生物基因组学研究的重要手段和分析方法[22]。本研究通过对仔猪肠道内容物中分离的丁酸梭菌进行重测序及二代框架测序,有利于在分子水平揭示丁酸梭菌的基因架构及功能特性的本质,为其在生物发酵饲料、功能食品、生物医药领域的应用提供理论基础。总之,本研究所获得的丁酸梭菌LY33菌株有望作为一种新的微生物资源用于微生态制剂产品的生产,并能与乳酸菌、芽孢菌、双歧杆菌等益生菌复配使用,增强此类产品的功效[23-24]。与目前广泛应用的非芽孢类活菌制剂相比,丁酸梭菌及其复合菌制剂更具广阔的市场前景。
4 结 论
本研究从饲喂4%乳酸菌发酵饲料的健康仔猪肠道内容物中分离到1株利用乳酸快、丁酸转化率高的LY33菌株,经鉴定为丁酸梭菌。LY33菌株的重测序表明,其基因组特别是编码基因整体变异较小,突变基因主要为与菌株生长、 能量代谢调节、 维生素合成(特别是生物素)有关的基因。LY33菌株框架图测序及其编码基因蛋白的功能注释表明,其基因组序列长度4 627 127 bp、GC含量28.60%,含有4 171个基因,编码区总长度占比84.12%,参与了糖代谢、膜转运和氨基酸代谢等多种代谢途径转化过程,基因组中抗大环内酯类药物、抗杆菌肽、VanI糖肽抗性基因个数较多。
