杉木NAC基因家族基因的鉴定及生物信息学分析
2022-03-02高文杰马祥庆
高文杰,刘 娇,马祥庆,帅 鹏
(福建农林大学 林学院,福建 福州 350002)
杉木Cunninghamia lanceolata是我国南方重要造林用材树种,距今已有2 000 多年的栽培历史,其生长迅速、主干通直、材质优良,具有很高的经济价值,目前杉木种植面积和蓄积量均居我国人工林树种的首位,但杉木多代连栽导致地力衰退,严重影响杉木的产量,制约杉木林的可持续经营[1]。转录因子对植物的生长发育以及逆境胁迫应答发挥重要作用,通过与顺式原件特异性结合,调控下游功能功能基因的表达[2]。除此之外,NAC转录因子家族中大部分的成员是通过植物生长发育的不同时期、外界环境中的因素或外源激素诱导而表达的,同时NAC基因的表达具有组织特异性。至今,NAC转录因子已经被验证在植物体生长发育、响应生物胁迫与非生物胁迫中扮演重要的角色。因此进一步挖掘和分析杉木NAC基因家族对选育有优良抗性的杉木品种、提高杉木的生长速度和提高杉木人工林的产量具有重大意义。
Souer 等[5]于1996年在矮牵牛(Petunia hybrida)中克隆得到的NAM(No apical meristem);Hirt 和Aida 等[6]于1997年分别克隆得到拟南芥的ATAF1/2和CUC2,发现它们与NAM 具有类似的功能并且编码蛋白区域N 端都包含了一段高度保守的氨基酸序列,因此这类基因取这3 个基因的首字母命名为NAC,之后便将含有NAC保守结构域的蛋白统称为NAC转录因子。大量植物基因组测序的完成和生物信息学的不断完善为NAC转录因子的研究提供了更加广泛的数据基础。近年来,对NAC转录因子家族的研究进入了热潮,目前已在拟南芥Arabidopsis thaliana中鉴定得到167 个NAC转录因子[7];水稻Oryza sativa中包含140 个NAC转录因子成员[8];大豆Glycine max中鉴定得到101 个NAC 转录因子成员[9];对木本植物NAC基因的研究起步较晚,但也取得了不少进展,例如在毛果杨Populus trichocarpa中鉴定得到163 个NAC转录因子成员[10];茶树Camellia sinensis中鉴定得到49 个NAC转录因子成员[11],进一步说明NAC转录因子的普遍存在性。
对于杉木NAC转录因子的也有研究报道,刘琼瑶[12]从杉木中转录组测序数据中克隆出8 条具有完整开放阅读框的杉木NAC基因序列:Cl-NAC1~Cl-NAC8,并且根据基因生物信息学分析推测,ClNAC1、Cl-NAC6 和Cl-NAC8 可能与次生细胞壁合成有关。魏明科等[13]克隆出与杉木次生壁形成相关的Cl-NAC1 基因,在组织表达特异性分析基础上开展该基因的SNP及其连锁不平衡分析。由于杉木基因组数据尚未破译,随着数据库的不断更新,进一步挖掘和分析杉木NAC基因家族十分有必要,本研究在杉木转录组测序获得的相关数据基础上,筛选鉴定出45 条杉木NAC基因序列,并运用多重序列比对、构建系统进化树等生物信息学分析技术对其进行结构和保守性研究,为进一步验证基因相关功能提供依据和基础。
1 材料与方法
1.1 数据库
为了最大限度的鉴定杉木NAC基因序列,本研究搜集到以下4 套包含不同组织来源的杉木转录组数据,包括课题组前期Li 等[14]采集32 号家系杉木幼苗的根、茎和叶组织,由通过Illumina HiSeq 2000 平台进行配对测序;北京林业大学Hu 等[15]采集GZ7 号家系杉木苗顶端针叶,由Illumina Solexa mRNA-Seq 平台进行测序;浙江农林大学Huang 等[16]通过Illumina HiSeqTM2000 平台对Zhelin21 号杉木的茎、幼叶、成熟叶、球果和幼根进行测序;北京林业大学Qiu 等[17]通过Illumina HiSeqTM2000 平台对杉木的根茎叶进行测序。
文章中涉及到的拟南芥、水稻和挪威云杉的NAC 家族成员来自北京大学开放研究数据平台——植物转录因子数据库PlantTFDB v4.0(http://planttfdb.cbi.pku.edu.cn/family.php?fam=NAC)。
1.2 生物学信息分析方法
利用PlantFDB 植物转录因子数据库上的Prediction 功 能(http://planttfdb.cbi.pku.edu.cn/prediction.php)对搜集到的杉木转录组中基因序列进行预测分析鉴定,初步筛选出NAC基因;将搜集到的杉木NAC基因序列导入ClustalX2.1 进行多序列比对,筛选去除重复的序列;使用DNAMAN v8.0 软件预测杉木NAC基因CDS 序列和氨基酸序列;在PlantTFDB 植物转录因子数据库上的BLAST 功 能(http://planttfdb.cbi.pku.edu.cn/blast.php)确定所得序列是否为NAC基因,确定得到Cl-NAC基因后分别与拟南芥和挪威云杉进行比对,获得与各Cl-NAC基因序列相似度最高的拟南芥与挪威云杉NAC基因[18]。
利用在线软件ExPASy-ProtParam(https://web.expasy.org/protparam/)分析蛋白的理化性质,包括分子量(kDa)、等电点(pI)等;使用GOR4(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_gor4.html)预测蛋白的二级结构;不同序列间相似度和NAC 结构域氨基酸序列比对用DNAMAN v8.0 中的多序列比对功能测定,结合MEGA 7.0 的ClustalW 功能和在线绘制软件WebLogo3.50(http://weblogo.threeplusone.com/)分析杉木NAC保守序列并作图;使用在线分析软件MEME(http://meme.sdsc.edu/meme/ meme-intro.html)对Cl-NACs 蛋白Motif 保守域进行分析[19-20],结合TBtools v0.665 作图; 使用ClustalX2.1 软件构建杉木、拟南芥同水稻NAC家族基因的进化树(依据N-J 法,其中bootstrap(1000 次重复));NAC 蛋白进化树亚家族的分类方法参考Ooka[21]的分族方法;在iTOL 网站(https://itol.embl.de/)对构建所得的进化树进行美化;采用在线psRNATarget 植物小RNA 靶基因预测分析 系 统(https://plantgrn.noble.org/psRNATarget/analysis?function=3),将杉木NAC基因与已发表的miR164 序列进行靶基因的预测分析,期望值选择<3.0,其他选项保持默认值。
1.3 计算公式
为了验证鉴定得到的Cl-NACs基因的完整性,本研究引入氨基酸序列覆盖度来评价其序列完整性,计算公式为:氨基酸序列覆盖度=目标氨基酸序列长度/比对氨基酸序列长度。
2 结果与分析
2.1 杉木NAC基因的鉴定与序列分析
共鉴定得到45 条Cl-NACs 家族基因序列(Cl-NAC1~Cl-NAC45),具体序列见表1。如表1所示,预测所得CDS 序列长度在156~3 033 bp 之间,由51~1 010 个氨基酸残基构成,由于转录组数据来源不同,测序方法间存在差异,部分基因序列非全长,部分序列长度差异较大;分子量在6.19~114.28 kDa 之间变化,等电点的范围在4.32~10.17 之间,分子量最大的为Cl-NAC23,最小的为Cl-NAC19,等电点最大的为Cl-NAC12,最小的为Cl-NAC3;通过对蛋白的理化性质分析,其中有34 个Cl-NACs 的蛋白不稳定系数高于40,属于不稳定蛋白,Cl-NAC43的不稳定系数为66.89,稳定性最差;11 个Cl-NACs 的蛋白不稳定系数低于40,为稳定蛋白,Cl-NAC45 不稳定系数为30.58,稳定性最高;脂溶系数在48.65~103.51 之间,总平均疏水指数在-1.033~0.206 之间;预测蛋白二级结构结果显示:α-螺旋(Alpha helix)在二级结构中占的比例在 0%~41.96%之间,延伸链(Extended strand)在3.09%~36.54% 之间, 无规卷曲(Random coil)所占比例最高,在37.05%~68.60%之间。

表1 Cl-NACs 基因序列分析及氨基酸的理化性质和二级结构Table 1 Sequence analysis and physicochemical properties and secondary structures of Cl-NACs
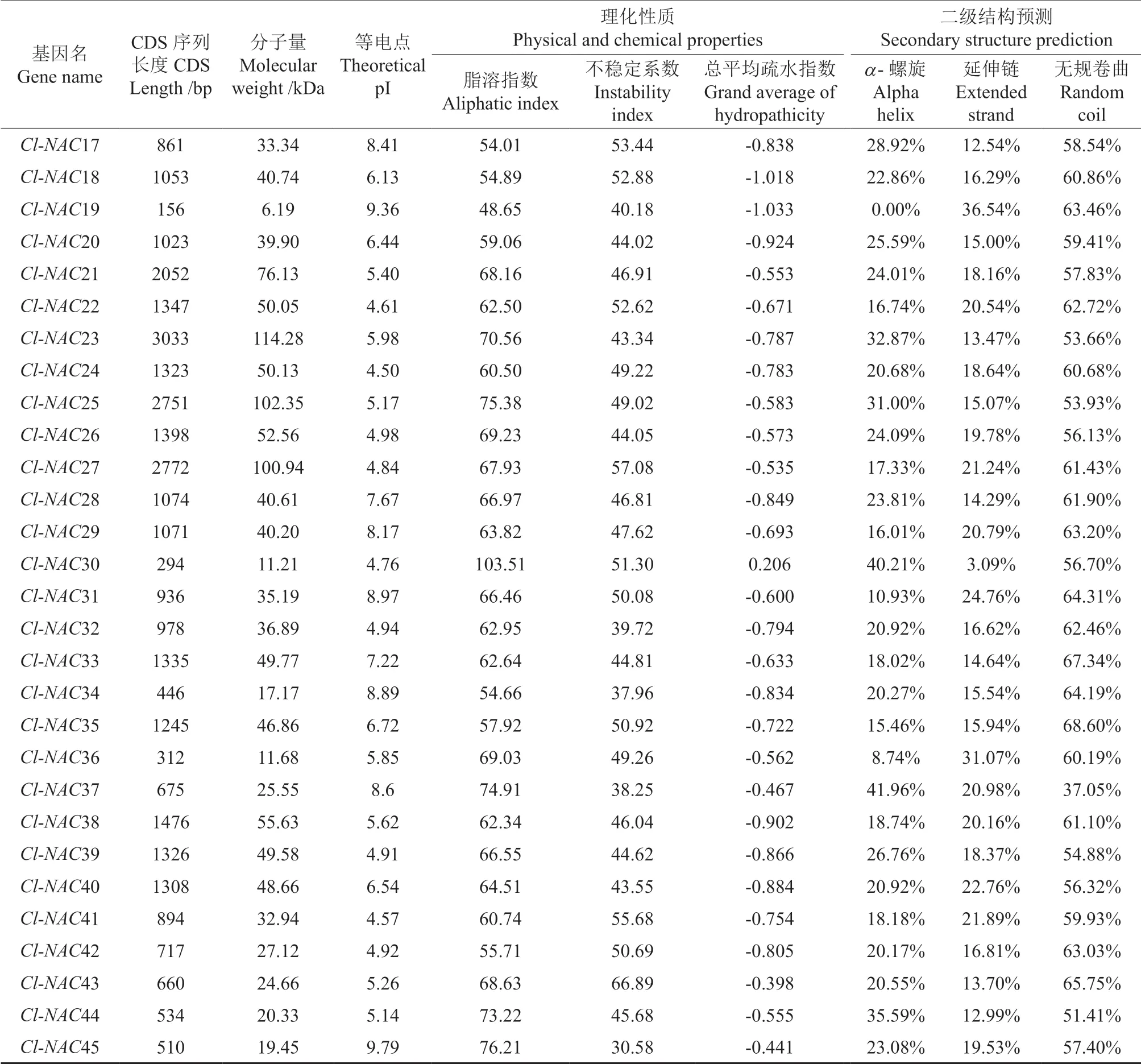
续表1Continuation of table 1
2.2 Cl-NACs 基因与拟南芥和挪威云杉序列的比对
将45 条Cl-NACs 基因分别与拟南芥和挪威云杉NAC基因进行BLAST 比对,挑选相似度最高的目标序列,并测得相似度,结果如表2所示,对比发现,Cl-NACs 基因与拟南芥NAC基因相似度在2.32%~52.38%之间变化,与挪威云杉NAC基因相似度在4.66%~73.73%之间变化;此外,对比同一条Cl-NAC基因与两种植物的相似度发现除了Cl-NAC19、20、25、27、31、34、36 和45这8 条基因与拟南芥NAC基因比对的相似度大于挪威云杉外,其余37 条基因均与挪威云杉NAC基因的相似度更高,其中Cl-NAC3 的差异最明显,与MA_103386g0010 的相似度为AT4G14960.1 的5 倍。

表2 Cl-NACs 基因与拟南芥和挪威云杉比对结果†Table 2 Comparison of Cl-NACs with NAC genes in Arabidopsis and Norway spruce
为了进一步区分已经鉴定出来的基因的完整度,本研究引入氨基酸序列覆盖度来描述获得基因的完整性。对比Cl-NACs 与拟南芥NAC 的蛋白覆盖度发现,有6 条Cl-NACs 的氨基酸序列覆盖度低于50%,最低的为Cl-NAC37,仅14.02%,有24 条Cl-NACs 的氨基酸序列覆盖度介于50%~100%之间,序列可能较为完整,而有15条Cl-NACs 的氨基酸序列覆盖度超过100%,最高为Cl-NAC13,达318.29%;对比Cl-NACs 蛋白与挪威云杉NAC的氨基酸序列覆盖度发现,有7 条Cl-NACs 的氨基酸序列覆盖度低于50%,最低为Cl-NAC19,仅11.56%,有28 条Cl-NACs 的氨基酸序列覆盖度介于50%~100%之间,序列可能较为完整,而有10 条Cl-NACs的氨基酸序列覆盖度超过100%,最高为Cl-NAC27,达250.14%。
2.3 Cl-NACs 蛋白结构域氨基酸序列多重比对及保守域鉴定与分析
45 条Cl-NACs 蛋白的多序列比对相似度(Identity)为8.24%,对Cl-NACs 蛋白的N 端结构域进行多重比对结果如图1所示,除了长度不足的序列,所有的NAC 蛋白都具有A、B、C、D、E 5 个亚结构域,并且位置一致。

图1 Cl-NACs 蛋白N 端序列比对Fig.1 Cl-NACs protein N-terminal sequence alignment
通过MEME 在线分析软件对Cl-NACs 蛋白进行保守序列区域分析,得出了最有相关性的7 个保守元件(Motif1~Motif7),各保守元件所在Cl-NACs 蛋白上的位置如图2所示,保守元件长度在11~29 aa 之间,具体信息如表3所示。

表3 所发现的Motif 结构域性质Table 3 Motif properties

图2 Cl-NACs 蛋白保守序列区域预测Fig.2 Prediction of conserved sequences in Cl-NAC proteins
2.4 Cl-NACs 蛋白进化树分析
为了研究45 条Cl -NACs 蛋白与拟南芥和水稻NAC 蛋白的进化关系,利用ClustalX2.1 的Neighbor-Joining(NJ)将预测所得的Cl-NACs 蛋白与来自拟南芥的105 条、来自水稻的75 条NAC蛋白共225 条氨基酸序列构建进化树,共分为18个亚家族,结果如图3所示。45 条Cl-NAC 蛋白分布在10 个NAC 亚家族中,分别是ONAC003、ONAC001、ANAC063、ONAC022、NAP、AtNAC3、ATAF、ANAC011、OsNAC7 和NAM,8 个亚家族中未发现有Cl-NAC 蛋白分布,分别是ANAC001、TERN、SENU5、OsNAC3、NAC2、TIP、OsNAC8 和NAC1 亚家族,Ⅰ-ONAC022 族中有11 条Cl-NAC 蛋白,数量最多,Ⅰ-ANAC011族中有7 条,Ⅰ-NAP 族中有6 条,Ⅱ-ANAC063族中有5 条,Ⅰ-OsNAC7 和Ⅰ-NAM 族中各有4条,Ⅱ-ONAC003 和Ⅰ-AtNAC3 族中各有3 条,Ⅱ-ONAC001 和Ⅰ-ATAF 族中各有1 条,具体分族情况如表4所示。

表4 NAC 亚家族分族情况Table 4 NAC subfamilies
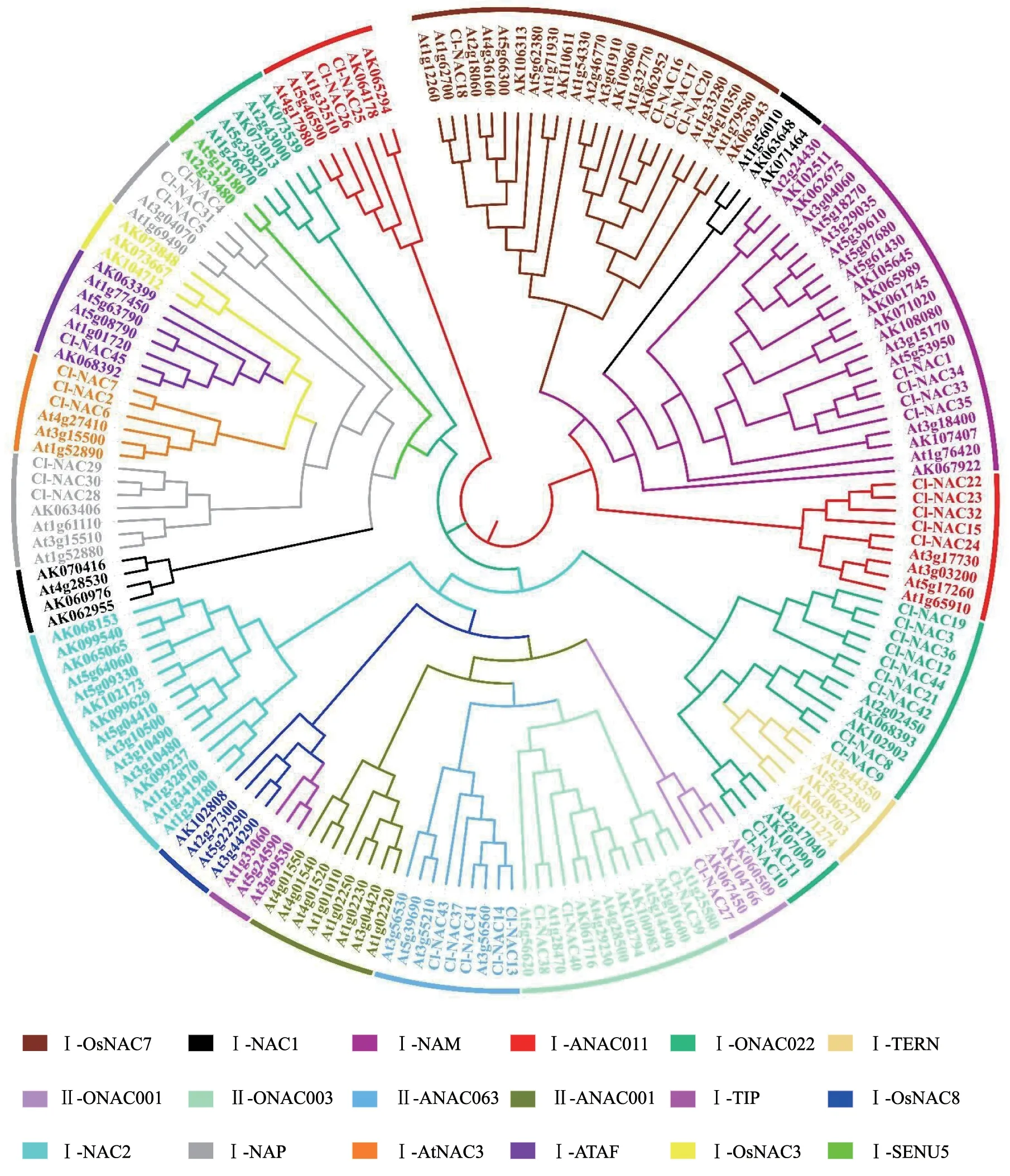
图3 杉木、拟南芥和水稻NAC 蛋白进化树Fig.3 Evolutionary tree of Chinese fir,Arabidopsis and rice NAC proteins
2.5 Cl-NACs 基因与miR164 的可能靶向关系分析
通过小RNA 靶基因预测系统分析,结果如图4所示, 发现cln-miR164与Cl-NAC33 和Cl-NAC35 之间找出了可能的目标位点,期望值分别为1.0 与1.5,分别对应Cl-NAC33 的641~661 bp区间和Cl-NAC35 的656~676 bp 区间,并且Cl-NAC33 和Cl-NAC35 同为Ⅰ-NAM 亚家族。

图4 杉木NAC基因和miR164 的可能靶向关系Fig.4 Possible targeting relationships between Chinese fir NACgenes and miR164
3 讨论
3.1 杉木NAC基因鉴定的准确性与完整性评估
目前关于杉木NAC基因鉴定的报道较少,刘琼瑶[12]克隆出8 个具有完整开放阅读框的杉木NAC基因(Cl-NAC1~Cl-NAC8);魏明科等[13]克隆出与杉木次生壁形成相关的Cl-NAC1 基因。将这8 条杉木NAC引物序列与本试验鉴定得到的45 个Cl-NACs 序列进行比对发现,本试验鉴定出的Cl-NACs 基因包括了已被报道的这8 条杉木NAC基因,具体对应情况见表2,这不仅大大丰富了NAC转录因子在杉木中的研究,同时能够为培育抗逆境胁迫杉木材料打下初步基础。
但由于杉木基因组尚未破解,本试验中使用的是转录组数据对杉木NAC基因进行的鉴定,其准确度相比基因组来说有所差距,可能存在着加工型假基因,也可能存在部分基因结构的不完整与低表达基因的缺失,也就是说可能还存在未被测序出的杉木NAC基因。对比Cl-NACs 与拟南芥和挪威云杉NAC基因序列的覆盖度发现部分Cl-NACs 氨基酸序列覆盖度低于50%,说明本试验鉴定得到的部分NAC基因序列可能并不完整或者测序存在错误导致得到的序列与真实序列存在偏差,需通过完成杉木基因组测序或不断丰富杉木转录组数据进行验证和补充。
3.2 通过转录因子相似度推测物种间亲缘性
通过对比Cl-NACs 基因与拟南芥和挪威云杉中最相似NAC基因的相似度发现(表2),大部分Cl-NACs 基因与挪威云杉NAC基因序列相似度明显大于拟南芥,并且45 条基因同挪威云杉NAC基因序列相似度平均值(36.69%)是同拟南芥NAC基因(28.46%)的1.29 倍,这与曾铭等[22]在对杉木WRKY 基因的研究结果一致,表明亲缘性较高的物种间的转录因子相似度较高。
3.3 Cl-NACs基因亚家族分布情况
Ⅱ-ANAC001、Ⅰ-TERN、Ⅰ-SENU5、Ⅰ-OsNAC3、Ⅰ-NAC2、Ⅰ-TIP、Ⅰ-OsNAC8 和Ⅰ-NAC1 8 个亚家族中未发现Cl-NACs 基因,这与Hu 等[10]和张晓孟[23]的研究结果类似,由于杉木为裸子植物,而杨树、黄瓜与番茄同为被子植物,鉴定得到的NAC基因数量较多,并且在亚家族中分布较为普遍。在上述8 个亚家族中,Ⅱ-ANAC001、Ⅰ-SENU5、Ⅰ-OsNAC3 和Ⅰ-TIP 4个亚家族中只有拟南芥NAC基因,没有水稻和杉木的同源基因,这表明4 个亚家族的NAC成员可能是在与单子叶植物分化后获得的,而且在双子叶植物和单子叶植物分化后,这些基因特异地在单子叶植物中产生。由于NAC亚家族的分类方法种类繁多、算法多样,导致不同的植物在构建NAC基因进化树结果差异大,对进化树分析产生一定的影响。
3.4 Cl-NACs 基因功能初步推测
本试验共鉴定出了45 个Cl-NACs 基因,根据系统进化树的分析,发现在两个亚族(Ⅰ和Ⅱ)族中均有分布Cl-NACs 基因,而Cl-NACs 蛋白共分布在10 个NAC亚家族中,有8 个亚家族中未发现Cl-NACs 基因。大量的研究表明Ⅰ-ATAF 亚家族中大量NAC基因都参与植物响应环境胁迫分子调控机制[24-25],而在鉴定得到的Cl-NACs基因中Cl-NAC45 属于Ⅰ-ATAF 亚家族,前人的研究表明同一亚家族内的基因功能通常类似,因此它可能与杉木在响应逆境胁迫过程中发挥重要作用。Ⅰ-NAM 亚家族中大量NAC基因如NAM和CUC2都被验证参与茎尖分生组织的形成[26-27],而有4 个Cl-NACs基因属于这个亚家族,分别是Cl-NAC1、Cl-NAC33、Cl-NAC34、Cl-NAC35, 它们都有可能参与杉木生长发育中的调节作用。
刘琼瑶[12]和魏明科等[13]的研究表明杉木NAC基因Cl-NAC1、Cl-NAC6 和Cl-NAC8 可能与次生细胞壁的合成有关,Cl-NAC8 转基因烟草植株叶片相对于对照组较小,叶片较厚,叶缘褶皱多,进一步验证了Cl-NAC8 可能与参与植物生长发育过程的调节。本试验验证得到的杉木NAC基因与这3 个基因对应的分别是Cl-NAC40、Cl-NAC38和Cl-NAC17,这3 个基因极有可能在杉木生长发育过程中发挥重要作用。
3.5 Cl-NAC33 和Cl-NAC35 受cln-miR164 调控参与非生物胁迫应答过程
近年来,国内外对miRNA 功能的研究掀起了热潮,越来越多的研究表明miRNA 参与植物生长发育、激素水平调节和逆境胁迫响应等过程[28-30]。唐芳等[31]通过序列比对、不同发育时期及不同组织中的表达模式以及5′-RACE 检测等方面对杨树miR393 与FBL基因家族成员的调控作用进行鉴定,揭示了miRNA 对靶基因的剪切调控功能。miR164 是其中较为保守的一个家族,并且大量研究证明miR164 的最主要的靶标是NAC家族基因[32-36]。Li 等[37]在研究中发现马铃薯的stu-miR164在NAC转录因子的CDS 序列中具有互补序列,并且在干旱胁迫下stu-miR164表达量显著下降,而其靶向NAC基因表达量显著升高,此外通过试验发现过表达stu-miR164的马铃薯植株在干旱胁迫下StNAC262的表达量明显降低,侧根数量也较对照组减少,进一步证实stu-mi164对NAC 转录因子具有调控作用。本试验通过小RNA 靶基因预测系统分析,在cln-miR164与Cl-NAC33 和Cl-NAC35 之间找出了可能的假定目标位点,这与Li等[37]的研究结果类似。此外,张莉[38]对马铃薯StNAC262 基因进化分析其属于NAM 亚家族,而Cl-NAC33 和Cl-NAC35 同属NAM 亚家族,说明Cl-NAC33 与Cl-NAC35 的功能可能与StNAC262类似受cln-miR164的调控,通过其表达量变化参与调控杉木应答干旱胁迫或其他非生物胁迫。
通过BLAST 功能发现与Cl-NAC33 最相似的拟南芥NAC基因AT5G53950(即CUC2)已经被验证为miR164a的靶基因,研究表明植物叶片锯齿化程度由miR164a 介导抑制CUC2的表达量来调节,并且miR164/CUC2在植物心皮发育中发挥重要的作用[39-40],Cl-NAC33 与CUC2基因序列相似度高达38.96%,且同为miR164 的靶基因,说明Cl-NAC33 与CUC2可能具有相似的功能,在杉木叶片形态形成与生长发育阶段起重要调控作用;而与Cl-NAC35 最相似的拟南芥NAC基因AT3G18400(即ANAC058),目前尚未发现其具体功能,待进一步研究。
本研究利用多套杉木转录组数据来鉴定杉木NAC基因。由于没有基因组数据,结果分析中难免会出现大量杉木NAC基因不完整的现象,而且一些表达量低或具有杉木组织特异性表达的NAC基因可能未鉴定到。下一步将基于杉木基因组数据来完善鉴定NAC基因鉴定。但本研究所鉴定的不完整杉木NAC基因仍然可以通过RACE 实验等技术获得其全长序列,为下一步深入研究提供一定的基础。
4 结论
本研究在杉木转录组测序获得的相关数据的基础上,筛选鉴定出45 条杉木NAC基因序列,分别命名为Cl-NAC1~Cl-NAC45,CDS 序列长度在156~3 033 bp 之间,由51~1 010 个氨基酸残基构成。将45 条Cl-NACs 分别与拟南芥和挪威云杉NAC基因进行BLAST 比对,挑选相似度最高的目标序列对比发现,Cl-NACs 基因与拟南芥NAC基因相似度区间为2.32%~52.38%,与挪威云杉NAC基因相似度区间为4.66%~73.73%。对Cl-NACs 蛋白进行保守序列区域分析,获得7 个最有相关性的保守元件(Motif1-Motif7),7 个保守元件长度为11~29 aa。通过构建Cl-NACs 蛋白与拟南芥和水稻NAC 蛋白的进化树发现,45 个Cl-NACs 蛋白分布在10 个NAC 亚家族中。此外,研究还发现Cl-NAC33 和Cl-NAC35 可能为clnmiR164 的靶基因,参与生物胁迫响应途径。
