中华蜜蜂幼虫肠道中微小RNA的鉴定及分析
2022-02-22冯睿蓉付中民杜宇张文德范小雪王海朋万洁琦周紫彧康育欣陈大福郭睿史培颖
冯睿蓉,付中民,2,杜宇,张文德,范小雪,王海朋,万洁琦,周紫彧,康育欣,陈大福,2,郭睿,2,史培颖
中华蜜蜂幼虫肠道中微小RNA的鉴定及分析
冯睿蓉1,付中民1,2,杜宇1,张文德1,范小雪1,王海朋1,万洁琦1,周紫彧1,康育欣1,陈大福1,2,郭睿1,2,史培颖1
1福建农林大学动物科学学院(蜂学学院),福州 350002;2福建农林大学蜂疗研究所,福州 350002
【】利用small RNA-seq(sRNA-seq)技术和生物信息学方法对中华蜜蜂(,简称中蜂)幼虫肠道的微小RNA(microRNA,miRNA)进行全转录组鉴定和分析,旨在丰富中蜂的miRNA信息,并为深入研究miRNA调控中蜂幼虫肠道发育的分子机理提供依据。利用sRNA-seq技术对中蜂4、5和6日龄幼虫肠道样品(Ac1、Ac2和Ac3)进行测序,通过数据质控获得有效标签序列(clean tags)。采用Blast工具将clean tags连续比对东方蜜蜂()基因组和miRBase数据库,以鉴定保守miRNA和新miRNA。采用TPM法对miRNA的表达量进行归一化处理。通过GraphPad Prism 7软件统计各组肠道样品中sRNA占比、miRNA长度分布及首位碱基偏向性。利用相关软件预测上述miRNA靶向的mRNA并进行GO和KEGG数据库注释。进一步根据靶向结合关系构建和分析注释到发育和免疫相关通路的基因及其靶向miRNA的调控网络,并利用Cytoscape软件进行可视化。利用茎环反转录PCR(Stem-loop RT-PCR)、分子克隆和Sanger测序验证miRNA的表达和序列的真实性。共鉴定到中蜂的371个保守miRNA和64个新miRNA;这些miRNA的长度介于18—25 nt且首位碱基主要偏向于U;上述miRNA共靶向14 750条mRNA,涉及离子结合、金属离子结合、细胞膜、细胞膜组件和单一有机体进程等2 270个GO条目,以及内吞作用、细胞凋亡、mTOR信号通路、RNA转运和昆虫激素的生物合成等332条KEGG通路。进一步分析结果显示156个miRNA与注释到Wnt、Hippo、Notch和mTOR等生长发育相关通路的67个靶基因存在调控关系,145个miRNA与注释到Toll、Imd/JNK、Jak-STAT和抗菌效应因子等免疫相关途径的21个靶基因存在调控关系。Stem-loop RT-PCR 结果显示miR-8-y、miR-9-z、miR-14-y、miR-281-y、miR-283-x和miR-306-x均能扩增出预期的特异性片段;Sanger测序结果显示上述6个miRNA的序列与深度测序结果一致。提供了中蜂miRNA的数量、结构特征和表达谱;揭示中蜂幼虫肠道miRNA潜在调控诸多生命进程与细胞活动;中蜂幼虫肠道的部分miRNA可通过靶向结合相应的mRNA参与调节发育和免疫相关途径。
东方蜜蜂;中华蜜蜂;幼虫;肠道;微小RNA;调控网络
0 引言
【研究意义】中华蜜蜂(,简称中蜂)是高度适应我国自然环境的东方蜜蜂()亚种,具有特殊的生态价值和经济价值。微小RNA(microRNA,miRNA)作为一类重要的基因表达调控因子,通过在转录后水平调控基因表达而发挥诸多重要的生物学功能[1]。目前,中蜂的miRNA研究较为滞后。对中蜂幼虫肠道的miRNA进行全转录组鉴定和分析,可丰富中蜂的miRNA信息,也为深入开展中蜂miRNA的功能研究打下基础。【前人研究进展】研究表明,miRNA参与调控昆虫的生长、发育、生殖、昼夜节律、学习记忆、免疫应答及抗药性等重要生物学过程[2-3]。目前,miRBase数据库(http://www.mirbase.org/)已收录黑腹果蝇()、家蚕()、赤拟谷盗()等26种昆虫的3 000余个miRNA,包括西方蜜蜂()的262个miRNA,但尚未收录东方蜜蜂miRNA。前人对西方蜜蜂的miRNA进行了较多研究[4-5]。例如,CRISTINO等[6]研究发现,miR-932可靶向调控西方蜜蜂体内肌动蛋白基因的表达,从而影响蜜蜂大脑的记忆产生过程。笔者团队前期对意大利蜜蜂(,简称意蜂)4、5和6日龄幼虫肠道进行深度测序和分析,共预测到560个miRNA,并发现ame-bantam和ame-miR-927a等潜在调控Wnt、Hippo和Notch等信号通路进而参与意蜂幼虫肠道的发育[7];差异表达谱分析揭示ame-miR-6001-3p和miR-342-y等差异表达miRNA(differentially expressed miRNA,DEmiRNA)通过调控生长发育及物质能量代谢相关的多条通路潜在影响意蜂工蜂中肠的发育过程[8]。相比于西方蜜蜂,东方蜜蜂的miRNA研究较为滞后,相关信息有限[9-11]。笔者团队前期发现中蜂工蜂中肠内miR-8、miR-14和bantam等miRNA维持较高表达水平,而miR-1、miR-92和miR-194等miRNA在宿主响应东方蜜蜂微孢子虫()胁迫的过程中不同程度的差异表达,并潜在调节应激反应、能量代谢和细胞凋亡等途径进而参与宿主免疫应答[11]。【本研究切入点】目前,东方蜜蜂的miRNA信息总体有限,中蜂的miRNA信息更为匮乏。【拟解决的关键问题】利用small RNA-seq(sRNA-seq)技术对中蜂4、5和6日龄幼虫肠道样品进行深度测序,通过生物信息学方法对中蜂miRNA进行全转录组鉴定,并构建和分析肠道发育和免疫相关调控网络,进而对部分miRNA进行表达和序列验证,为东方蜜蜂和中蜂的miRNA信息提供有益补充,并为进一步研究miRNA调控中蜂幼虫肠道发育的分子机理打下基础。
1 材料与方法
试验于2018年11月至2020年1月在福建农林大学动物科学学院(蜂学学院)蜜蜂保护实验室完成。
1.1 供试蜜蜂幼虫
中蜂幼虫取自福建农林大学动物科学学院(蜂学学院)教学蜂场。
1.2 中蜂幼虫的人工饲养与肠道样品制备
参照笔者所在实验室前期建立的技术流程[12-13],从蜂群群势较强且整体健康的中蜂蜂群中提取已限王产卵的巢脾,将2日龄幼虫用移虫针移至已预置50 μL饲料的48孔细胞培养板中,并将培养板放置于温度为35℃、相对湿度(RH)为90%的培养箱内。幼虫饲料按照蜂王浆63%、无菌水30%、蜂蜜6%和酵母提取物1%的质量比例配制;每24 h更换一次饲料,并及时移除死亡幼虫。分别剖取4、5和6日龄幼虫肠道(分别命名为Ac1、Ac2和Ac3),置于RNA-Free的EP管,放入液氮速冻,然后-80℃超低温冰箱保存备用。Ac1-1、Ac1-2和Ac1-3为Ac1组的3个生物学重复;Ac2-1、Ac2-2和Ac2-3为Ac2组的3个生物学重复;Ac3-1、Ac3-2和Ac3-3为Ac3组的3个生物学重复。
1.3 sRNA-seq与数据质控
按照陈华枝等[14]的方法,利用Trizol法提取上述9个幼虫肠道样品的总RNA;通过琼脂糖凝胶电泳、切胶回收并连接3′接头序列;15%的变性聚丙烯酰胺凝胶电泳进行分离,切胶回收连接5′接头序列;反转录的产物经3.5%的琼脂糖凝胶电泳分离,进而切胶回收140—160 bp区域条带,产物即为终文库。委托广州基迪奥生物科技有限公司进行单端测序,测序结果已上传至NCBI SRA数据库,BioProject号:PRJNA395108。参照杜宇等[10]的方法对下机的原始读段(raw reads)进行过滤,进一步对得到的有效读段(clean reads)进行严格质控,将得到有效标签序列(clean tags)用于后续分析。
1.4 miRNA的生物信息学预测与分析
参照熊翠玲等[15]的方法进行miRNA的预测和分析,先利用Blast工具将各样品的clean tags序列比对到GenBank(https://www.ncbi.nlm.nih.gov/genbank/)及Rfam(http://rfam.xfam.org/)数据库,去除可注释上的clean tags,再将未注释上的clean tags比对到东方蜜蜂的参考基因组(Assembly ACSNU-2.0),以获得参考基因组上的位置信息。进一步将比对上参考基因组的clean tags与miRBase数据库中已知miRNA的前体序列进行比对,以鉴定保守miRNA(known miRNA),保守miRNA后缀“-x”和“-y”分别代表从miRNA前体的5′臂或3′臂加工而来,“-z”代表来源位置尚不确定;根据miRNA是否具有经典的茎环结构鉴定新miRNA(novel miRNA)。根据公式TPM=T×106/N(T表示miRNA的tags,N表示总miRNA的tags)对表达量进行归一化处理,从而得到各组样品中全部miRNA的表达谱。通过GraphPad Prism 7软件统计各组肠道样品中sRNA的占比、miRNA的长度分布和首位碱基偏向性。利用OmicShare在线工具集合(www.omicshare.com)对各组肠道样品中的miRNA进行Venn分析。
1.5 miRNA的靶向预测及调控网络构建与分析
参照耿四海等[16]和陈华枝等[17]的方法,联用TargetFinder/RNAhybrid(v2.1.2)+svm_light(v6.01)、Miranda(v3.3a)、TargetScan(v7.0)软件预测上述miRNA靶向的mRNA,均采用默认参数,将3种预测结果的交集作为可靠的靶标集合。利用Blast工具将预测得到的靶mRNA序列与GO和KEGG数据库比对,获得靶mRNA的功能注释信息。
鉴于中蜂的全部miRNA与靶向mRNA之间的调控网络十分复杂,本研究根据KEGG数据库及Nr数据库注释结果筛选出注释到发育和免疫相关通路的mRNA,将上述mRNA对应的基因命名为靶基因,根据miRNA与靶基因之间的结合关系构建相关调控网络,进而通过Cytoscape软件(v3.6.1)进行可视化。
1.6 miRNA的Stem-loop RT-PCR验证
参照杜宇等[10]的方法,随机挑选6个miRNA(miR-8-y、miR-9-z、miR-14-y、miR-281-y、miR-283-x和miR-306-x)进行表达验证。根据相应的核酸序列,设计特异性Stem-loop引物和上游引物以及通用下游引物,委托上海生工生物工程股份有限公司合成。利用RNA抽提试剂盒(Promega,美国)分别提取中蜂4、5和6日龄幼虫肠道的总RNA,然后各0.5 μg进行混合作为反转录模板,再利用Stem-loop引物进行反转录得到相应的cDNA,作为模板进行PCR扩增(翊圣,中国)。PCR产物经1.5%琼脂糖凝胶电泳检测。胶回收100 bp附近的片段,连接pMD-19T载体(Takara,中国),转化DH5大肠杆菌(天根,中国)后挑斑摇菌,取菌液PCR呈阳性的菌液送上海生工进行单端测序。
2 结果
2.1 数据质控与评估
Ac1、Ac2和Ac3组幼虫肠道样品测得的clean reads数分别平均为11 273 306、11 349 964和11 122 092条,经质控得到的clean tags数分别平均为9 791 926、9 402 531和9 394 648条,占比均≥82.77%(表1)。上述结果说明本研究中sRNA-seq数据质量良好,可满足后续分析需要。
2.2 中蜂幼虫肠道miRNA结构特征与表达谱
共预测出435个miRNA,包含371个保守miRNA和64个新miRNA,长度分布介于18—25 nt,分布数量最多的长度为18 nt和22 nt(图1-A);此外上述miRNA的首位碱基主要偏向于U,其次偏向于A(图1-B)。

表1 sRNA-seq数据信息概览

A:miRNA的长度分布Length distribution of miRNAs;B:miRNA的首位碱基偏向性First nucleotide bias of miRNAs
表达谱分析结果显示,上述435个中蜂miRNA的表达量介于0.08—192 644,表达量最高的前10位miRNA分别为miR-8-y(TPM=192 644)、miR-750-y(TPM=104 019)、miR-184-y(TPM=70 814)、bantam-y(TPM=47 727)、miR-275-y(TPM=44 336)、miR-14-y(TPM=43 636)、miR-9-z(TPM=39 580)、miR-281-y(TPM=36 366)、miR-306-x(TPM=33 676)和miR-283-x(TPM=31 896)。
2.3 中蜂幼虫肠道miRNA的Stem-loop RT-PCR验证
Stem-loop RT-PCR结果显示,随机选择的6个miRNA(miR-8-y、miR-9-z、miR-14-y、miR-281-y、miR- 283-x和miR-306-x)均真实表达。进一步的分子克隆和Sanger测序结果与其测序结果中的序列完全一致(图2)。上述结果证明了测序数据和预测结果的可靠性。
2.4 中蜂幼虫肠道miRNA的功能注释
靶向预测结果显示,中蜂幼虫肠道的435个miRNA共靶向14 750条mRNA,涉及分子功能、细胞组分和生物学进程相关的2 270个GO条目;其中,有941条靶mRNA可注释到离子结合(GO:0043167)和565条靶mRNA可注释到金属离子结合(GO:0046872)等570个分子功能相关条目,有874条靶mRNA可注释到细胞膜(GO:0016020)和826条靶mRNA可注释到细胞膜组件(GO:0044425)等241个细胞组分相关条目,有1 509条靶mRNA可注释到单一有机体进程(GO: 0044699)和281条靶mRNA可注释到单一有机体定位(GO:1902578)等1 459个生物学进程相关条目(图3)。

A:miRNA的Stem-loop RT-PCR Stem-loop RT-PCR of miRNAs;B:miRNA的Stem-loop RT-PCR产物的Sanger测序结果 Sanger sequencing result of Stem-loop RT-PCR product from miRNAs;黄色区域表示相应miRNA的成熟序列 Yellow regions indicate mature sequences of corresponding miRNAs

图3 中蜂幼虫肠道miRNA靶向mRNA的GO条目富集圈图
上述靶mRNA还可注释到332条通路,包括内吞作用(ko04144)和细胞凋亡(ko04210)等细胞进程相关通路,FoxO信号通路(ko04068)和mTOR信号通路(ko04150)等环境信息加工相关通路,泛素介导的蛋白水解(ko04120)和RNA转运(ko03013)等遗传信息进程相关通路,Toll和Imd信号通路(ko04624)等有机系统相关通路,2-氧代羧酸代谢(ko01210)和昆虫激素的生物合成(ko00981)等与代谢相关通路(图4)。
2.5 中蜂幼虫肠道miRNA及其靶基因的调控网络
共有156个miRNA靶向注释在Wnt、Hippo、Notch和mTOR等发育相关通路的67个靶基因;在miRNA-靶基因调控网络中,有95个miRNA靶向的33个靶基因涉及Wnt信号通路,76个miRNA靶向的22个靶基因涉及Hippo信号通路,22个miRNA靶向的7个靶基因涉及Notch信号通路,31个miRNA靶向的7个靶基因涉及mTOR信号通路(图5)。进一步分析发现,miR-466-x和miR-1277-x等5个miRNA的靶基因同时注释到Wnt、Hippo、Notch和mTOR信号通路,miR-9394-y和miR-466-y等6个miRNA的靶基因同时注释到Wnt、Hippo和mTOR信号通路,miR-3759-y、miR-6873-y和miR-6528-x的靶基因同时注释到Wnt、Hippo和Notch信号通路,miR-6001-y和miR-8109-x等17个miRNA的靶基因同时注释到Wnt和Hippo信号通路,bantam-y和miR-195-x等9个miRNA的靶基因同时注释到Wnt和Notch信号通路,miR-4787-x和novel-m0035-3p等5个miRNA的靶基因同时注释到Wnt和mTOR信号通路,miR-228-x、miR-263-x和miR-183-x的靶基因同时注释到Hippo和mTOR信号通路,miR-6538-x的靶基因同时注释到Notch和mTOR信号通路(图5)。
此外,共有145个miRNA靶向Toll、Imd/JNK、Jak-STAT和抗菌效应因子等免疫通路相关的21个靶基因,88个miRNA靶向Toll信号通路相关的7个基因,60个miRNA靶向Imd/JNK信号通路相关的7个基因,11个miRNA靶向Jak-STAT信号通路相关的2个基因,14个miRNA靶向抗菌效应因子信号通路相关的5个基因(图6)。进一步分析发现,miR-466-y和miR-1277-x等4个miRNA的靶基因同时注释到Toll、Imd/JNK和抗菌效应因子信号通路,miR-235-y和miR-92-y的靶基因同时注释到Toll、Imd/JNK和Jak-STAT信号通路,miR-283-x和miR-3759-y等16个miRNA的靶基因同时注释到Toll和Imd/JNK信号通路,miR-4206-y和miR-6516-y等6个miRNA的靶基因同时注释到Toll和Jak-STAT信号通路,miR-2480-x和novel-m0032-5p等4个miRNA的靶基因同时注释到Toll和抗菌效应因子信号通路,miR-25-y和miR-653-x的靶基因同时注释到Imd/JNK和Jak-STAT信号通路,novel-m0033-5p的靶基因同时注释到Imd/JNK和抗菌效应因子信号通路(图6)。
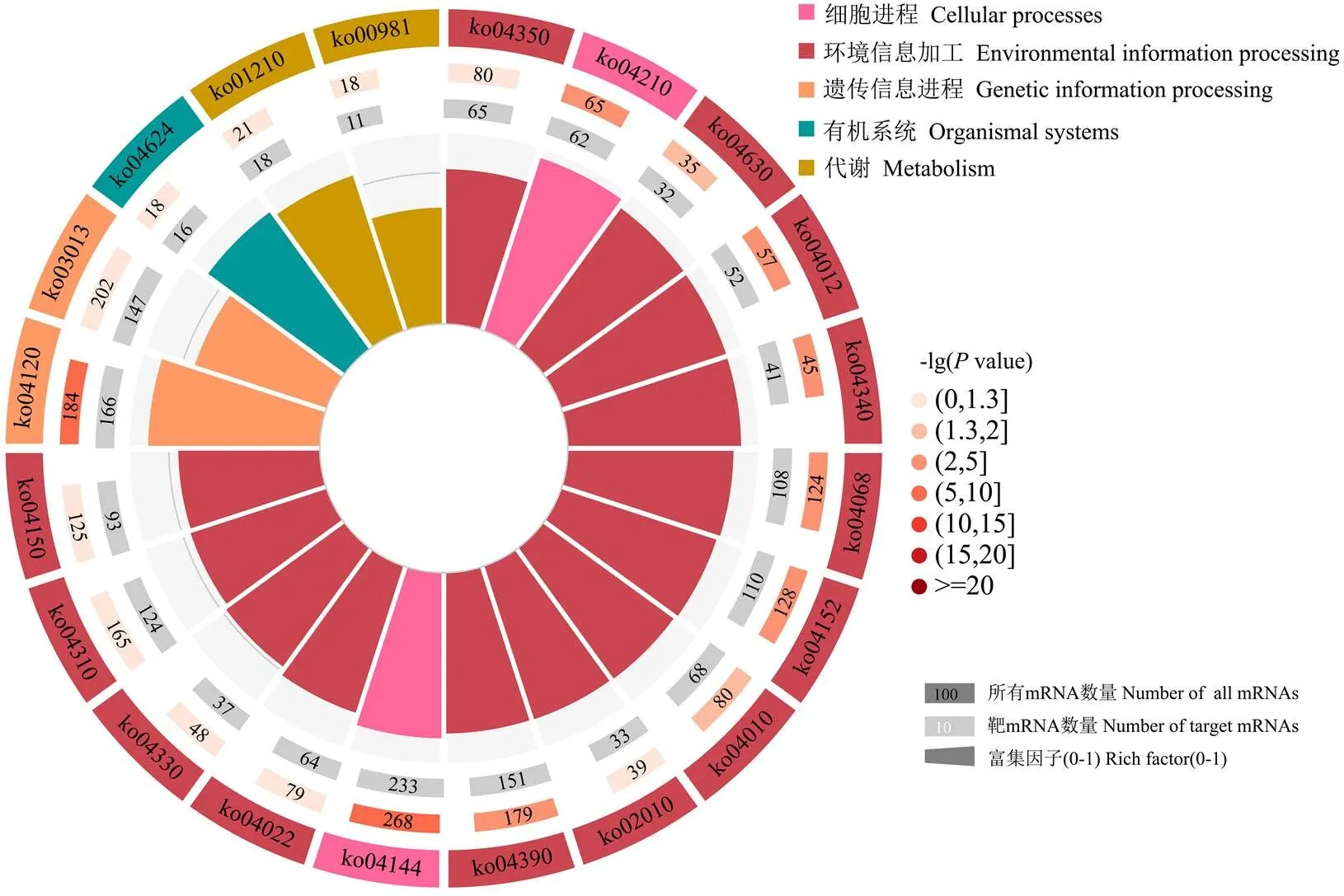
图4 中蜂幼虫肠道miRNA靶向mRNA的KEGG通路富集圈图

图5 中蜂幼虫肠道miRNA及其靶向发育相关基因的调控网络

图6 中蜂幼虫肠道miRNA及其靶向免疫相关通路基因的调控网络
3 讨论
3.1 鉴定到的中蜂miRNA丰富了东方蜜蜂的miRNA信息
东方蜜蜂与西方蜜蜂同属于蜜蜂科蜜蜂属,是亲缘关系较近的两个蜂种。西方蜜蜂的miRNA研究起步较早且研究较多,但有关东方蜜蜂的miRNA研究比较滞后且信息匮乏。笔者团队前期在意蜂4、5和6日龄幼虫肠道中共鉴定到560个miRNA,包括515个保守miRNA和45个新miRNA;其中有331个miRNA为3个日龄幼虫肠道共有且稳定表达[7]。本研究利用sRNA-seq技术对中蜂4、5和6日龄幼虫肠道进行测序,通过生物信息学分析方法鉴定到371个保守miRNA和64个新miRNA,略少于意蜂幼虫肠道内鉴定得到的保守miRNA[7],其中257个miRNA为中蜂4、5和6日龄幼虫肠道共有且稳定表达。进一步对本研究鉴定到的中蜂幼虫肠道miRNA与前期鉴定到的意蜂幼虫肠道miRNA进行比较分析,发现有多达174个中蜂幼虫肠道miRNA(如miR-8-y、bantam-y、miR-275-y、miR-9-z和miR-12-z等)的核酸序列与意蜂幼虫肠道内鉴定到的保守miRNA核酸序列[7]完全一致,表明这些miRNA在亲缘关系较近的两个蜂种中具有较强的保守性。这暗示上述保守miRNA在中蜂幼虫肠道和意蜂幼虫肠道中可能发挥相近的调控功能。此外,笔者发现中蜂miRNA长度介于18—25 nt,并主要富集在18 nt和22 nt,且首位碱基主要偏向于U,与意蜂及其他昆虫的miRNA结构特征相似[14,18]。前期研究发现意蜂幼虫肠道miRNA潜在调控Wnt、Hippo、FoxO和Hedgehog信号通路相关靶mRNA[7]。本研究中,中蜂miRNA与涉及内吞作用、细胞凋亡、泛素介导的蛋白水解、Toll和Imd、FoxO和mTOR信号通路等相关的靶mRNA具有潜在调控关系,可形成较复杂的调控网络。这说明在意蜂幼虫肠道和中蜂幼虫肠道中miRNA均发挥广泛的调控作用。本研究鉴定到的miRNA较好地补充了中蜂的miRNA信息,也为东方蜜蜂其他亚种的miRNA研究提供了宝贵的数据资源。
3.2 miR-306在中蜂和其他昆虫中具有很高的保守性
较多的研究表明miRNA在不同物种中具有较高的保守性[19]。通过与miRBase数据库中收录的miRNA相比较,笔者发现中蜂miR-306-x的5′端第2—8位种子区碱基序列(CAGGTAC)与黑腹果蝇dme-miR-306-5p、家蚕bmo-miR-306a-5p、西方蜜蜂ame-miR-306-5p和豌豆长管蚜()api-miR-306的种子区碱基序列完全一致,表明中蜂和上述几种昆虫的miR-306具有很高的保守性。研究表明miR-306负向调控,并影响黑腹果蝇的翅脉形成过程[20];miR-306在意蜂处女蜂王和产卵蜂王卵巢中差异表达,并可能参与卵巢激活过程[21]。miR-306-x在中蜂体内的生物学功能尚不清楚,可作为候选靶标用于下一步的功能研究。
3.3 中蜂幼虫肠道miRNA潜在参与调节生长发育相关生命进程
昆虫等真核生物可通过复杂而精密的信号转导途径调控各类生命活动[22]。Wnt、Hippo、mTOR及Notch等关键信号通路上的多个基因已被证实参与昆虫的生长发育及器官形成等过程[23-25]。飞蝗()miR-2/13/71簇通过抑制靶基因的表达,降低卵黄原蛋白(Vg)mRNA的水平,进而影响卵巢的发育和生殖[24]。植物花粉来源的miR-162a可靶向抑制西方蜜蜂的和黑腹果蝇的,进而延缓虫体的生长发育过程[25]。本研究发现共有156个miRNA靶向Wnt、Hippo、mTOR及Notch信号通路相关的67个基因,此外许多miRNA潜在调控两条及以上发育相关通路。在前期关于意蜂幼虫肠道和工蜂中肠的miRNA相关研究中,发现ame-bantam、ame-miR-14和ame-miR-8等大量miRNA参与对意蜂幼虫肠道内Wnt信号通路相关基因的调控[7]。ame-miR-6001-3p在意蜂工蜂肠道发育过程中差异表达,并与Hippo信号通路相关基因存在靶向调控关系[8]。这表明miRNA作为关键调控因子,可能通过调控靶基因表达参与关键信号通路的调节,进而影响中蜂幼虫肠道发育过程。
3.4 中蜂幼虫肠道miRNA潜在参与调控免疫途径应答病原侵染
在与病原的长期协同进化与相互作用中,蜜蜂进化出多条细胞和体液免疫途径以应答和抵御病原侵染,其中Toll、Imd、JNK和Jak-STAT等体液免疫途径可通过激活抗菌肽基因的表达增强defensin、hymenoptaecin、apidaecin、abaecin和apisimin的合成与释放,并与体内多酚氧化酶(PPO)和溶菌酶(Lys)共同参与对多种病原的免疫过程[26-27]。LOURENÇO等[28]研究发现,ame-miR-2、ame-miR-184和ame-bantam等miRNA在被黏质沙雷氏菌()和藤黄微球菌()接种感染的西方蜜蜂工蜂体内显著差异表达,可能通过共同调控Imd、JNK和Toll等体液免疫途径,促进抗菌肽的合成以应答细菌的感染。本研究中,中蜂幼虫肠道的145个miRNA与Toll、Imd/JNK、Jak-STAT和抗菌效应因子等免疫途径相关的21个基因存在靶向结合关系,例如miR-9-z靶向Imd/JNK信号通路上的(),miR-184-y靶向调控Toll信号通路上的(),miR-283-x同时靶向通路Toll信号通路上的()和();此外,许多miRNA潜在调控两条及以上免疫途径。上述结果表明相关miRNA与中蜂幼虫肠道免疫防御的潜在调控关系。鉴于蜜蜂幼虫肠道不仅是重要的免疫器官,也是其与蜜蜂球囊菌()等病原互作的主要部位[29]。上述潜在调控中蜂幼虫肠道免疫防御的miRNA值得进一步深入研究。
4 结论
在中蜂幼虫肠道中鉴定到371个保守miRNA和64个新miRNA,这些miRNA具有与其他昆虫miRNA类似的结构特征;通过分子生物学手段验证了6个miRNA的表达和序列真实性;上述miRNA潜在调控Wnt、Hippo、mTOR和Notch等发育相关通路,以及Toll、Imd/JNK、Jak-STAT和抗菌效应因子等免疫途径。
[1] HUSSAIN M, ASGARI S. MicroRNAs as mediators of insect host-pathogen interactions and immunity. Journal of Insect Physiology, 2014, 70: 151-158.
[2] ASGARI S. MicroRNA functions in insects. Insect Biochemistry and Molecular Biology, 2013, 43(4): 388-397.
[3] 杨婕, 谢苗, 徐雪娇, 白建林, 尤民生. 昆虫miRNA研究进展. 昆虫学报, 2021, 64(2): 259-280.
YANG J, XIE M, XU X J, BAI J L, YOU M S. Research progress of insect miRNAs. Acta Entomologica sinica, 2021, 64(2): 259-280. (in Chinese)
[4] 施腾飞, 余林生, 刘芳, 宗超, 汪天澍. 蜜蜂microRNA的研究进展. 昆虫学报, 2014, 57(5): 601-606.
SHI T F, YU L S, LIU F, ZONG C, WANG T S. Progress in microRNAs in honey bees. Acta Entomologica sinica, 2014, 57(5): 601-606. (in Chinese)
[5] LIU F, SHI T, YIN W, SU X, QI L, HUANG Z Y, ZHANG S, YU L. The microRNA ame-miR-279a regulates sucrose responsiveness of forager honey bees (). Insect Biochemistry and Molecular Biology, 2017, 90: 34-42.
[6] CRISTINO A S, BARCHUK A R, FREITAS F C, NARAYANAN R K, BIERGANS S D, ZHAO Z, SIMOES Z L, REINHARD J, CLAUDIANOS C. Neuroligin-associated microRNA-932 targets actin and regulates memory in the honeybee. Nature communications, 2014, 5: 5529.
[7] 熊翠玲, 杜宇, 陈大福, 郑燕珍, 付中民, 王海朋, 耿四海, 陈华枝, 周丁丁, 吴素珍, 石彩云, 郭睿. 意大利蜜蜂幼虫肠道的miRNAs的生物信息学预测及分析. 应用昆虫学报, 2018, 55(6): 1023-1033.
XIONG C L, DU Y, CHEN D F, ZHENG Y Z, FU Z M, WANG H P, GENG S H, CHEN H Z, ZHOU D D, WU S Z, SHI C Y, GUO R. Bioinformatic prediction and analysis of miRNAs in thelarval gut. Chinese Journal of Applied Entomology, 2018, 55(6): 1023-1033. (in Chinese)
[8] 杜宇, 范小雪, 蒋海宾, 王杰, 范元婵, 祝智威, 周丁丁, 万洁琦, 卢家轩, 熊翠玲, 郑燕珍, 陈大福, 郭睿. 微小RNA及其介导的竞争性内源RNA调控网络在意大利蜜蜂工蜂中肠发育过程中的潜在作用. 中国农业科学, 2020, 53(12): 2512-2526.
DU Y, FAN X X, JIANG H B, WANG J, FAN Y C, ZHU Z W, ZHOU D D, WAN J Q, LU J X, XIONG C L, ZHENG Y Z, CHEN D F, GUO R. The potential role of microRNAs and microRNA-mediated competing endogenous networks during the developmental process ofworker’s midgut. Scientia Agricultura Sinica, 2020, 53(12): 2512-2526. (in Chinese)
[9] 吴小波, 王子龙, 石元元, 张飞, 曾志将. 婚飞对中华蜜蜂性成熟处女蜂王sRNAs表达的影响. 中国农业科学, 2013, 46(17): 3721-3728.
WU X B, WANG Z L, SHI Y Y, ZHANG F, ZENG Z J. Effects of mating flight on sRNAs expression in sexual matured virgin queens (). Scientia Agricultura Sinica, 2013, 46(17): 3721-3728. (in Chinese)
[10] 杜宇, 童新宇, 周丁丁, 陈大福, 熊翠玲, 郑燕珍, 徐国钧, 王海朋, 陈华枝, 郭意龙, 隆琦, 郭睿. 中华蜜蜂幼虫肠道响应球囊菌胁迫的microRNA应答分析. 微生物学报, 2019, 59(9): 1747-1764.
DU Y, TONG X Y, ZHOU D D, CHEN D F, XIONG C L, ZHENG Y Z, XU G J, WANG H P, CHEN H Z, GUO Y L, LONG Q, GUO R. MicroRNA responses in the larval gut oftostress. Acta Microbiologica Sinica, 2019, 59(9):1747-1764. (in Chinese)
[11] CHEN D F, DU Y, CHEN H Z, FAN Y C, FAN X X, ZHU Z W, WANG J, XIONG C L, ZHENG Y Z, HOU C S, DIAO Q Y, GUO R. Comparative identification of microRNAs inworkers’ midguts in response toinvasion. Insects, 2019, 10(9): 258.
[12] 郭睿, 杜宇, 熊翠玲, 郑燕珍, 付中民, 徐国钧, 王海朋, 陈华枝, 耿四海, 周丁丁, 石彩云, 赵红霞, 陈大福. 意大利蜜蜂幼虫肠道发育过程中的差异表达microRNA及其调控网络. 中国农业科学, 2018, 51(21): 4197-4209.
GUO R, DU Y, XIONG C L, ZHENG Y Z, FU Z M, XU G J, WANG H P, CHEN H Z, GENG S H, ZHOU D D, SHI C Y, ZHAO H X, CHEN D F. Differentially expressed microRNA and their regulation networks during the developmental process oflarval gut. Scientia Agricultura Sinica, 2018, 51(21): 4197-4209. (in Chinese)
[13] 郭睿, 杜宇, 童新宇, 熊翠玲, 郑燕珍, 徐国钧, 王海朋, 耿四海, 周丁丁, 郭意龙, 吴素珍, 陈大福. 意大利蜜蜂幼虫肠道在球囊菌侵染前期的差异表达microRNA及其调控网络. 中国农业科学, 2019, 52(1): 166-180.
GUO R, DU Y, TONG X Y, XIONG C L, ZHENG Y Z, XU G J, WANG H P, GENG S H, ZHOU D D, GUO Y L, WU S Z, CHEN D F. Differentially expressed microRNAs and their regulation networks inlarval gut during the early stage ofinfection. Scientia Agricultura Sinica, 2019, 52(1): 166-180. (in Chinese)
[14] 陈华枝, 熊翠玲, 祝智威, 王杰, 范小雪, 蒋海宾, 范元婵, 万洁琦, 卢家轩, 郑燕珍, 付中民, 徐国钧, 陈大福, 郭睿. 基于small RNA组学分析揭示意大利蜜蜂响应东方蜜蜂微孢子虫胁迫的免疫应答机制. 微生物学报, 2020, 60(7): 1458-1478.
CHEN H Z, XIONG C L, ZHU Z W, WANG J, FAN X X, JIANG H B, FAN Y C, WAN J Q, LU J X, ZHENG Y Z, FU Z M, XU G J, CHEN D F, GUO R. Unraveling the mechanism underlying the immune responses oftostress based on small RNA omics analyses. Acta Microbiologica Sinica, 2020, 60(7): 1458-1478. (in Chinese)
[15] 熊翠玲, 杜宇, 冯睿蓉, 蒋海宾, 史小玉, 王海朋, 范小雪, 王杰, 祝智威, 范元婵, 陈华枝, 周丁丁, 郑燕珍, 陈大福, 郭睿. 侵染中华蜜蜂6日龄幼虫的蜜蜂球囊菌的微小RNA差异表达谱及调控网络. 微生物学报, 2020, 60(5): 992-1009.
XIONG C L, DU Y, FENG R R, JIANG H B, SHI X Y, WANG H P, FAN X X, WANG J, ZHU Z W, FAN Y C, CHEN H Z, ZHOU D D, ZHENG Y Z, CHEN D F, GUO R. Differential expression pattern and regulation network of microRNAs ininvading6-day-old larvae. Acta Microbiologica Sinica, 2020, 60(5): 992-1009. (in Chinese)
[16] 耿四海, 石彩云, 范小雪, 王杰, 祝智威, 蒋海宾, 范元婵, 陈华枝, 杜宇, 王心蕊, 熊翠玲, 郑燕珍, 付中民, 陈大福, 郭睿. 微小RNA介导东方蜜蜂微孢子虫侵染意大利蜜蜂工蜂的分子机制. 中国农业科学, 2020, 53(15): 3187-3204.
GENG S H, SHI C Y, FAN X X, WANG J, ZHU Z W, JIANG H B, FAN Y C, CHEN H Z, DU Y, WANG X R, XIONG C L, ZHENG Y Z, FU Z M, CHEN D F, GUO R. The mechanism underlying microRNAs-mediatedinfection toworker. Scientia Agricultura Sinica, 2020, 53(15): 3187-3204. (in Chinese)
[17] 陈华枝, 祝智威, 蒋海宾, 王杰, 范元婵, 范小雪, 万洁琦, 卢家轩, 熊翠玲, 郑燕珍, 付中民, 陈大福, 郭睿. 蜜蜂球囊菌菌丝和孢子中微小RNA及其靶mRNA的比较分析. 中国农业科学, 2020, 53(17): 3606-3619.
CHEN H Z, ZHU Z W, JIANG H B, WANG J, FAN Y C, FAN X X, WAN J Q, LU J X, XIONG C L, ZHENG Y Z, FU Z M, CHEN D F, GUO R. Comparative analysis of microRNAs and corresponding target mRNAs inmycelium and spore. Scientia Agricultura Sinica, 2020, 53(17): 3606-3619. (in Chinese)
[18] XIAO S, WANG B, LI K, XIONG S, YE X, WANG J, ZHANG J, YAN Z, WANG F, SONG Q, STANLEY D W, YE G, FANG Q. Identification and characterization of miRNAs in an endoparasitoid wasp,. Archives of insect biochemistry and physiology, 2020, 103(2): e21633.
[19] ROUSH S, SLACK F J. The let-7 family of microRNAs. Trends in cell biology, 2008, 18(10): 505-516.
[20] SIMOES DA SILVA C J, SOSPEDRA I, APARICIO R, BUSTURIA A. The microRNA-306/abrupt regulatory axis controls wing and haltere growth in. Mechanisms of Development, 2019, 158: 103555.
[21] MACEDO L M, NUNES F M, FREITAS F C, PIRES C V, TANAKA E D, MARTINS J R, PIULACHS M D, CRISTINO A S, PINHEIRO D G, SIMÕES Z L. MicroRNA signatures characterizing caste- independent ovarian activity in queen and worker honeybees (L.). Insect Molecular Biology, 2016, 25(3): 216-226.
[22] KLEINO A, SILVERMAN N. Regulation of theImd pathway by signaling amyloids. Insect biochemistry and molecular biology, 2019, 108: 16-23.
[23] GOU J, LIN L, OTHMER H G. A model for the Hippo pathway in thewing disc. Biophysical journal, 2018, 115(4): 737-747.
[24] SONG J, LI W, ZHAO H, ZHOU S. Clustered miR-2, miR-13a, miR-13b and miR-71 coordinately targetgene to regulate oogenesis of the migratory locust. Insect biochemistry and molecular biology, 2019, 106: 39-46.
[25] ZHU K, LIU M, FU Z, ZHOU Z, KONG Y, LIANG H, LIN Z, LUO J, ZHENG H, WAN P,. Plant microRNAs in larval food regulate honeybee caste development. PLoS genetics, 2017, 13(8): e1006946.
[26] Hu Y T, Wu T C, Yang E C, Wu P C, Lin P T, Wu Y L. Regulation of genes related to immune signaling and detoxification inby an inhibitor of histone deacetylation. Scientific reports, 2017, 7: 41255.
[27] BRUTSCHER L M, DAUGHENBAUGH K F, FLENNIKEN M L. Antiviral defense mechanisms in honey bees. Current opinion in insect science, 2015, 10: 71-82.
[28] LOURENÇO A P, GUIDUGLI-LAZZARINI K R, FREITAS F C, BITONDI M M, SIMÕES Z L. Bacterial infection activates the immune system response and dysregulates microRNA expression in honey bees. Insect Biochemistry and Molecular Biology, 2013, 43(5): 474-482.
[29] 李江红, 郑志阳, 陈大福, 梁勤. 影响蜜蜂球囊菌侵染蜜蜂幼虫的因素及侵染过程观察. 昆虫学报, 2012, 55(7): 790-797.
LI J H, ZHENG Z Y, CHEN D F, LIANG Q. Factors influencinginfection on honeybee larvae and observation on the infection process. Acta Entomologica sinica, 2012, 55(7): 790-797. (in Chinese)
Identification and analysis of microRNAs in the larval gut of
FENG RuiRong1, FU ZhongMin1,2, DU Yu1, ZHANG WenDe1, FAN XiaoXue1, WANG HaiPeng1, WAN JieQi1, ZHOU ZiYu1, KANG YuXin1, CHEN DaFu1,2, GUO Rui1,2, SHI PeiYing1
1College of Animal Sciences (College of Bee Science), Fujian Agriculture and Forestry University, Fuzhou 350002;2Apitherapy Research Institute, Fujian Agriculture and Forestry University, Fuzhou 350002
【】In this study, transcriptome-wide identification and analysis of miRNAs in the larval guts ofwas conducted using a combination of small RNA-seq (sRNA-seq) technology and bioinformatic method, aiming to enrich the information ofmiRNAs and offer a basis for further investigation of miRNA-regulated molecular mechanism underlyinglarval gut development.【】Gut samples of4-, 5-, and 6-day-old larvae (Ac1, Ac2, and Ac3 ) were sequenced using sRNA-seq technology, and clean tags were obtained after quality control. By using Blast tool, clean tags were continuously mapped togenome and miRBase database to identify known miRNAs and novel miRNAs. TPM method was used to perform normalization of miRNAs’ expression. The ratio of sRNAs, length distribution of miRNAs and first base bias were calculated with GraphPad Prism 7 software. Using related software, target mRNAs of above-mentioned miRNAs were predicted followed by GO and KEGG database annotation. Further, regulatory networks between genes associated with development and immune-related pathways and corresponding target miRNAs were constructed and analyzed, followed by visualization of regulatory networks with Cytoscape software. The authenticity of miRNA expression and sequence was verified by using Stem-loop RT-PCR, molecular cloning and Sanger sequencing.【】In total, 371 known miRNAs and 64 novel ones ofwere identified; their length was distributed among 18-25 nt, and the first base had an U bias. The aforementioned miRNAs could target 14 750 mRNAs, involving 2 270 GO terms such as ion binding, metal ion binding, membrane, membrane part and single-organism process, as well as 332 KEGG pathways including endocytosis, apoptosis, mTOR signaling pathway, RNA transport and insect hormone biosynthesis. Further investigation suggested that 156 miRNAs could target 67 genes relative to development-associated pathways such as Wnt, Hippo, Notch and mTOR signaling pathways, while 145 miRNAs could target 21 genes relevant to immune-associated pathways such as Toll, Imd/JNK, Jak-STAT signaling pathways and antimicrobial effectors. Stem-loop RT-PCR result indicated that specific fragments with expected sizes could be amplified from miR-8-y, miR-9-z, miR-14-y, miR-281-y, miR-283-x and miR-306-x; Sanger sequencing result demonstrated that sequences of above-mentioned six miRNAs were in accordance with those in deep sequencing result.【】Our findings provide number, structural characteristics and expression profile ofmiRNAs, and unraveled that miRNAs inlarval gut potentially regulate a lot of life processes and cellular activities, part of miRNAs can participate in regulation of development-related and immune-related pathways by targeting corresponding mRNAs.
;; larva; gut;microRNA; regulatory network

10.3864/j.issn.0578-1752.2022.01.017
2021-04-28;
2021-06-07
国家现代农业产业技术体系建设专项(CARS-44-KXJ7)、福建农林大学杰出青年科研人才计划(xjq201814)、福建农林大学硕士生导师团队项目(郭睿)、福建省大学生创新创业训练计划(202010389012,202010389016)
冯睿蓉,E-mail:frr18705911634@163.com。付中民,E-mail:369699776@qq.com。冯睿蓉和付中民为同等贡献作者。通信作者史培颖,E-mail:peiyshi@126.com。通信作者郭睿,E-mail:ruiguo@fafu.edu.cn
(责任编辑 岳梅)
