基于TCGA数据库代谢相关基因构建乳腺癌预后模型
2022-01-28李伟华张广凤马骊骊何品王雯李顶夫
李伟华 张广凤 马骊骊 何品 王雯 李顶夫
乳腺癌是全球女性最常见的恶性肿瘤之一,其发病率逐年上升,2020年已超过肺癌成为全球第一大癌症[1]。乳腺癌是一组高度异质性的疾病,每个病例的临床表现、形态学、分子特性、治疗效果及预后差异很大[2]。临床上,常用肿瘤分期、组织学分级和分子亚型作为评估乳腺癌患者的预后因素。然而,这些临床病理特征并不能准确地预测患者预后的信息。这可能导致对患者预后的判断不准确,一些低风险患者可能接受不必要或过度的治疗,而其他高风险患者可能因治疗不当而面临复发或转移[3]。因此,迫切需要寻找新的分子标记物来预测乳腺癌患者的预后,从而有利于患者的精确治疗。
肿瘤通过代谢为肿瘤细胞提供重要的能量来源,如以“有氧糖酵解”或“Warburg效应”为代表的糖代谢,可以增加大分子和中间产物的合成以维持肿瘤的增殖[4]。在肿瘤细胞内,产生了许多代谢的变化以满足肿瘤的能量和合成要求,这也成为肿瘤的一个重要特征[5]。本研究构建了一个结合多个乳腺癌代谢相关基因的模型来预测乳腺癌患者的预后。通过对癌症基因组图谱计划(The Cancer Genome Atlas Program,TCGA)数据库中乳腺癌代谢相关基因进行差异分析,筛选出代谢相关的差异基因,通过lasso回归去掉过度拟合的基因用作预后模型的构建。对高低风险两组进行生存比较,对风险得分进行了单因素和多因素Cox回归分析,计算出每个基因的风险系数和在样本中的表达情况。根据基因表达量分析不同风险分组的样本的生存状态。
1 材料与方法
1.1 数据收集
TCGA(https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga)下载了113例正常对照和1109例肿瘤样本的mRNA表达数据,并下载与之对应的患者临床信息用作预后分析。从京都基因与基因组百科全书(Kyoto Encyclopedia of Genes and Genomes,KEGG)(https://www.genome.jp/kegg/)下载41条代谢相关的通路,将其中的944个基因提取出来作为代谢相关基因。
1.2 预后相关的基因筛选
通过limma包对KEGG中提取的代谢基因进行差异分析,FDR<0.05,│logFC│>1作为筛选标准,并通过Cox回归筛选出P<0.05的预后基因。随机抽取70%的基因作为训练集,其余样本作为验证集,进一步通过Lasso回归防止过度拟合,13个代谢相关预后基因用作预后模型构建。
1.3 构建预后模型
利用Cox回归得到的代谢预后相关基因权重进行预后模型构建,通过风险得分中位值将样本分为高低风险两组,并利用survival包及survminer包绘制生存曲线。利用pheatmap包进行风险得分与生存状态之间的热图绘制。
风险得分=代谢相关风险基因表达值(FPKM)×风险系数
1.4 基因集富集分析(Gene set enrichment analysis,GSEA)
GSEA可以通过将预定义的基因集与特定表型进行比对从而找出与预定义基因集相关的表型。根据代谢相关预后基因的风险得分进行分组,并与KEGG的基因集进行比对,对代谢相关预后基因在乳腺癌中可能的作用机制进行探索。
1.5 统计分析
所有数据的统计分析均使用R软件(版本4.0.2),P值多重检验校正采用Benjamini & Hochberg(BH)方法,P<0.05认为具有统计学意义。
2 结果
2.1 代谢相关预后基因筛选
通过从TCGA数据库下载得到113例正常对照和1 109例乳腺癌患者样本的mRNA表达谱数据。从中提取KEGG通路中代谢相关的基因用作进一步差异分析。最终筛选出168个代谢相关的差异基因(图1)。并通过单因素Cox回归确定与预后相关的14个基因(图2)。
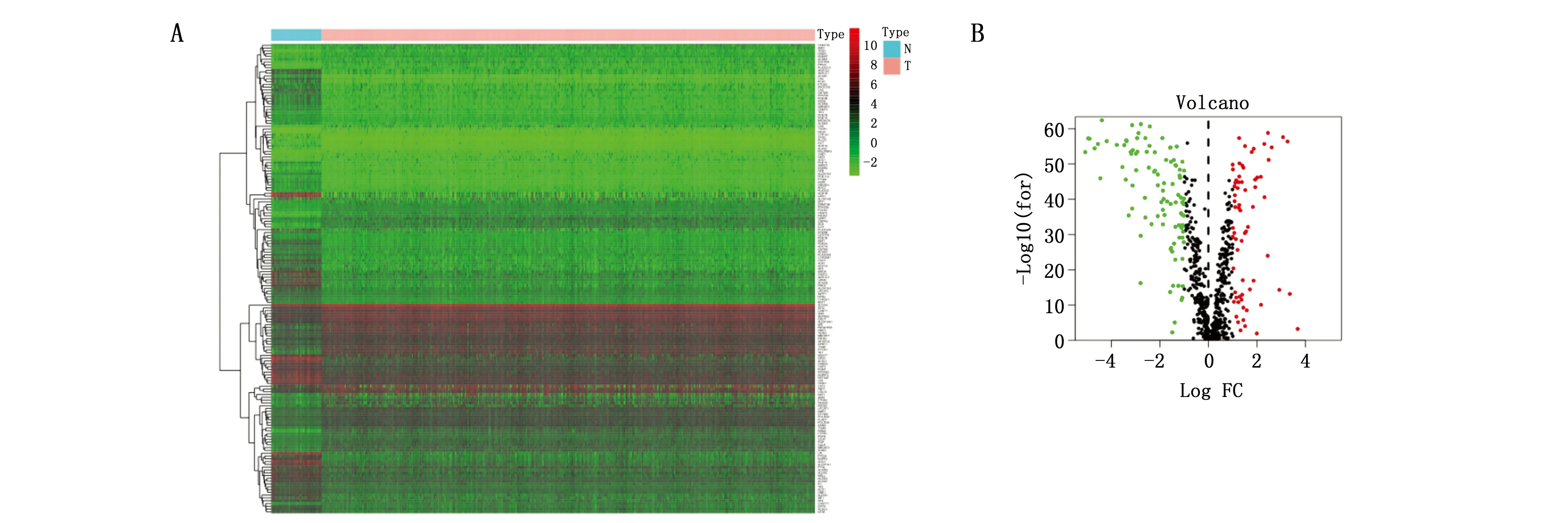
图1 代谢相关的差异表达基因Figure 1 Metabolism-related differentially expressed genesNote:A.Differential gene expression;B.Volcano map showing the differential gene distribution.
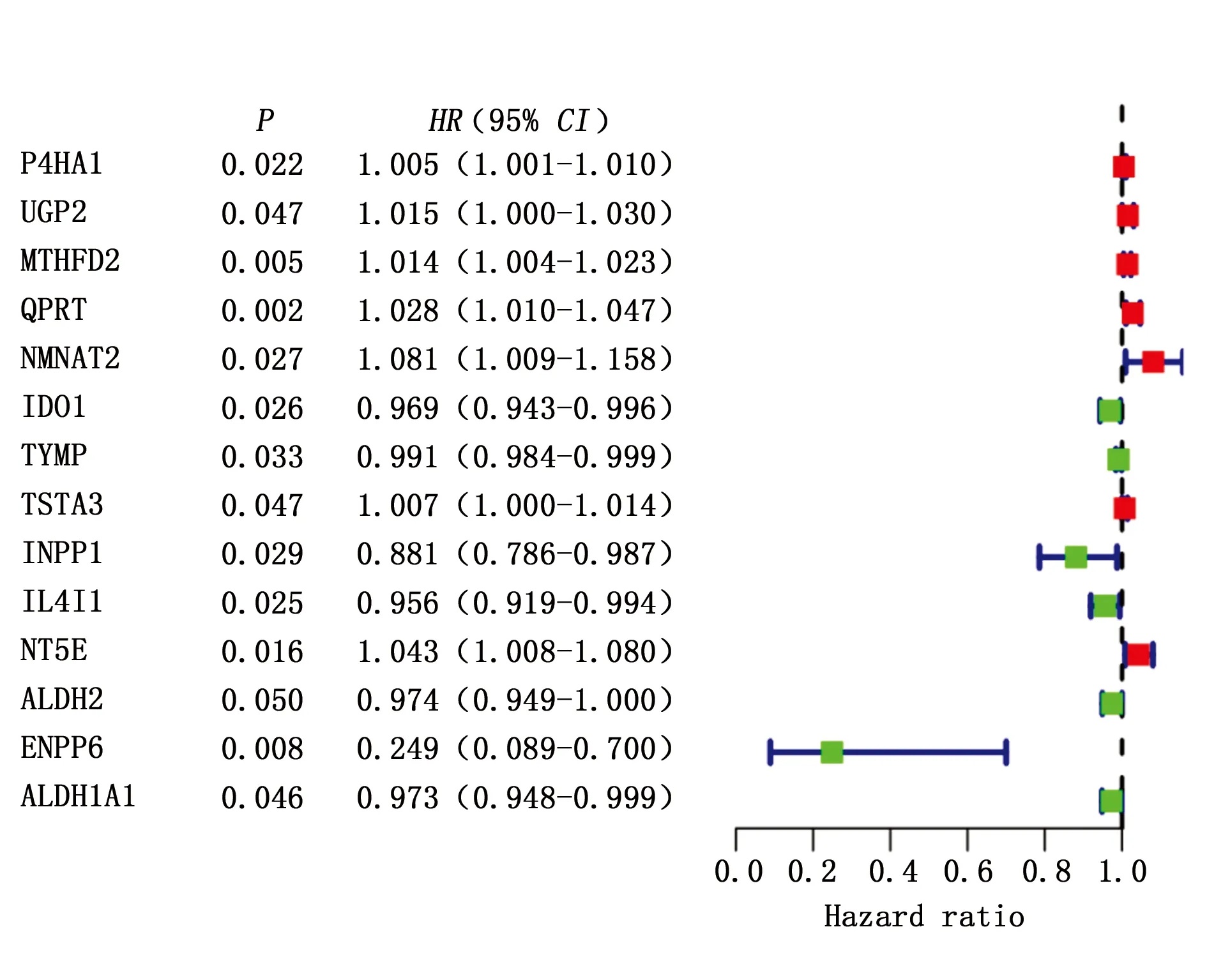
图2 森林图 Cox回归单因素分析筛选出7个高风险基因和7个保护性基因Figure 2 Forest map Cox regression single factor analysis screened out 7 high-risk genes and 7 protective genes
2.2 Lasso回归筛选关键基因及预后模型的构建
进一步通过lasso回归去掉过度拟合的基因,最终确定了13个基因(NMNAT2、NT5E、QPRT、UGP2、MTHFD2、TSTA3、TYMP、ALDH2、ALDH1A1、IDO1、IL4I1、INPP1和ENPP6)用作预后模型的构建(图3)。并计算出每个基因的风险系数(表1)。展示每个基因在样本中的表达情况(图4A)。根据基因表达量将样本分为高低风险两组,展示不同风险分组的样本的生存状态。随着风险得分的增加,相同时间节点,患者死亡率增高(图4B,C)。
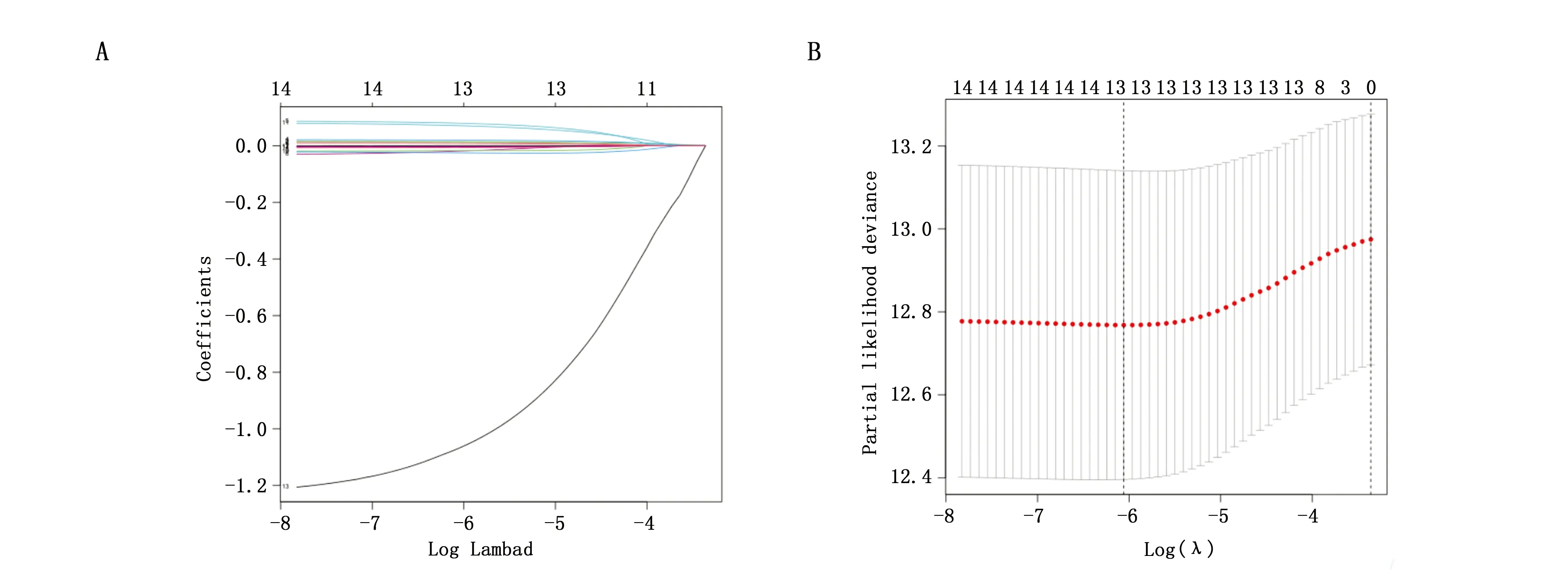
图3 比例风险模型构建Figure 3 The construction of proportional risk modelNote:A.Adjust the cross-validation of parameter selection in the proportional hazard model;B.LASSO coefficient spectrum of 14 genes in breast cancer.
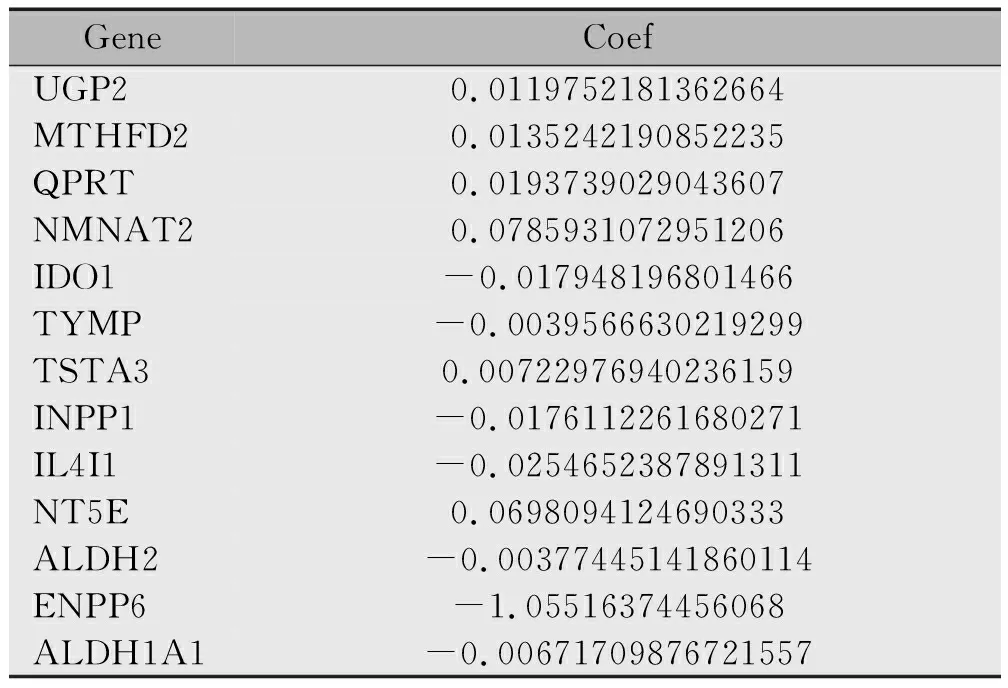
表1 风险基因的风险系数
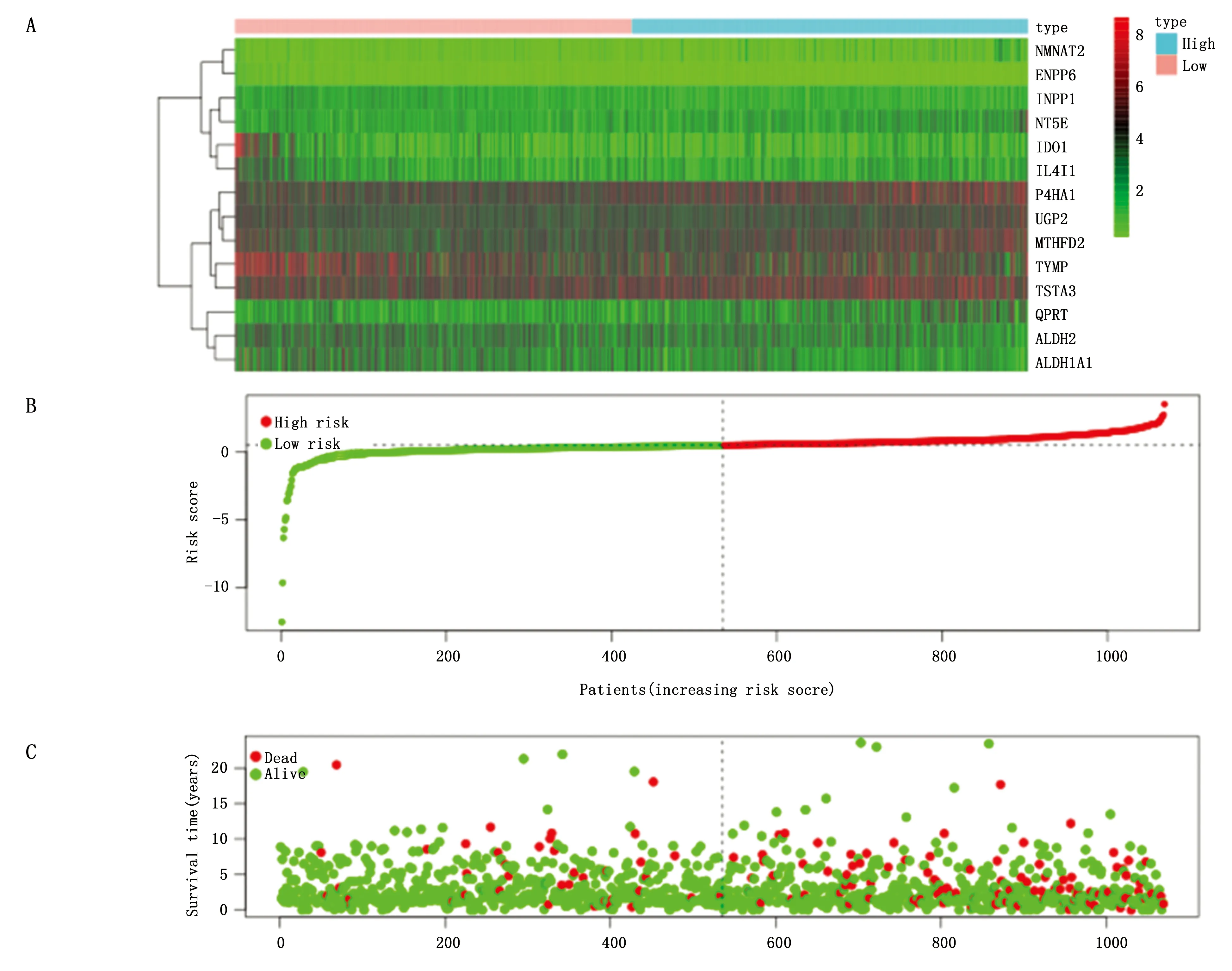
图4 高低风险分组基因表达及生存状态Figure 4 The high-and low-risk group gene expression and survival status of breast cancer patientsNote:A.The expression of risk genes in different groups;B.The distribution of risk scores in different groups;C.The distribution of survival status of patients in different groups.
2.3 预后模型的检验
通过对高低风险两组进行生存比较,发现高风险组患者的生存率更低(图5)。并对风险得分进行了单因素和多因素的Cox回归分析,结果表明,利用代谢相关基因构建的风险得分能够作为患者的一个独立危险因素去预测患者的预后(图6),并利用ROC曲线下面积去检验了风险得分去预测患者预后是较为准确的(图7)。
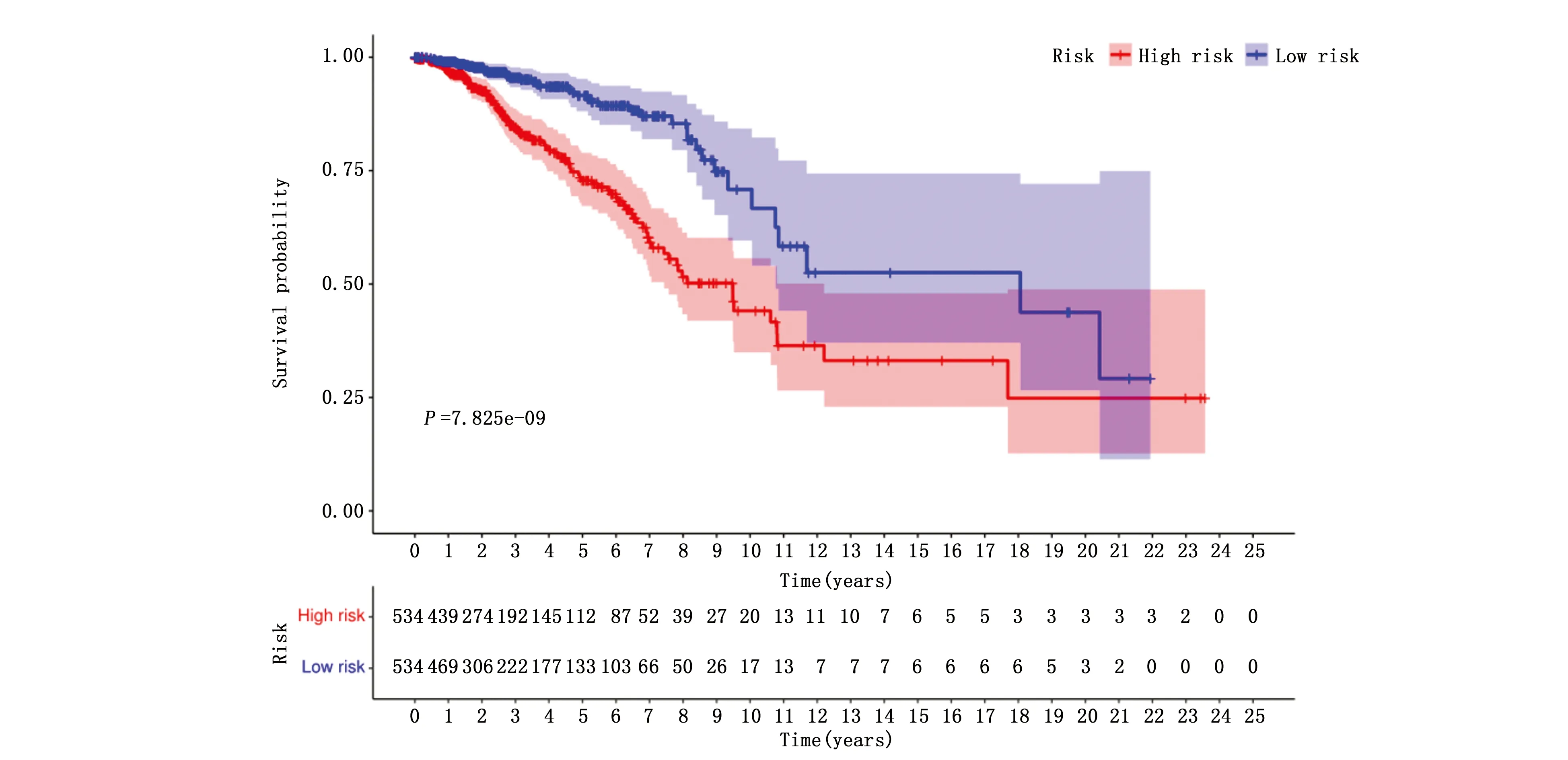
图5 高风险组乳腺癌患者生存时间更短Figure 5 A shorter survival time in breast cancer patients from the high-risk group
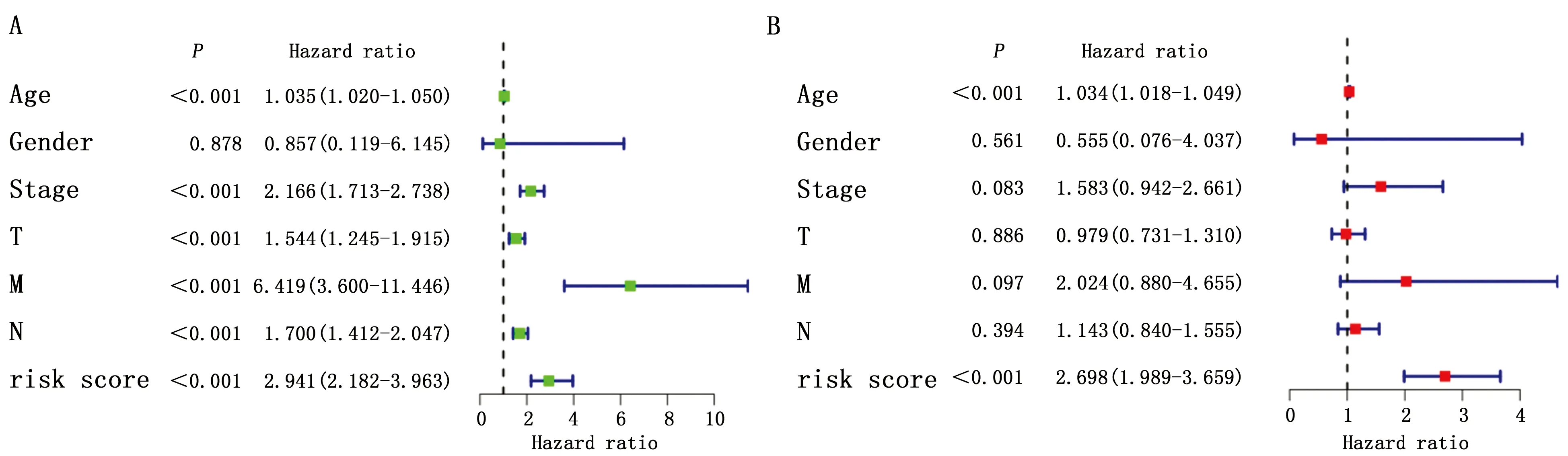
图6 单因素和多因素Cox回归确定风险得分为独立预后因素Figure 6 Univariate and multivariate Cox regression determined the risk score as an independent prognostic factor
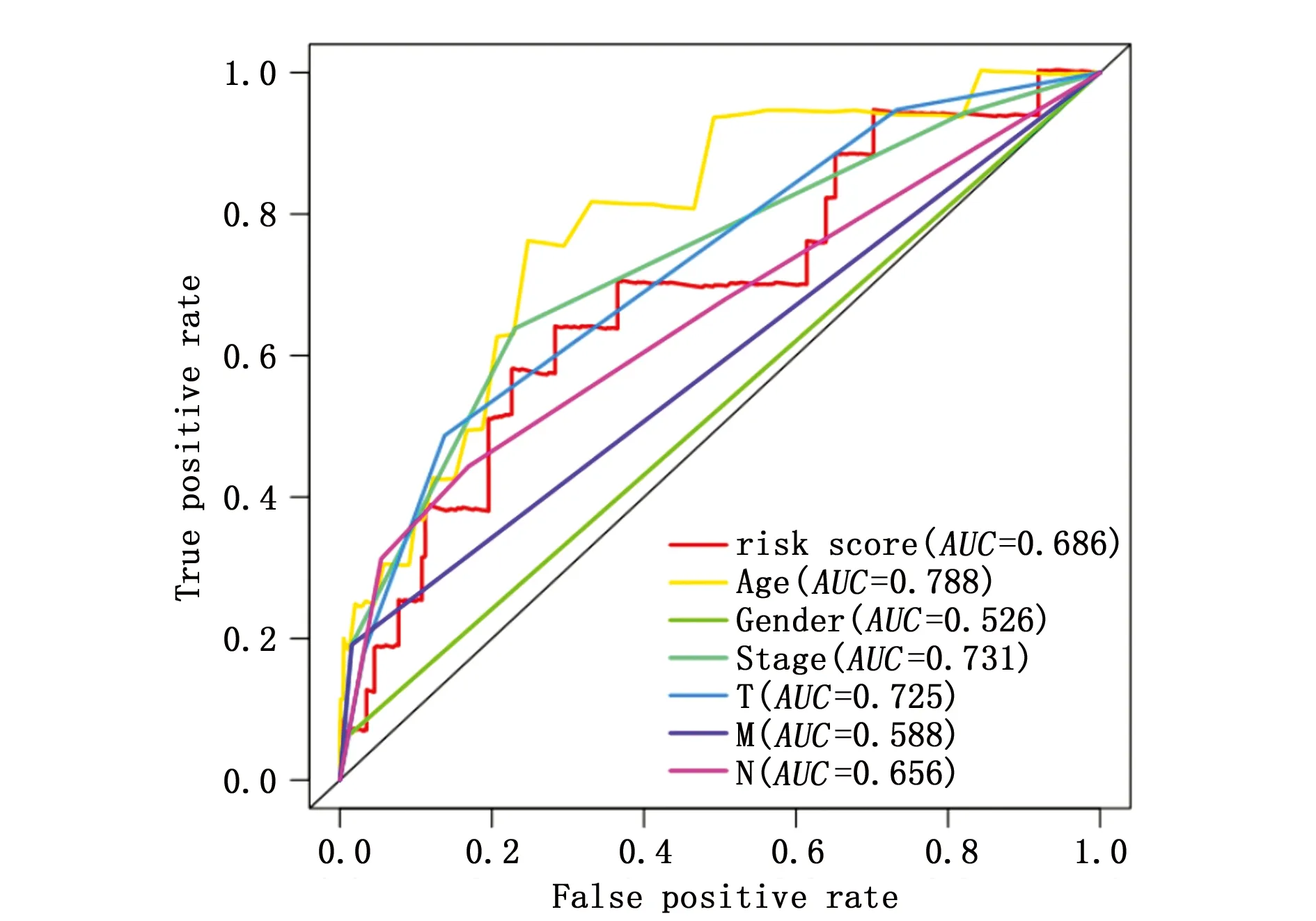
图7 风险评分对预后具有较高的预测率Figure 7 The risk score had a high predictive rate for prognosis
此外,根据风险得分进行GSEA分析(图8),发现其显著富集到代谢相关的通路(KEGG_ARGININE_AND_PROLINE_METABOLISM、KEGG_GLYCOLYSIS_GLUCONEOGENESIS、KEGG_PYRIMIDINE_METABOLISM、KEGG_STARCH_AND_SUCROSE_METABOLISM)。
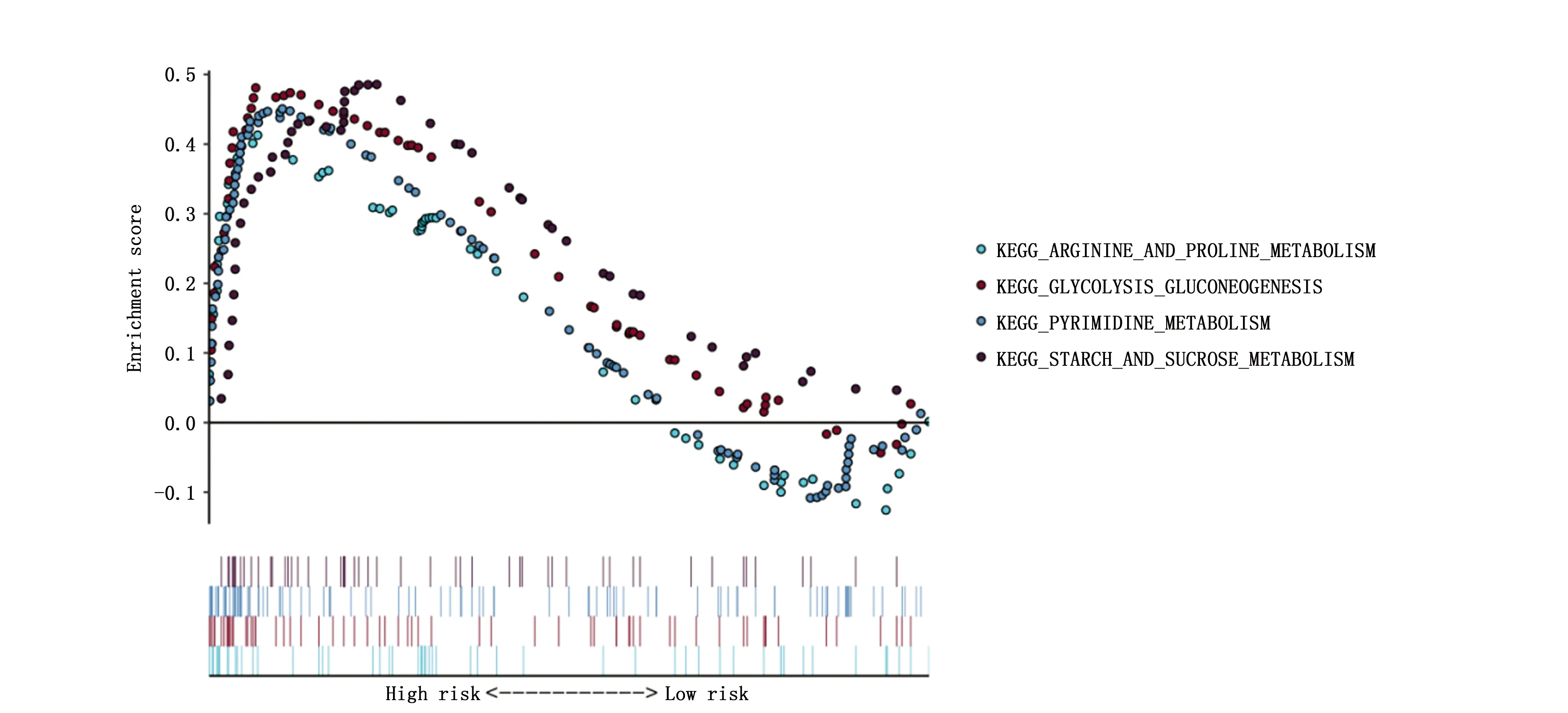
图8 风险得分相关基因显著富集在代谢相关通路Figure 8 Risk score-related genes were significantly enriched in metabolic pathways
3 讨论
肿瘤细胞为适应其对能量和物质的需求而发生了许多改变,而代谢变化成为其一个重要特征,对代谢相关基因的研究来判断肿瘤的特征和预后被越来越多的人所重视[6]。因此,本研究把代谢相关基因作为乳腺癌的预后的判断。本研究从TCGA数据库下载了乳腺癌患者和对照样本的mRNA表达谱数据。从中提取KEGG通路中代谢相关的基因用作进一步差异分析。最终筛选出168个代谢相关的差异基因。并通过单因素Cox回归确定与预后相关的13个基因。
用于构建预后模型的13个基因包括NMNAT2、NT5E、QPRT、UGP2、MTHFD2、TSTA3、TYMP、ALDH2、ALDH1A1、IDO1、IL4I1、INPP1和ENPP6。NMNAT2对合成烟酰胺腺嘌呤二核苷酸(NAD+)、调节细胞代谢具有重要的作用[7]。NMNAT2前体缺失的神经元减弱了小鼠他莫昔芬对葡萄糖代谢和胰岛素耐受的影响,最后证实NMNAT2参与脂质和糖代谢[8]。NT5E是一种糖基磷脂酰肌醇锚定的细胞表面蛋白,在细胞外单磷酸腺苷(AMP)分解代谢为腺苷的过程中起着关键作用,有研究表明NT5E基因甲基化与乳腺癌的发生有关,并与乳腺癌的不良预后因素相关[9]。NT5E表达变化和相关代谢组学变化也与肿瘤对包括铂类药物在内的几种化疗药物的敏感性相关[10]。QPRT催化色氨酸转移生成烟酸单核苷酸,这是关键辅酶烟酰胺腺嘌呤二核苷酸(NAD)从头生物合成的前体,对细胞呼吸链中的氧化磷酸化至关重要[11]。乳腺癌中DSCAM-AS1基因敲除降低了QPRT表达,从而抑制乳腺癌的生长、迁移和侵袭[12]。UGP2是一种由508个氨基酸残基组成的酶,在糖原生物合成中起着至关重要的作用[13]。MTHFD2是一种线粒体单碳代谢酶,已证明和包括乳腺癌在内的多种恶性肿瘤发展密切相关[14]。TSTA3岩藻糖合成酶在糖代谢的过程中发挥了重要的作用[15]。TSTA3在乳腺癌中是一种致癌基因,miR-125a-5p/miR-125b抑制乳腺癌细胞中TSTA3的表达,TSTA3通过调节CXCR4的表达来控制细胞增殖和侵袭[16]。TYMP是胸苷酸合成酶,通过促进胸苷酸合成而调节代谢过程。在乳腺癌的研究中发现TYMP高表达和化疗药卡培他滨反应显著相关[17]。ALDH2是一种乙醛脱氢酶,ALDH2多态性对乙醛代谢产生影响,具有ALDH2的Lys/Lys基因型的亚裔女性中,乳腺癌患病风险增加[18]。ALDH1A1可促进维生素A和视黄醇合成全反式维甲酸,ALDH1A1敲除的小鼠对饮食诱导的肥胖具有抵抗力[19]。ALDH1A1降低乳腺癌细胞内pH值,促进TAK1磷酸化,激活NFκB信号促进了肿瘤的生长[20]。IDO1是一种含血红素的酶,催化色氨酸分解为犬尿氨酸。IDO1已证明是女性癌症中重要一个免疫相关基因,参与了抗肿瘤免疫过程,并与突变负荷相关[21]。IL4I1催化苯丙氨酸氧化脱氨基生成苯丙酮酸,同时生成H2O2和NH3。IL4I1通过重塑乳腺癌细胞中的糖代谢来加强免疫治疗的潜力[22]。INPP1是一种使游离多磷酸肌醇去磷酸化的酶,参与磷脂酰肌醇信号通路的6种关键分子合成[23]。ENPP6对含有胆碱的化合物(如GPC22和O-磷酰胆碱N-酰基乙醇胺23)具有磷酸二酯酶活性,以产生磷酸胆碱[24]。
进一步计算每个基因的风险系数,展示每个基因在样本中的表达情况。根据基因表达量将样本分为高低风险两组,展示不同风险分组样本的生存状态。随着风险得分的增加,相同时间节点,患者死亡率增高。最后对高低风险两组进行生存比较,发现高风险组患者的生存率更低。对风险得分进行了单因素和多因素的Cox回归分析,结果表明,利用代谢相关基因构建的风险的得分能够作为患者的一个独立危险因素去预测患者的预后,利用ROC曲线下面积检验了风险得分预测患者预后的结果是较为准确的。根据风险得分进行GSEA分析,发现风险得分相关基因显著富集到代谢相关的通路。
综上所述,通过筛选得到的代谢相关基因显著影响着乳腺癌患者的代谢情况,进而对患者的预后产生影响。通过单因素和多因素Cox回归分析,得到影响乳腺癌患者预后的代谢相关基因,富集分析表明这些基因通过多种通路影响乳腺癌患者的预后情况,这为研究乳腺癌患者代谢与生存的关系提供了证据。研究构建的预后模型可以帮助临床医师对患者的预后提供有益的参考。
