茶树开花相关基因家族的克隆及CsMFT基因的可变剪切分析
2022-01-26夏华富代洪苇袁连玉童华荣
黄 瑞,夏华富,代洪苇,袁连玉,童华荣
(西南大学 食品科学学院,重庆 400715)
茶树[Camelliasinensis(L.) O. Kuntze]是双子叶植物山茶属多年生常绿木本经济植物,起源于中国西南地区。茶树叶片和茶树花分别作为茶树组织器官和生殖器官在生产和应用上起着重要作用,茶树叶片经加工后因其独特的风味和有益健康的特性受到广泛欢迎,茶树花也因为其丰富的内含成分应用于食品、医药、园艺鉴赏等领域[1]。茶树生长过程包括营养生长和生殖生长两种生长状态,这两种状态相互竞争水分和营养物质[2]。营养生长有益于茶树叶片的生长发育,获取高品质的茶叶,生殖生长有益于茶花生长发育,维持物种多样性,但生殖生长旺盛消耗茶树养分抑制营养生长,导致茶叶产量质量下降,降低经济效益[3]。开花诱导是植物从营养生长到生殖生长的开端,研究茶树成花调控机理和花芽分化相关机制有益于调控茶树营养生长和生殖生长,提高茶叶和茶树花的产量和品质,为茶叶经济发展以及良种选育提供依据。
植物开花是一个受多通路调节且连续、复杂的过程,植物进入成花诱导阶段,经信号传导后再进入成花决定状态,开花整合子激活花分生组织相关基因的表达,进行花芽分化和花器官发育形成[4]。开花诱导是植物从营养生长到生殖生长的重要过渡,研究表明植物开花诱导调控途径分为5类,分别是光周期途径(photoperiod pathway)、春化途径(vernilization pathway)、自主途径(autonomous pathway)、赤霉素途径(GA pathway)和年龄途径(aging pathway)[5-6]。其中,光周期、春化途径与受外界刺激相关,赤霉素、自主和年龄途径受到内源信号的调节[7]。拟南芥(Arabidopsisthaliana)开花调控途径和相关基因的研究较其他植物更为成熟,FT(FLOWERINGLOCUST)基因和TFL1(TERMINALFLOWER1)基因这一对同源基因最先在拟南芥中被鉴定出来[5],是拟南芥开花调控途径中的关键基因[8]。FT基因与TFL1基因属于PEBP基因家族,共同编码磷脂酰乙醇胺结合蛋白(phosphatidyl ethanolamine-binding protein, PEBP)。在被子植物中,PEBP蛋白家族成员既能参与开花过程的调控,也能参与植物的形态建成,可分为3个亚家族FT-like、MFT-like和TFL1-like,包括FT(FLOWERING LOCUS T)、TFL1(TERMINAL FLOWER 1)、TSF (TWIN SISTER OF FT)、BFT (BROTHER FT AND TFL1)、 ATC (ARABIDOPSIS THALIANA CENTRORADIALIS)及MFT (MOTHER OF FT AND TFL1)[9]。研究表明,在拟南芥中AtFT、AtTSF和AtMFT基因促进开花,而AtTFL1、AtATC和AtBFT基因抑制开花[10-13]。
FT基因编码的FT蛋白是“成花素”的主要构成部分,它可通过植物韧皮部被运输至茎顶端分生组织与FD蛋白作用,形成促使花分生组织表达的复合体,促进植物开花[14-15]。FT基因是成花调控多种途径的关键结合点,它整合了光周期、春化、赤霉素、自主等途径的诱导信号,是植物成花过程中重要的整合因子[16]。目前,FT基因在其他植物中也有相关研究,如小麦(Triticumaestivum)VRN3基因[17]、水稻(Oryzasativa)Hd3a基因[15]、苹果(Malusdomestica)MdFT1和MdDF2基因[18-19]、番茄(Solanumlycopersicum)SP基因和玉米(Zeamays)ZCN8基因[20]等。TFL1基因与植物营养生长相关,TFL1基因与FT基因具有高度相似性,但TFL1蛋白与FD蛋白作用,其功能与FT蛋白相拮抗,表现为抑制花原基形成,能够延迟开花[21-22]。TFS基因与FT基因也具有高相似性,其表达模式和组织表达部位相似,在拟南芥中TSF基因过表达使开花提前,表明TSF基因具有成花诱导作用[23]。MFT基因与FT基因功能相似,有较弱的开花诱导作用,但主要作用是通过赤霉素和脱落酸调控种子萌发[4, 24-25]。
可变剪切(alternative splicing, AS)是指不均一核RNA(hnRNA)转录位点被剪切体选择性剪切产生不同的mRNA异构体的现象[26]。在真核生物中普遍存在可变剪切现象,超过60%的外显子基因经可变剪切后形成不同的转录本类型,增加了蛋白质类型和功能的多样性[27]。可变剪切在植物生长中起重要作用,在转录后对调节植物的生长、发育、非生物胁迫的响应起作用,有利于植物适应环境和改善植物表型[28]。外显子跳跃(exon skipping)、内含子保留(intron retention)、5′端可变剪接(alternative 5′ splice site) 和 3′端可变剪接(alternative 3′ splice site)是植物中主要的剪切方式[29]。目前,茶树开花调控的相关作用机制尚未完全明确,关于茶树开花调控的相关基因的报道也较少。本研究鉴定并克隆了茶树中的5个PEBP基因家族成员,并进行了全面的生物信息学、启动子及时空表达特异性分析。并在实验过程中发现茶树CsMFT基因存在2个不同的转录本,可为进一步研究茶树开花相关基因的功能提供一定的理论参考。
1 材料和方法
1.1 材 料
本研究采用的试验材料为3年生‘南川大茶树’扦插苗,种植于西南大学校内茶树种质资源圃。转录组数据库来源于TPIA数据库(http://tpdb.shengxin.ren/Blast.html),所用茶树品种为‘舒茶早’。不同开放程度的茶树花(花苞、露白、半开、全开、盛开)用于基因表达模式分析,取样地点为重庆市南川区,品种为‘南川大茶树’,液氮速冻后于-80 ℃冷冻保存。
1.2 方 法
1.2.1 茶树开花相关基因的鉴定及克隆根据拟南芥中开花相关基因的研究,在TAIR数据库(http://arabidopsis.org)中下载6个开花相关基因的序列信息,在茶树基因组数据库TPIA中利用本地Blast检索获得5个茶树开花相关基因。以3年生扦插苗的芽头、叶片、嫩茎、根部作为材料,品种‘南川大茶树’通过艾德莱EASY spin 植物RNA快速提取试剂盒,提取其总RNA,用NoVoScript Plus All-in-one 1st strand cDNA Synthesis Super Mix合成第一链cDNA。采用Primer3对获取到的基因CDS序列设计相应的引物(表1),使用Prime STAR Max DNA Polymer酶进行基因扩增,经1%琼脂糖凝胶检验后,用TAINgel MiDi Purification Kit琼脂糖凝胶DNA回收试剂盒回收相应的片段;将回收到的片段连接到pMD18-T载体上,通过热激转化大肠杆菌EcoliDH5,挑取单菌落至含AMP抗生素的LB培养基上划线培养,经菌落PCR扩增后将阳性克隆菌液送至生工公司测序。

表1 引物序列信息
1.2.2 生物信息学分析采用软件ProtParam tool(https://web.expasy.org/protparam/)分析茶树开花相关基因编码蛋白的氨基酸数目、分子量、等电点,并在 Plant-mPLoc(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)网站上对蛋白质的亚细胞定位进行分析。分别利用在线软件SOPM (https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopm.html)和SWISS-MODEL (http://swissmodel.expasy.org/interactive/)对5个茶树开花相关基因蛋白质的二、三级结构模型进行分析。采用DNAMAN软件对茶树开花相关基因的氨基酸序列进行多序列比对,并基于邻接法在MEGA 4软件中构建6个物种(拟南芥、水稻、甜橙、葡萄、杨树及茶树)PEBP基因家族的系统发育进化树。通过在线软件MEME(http://memesuite.org/)对茶树开花相关基因保守基序的进行分析,其中motif基序最大值设定10。在茶树基因组数据库TPIA中,获取茶树开花相关基因在茶树不同组织部位以及在不同逆境胁迫条件下(高盐、低温、干旱和外源激素MeJA)的表达水平等数据,通过软件Tbtools制作热图。根据chen[30]构建的染色体规模基因组信息,在其文献中下载染色体规模基因组模型信息,通过基因编号检索基因在染色体中的位置,获取染色体起始位点信息,在MAPchart软件中对基因染色体位置信息进行可视化。在茶树基因组TPIA中下载茶树开花相关基因翻译起始密码子前2 kb的启动子区域序列,利用在线软件Plantcare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html/)预测顺式作用元件的类型和数量,并采用TBtools软件绘制茶树开花相关基因启动子中的顺式作用调控元件。
1.2.3 基因表达模式分析以不同开放程度的茶树花(花苞、露白、半开、全开、盛开)为实验材料,使用EASY spin植物RNA快速提取试剂盒提取其RNA,以2 μg RNA为模板采用NoVoScript Plus All-in-one 1st strand cDNA Synthesis Super Mix逆转录合成cDNA,稀释20倍作为荧光定量PCR模板。利用Primer3软件设计荧光定量PCR引物(表1),选用已登录的TUBA基因为内参基因,采用荧光定量PCR方法对其进行表达分析。荧光定量PCR 反应体系:SsoFastTMEvaGreen®Supermix 5 μL,上、下游引物各(10 μmol/L) 0.25 μL,cDNA 1 μL,ddH2O补足至10 μL。PCR程序:95 ℃ 3 min,95 ℃ 10 s,60 ℃ 60 s,72 ℃ 30 s,共40个循环。试验进行3次重复,采用2-ΔΔCT法对基因相对表达量进行计算,采用Origin 2021绘图。
2 结果与分析
2.1 茶树开花相关基因的鉴定及克隆
以模式植物拟南芥中开花相关基因的序列信息为参考,在茶树基因组数据库TPIA中利用Blast检索功能获得5个茶树开花相关基因,并在基因编码区设计特异性引物(表1),对5个基因进行克隆,克隆到5个CsPEBP家族基因(图 1),其长度均在500 bp左右,分别命名为CsFT、CsATC、CsMFT、CsTFL1和CsBFT。分析结果显示:CsTFL1、CsBFT、CsATC和CsMFT基因的核苷酸序列与TPIA数据库中的序列一致, 但CsFT基因的克隆序列与TPIA数据库序列相比缺失了42 bp的序列,可能是因为本研究克隆过程中采用的茶树品种为‘南川大茶树’,与TPIA数据中使用的‘舒茶早’茶树品种不同所致。
M. DL2000; 1. CsTFL1; 2. CsFT; 3. CsMFT; 4. CsBFT; 5. CsATC图1 茶树开花相关基因的PCR扩增Fig.1 PCR amplification of flowering-related genes in tea plant
2.2 茶树开花相关基因的生物信息学分析
2.2.1 茶树开花相关基因的基本信息茶树开花相关基因的基本信息和蛋白质理化性质如表2所示,5个茶树开花相关基因长度为519~525 bp,编码氨基酸数目为172~174 aa,蛋白质的分子量在19.17~19.64 kD之间,等电点的范围在7.74~8.93,均为碱性蛋白。蛋白质亚细胞定位预测显示,CsATC、CsMFT、CsBFT定位于细胞质, CsFT定位于细胞核, CsTFL1定位于细胞核和细胞质。

表2 茶树和拟南芥中开花相关基因的基本信息
2.2.2 茶树开花相关基因蛋白的系统进化分析根据拟南芥、水稻、甜橙、葡萄、杨树及茶树PEBP家族成员的蛋白质序列,用 MEGA 4软件构建系统进化树。PEBP蛋白家族可分为MTF-like、FT-like和TFL1-like 3个亚家族,其中,MFT-like亚家族中有7个成员PEBP蛋白,包括茶树、甜橙、葡萄、杨树和拟南芥各1个,水稻2个;FT-like亚家族有24个PEBP蛋白,包括水稻13个,甜橙4个,杨树4个,拟南芥2个和茶树1个;TFL1-like亚家族有19个PEBP蛋白,其中拟南芥3个,茶树3个,甜橙3个,杨树3个,葡萄3个和水稻4个(图2)。
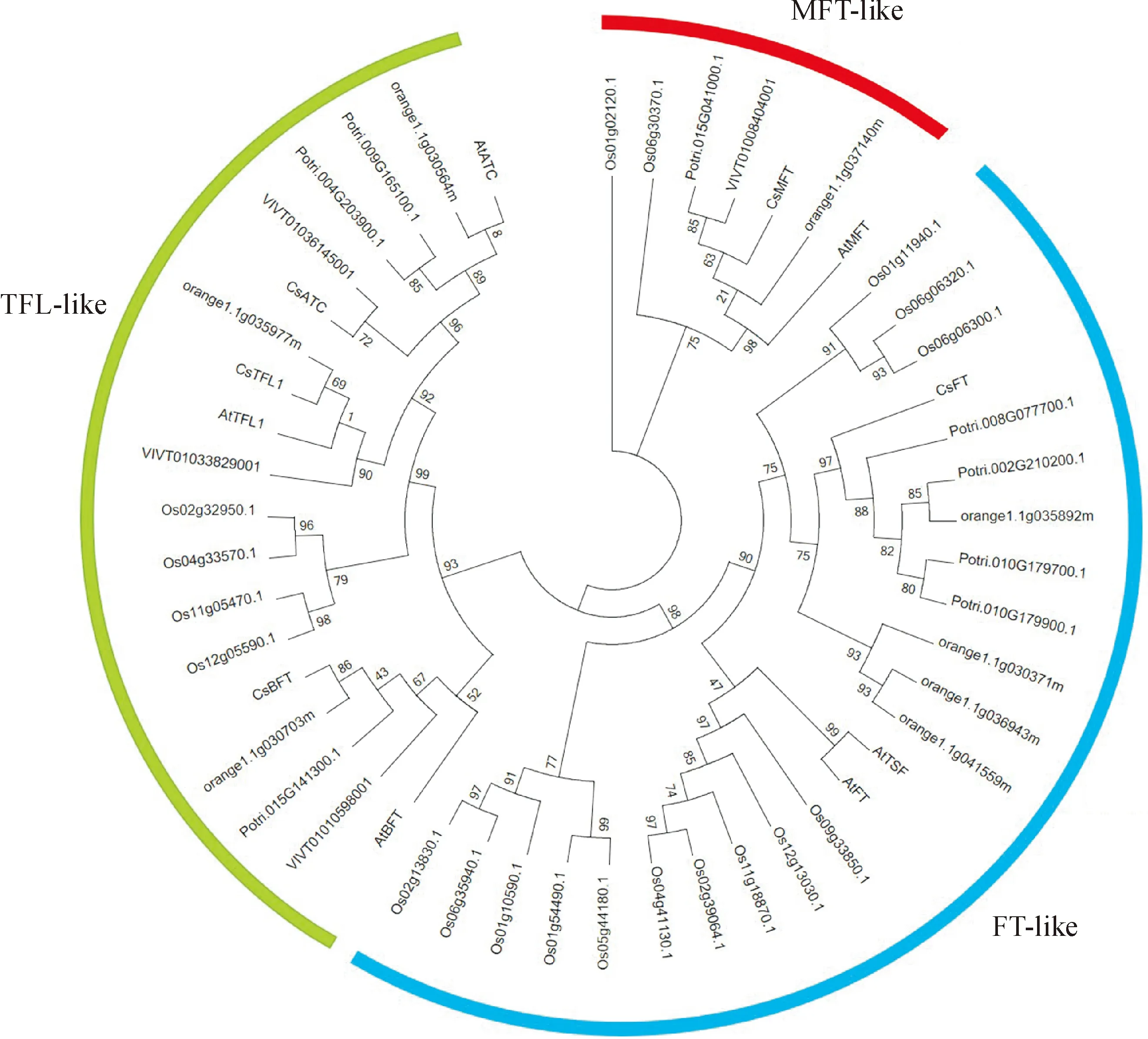
Cs. 茶树;Os. 水稻;orange. 甜橙;At. 拟南芥;VIVT. 葡萄;Potri. 杨树图2 茶树与拟南芥等植物中开花相关蛋白的系统进化树分析Cs. Tea plant; Os. Oryza sativa; Orange. Citrus sinensis; At. Arabidopsis; VIVT. Vitis vinifera; Potri. PoplarFig.2 Phylogeny of flowering-related proteins in tea plant and Arabidopsis et al
MTF-like亚家族中茶树CsMFT与杨树Potri.015G041000.1、葡萄VIVT01008404001聚为一支;FT-like亚家族中茶树CsFT与杨树Potri.008G077700.1、Potri.002G210200.1、Potri.010G179700.1、Potri.010G179900.1和甜橙Orange1.1g035892m聚到一支;TFL1-like亚家族中的CsATC与葡萄的VIVT01036145001,CsTFL1、CsBFT分别与甜橙的Orange1.1g035977m、Orange1.1g030703m聚到同一分支。综上所述,茶树中开花相关蛋白与杨树、葡萄、甜橙等木本植物的亲缘关系更近,可能具有相似的蛋白功能。
2.2.3 保守基序分析为进一步研究茶树开花相关基因序列的特征,分析了5个茶树开花相关基因的结构和保守基序组成。茶树开花相关基因结构分析显示(图3):此家族5个基因均由3个内含子和4个外显子组成与拟南芥PEBP基因家族的基因结构相似,此家族基因外显子长度相似但内含子长度差异较大;5个编码茶树开花相关蛋白与拟南芥均含有motif 1~5基序元件,元件顺序一致,具有高度的保守性。但茶树CsFT蛋白序列中多了motif 6和motif 7两个基序元件,具有独特的序列特征,推测其可能还具有其他特异性的结构功能。

图3 茶树开花相关基因的基因结构和保守基序Fig.3 The gene structure and conservative motif analysis of flowering-related genes in tea plant
2.2.4 基因的染色体定位与蛋白多序列对比茶树开花相关基因进行染色体定位分析显示(图4),5个茶树开花相关基因分别定位于5条不同的染色体上,CsTFL1基因定位于1号染色体,CsFT基因定位于3号染色体,CsMFT基因定位于15号染色体,CsBFT基因定位于9号染色体,CsATC基因定位于7号染色体。5个茶树开花相关基因编码的蛋白的多序列比对结果发现(图5),5个蛋白的氨基酸序列存在高度相似性,其比对结果的一致性高达72.7%,推测其具有类似的功能,可能存在功能冗余。

图4 茶树开花相关基因的染色定位Fig.4 Chromosome location of flowering-related genes in tea plant

图中方框为3个保守结构域D-P-D-x-P 、H和G-x-H-R图5 茶树开花相关蛋白的多序列比对The three conserved domains labeled in the figure were D-P-D-x-P, H and G-x-H-RFig.5 Multiple sequence alignment of flowering-related proteins in tea plant
2.2.5 茶树开花相关基因家族编码蛋白的结构分析对该蛋白家族的蛋白二级和三级结构进行分析发现,自由卷曲是该5个蛋白的主要组成成分,所占比例在39.88%~49.07%之间;其次是延长链和α螺旋,所占比例为23.15%~27.75%;β转角在蛋白结构中所占的比例最低。三级结构预测结果显示,茶树CsBFT、CsATC、CsMFT与拟南芥AtMFT蛋白,CsTFL1与AtTFL1、AtBFT蛋白的三级结构均具有高度的相似性,推测其具有相似的生理调节功能; 此外,茶树CsFT与AtFT蛋白三级结构有极大部分的相似性外,还有独特的结构特征(图6),说明茶树CsFT蛋白可能还具有其他的生物学功能。

图6 茶树开花相关蛋白的二、三级结构Fig.6 The secondary and tertiary structure of flowering-related proteins in tea plant
2.2.6 茶树开花相关基因的启动子元件分析为了解茶树开花相关基因的作用机制,本研究分析了启动子顺式作用元件(图7),5个茶树开花相关基因启动子中均包含有光响应元件、激素响应元件(赤霉素、生长素、脱落酸、水杨酸、茉莉酸响应元件)、逆境胁迫响应元件(干旱胁迫响应元件、低温响应元件、创伤响应元件、厌氧诱导相关元件,防御和应激反应的顺式作用元件),还有一些与分生组织表达相关的顺式调控元件、参与细胞周期调控元件和参与胚乳表达的顺式调节元件,其中光响应元件和激素响应元件最多。
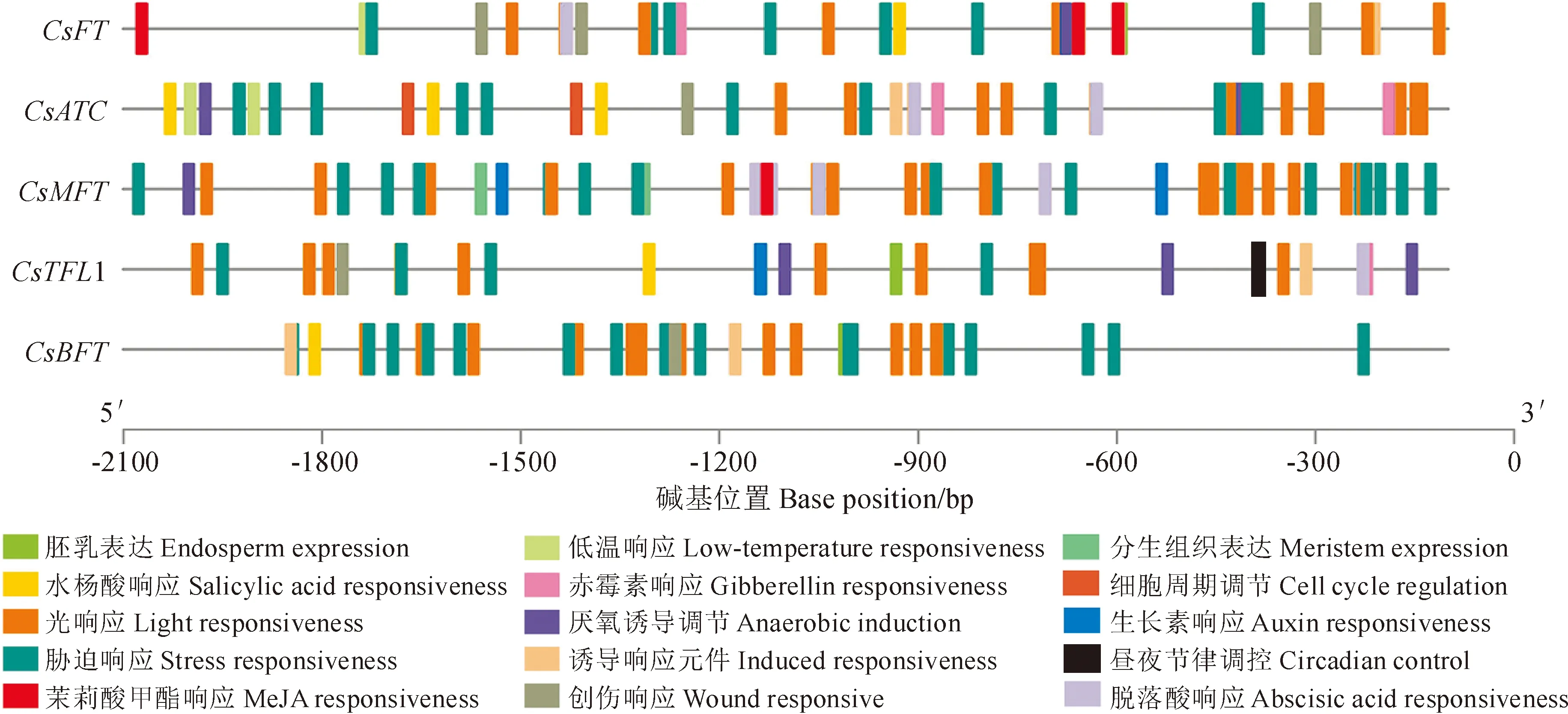
图7 茶树开花相关基因启动子的顺式作用元件分析Fig.7 Cis-acting element analysis of the promoter for flowering-related genes in tea plant
2.3 茶树开花相关基因的时空表达特异性分析
本研究从茶树基因组TPIA数据库下载了茶树开花相关基因在不同组织的转录表达数据,分析其在生长发育过程中的功能。CsFT、CsATC、CsTFL、CsBFT基因在大部分组织中表达量都低,但CsMFT基因在茶树各个部位表达量都高于其他4个基因,且在茶树芽头、嫩叶和茎的表达量较高(图8),由此推测基因CsMFT在茶树开花调控中可能起主要的调控作用。

颜色代表log2值,红色代表高表达,蓝色代表低表达。下同图8 茶树开花相关基因的组织特异性表达分析The color represents the high (red) and low (blue) expression levels. The same as below.Fig.8 Tissue specific expression of flowering-related genes in tea plant
2.4 茶树开花相关基因在非生物胁迫下的表达分析
为探究茶树开花相关基因在非生物逆境胁迫下的响应机制,本研究还从茶树基因组数据库TPIA获取茶树开花相关基因在非生物胁迫下的转录表达数据并制成热图。结果表明(图9),在4种非生物胁迫下茶树CsFT、CsATC、CsTFL1、CsBFT基因响应灵敏度均较低,而茶树CsMFT基因有较高的表达。在茉莉酸甲酯(MeJA)处理条件下,CsMFT基因经外源激素茉莉酸甲酯处理后表达下调,且在处理24 h时表达量最低。在冷驯化处理条件下,茶树CsMFT基因表达均受到抑制,CsFT基因表达量有少量上调。在干旱胁迫条件下,茶树CsFT基因的表达量随PEG处理时间的延长表现上调趋势,而CsMFT基因的表达量受到抑制。在盐胁迫下,CsFT基因表达量在NaCl处理72 h有轻微的上调,而CsMFT基因的表达受到抑制,且随处理时间的增加抑制程度也增加。推测在茶树开花相关基因中,CsMFT基因是主要参与非生物胁迫相应过程的基因。

CK、CA1、CA2分别代表未冷驯化处理、完全驯化和去驯化图9 不同非生物胁迫处理下茶树开花相关基因的表达CK, CA1 and CA2 mean non-cold domestication control, complete domestication and de-tamedFig.9 Expression analysis of flowering-related genes under different abiotic stresses in tea plant
2.5 茶树开花相关基因花期表达分析
本研究采用荧光定量PCR法分析了基因在不同开放时期的茶树花中表达情况,结果显示(图10),CsFT、CsATC基因在整个开花期均呈现高表达,CsTFL1基因在整个开花期的表达量均较低。茶树CsFT、CsATC、CsMFT基因的表达量均呈现相同水平,即在茶树花半开时表达量达到最高,并且随着茶树花开放时间的推移表达量下降,花盛开时几乎不表达。推测5个茶树开花相关基因是在茶树花半开时表达量高。
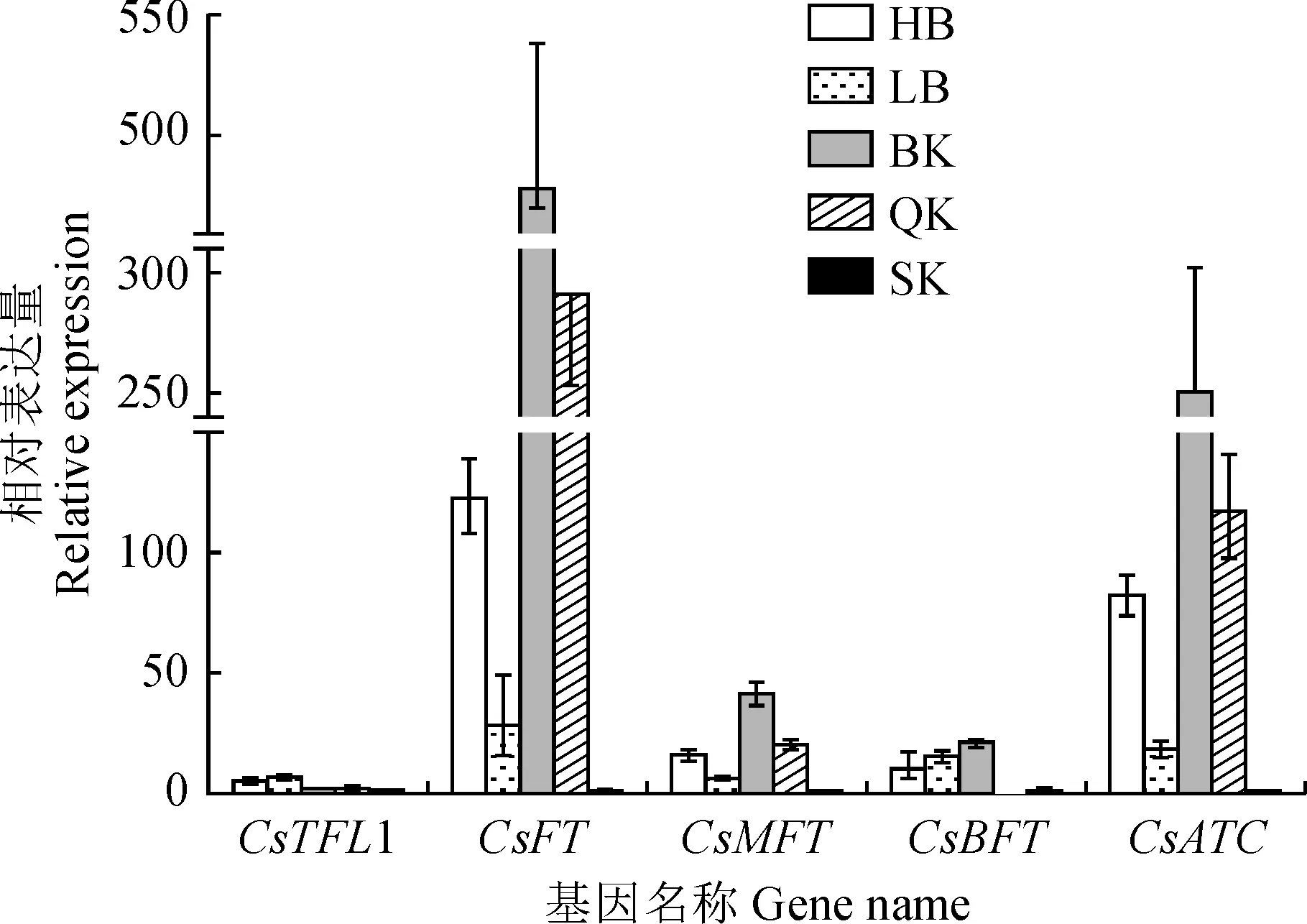
HB. 花苞;LB. 露白;BK. 半开;QK.全开;SK.盛开图10 不同开花时期茶树开花相关基因的表达HB. Bud stage; LB. Initial opening; BK. Half opening; QK. Full opening; SK. BloomingFig.10 Gene expression of flowering-related genes in different flowering periods of tea plant
2.6 CsMFT的可变剪切
在克隆CsMFT基因的过程中发现有2条带(图11),条带大小分别在500 bp与750 bp左右,推测此基因可能存在2个不同的转录本。对这两条带进行了测序和比对分析发现,CsMFT基因(525 bp)与茶树基因组数据库TPIA中CsMFT基因的序列基本一致,长度与下载序列信息相同,在75、97、416三个位点发生单碱基变异,但其编码蛋白序列未发生变异。689 bp大小的CsMFT基因序列与下载序列信息相比,在581处存在一个单碱基变异(A→G),在200 bp处多了一段164 bp的基因片段,此基因片段含有7个终止密码子。经基因结构分析发现(图12),此增加的片段为第一、二外显子之间的区域,并与前后2个外显子合并形成1个外显子,属于内含子保留的剪切方式。

M.DL2000图11 CsMFT基因的可变剪切Fig.11 Alternative splicing of CsMFT gene

图12 CsMFT基因的可变剪切的结构比较Fig.12 Gene structure of alternative splicing of CsMFT gene
3 讨 论
开花是植物从营养生长到生殖生长转变的关键过程,PEBP家族基因参与了植物花器官形成和其他生长发育过程[31]。目前,已经有多种植物的PEBP基因被鉴定,茶树PEBP基因家族有5个成员,除了CsFT比基因组组装序列少了42 bp碱基序列外,其余4个基因与基因组TPIA数据库中提供的核苷酸序列一致。CsPEBPs蛋白的空间结构、保守结构域等与拟南芥该家族蛋白类似,具有高度保守性,均含有D-P-D-x-P 基序、80位的his和G-x-H-R 基序等PEBP蛋白家族的特征保守结构[32]。FT与TFL1蛋白质序列高度相似,但功能相反,在其他植物中,决定其功能的关键位点在FT蛋白中是Tyr(Y85),在TFL1蛋白中是His(H88)[33],在茶树中CsFT蛋白存在Tyr(Y84),CsTFL1蛋白中存在His(H85),因此,茶树CsFT基因与CsTFL基因具有高度保守性,在茶树进化过程中并未发生过多改变。另外,与已报道的拟南芥、辣椒、甘蓝、棉花和月季等植物中的PEBP基因结构相同[34-37],CsPEBPs基因也有4个外显子和3个内含子。与已知其他植物相比,茶树中该基因家族的成员数量较少,只有5个,可能是由于茶树在进化过程中发生了基因的丢失,并且与同为木本植物的杨树PEBP家族具有最近的亲缘关系。
植物PEBP基因家族成员的基因表达具有组织表达特异性,同一亚家族基因表达模式相似。棉花GhMFT3D、GhMFT3A和GhFTL2A基因在花瓣或雄蕊中的表达量最高[38];梨PbFT基因在叶片中表达量最高,其他基因在各个组织中表达量均较低[39];小麦TaPEBP基因表达水平不同,在同一亚家族内,基因具有相似的表达模式[40];茶树CsPEBP基因家族成员也具有组织表达特异性,且CsMFT基因的表达量整体高于CsFT、CsTFL1、CsBFT、CsATC,CsMFT基因在茶树生长中发挥主要调控作用。
关于植物PEBP基因家族的功能研究主要集中在植物开花调控和形态构建方面[41]。TFL1突变体被认为具有早开花和促进末端花分生组织形成的作用[42-43];CEN(Centroradialis)基因在花序顶端表达,并与FLO(Floricaula)基因相互作用共同调控花的位置和形态发育[44]。FT基因除具有促进开花的作用外,还可参与植物营养生长和贮藏器官分化过程调控[13, 45]。在花的不同发育时期,茶树CsFT基因的表达量均远高于其他基因,且在半开和全开时期达到最高,冬枣FT基因在开花期较花苞期的表达量高[46],说明CsFT基因的开花调控功能主要作用于花的半开和全开时期。茶树花期的表达分析中CsMFT、CsFT和CsATC基因有相同表达模式,说明这3个基因可能存在部分功能相同,共同促进茶树开花。此前,MFT基因被认为对种子萌发有促进作用,同时对促进植物开花也有一定作用[24-25]。本研究中CsMFT基因在芽头、嫩叶的表达量高于花、果等,且该基因存在可变剪切现象。TFL1基因被认为对植物开花起抑制作用[10],CsTFL1在花期表达分子中几乎无表达,与其功能预测相符合。PEBP基因家族还可参与植物非生物胁迫的响应过程,但这方面的研究较少。小麦TaPEBP基因的表达对冷、热、干旱、白粉病、条锈病、麦瘟病等非生物胁迫均有响应[47];青杄PwPEBP在干旱、低温和高温下均有明显响应,对盐胁迫响应较低[48];茶树5个CsPEBP基因的表达也受到非生物胁迫的影响,其中,CsMFT基因受到盐、干旱、冷及MeJA等非生物逆境的诱导,基因表达水平高,而CsFT、CsTFL1、CsBFT、CsATC基因的表达水平较低。绝大多数植物PEBP基因的启动子都包含光响应元件。苹果MdPEBP基因家族含有较多的光响应元件和激素响应元件[49];光响应元件在棉花GhPEBP家族基因启动子区域普遍存在[50];茶树CsPEBP基因家族启动子含有较多的光响应元件与激素响应元件,均表明其功能与光调控和激素调节的关联性较强。
可变剪切广泛存在于各种植物转录过程中,是增加基因表达调控方式的重要途径之一,有利于植物生长和环境适应[28]。内含子保留(IR)出现在植物中频率远高于动物,一些研究认为IR现象的发生是因为不准确的剪切信号影响剪切效率,在另外的研究中IR也被证实可通过外部刺激触发作用于特定发育阶段和组织类型,并呈现性别二态性[51-52]。本研究发现茶树CsMFT基因存在可变剪切的现象,第一、二外显子之间的内含子部分未被剪切,属于内含子保留(IR)。其中,长度为689 bp的转录本并不是完整的开放阅读框(ORF),在位点285提前终止翻译,不能够形成完整的CsMFT蛋白,而长度为525 bp的转录本可以表达成完整的CsMFT蛋白,该现象的存在在茶树开花调控可能起到开关的作用,本课题组将会继续深入研究茶树中存在该机制的生物学意义。
综上所述,本研究克隆了5个茶树CsPEBP家族基因,并利用生物信息学的方法全面分析了其编码蛋白的结构特点、组织表达特性及其启动子元件等信息,用荧光定量PCR的方法分析了CsPEBPs开花过程中的表达特异性。此外,茶树CsMFT基因存在可变剪切,存在两个不同长度的转录本。本研究结果可为茶树开花调控的机理研究提供新思路。
