基于深度神经网络融合稀疏分组lasso的预测模型研究*
2022-01-19哈尔滨医科大学卫生统计教研室150081卢宇红宋佳丽
哈尔滨医科大学卫生统计教研室(150081) 卢宇红 宋佳丽 王 萌 侯 艳
【提 要】 目的 探索深度神经网络(DNN)联合不同正则化方法后模型预测准确性的差异;探索模型预测准确性较高时的样本特征规律。方法 R软件产生不同分组、不同样本量的模拟数据集,在不同数据特征下比较DNN模型及融合正则化后模型的预测能力。通过真实数据分析进一步评价两种模型的预测能力。结果 DNN融合不同正则化方法的结果均优于单纯DNN模型,其中DNN融合稀疏分组lasso(SDP)效果最好。稀疏组别组内变量个数的大小及样本量会影响预测准确性,组内变量个数≥8,样本量≥700时,SDP模型预测准确性较高。结论 与单纯DNN模型相比,SDP模型预测准确性得到显著改善;考虑不同样本量和分组方式的情况,SDP模型的预测能力均有明显提高,并且其对预测相关重要特征的提取较为准确。在实际案例分析中发现在小样本的高维组学数据中,SDP模型预测准确性和防止过拟合的能力均有明显提升。
随着高通量检测技术的快速发展,产生了大量的组学数据,其越来越普遍地用于疾病与健康的相关研究,但组学数据具有维度高、样本量小、结构复杂的特点,分析起来较为复杂,而深度神经网络(deep neural networks,DNN)模型可拟合任意函数,适用于分析此类数据,但在进行模型训练时,组学数据中存在大量与预测不相关的冗余特征,训练过程中使用全部特征可能会导致模型出现过拟合问题,影响模型预测的准确性,而在DNN模型中,可以通过删除冗余参数达到压缩模型的目的[1-2]。在DNN压缩方法中基于梯度正则化方法具有更高的优势,具体表现为该方法可在训练网络调节网络参数的同时,进行特定结构的稀疏[3],并且对变量数目没有限制、计算速度较快,可用于处理高维、低样本量的数据[4]。目前基于梯度正则化方法主要用于对DNN结构的调整,通过修剪或合并网络结构以简化模型,降低模型的复杂度和过拟合程度,而未用于输入层特征的稀疏[5-7]。
本文提出基于深度神经网络融合稀疏分组lasso的预测模型(prediction model based on deep neural network together with sparse group lasso,SDP),该方法基于梯度正则化方法压缩DNN结构的思想,在每次迭代调整模型参数的过程中,修剪掉输入层中不重要的特征,使模型充分学习重要特征,以提高预测准确性、避免过拟合。本研究将通过模拟不同样本特征的数据评估SDP模型的预测能力是否优于传统的DNN模型,并探索SDP模型预测能力较高时样本特征规律。采用肿瘤基因组图谱计划(the cancer genome atlas,TCGA)数据库中乳腺癌数据,按通路分组进行实例分析,进一步评价SDP模型与DNN模型的预测能力。
原 理
1.深度神经网络融合稀疏分组lasso模型(SDP)
该方法的基本原理是把额外的惩罚项加到已有模型的损失函数上,稀疏模型中的特定结构,以防止过拟合现象发生。在传统的DNN基础上,将正则化方法应用于深度神经网络的输入层与第一隐藏层间,用以对输入层特征进行稀疏,将Cox模型连接于深度神经网络的输出层,即图1所示。
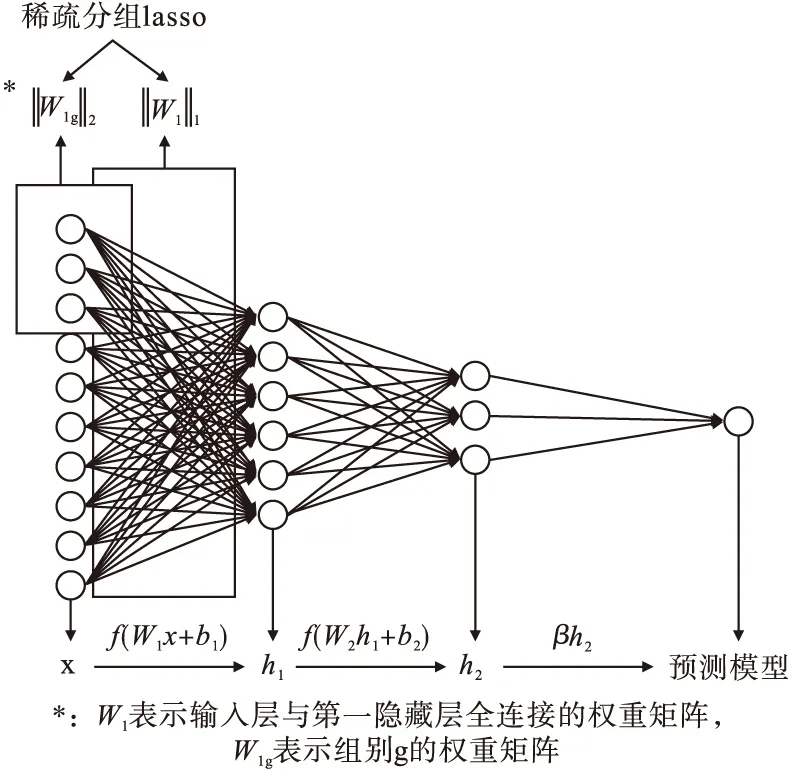
图1 基于深度神经网络融合稀疏分组lasso模型(SDP)基本结构
在深度神经网络中,第一隐藏层h1表示为:
h1=f(W1x+b1)
(1)
第二隐藏层h2表示为:
h2=f(W2h1+b2)
(2)
其中,f(z)为激活函数,本研究中采用常用的ReLU函数,表达式为:
f(x)=max(0,x)
(3)
将深度神经网络第二隐藏层的输出作为Cox模型的输入,则有
h(t|x)=h0(t)exp(βh2)
(4)
其中h2表示第二隐藏层节点,β表示第二隐藏层节点与输出层之间的权重向量。βh2为预后指数(prognostic index,PI),PI越大则风险函数h(t|x)越大、预后越差,该指标不随时间的变化而变化。由于未对基础风险函数h0作任何假设,因此常规的最大似然估计无法估计回归系数向量β,对此可以构造对数偏似然函数对模型参数进行估计。
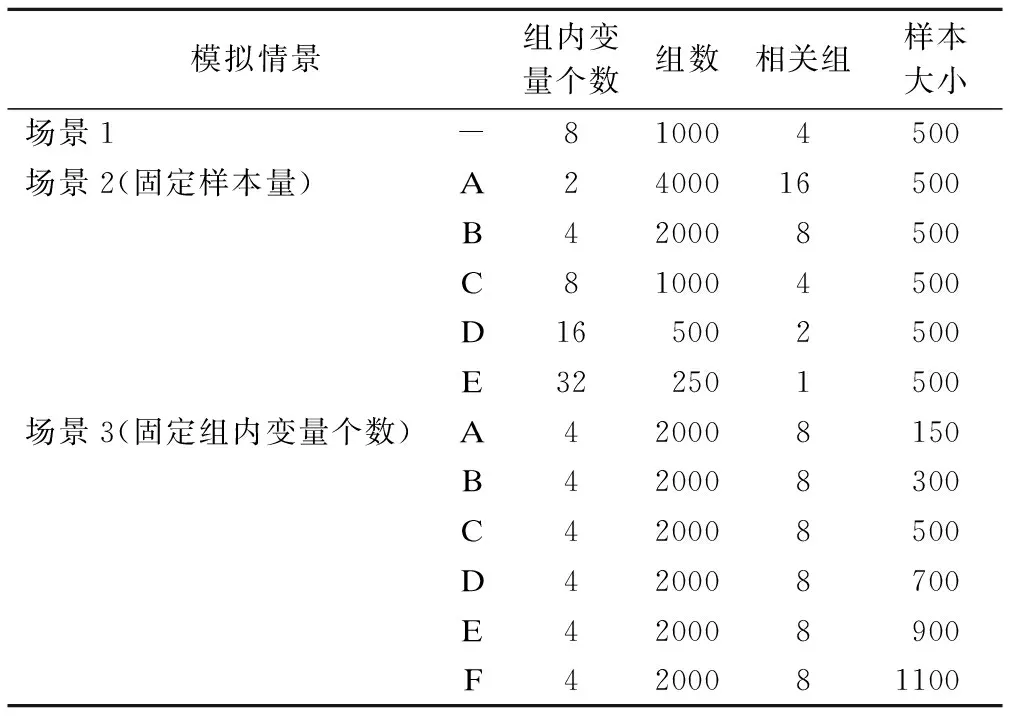
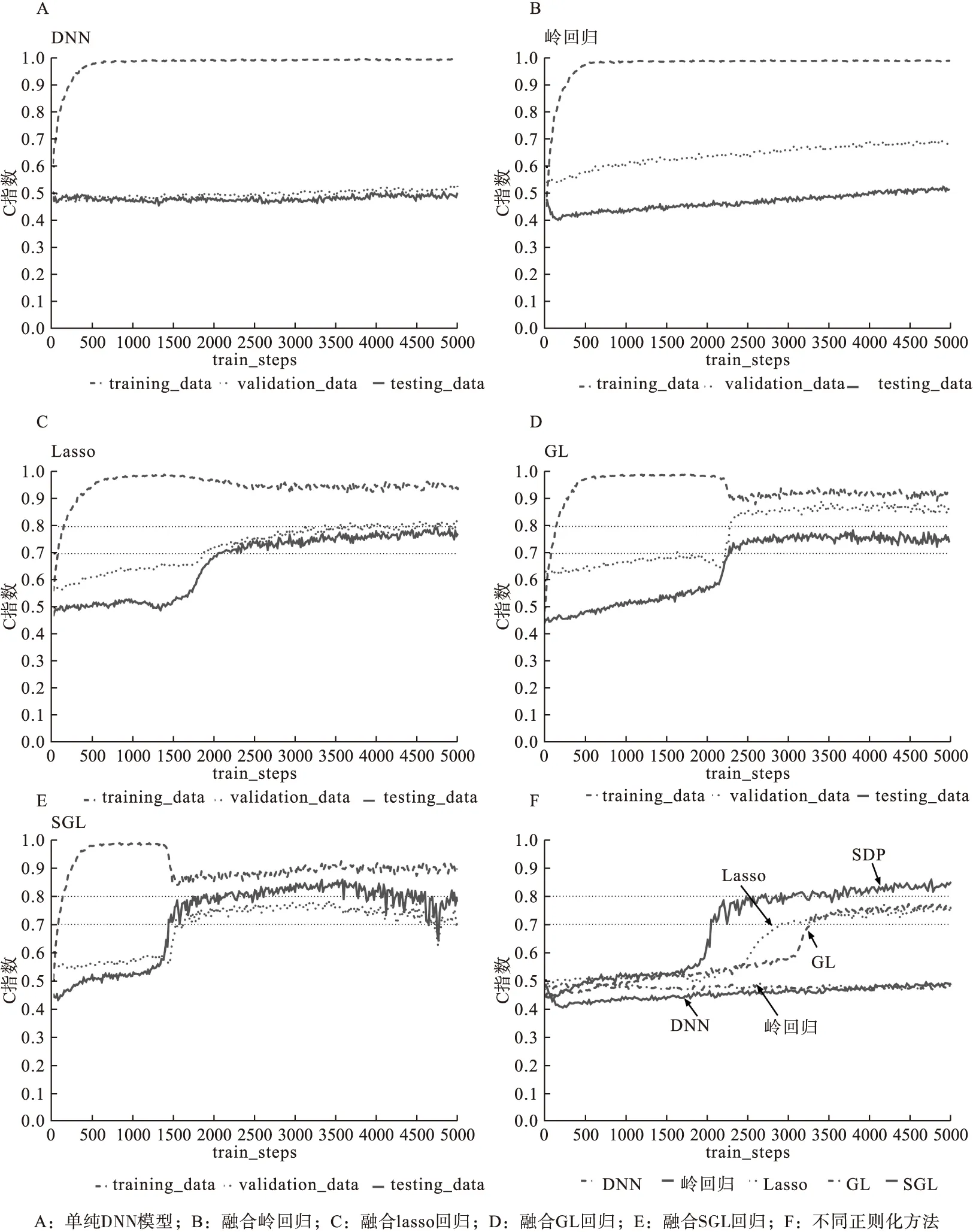
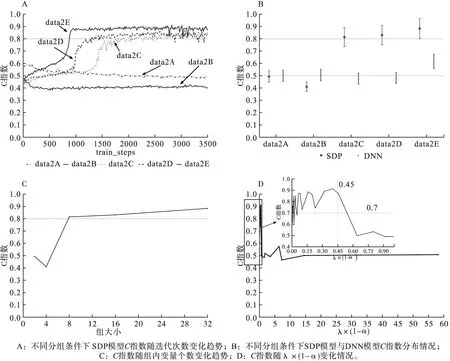
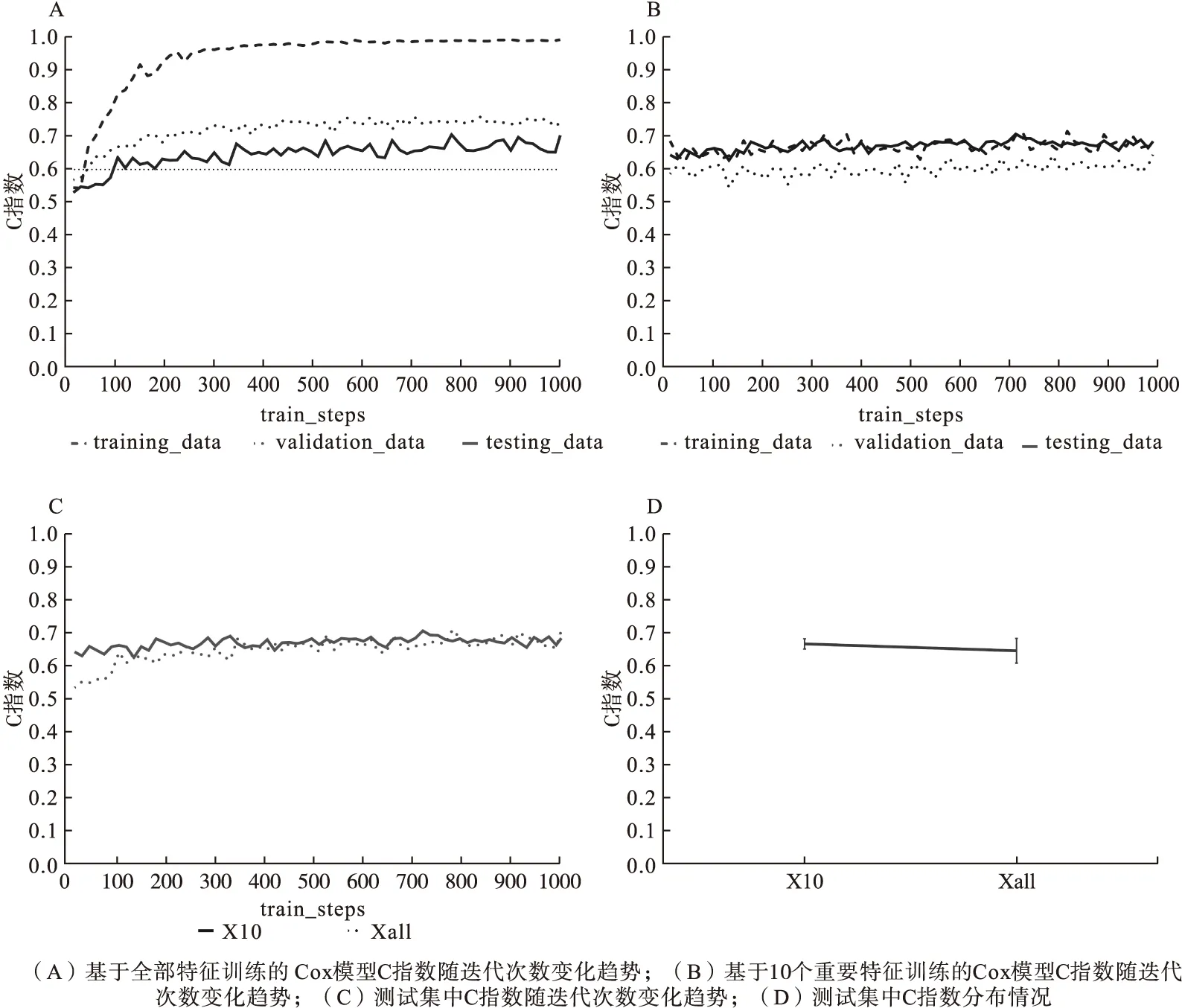
首先,将k个样本生存时间从小到大排序t1 (5) 则各死亡时间点累积死亡概率为: (6) 式中censor表示删失状态。对公式(6)取对数得到以下公式: (7) 此为SDP模型不考虑正则化惩罚时的损失函数。当考虑稀疏输入层成组特征和单个特征时,将SGL惩罚项加入式(7)中,则SDP模型的损失函数表达式为: (8) 2.模型训练及评价指标 根据样本量大小选取适当批尺寸的样本进行模型迭代训练,用60%的样本作为训练数据集(training dataset)进行模型训练,用20%的验证数据集(validation dataset)评估并选择出模型预测能力较高时的正则化参数α、λ,最终的模型预测能力通过另外20%测试数据集(testing dataset)进行比较。本文以生存分析为例评价SDP模型的预测准确性,评价指标选用常规生存分析预测准确性评价指标C指数,C指数越高说明模型预测准确性越高。 1.模拟数据产生原理及特征 通常分三步产生包含随机删失的生存数据:首先采用Bender等提出的模拟方法[8],产生完整生存时间T。第二步产生删失时间Tc,观察时间T0=min(Tc,T)。第三步判断样本状态,如果T≤TC,则观察时间为T,该样本状态为死亡;如果T>TC,则观察时间为Tc,此时样本为删失状态。 本研究模拟在固定总特征不变的前提下,考虑不同分组信息以及每组包含不同的变量个数情况下,评价模型的预测能力。模拟数据共分为三个场景,主要探讨深度神经网络融合不同正则化方法模型的预测能力及过拟合改善情况,以及模型预测能力较好时样本特征规律。具体特征如表1。 表1 模拟数据特征* 2.模拟数据结果 (1)融合不同正则化方法模型预测效果比较 本次模拟改变输入层与第一隐藏层权重稀疏方法,比较DNN及其融合不同正则化方法时模型预测准确性。由图2A可知DNN模型预测C指数约为0.5,准确性较差,验证集、测试集与训练集间距离依然很大存在过拟合现象。由图2B~F可知,当融合lasso回归、岭回归、分组lasso回归及稀疏分组lasso(SGL)时,测试集C指数均值分别为0.5、0.72、0.77、0.8,除岭回归外模型预测能力均有显著改善;训练集与验证集和测试集的间距明显缩小,有效改善DNN模型的过拟合问题。其中融合SGL时预测准确性及过拟合问题改善最为明显。DNN模型测试集C指数约为0.5,与DNN融合稀疏分组lasso(SDP)模型的差异有统计学意义(t=-31.95,P<0.0001),SDP模型预测能力优于传统的DNN模型。 图2 DNN融合不同正则化方法后模型C指数随迭代次数的变化情况 从上述结果中可以看出,基于模拟数据data1应用SDP模型对生存风险的预测结果与实际情况一致性较好(0.8),为进一步探索模型对重要特征的提取能力,将基于data1数据集训练的SDP模型输入层与第一隐藏层的权重输出,对每个特征各节点权重加和后得到各个特征的权重,按绝对值大小进行排序,选取权重值前32的特征与模拟生存数据的特征做对比,32个特征中与相关特征(16个)一致的特征有15个,特征提取灵敏度为93.75%,可证明模型对重要特征提取的准确性较高。 (2)不同分组情况SDP模型预测效果比较 本次模拟固定样本量不变,假定先验分组方式不同,则组内变量个数不同,即模拟产生组内变量个数不同的data2A~data2E数据集,比较先验分组信息不同条件下SDP模型与DNN模型的预测准确性。当组内变量个数为2、4、8、16、32时,正则化参数的最佳组合分别为0.5/0.99、0.25/0.99、16/0.99、64/0.999、32/0.99。在图3A~B中,除组大小为2、4的C指数低于0.5,其余分组方式SDP模型C指数均高于0.8。说明应用SDP模型时,要选择合适的先验分组信息,注意控制组内变量个数不宜过低;图3C中,随着组大小的增大,C指数有逐渐增高的趋势,当组大小≥8时SDP模型C指数大于0.8,预测能力较好。为了进一步总结分组方式不同时,正则化参数设置的规律,以2E数据集为例,设λ×(1-α)为横轴,迭代次数2000~3500的C指数均值为纵轴(此时模型训练趋于稳定),观察随正则化参数变化C指数均值变化情况,如图3D,当λ×(1-α)大于1时,C指数均值低至0.5,模型预测能力不佳,将图3D的0~1部分放大,当λ×(1-α)小于0.45时,SDP模型的预测C指数高于0.7,预测能力较好。 图3 不同分组情况SDP模型的预测情况 (3)不同样本量SDP模型预测效果比较 本次模拟固定分组方式不变,改变样本量以比较不同样本量条件下,SDP模型与DNN模型的预测准确性。在不同研究、不同平台及不同疾病等中,可获得的模型训练数据样本量不同,而样本量大小可能影响模型的学习能力,进而影响其预测性能。如图4A所示,在各样本量条件下,SDP模型C指数均大于0.7,SDP模型预测能力较好;图4B中,Y轴为训练稳定后,各模拟数据集测试集C指数均值,SDP模型C指数均大于DNN模型,预测能力优于DNN模型;如图4C所示,随着样本量增大,SDP模型C指数有逐步上升的趋势,当样本量≥700时,C指数在0.8以上,模型预测能力较好。 图4 不同样本量SDP模型和DNN模型的预测情况 TCGA数据库中乳腺癌患者的mRNA数据(1217例)、临床表型数据(1284例)和生存数据(1260例)进行分析,考虑到样本删失率不能超过80%,否则测试时可能会导致无可比对子数进而无法计算C指数,因此排除部分删失数据,最终选择700例样本作为模型训练样本。考虑到京都基因与基因组百科全书(Kyoto encyclopedia of genes and genomes,KEGG)通路是一组包含生物系统信息的网络通路,其可整合生物分子及化学分子间的相互作用[9],因此将KEGG通路作为乳腺癌mRNA特征的分组信息。采用R软件中的“org.Hs.eg.db”软件包识别60483个KEGG-ID号,共有25880个mRNA成功获得KEGG-ID号;利用KEGG的API获取基因对应的通路信息,共有32605个mRNA(一个mRNA可能存在于多个通路中,共7921个mRNA)富集于337个通路,每个通路中包含的mRNA的范围是1到1487个;考虑到临床表型特征的实际意义以及缺失率,纳入8个临床表型特征进行分析,包括初始诊断年龄、TNM分期、样本来源、种族、初始诊断类型以及stage分期,将每个特征单独分为一组。 基于此数据训练模型,当正则化参数λ、α分别设为16、0.999时,SDP模型预测结果如图5所示,与DNN模型相比,SDP模型过拟合问题及预测能力有明显改善。随着迭代次数的增加SDP模型训练集C指数与验证集、测试集的C指数间距小于DNN模型训练集与测试集间距,说明SDP模型一定程度上改善了单纯DNN模型存在的过拟合现象(见图5A和图5B)。SDP模型测试集C指数均值为0.70,相较于DNN模型的0.58有明显提高,预测能力改善明显(见图5C和图5D)。 图5 SDP模型及DNN模型在乳腺癌数据中的预测情况 为了进一步说明SDP模型特征提取的准确性,在训练好的SDP预测模型中,根据输入层与第一隐藏层的权重求得各特征的平均权重,排序得到权重前10的重要特征,基于这10个特征建立Cox模型,与利用全部特征拟合的Cox模型进行预测能力、过拟合改善情况的对比。如图6A,基于全部特征训练Cox模型,其训练集C指数与验证集、测试集C指数间差距较大,模型存在过拟合问题。如图6B,基于提取特征训练的Cox模型,其训练集C指数与验证集、测试集C指数间差距明显缩小,有效改善了过拟合问题。由图6C和图6D可知,基于SDP模型中提取的重要特征训练的Cox模型C指数为0.67,与基于全部特征训练的模型的0.65相比,其预测能力差异不大。综上所述,基于SDP模型提取的特征训练Cox模型,可在不降低预测准确性的同时,有效改善由于变量过多导致的过拟合现象,提示提取的特征与预后相关较好,证明SDP模型在重要特征提取方面具有一定的优越性。 图6 基于乳腺癌数据不同特征拟合Cox模型的预测情况 基于小样本高维组学数据应用深度神经网络(DNN)模型时,存在的一个显著缺点是模型的“过拟合”问题,该问题可以通过减少过度参数化、简化、压缩DNN解决。压缩DNN模型一般有构建法和修剪法两种方法,修剪法学习速度较快,对初始条件的敏感度较低,并且泛化能力较强,因此修剪法更为常用。神经网络修剪方法,一般包括以下四种类型,即基于阈值的方法、基于结构灵敏度的方法、基于结构间相关性的方法以及基于梯度正则化的方法[10]。相对于其他三种方法,基于梯度正则化方法因以下优势而使用更加广泛:第一,正则化方法不需要预训练模型,模型参数调整与结构修剪可同时进行;第二,不需要计算灵敏度、特异度;第三,仅通过添加一个或多个正则化项稀疏网络结构;第四,可以考虑学习错误使模型获得更好的性能。目前基于梯度正则化的方法未用于输入层数据[7,11-12],然而应用DNN进行癌症预测问题时,输入数据中存在大量的对预测不重要的冗余数据,以及一些相对很重要的数据,若不加选择地全部应用于模型训练,势必会导致模型的过拟合问题,影响其预测准确性。 在本研究中,基于梯度正则化修剪网络的思想,在DNN模型损失函数中加上稀疏输入层特征的正则化项,在最小化误差调节参数的同时惩罚输入层特征的权重,不断加强对重要特征的学习,有效改善了模型过拟合问题、提高了其预测能力。目前常见的正则化方法中,岭回归是将每个变量系数变小;lasso可以使部分变量稀疏为0;分组lasso回归根据先验分组信息考虑组间特征的相关性,对分组变量进行筛选;SGL回归结合lasso和GL回归二者优势,可同时实现成组变量和单个变量的筛选,对特征的稀疏程度更加充分。由模拟实验结果可知,相对于其他正则化方法,融合SGL的SDP模型的C指数最高、预测能力最好,测试集和验证集C指数与训练集差距最小,过拟合改善情况最为明显。可能的原因是与其他的正则化方法相比,SGL将先验分组信息加入模型损失函数的正则化项,充分考虑了输入特征间的相关性,同时稀疏分组特征和单个特征,模型对重要特征提取能力可能更高。当组内变量个数≥8时,SDP模型预测能力优于单纯DNN模型;应用于不同样本量数据时,SDP模型的预测能力优于DNN模型,当样本量≥700时模型预测能力更佳,样本量越大SDP模型的学习越充分。 在实例分析中,通过联合TCGA乳腺癌的mRNA数据和临床表型数据,应用SDP模型和DNN模型进行死亡风险预测,显示SDP模型预测准确性和过拟合改善情况优于DNN模型。值得注意的是,有研究表明随着数据删失率的增大,Cox模型的偏倚性、准确性以及模型的拟合程度均会有所下降,且删失率较大时模型偏倚性有加速下降的趋势[13-15]。而在乳腺癌训练数据中删失率高达76%,但应用SDP模型其预测准确性较高,提示SDP模型对于删失率较高的数据可能具有一定的适用性。另外,在基于乳腺癌数据训练的SDP模型中,通过特征权重排序获得的重要特征训练Cox模型,与基于全部特征训练的Cox模型相比,过拟合现象有显著的改善,说明SDP模型在特征提取方面具有一定的优越性。 SDP模型也存在一定的缺陷,例如损失函数加入正则化项后降低了模型的运算速度,当数据变量较多时较为耗时;另一个问题是网络结构的设置,如网络层数、各层节点数,均可能影响模型的预测结果,如何设置网络结构有待进一步探讨。根据目的不同,可以将SDP模型的输出层部分的Cox模型替换,如当感兴趣结局为事件的分类时,像患病与否,疾病分型等,可连接logistic模型或者SVM模型等,但此时DNN融合正则化模型的预测能力是否依然优于DNN模型有待未来的研究具体验证。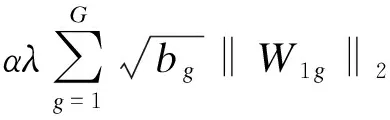
模拟数据

实例分析

讨 论
