水稻雄性核不育突变体ms7的遗传分析及基因定位
2022-01-18杨晋宇白琛丁小惠申红芳王磊应杰政鄂志国
杨晋宇 白琛 丁小惠 申红芳 王磊 应杰政, * 鄂志国, *
水稻雄性核不育突变体的遗传分析及基因定位
杨晋宇1, #白琛2, 3, #丁小惠2, 3申红芳1王磊1应杰政1, *鄂志国1, *
(1中国水稻研究所 水稻生物学国家重点实验室, 杭州 310006;2中国科学院 植物研究所 植物分子生理学重点实验室, 北京 100093;3中国科学院大学,北京 100049;#共同第一作者;*通信联系人, E-mail: yingjiezheng@caas.cn; ezhiguo@caas.cn)
【】通过对水稻雄性不育突变体的研究,可以鉴定更多与育性或花粉发育相关的基因,有助于解析水稻雄性生殖发育的整个调控网络。常规种植条件下,突变体()与对照种植于浙江富阳和海南陵水,比较它们的育性及主要农艺性状差异,利用混池关联分析和图位克隆方法进行目标基因定位。整个生育期,突变体生长速率与野生型一致,成熟期的株高、分蘖数、叶数、叶大小、穗长和每穗颖花数等性状与野生型相比也没有显著差异,但结实率为0,表现为完全雄性不育,花药瘦小且颜色发白,半薄切片显示绒毡层降解推迟,花粉镜检呈染败。遗传分析表明花粉败育受单个隐性基因控制,定位于第7染色体上BSA11与YD7045之间1.17 Mb的范围内。本研究为水稻雄性不育基因的克隆和功能研究打下了基础。
水稻;雄性不育;混池关联分析;基因定位
水稻是我国乃至东南亚最重要的粮食作物,在我国有着悠久的种植和驯化历史[1]。水稻细胞核雄性不育系的发现及应用,推动了杂交水稻从三系到两系的发展,为我国乃至世界粮食安全发挥了重要作用[2]。目前生产中利用的水稻核不育系多是光温敏雄性核不育系,相应的控制基因如[3]、/[4-5]、等也已鉴定。近年发现一种新型湿敏雄性核不育现象,突变体在低湿环境(相对湿度<60%)下因花粉不能有效防止脱水而无法完成粘附和水合作用,造成不育,而高湿环境(相对湿度>80%)下恢复育性[8-10]。除这种环境和遗传因子互作外,还有诸多影响小孢子和花粉发育的因素,而绒毡层发生和降解异常是重要原因之一。绒毡层位于花药壁最内层,通过细胞程序化死亡(programmed cell death, PCD),为小孢子的发生发育提供营养,分泌脂质参与花粉外壁建成和花药角质层合成[11]。F-box蛋白OsADF、半胱氨酸蛋白酶OsCP1[13]、天冬氨酸蛋白酶OsAP25和OsAP37[14]等,可通过蛋白水解通路直接参与绒毡层的PCD脂肪酸还原酶DPW[15]、α整合素样蛋白DPW3[16]、酰基辅酶A合成酶[17]、酰基转移酶DPW2[18]和酰基转移酶OsGPAT3[19],通过参与脂质代谢,影响绒毡层PCD和花粉外壁前体物质合成。此外,钙离子结合蛋白Ca。OsAGO2通过DNA甲基化直接下调己糖激酶基因表达,影响绒毡层活性氧积累,敲低或过表达都能导致PCD提早和花粉败育[21]。
在绒毡层发育调控网络中,MYB转录因子GAMYB[22-23]和BM1[24],bHLH转录因子UDT1[25]、TIP2[26-27]、TDR[28]和EAT1/DTD[14, 29],E3泛素连接酶OsHUB1和OsHUB2[30],PHD锌指蛋白PTC1/OsMS1[31-33]和凋亡抑制因子API5[34]等都起重要作用。TDR作用于GAMYB和UDT1下游,通过直接调节、和等基因的表达,激活蛋白水解通路从而引发绒毡层降解[12, 23, 28]。EAT1/DTD作用于TDR下游并能与TDR互作,通过上调和表达,正调控绒毡层PCD[14, 29]。TIP2/bHLH142能直接调控和的表达,且TIP2能与TDR互作,从而调控绒毡层PCD[26-27]。OsMS1和TIP3都具有转录激活活性,它们分别通过与TIP2和TDR互作,调控乌氏体发生和绒毡层降解[32-33]。OsHUB1和OsHUB2作为E3连接酶,通过组蛋白H2B单泛素化,正调控、和表达,影响绒毡层降解[30]。ATP柠檬酸裂合酶ACL催化细胞质中的柠檬酸生成草酰乙酸和乙酰辅酶A,通过影响、、和等基因的表达,调控绒毡层PCD。ACL突变体绒毡层细胞过早降解,花药中ATP水平和脂肪酸含量降低,不能形成有活性的花粉[35]。这些表明,在花药壁的分化、发生和降解过程中,TIP2、TDR和EAT1这3个bHLH转录因子可能起核心调控作用。编码一种AAA-ATP酶,与RAD51和DMC1互作,参与减数分裂的染色体双链断裂修复,但突变体绒毡层降解异常,是导致花药异常和小孢子败育的原因之一[36]。此外,生殖细胞也能调节绒毡层PCD。编码一种糖蛋白,被小孢子分泌到细胞外与绒毡层细胞表面受体互作,调控绒毡层PCD和小孢子发育[37]。
我们在对籼稻中恢161进行组培实验时,从其后代中鉴定出一个雄性不育突变体,也表现为绒毡层降解滞后和花粉败育,其他表型在整个生育期与野生型没有显著差异。进一步用潮霉素基因的分子标记对后代不育个体进行PCR鉴定,在无筛选标记的个体中依然表现为不育,表明不育性状并非由遗传转化载体结构T-DNA插入而产生,推测可能是组培过程中产生了新的遗传变异导致中恢161细胞核雄性不育。为了明确雄性不育产生的原因,我们用作母本,IR96671为父本构建遗传群体进行遗传分析,结果表明的不育性状受单个隐性核基因控制。经混池关联分析(bulked segregant analysis, BSA)和基因定位,将定位于第7染色体上BSA11-YD7045之间,这些结果为进一步的基因克隆和功能分析打下了基础。
1 材料与方法
1.1 供试材料
本研究采用的突变体是籼型恢复系中恢161经组织培养产生的,该突变体在杭州富阳和海南陵水均表现为雄性完全不育,不受温度和日照影响。常规籼稻IR96671作为杂交亲本构建遗传分析和定位的群体。
1.2 群体构建及遗传分析
突变体为母本,籼稻品种IR96671为父本,配制杂交组合,观察F1的表型,分单株收获种子。在F2群体中统计野生型和不育型的分离比例,用于遗传分析、BSA分析和基因定位。所有材料2018−2019年种植于中国水稻研究所杭州富阳实验基地和海南陵水基地,正常田间管理。
1.3 花粉育性调查
抽穗期当日开花前,田间取回野生型和不育型的颖花,在显微镜下观察花药表型并拍照。用1% I2-KI溶液进行染色,观察野生型和不育型花粉染色的情况并拍照。
灌浆后期观察单株灌浆结实情况,记录植株育性。
1.4 花药半薄切片观察
取野生型和不育型不同发育时期的小花,在FAA固定液(甲醛∶冰乙酸∶50%乙醇=5∶5∶90)中固定,抽真空至样品全部沉底,换用新的FAA常温保存备用。挑选不同时期的小花剥去颖壳露出花药后用酒精进行梯度脱水,接着在饱和番红水溶液中处理2~3 h,再使用Technovit 7100试剂盒进行渗透包埋,60℃恒温烘箱中烘烤4 d。修块后用半薄切片机(Leica RM2265)横切花药,切片厚度为2 μm,经甲苯胺蓝染色后用光学显微镜观察拍照。
1.5 全基因组测序
在×IR96671的F2群体中,根据育性的分离分为可育型和不育型两组,从中分别挑选出30株,采用简易DNA提取法分单株提取叶片DNA,检测每份DNA的品质与浓度后等量混合制备混池。利用超声波将DNA序列片段化形成随机片段,对片段化的DNA进行末端修复、3′端加A、连接测序接头后,富集400 bp左右的随机片段,经PCR扩增形成测序文库。文库经质检合格后,用Illumina HiSeqTM平台进行测序,测序策略为Illumina PE150,总测序读长为300 bp。
对测序数据进行质量控制,过滤掉低质量数据。利用BWA软件[38]将质控后数据比对到参考基因组(IRGSP1.0, http://rice.Plantbiology.msu.edu/)上,获得序列的位置。利用GATK软件对位置归属校正,并进行SNP检测[39],同时利用Samtools软件的Vcfutils工具进行过滤[40],获得高质量的变异位点。采用Annovar软件和参考基因组的基因预测信息进行注释。基于亲本和突变混池的数据特点,计算SNP指数进行BSA关联分析,定位目标突变位点。
1.6 差异位点的关联分析
对所得到的SNP和InDel位点进行亲本和突变混池的差异位点开发,计算每个差异SNP位点的指数,即突变基因型在突变池和野生池中所有基因型中所占的比例。由于突变位点与周围标记的连锁效应,在突变位点附近,SNP指数更接近于1,其余位点由于高通量测序的随机分布,应符合孟德尔分离比例,本研究的突变性状属于隐性突变,在F2群体中,突变位点的SNP指数为1,正常位点为0.5。
为了消除假阳性位点,利用标记在基因组上的位置,用滑窗的方法将SNP指数进行拟合,消除由于随机扩增导致的差异突变。计算每个变异位点的指数,以染色体物理位置为横坐标绘制曲线图,计算所有SNP指数在95%、99%及99.9%置信水平下的阈值,挑选高于阈值的区域作为候选区间。
1.7 DNA提取和定位分析
基于BSA分析得到的初步定位区间,根据亲本材料重测序结果,利用Primer 5.0软件设计位于该区域的InDel标记,标记分布区域完全覆盖BSA分析得到的关联区间,并向左右各扩展数兆区间。挑选多态性标记用于验证BSA测序结果及筛选F2群体中的重组单株。除大田种植外,F2群体同时也在光温培养箱中进行水培。大田材料在移栽一周后取回叶片使用简易法提取DNA,水培材料在播种10 d后使用简易板提法提取叶片DNA。
PCR扩增使用北京康为世纪生物科技有限公司的10 μL反应体系:模板DNA 1 μL、10 μmol/L前后引物各0.5 μL、2×Taq MasterMix预混液5 μL、灭菌水3 μL。扩增程序如下:94℃下预变性2 min;94℃变性30 s,55℃退火30 s(根据不同引物TM值修改),72℃延伸30 s,共计30个循环;最后72℃延伸2 min。PCR扩增产物依据片段大小采用2%或2.5%琼脂糖凝胶电泳分离后经Gel Red染色检测,然后拍照保存统计带型。挑出目标区域内的重组单株后重新取样鉴定,对最终确认的重组单株进行目标区域的其他标记检测,移栽到大田,待灌浆完成后记录育性。
2 结果与分析
2.1 突变体ms7表型分析
自然生长条件下,突变体全生育期生长速率与野生型中恢161一致,灌浆前表型没有明显异常,包括株高、分蘖数、叶数、叶大小、穗长和每穗颖花数等性状与野生型相比没有显著差异(图1-A)。但不灌浆,结实率为0。用野生型花粉授粉后,能正常结实,表明雌蕊发育正常,该突变体为雄性不育(图1-B)。与野生型相比,花药瘦小且颜色变淡发白(图1-C)。花粉镜检发现的花粉粒无法被1% I2-KI溶液染色或染色不均,为染败,表明花粉无活性,而野生型花粉粒能全部染成深色(图1-D)。
2.2 花药半薄切片分析
根据张大兵等[41]将花药发育所分成的14个时期,可通过花药半薄切片来比较观察野生型和突变型在花药发育过程中存在的差异。在1~8a时期,突变体的花药发育形态与野生型相比无明显差异,花粉母细胞进行正常的减数分裂并形成了二分体(图2-A、图2-G)。在8b时期,小孢子母细胞继续进行减数分裂,形成四分体。此时,野生型的绒毡层开始液泡化并伴随着细胞程序性死亡(图2-B),突变体的绒毡层出现大量空泡(图2-H)。第9时期,随着胼胝质壁的降解,自由的单倍体小孢子从四分体中释放了出来,游离在药室中。野生型的绒毡层中空泡逐渐被吸收,细胞质继续浓缩,颜色加深(图2-C),突变体的绒毡层仍可见空泡(图2-I)。第10时期,小孢子液泡化,野生型的绒毡层进一步退化(图2-D),突变型的绒毡层却明显增厚(图2-J)。第11时期,液泡化的小孢子进行第一次不对称的有丝分裂,产生一个较小的生殖细胞和一个较大的营养细胞。野生型的绒毡层继续降解(图2-E),而突变型的绒毡层仍无降解表现(图2-K)。第12时期,小孢子内的生殖细胞进行了第二次有丝分裂,野生型中成熟的花粉粒由于淀粉粒和油脂的积累,细胞内充满储存物,表皮和药室内壁进一步降解,绒毡层完全消失(图2-F);突变体仅有极少数小孢子填充有淀粉,但缺少油脂,绝大多数小孢子表现为形状不规则的空泡,无填充物(图2-L)。
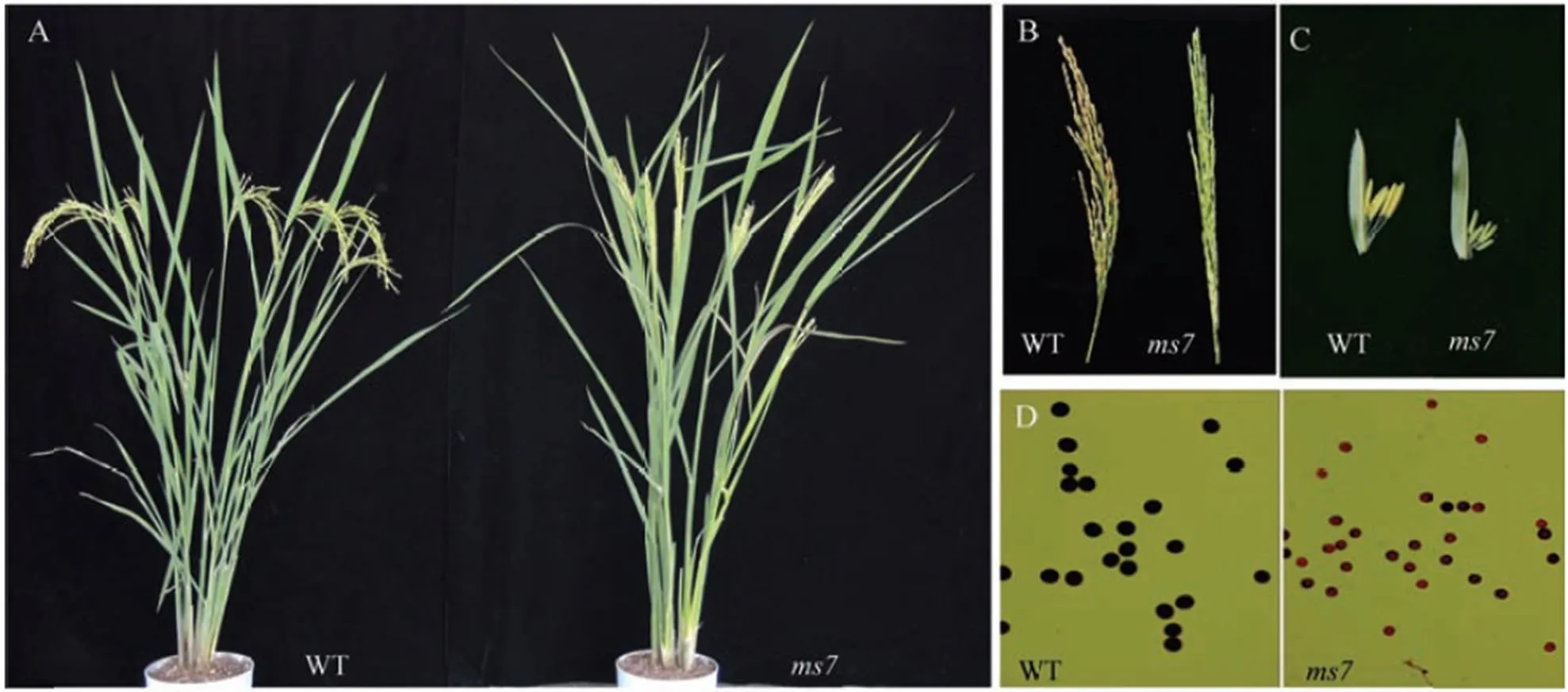
A―灌浆期的植株形态;B―ms7和野生型的结实情况;C―ms7和野生型的颖花形态;D―ms7和野生型的花粉碘化钾染色。
Fig. 1. Phenotype ofand its wild type Zhonghui 161.
2.3 突变体ms7雄性不育性状的遗传分析
以突变体为母本,IR96671为父本构建了×IR96671群体。考查F1植株的花粉育性,发现F1结实率与野生型相当,表明突变性状受隐性基因控制。统计了4个F2群体的可育型和不育型植株数,经卡方检验,≈0.43>0.05,正常育性与雄性不育个体的分离比符合3∶1(表1),说明的雄性不育性状受单个隐性核基因控制。
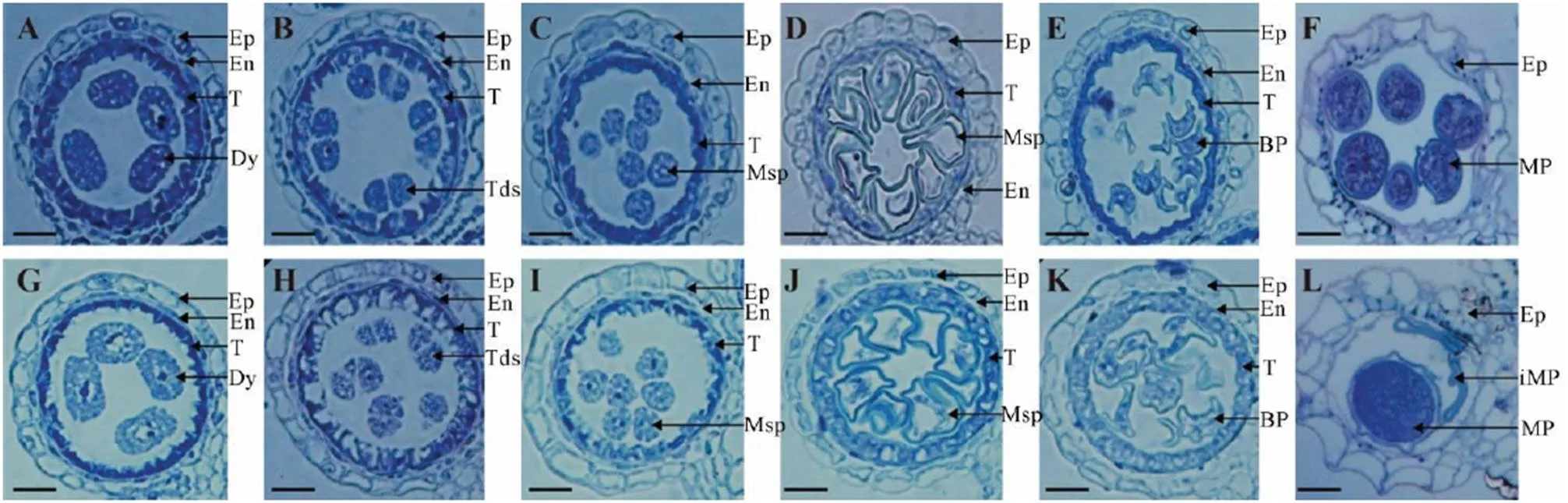
A~F为野生型花药在8a至12时期的切片;G~L为突变体花药在8a至12时期的切片。
A−F, Wild-type anthers from stages 8a to 12; G−L,anthers from stages 8a to 12.
Ep―表皮;En―内壁;T―绒毡层;Dy―二分体;Tds―四分体;Msp―小孢子;BP―二胞花粉;MP―成熟花粉粒;iMP―未成熟花粉粒。标尺为20 μm。
Ep, Epidermis; En, Endothecium; T, Tapetum; Dy, Dyad cell; Tds, Tetrads; Msp, Microspore; BP, Biceullar pollen; MP, Mature pollen; iMP, Immature pollen. Bar=20 μm.
图2 野生型中恢161和突变体不同发育时期花药半薄切片观察
Fig. 2. Contrast of slice of anther between themutant and its wild type.
2.4 BSA分析初定位ms7雄性不育基因
4个样本池测序并过滤后共获得87.59 G的数据,Q30达91.79%,样品平均覆盖深度43.72X,与参考基因组平均比对效率为79.49%,基因组覆盖度96.02%。这些数据表明测序数据充足,质量高,可用于下一步分析。
SNP分析表明,4个样本共获得3 905 483个SNP位点,其中非同义突变的SNP有1 482 935个。SNP信息统计显示,不同样品的转换与颠换两种类型SNP比率(i/v)大致相当(表2)。与亲本池相比,两个子代混池样本中纯合基因型比率明显降低,分别为38.00%和38.82%,表明杂交增加了子代的杂合SNP率。
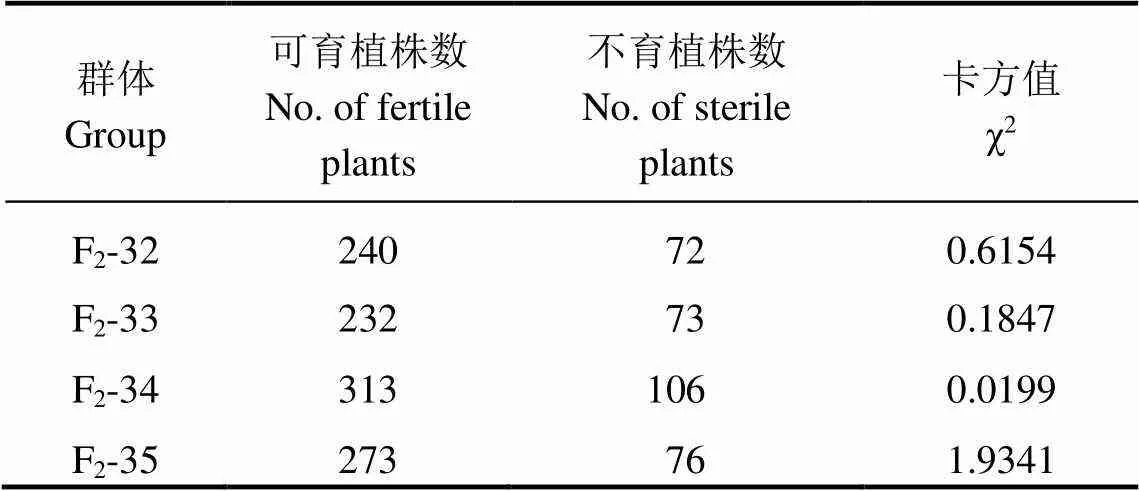
表1 ms7中雄性不育性状的遗传分析
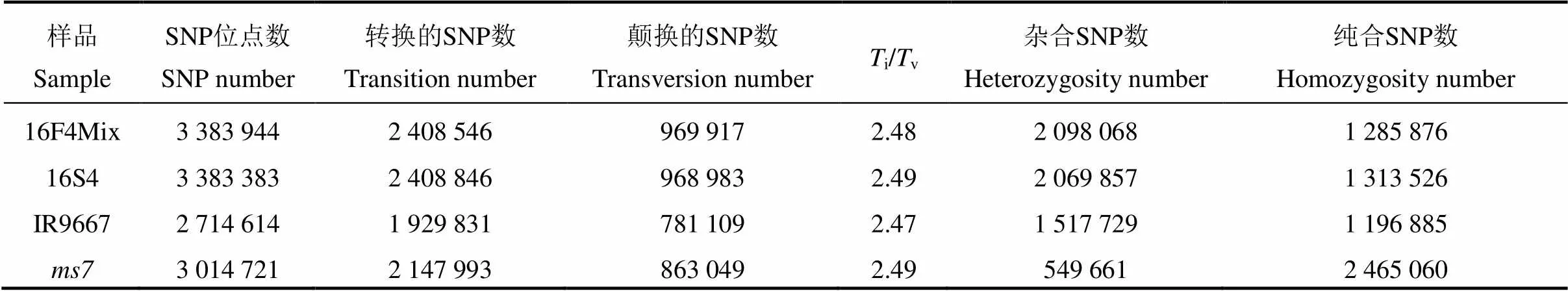
表2 SNP数据统计表
进一步对两个极端性状混池的SNP-index在染色体上的分布情况进行统计,计算两个子代混池的|ΔSNP-index|,结合性状进行关联分析后发现,当置信度为0.999时,第7染色体上15.5−16.7 Mb位置被筛选为候选区域(图3)。
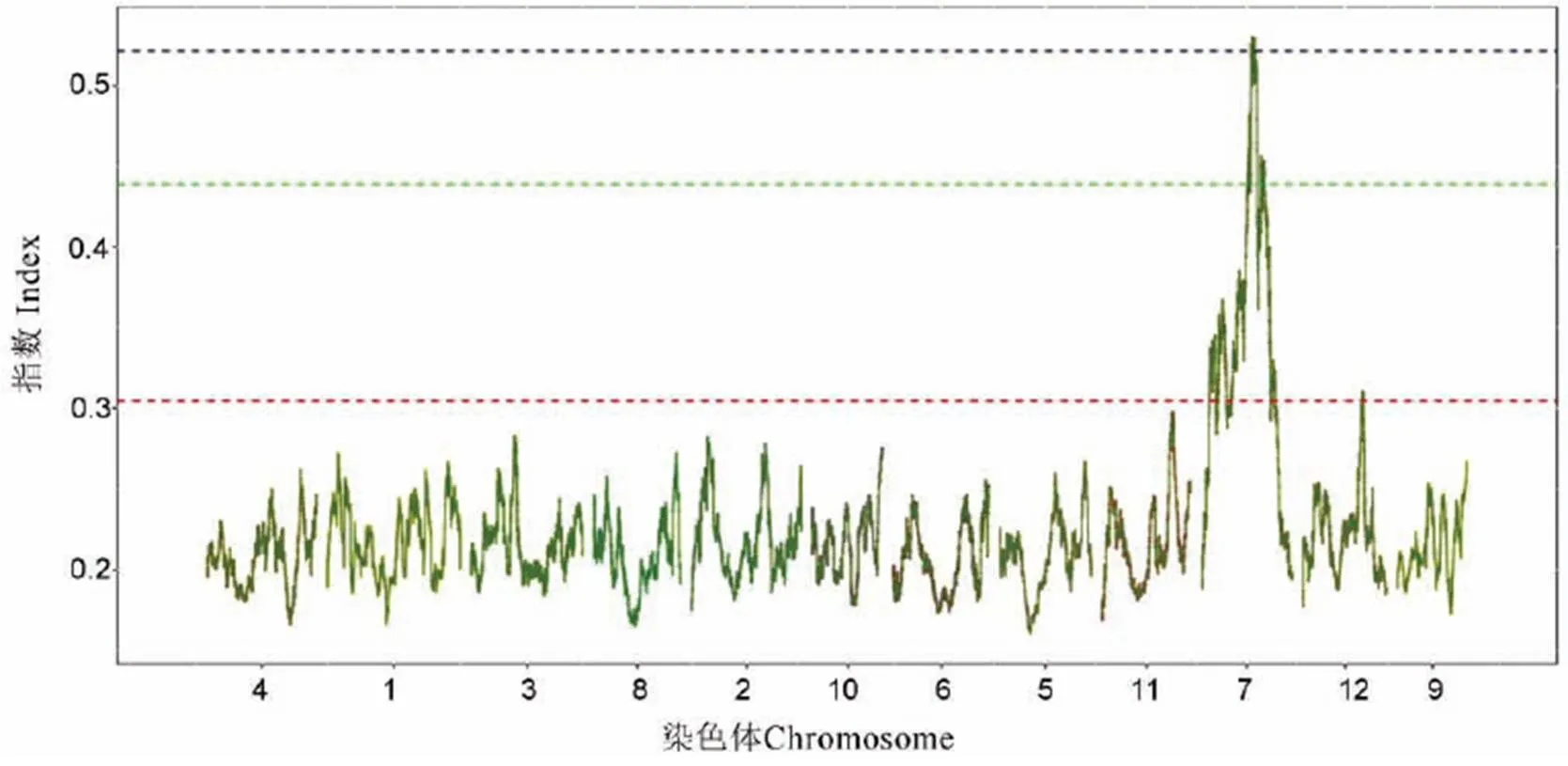
曲线是变异位点在染色体上所对应的Index值;红、绿和蓝三根线分别是95%、99%和99.9%置信区间阈值线。
Fig. 3. Distribution of SNP-index on the chromosomes.
2.5 雄性不育基因ms7的定位
为了验证测序结果并进行下一步的精细定位,我们根据两个亲本和IR96671的重测序结果在第7染色体长臂上设计了62个InDel标记,涵盖了BSA分析99.9%阈值对应的关联区间,并向两端延伸至区间7 968 748−21 483 477。对用于混池分析的材料按单株进行基因分型验证,确定基因的候选区间为15.0 Mb−18.4 Mb (15 032 966− 18 400 602)。从中筛选8个亲本间多态性明显的InDel标记对F2群体中剩余个体进行基因型鉴定,并结合各单株的育性表型,进一步缩小控制育性的定位区间(表3)。
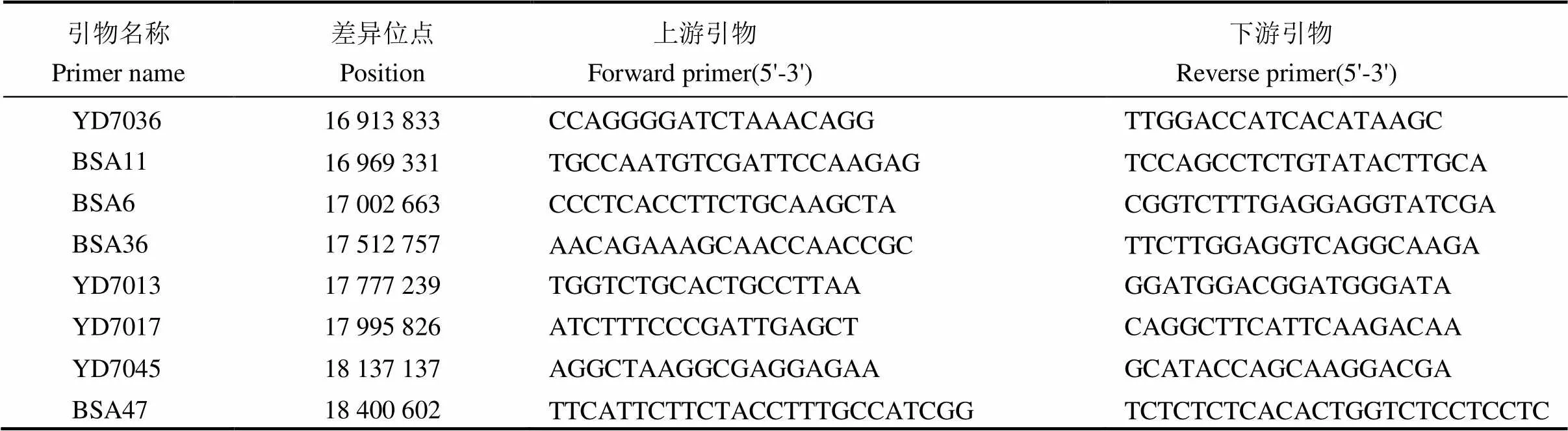
表3 8个InDel标记的引物信息
根据约14 000株F2群体的实验结果可挑选出7种类型重组单株,其基因型和表型数据如图4所示。其中,R1-R7是不同的单株,S表示该株不育,F表示该株可育。通过这7个单株我们将界定于第7染色体上BSA11(16.97 Mb)与YD7045 (17.99 Mb)之间约1.17 Mb的范围内。为了验证定位的结果,在BSA11−YD7045之间挑选杂合型材料发展群体在实验室培养箱种植,经标记检测,群体出现正常的基因型分离,从中挑选不同基因型的材料种植于温室,其结果显示,具有纯合型的单株均表现为不育,而杂合型和父本型均可育,此结果与定位结果一致。
2.5 GO分析
水稻第7染色体BSA11和YD7045间约1.17 Mb的物理区间内,根据日本晴参考基因组预测,共有137个基因位点。其中,已克隆的仅()[41]。剩下的136个位点中,根据基因本体(gene ontology, GO)注释分析,转座子或逆转录转座子合计51个,其他预测具有生物学功能的位点42个。从分子功能看,能与蛋白结合(GO: 0005515)的9个,具有转移酶活性(GO: 0016740)的8个,具有水解酶活性(GO: 0016787)的7个,等等;从生物学进程看,和预测参与花发育进程(GO:0009908),和预测参与细胞死亡过程(GO: 0008219),另有参与核酸类物质代谢(GO: 0006139)的11个,参与脂质代谢(GO: 0006629)的4个,参与碳水化合物代谢(GO: 0005975)的2个,参与底物运输(GO: 0006810)的4个。
3 讨论
本研究利用组培获得了水稻雄性不育突变体,除花粉败育外其他表型与野生型没有差异,遗传分析表明该性状受单个隐性核基因控制。水稻中已鉴定的影响育性的核基因中,如这样造成花粉完全败育,但又不影响胚囊或其他营养器官发育的基因,并不多见。
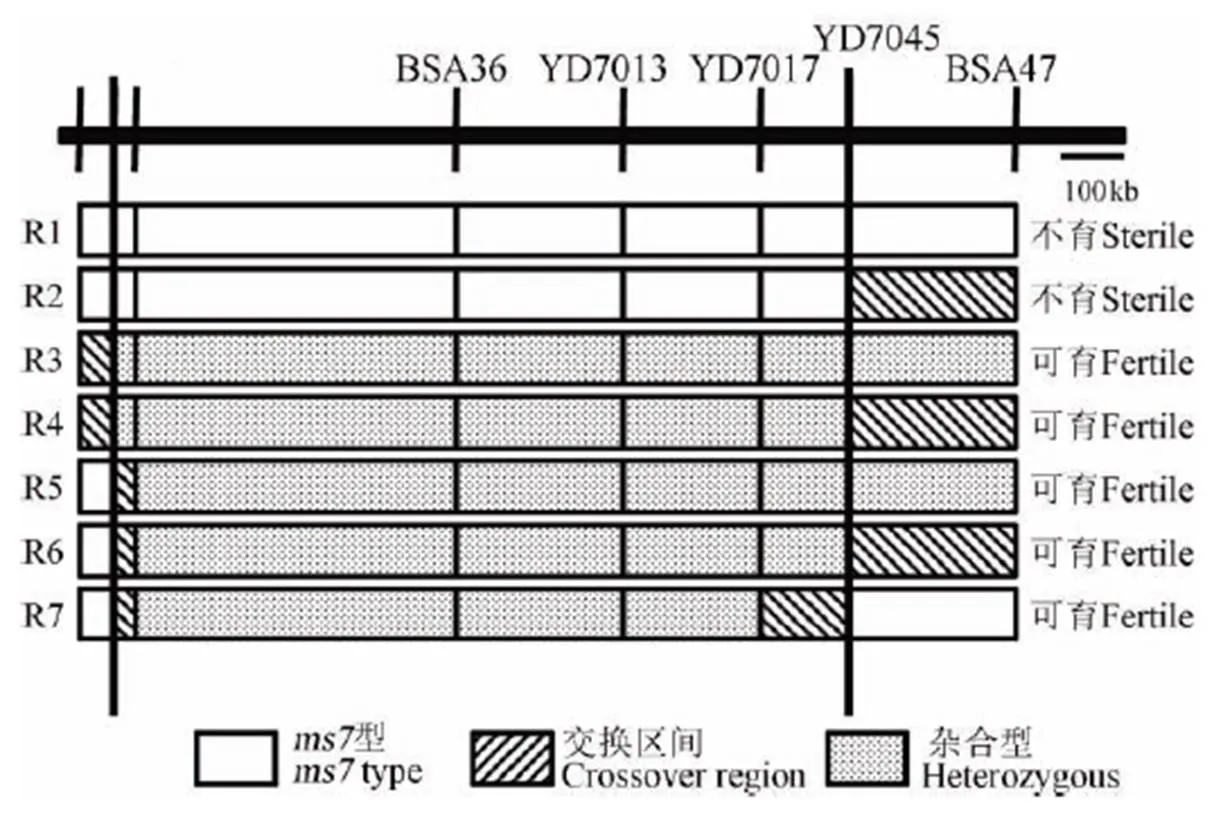
图4 重组单株的基因型组成和育性表现
Fig 4. Genotypic composition and fertility performance of recombinant plants.
突变体中控制雄性不育的基因定位在第7染色体上的InDel标记BSA11和YD7045之间,该区间内预测有基因位点137个。根据RiceXPro表达谱数据库(https://ricexpro.dna.affrc. go.jp/),共有42个基因在花药中有表达,相对表达量最高的是,预测编码丝氨酸羧肽酶,较高的有、、、和等,未发现花药特异表达基因。这些位点中,仅/得到鉴定。据报道,OsMSH4能与OsMSH5互作,形成异源二聚体,调控减数分裂过程中交叉的形成。突变体从终变期开始异常,导致雌雄配子均没有功能,花粉和胚囊均不育[42]。由于仅雄配子异常,与不同,推测它们不是同一位点突变。
本文对定位的物理区间1.17 Mb,仍然偏大,当中遗传因子众多,那些预测有生物学功能的位点,以及其他尚未注释的位点,都可能是的候选基因。为了确认基因在中是否有变异,我们将中恢161和中的DNA序列分成13段,通过PCR扩增后送公司测序,初步分析发现两者无差异。此外,为了能快速定位到目标位点,我们正对中恢161和进行第三代测序,尝试通过长序列的拼装,结合BSA二代测序结果,希望能直接找出与野生型在目标DNA区间上的差异。
[1] 鄂志国, 程本义, 孙红伟, 汪玉军, 朱练峰, 林海, 王磊, 童汉华, 陈红旗. 近40年我国水稻育成品种分析[J]. 中国水稻科学, 2019, 33(6): 523-531.
E Z G, Cheng B, Sun H, Wang Y, Zhu L, Lin Hai, Wang L, Tong H, Chen H. Analysis on Chinese improved rice varieties in recent four decades[J]., 2019, 33(6): 523-531. (in Chinese with English abstract)
[2] 范优荣, 曹晓风, 张启发. 光温敏雄性不育水稻的研究进展[J]. 科学通报, 2016, 61(35): 3822-3832.
Fan Y R, Cao X F, Zhang Q F. Progress on photoperiod thermo-sensitive genic male sterile rice[J]., 2016, 61(35): 3822-3832. (in Chinese with English abstract)
[3] Fan Y, Yang J, Mathioni S M, Yu J, Shen J, Yang X, Wang L, Zhang Q, Cai Z, Xu C, Li X, Xiao J, Meyers B C, Zhang Q., producing phased small-interfering RNAs, regulates photoperiod-sensitive male sterility in rice [J]., 2016, 113(52): 15144.
[4] Zhou H, Liu Q, Li J, Jiang D, Zhou L, Wu P, Lu S, Li F, Zhu L, Liu Z, Chen L, Liu Y G, Zhuang C. Photoperiod- and thermo-sensitive genic male sterility in rice are caused by a point mutation in a novel noncoding RNA that produces a small RNA[J]., 2012, 22(4): 649-660.
[5] Ding J, Lu Q, Ouyang Y, Mao H, Zhang P, Yao J, Xu C, Li X, Xiao J, Zhang Q. A long noncoding RNA regulates photoperiod-sensitive male sterility, an essential component of hybrid rice[J]., 2012, 109(7): 2654-2659
[6] Zhou H, Zhou M, Yang Y, Li J, Zhu L, Jiang D, Dong J, Liu Q, Gu L, Zhou L, Feng M, Qin P, Hu X, Song C, Shi J, Song X, Ni E, Wu X, Deng Q, Liu Z, Chen M, Liu Y G, Cao X, Zhuang C. RNase ZS1processesUbmRNAs and controls thermosensitive genic male sterility in rice [J]., 2014, 5: 4884.
[7] Pitnjam K, Chakhonkaen S, Toojinda T, Muangprom A. Identification of a deletion inand development of gene-based markers for selection[J]., 2008, 228(5): 813-822.
[8] Yu B, Liu L, Wang T. Deficiency of very long chain alkanes biosynthesis causes humidity-sensitive male sterility via affecting pollen adhesion and hydration in rice [J]., 2019, 42(12): 3340.
[9] Xue Z, Xu X, Zhou Y, Wang X, Zhang Y, Liu D, Zhao B, Duan L, Qi X. Deficiency of a triterpene pathway results in humidity-sensitive genic male sterility in rice[J]., 2018, 9: 604.
[10] Chen H, Zhang Z, Ni E, Lin J, Peng G, Huang J, Zhu L, Deng L, Yang F, Luo Q, Sun W, Liu Z, Zhuang C, Liu YG, Zhou H. HMS1 interacts with HMS1I to regulate very-long-chain fatty acid biosynthesis and the humidity-sensitive genic male sterility in rice () [J]., 2020, 225(5): 2077-2093.
[11] 王多祥, 祝万万, 袁政, 张大兵. 水稻雄性发育功能基因的发掘及应用[J]. 生命科学, 2016, 28(10): 1180-1188.
Wang DX, Zhu WW, Yuan Z, Zhang DB. Functional research of rice male reproduction and its utilization in breeding[J]., 2016, 28(10): 1180-1188. (in Chinese with English abstract)
[12] Li L, Li Y, Song S, Deng H, Li N, Fu X, Chen G, Yuan L. An anther development F-box (ADF) protein regulated by tapetum degeneration retardation (TDR) controls rice anther development [J]., 2015, 241(1): 157-166.
[13] Lee S, Jung K H, An G, Chung Y Y. Isolation and characterization of a rice cysteine protease gene,, using T-DNA gene-trap system[J]., 2004, 54(5): 755-765.
[14] Niu N, Liang W, Yang X, Jin W, Wilson Z A, Hu J, Zhang D. EAT1 promotes tapetal cell death by regulating aspartic proteases during male reproductive development in rice [J]., 2013, 4 : 1445.
[15] Shi J, Tan H, Yu X H, Liu Y, Liang W, Ranathunge K, Franke R B, Schreiber L, Wang Y, Kai G, Shanklin J, Ma H, Zhang D.is required for anther and microspore development in rice and encodes a fatty acyl carrier protein reductase [J]., 2011, 23(6): 2225-2246.
[16] Mondol P C, Xu D, Duan L, Shi J, Wang C, Chen X, Chen M, Hu J, Liang W, Zhang D. Defective Pollen Wall 3 (DPW3), a novel alpha integrin-like protein, is required for pollen wall formation in rice [J]., 2020, 225(2): 807-822.
[17] Yang X, Liang W, Chen M, Zhang D, Zhao X, Shi J. Rice fatty acyl-CoA synthetase OsACOS12 is required for tapetum programmed cell death and male fertility [J]., 2017, 246(1): 105-122.
[18] Xu D, Shi J, Rautengarten C, Yang L, Qian X, Uzair M, Zhu L, Luo Q, An G, Waßmann F, Schreiber L, Heazlewood J L, Scheller H V, Hu J, Zhang D, Liang W.() encodes an acyl transferase required for rice pollen development[J]., 2017, 173(1): 240-255.
[19] Men X, Shi J, Liang W, Zhang Q, Lian G, Quan S, Zhu L, Luo Z, Chen M, Zhang D. Glycerol-3-phosphate acyltransferase 3 (OsGPAT3) is required for anther development and male fertility in rice[J]., 2017, 68(3): 513-526.
[20] Yu J, Meng Z, Liang W, Behera S, Kudla J, Tucker MR, Luo Z, Chen M, Xu D, Zhao G, Wang J, Zhang S, Kim YJ, Zhang D. A rice Ca2+binding protein is required for tapetum function and pollen formation[J]., 2016, 172(3): 1772-1786.
[21] Zheng S, Li J, Ma L, Wang H, Zhou H, Ni E, Jiang D, Liu Z, Zhuang C.controls ROS production and the initiation of tapetal PCD by epigenetically regulatingexpression in rice anthers[J]., 2019, 116(15): 7549-7558.
[22] Aya K, Ueguchi-Tanaka M, Kondo M, Hamada K, Yano K, Nishimura M, Matsuoka M. Gibberellin modulates anther development in rice via the transcriptional regulation of GAMYB[J]., 2009, 21(5): 1453.
[23] Liu Z, Bao W, Liang W, Yin J, Zhang D. Identification ofand analysis of the regulatory role ofin rice anther development[J]., 2010, 52(7): 670-678.
[24] Xiang X J, Sun L P, Yu P, Yang Z F, Zhang P P, Zhang Y X, Wu W X, Chen D B, Zhan X D, Khan R M, Abbas A, Cheng S H, Cao L Y. The MYB transcription factor Baymax1 plays a critical role in rice male fertility[J]., 2021, 134(2): 453-471.
[25] Jung K H, Han M J, Lee Y S, Kim Y W, Hwang I, Kim M J, Kim Y K, Nahm B H, An G. Riceis a major regulator of early tapetum development[J]., 2005, 17(10): 2705-2722.
[26] Ko S S, Li M J, Ku M S B, Ho Y C, Lin Y J, Chuang M H, Hsing H X, Lien Y C, Yang H T, Chang H C, Chan M T. The bHLH142 transcription factor coordinates with TDR1 to modulate the expression ofand regulate pollen development in rice[J]., 2014, 26(6): 2486-2504.
[27] Fu Z, Yu J, Cheng X, Zong X, Xu J, Chen M, Li Z, Zhang D, Liang W. The rice basic Helix-Loop-Helix transcription factor TDR INTERACTING PROTEIN2 is a central switch in early anther development., 2014, 26(4): 1512-1524.
[28] Li N, Zhang D S, Liu H S, Yin C S, Li X X, Liang W Q, Yuan Z, Xu B, Chu H W, Wang J, Wen T Q, Huang H, Luo D, Ma H, Zhang D B. The ricegene is required for tapetum degradation and anther development[J]., 2006, 18(11): 2999-3014.
[29] Ji C, Li H, Chen L, Xie M, Wang F, Chen Y, Liu YG. A novel rice bHLH transcription factor, DTD, acts coordinately with TDR in controlling tapetum function and pollen development[J]., 2013, 6(5): 1715-1718.
[30] Cao H, Li X, Wang Z, Ding M, Sun Y, Dong F, Chen F, Liu L, Doughty J, Li Y, Liu Y X. Histone H2B monoubiquitination mediated by HISTONE MONOUBI-QUITINATION1 and HISTONE MONOUBIQUITI-NATION2 is involved in anther development by regulating tapetum degradation-related genes in rice[J]., 2015, 168(4): 1389.
[31] Li H, Yuan Z, Vizcay-Barrena G, Yang C, Liang W, Zong J, Wilson Z A, Zhang D.encodes a PHD-finger protein that is required for tapetal cell death and pollen development in rice[J]., 2011, 156(2): 615-630.
[32] Yang Z, Liu L, Sun L, Yu P, Zhang P, Abbas A, Xiang X, Wu W, Zhang Y, Cao L, Cheng S.functions as a transcriptional activator to regulate programmed tapetum development and pollen exine formation in rice[J]., 2019, 99(1-2): 175-191.
[33] Yang Z, Sun L, Zhang P, Zhang Y, Yu P, Liu L, Abbas A, Xiang X, Wu W, Zhan X, Cao L, Cheng S., encoding a PHD-finger transcription factor, regulates Ubisch bodies and pollen wall formation in rice[J]., 2019, 99(5): 844-861.
[34] Li X, Gao X, Wei Y, Deng L, Ouyang Y, Chen G, Li X, Zhang Q, Wu C. Rice APOPTOSIS INHIBITOR5 coupled with two DEAD-box adenosine 5’-triphosphate-dependent RNA helicases regulates tapetum degeneration [J]., 2011, 23(4): 1416-1434.
[35] Bai W, Wang P, Hong J, Kong W, Xiao Y, Yu X, Zheng H, You S, Lu J, Lei D, Wang C, Wang Q, Liu S, Liu X, Tian Y, Chen L, Jiang L, Zhao Z, Wu C, Wan J.() encodes an ATP-citrate lyase required for tapetum programmed cell death[J]., 2019, 181(3): 1223-1238.
[36] Zhang P, Zhang Y, Sun L, Sinumporn S, Yang Z, Sun B, Xuan D, Li Z, Yu P, Wu W, Wang K, Cao L, Cheng S. The rice AAA-ATPase OsFIGNL1 is essential for male meiosis[J]., 2017, 8: 1639.
[37] Tan H, Liang W, Hu J, Zhang D.encodes a secretory fasciclin glycoprotein required for male reproductive development in rice[J]., 2012, 22(6): 1127-1137.
[38] Li H, Durbin R. Fast and accurate long-read alignment with Burrows-Wheeler transform[J]., 2009, 25(14), 1754-1760.
[39] McKenna A, Hanna M, Banks E, Sivachenko A, Cibulskis K, Kernytsky A, Garimella K, Altshuler D, Gabriel S, Daly M, DePristo M A. The genome analysis toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data[J]., 2010, 20(9): 1297-1303.
[40] Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, Marth G, Abecasis G, Durbin R. The sequence alignment/map format and SAMtools[J]., 2009, 25(16): 2078-2079.
[41] Zhang D, Luo X, Zhu L. Cytological analysis and genetic control of rice anther development[J]., 2011, 38(9): 379-390.
[42] Wang C, Wang Y, Cheng Z, Zhao Z, Chen J, Sheng P, Yu Y, Ma W, Duan E, Wu F, Liu L, Qin R, Zhang X, Guo X, Wang J, Jiang L, Wan J. The role of OsMSH4 in male and female gamete development in rice meiosis[J]., 2016, 67(5): 1447-1459.
Genetic Analysis and Gene Mapping of a Male Sterile Mutantin Rice
YANG Jinyu1,#, BAI Chen2,3,#, DING Xiaohui2,3, SHEN Hongfang1, WANG Lei1, YING Jiezheng1,*, E Zhiguo1,*
(1State Key Laboratory of Rice Biology, China National Rice Research Institute, Hangzhou 310006, China;2Key Laboratory of Plant Molecular Physiology, Institute of Botany, Chinese Academy of Sciences, Beijing 100093, China;3University of Chinese Academy of Sciences, Beijing 100049, China;#These authors contributed equally to the work;*Corresponding author, E-mail: yingjiezheng@caas.cn; ezhiguo@caas.cn)
【】Genes related to pollen development and fertility can be identified through the study on male sterile mutants, which is helpful to analyze the whole regulatory network of male reproductive development in rice. 【】The fertility and agronomic traits of the() mutant and its wild type were compared under conventional planting conditions in Fuyang, Zhejiang and Lingshui, Hainan. The sterile gene was mapped by bulked segregant analysis and map-based cloning.【】During the whole growth period, the growth rate ofmutant was the same as that of wild type, and there was no significant difference in plant height, tiller number, leaf number, leaf size, panicle length and spikelets per panicle betweenand its wild type. However,exhibited slender and white anthers with inactive pollen grains which couldn’t be stained with I2-KI, and no seeds were produced. Observation results of anther cross-sections exhibited that tapetum programmed cell death (PCD) was delayed. Genetic analysis showed that pollen abortion was controlled by a single recessive nuclear gene, which was mapped to a 1.17 Mb region between BSA11 and YD7045 on chromosome 7. 【】It will contribute to the further molecular cloning and functional analysis of male sterile genein rice.
rice; male sterile; bulked segregant analysis; gene mapping
10.16819/j.1001-7216.2022.210302
2021-03-03;
2021-04-19。
浙江省自然科学基金资助项目(LY17G030031); 国家自然科学基金面上项目(32072050, 71773140); 中央级公益性科研院所基本科研业务费专项(CPSIBRF-CNRRI-202129)。
