番茄转录因子SlNAC29在调控植株衰老中的作用及机理
2022-01-17王萍郑晨飞王娇胡璋健邵淑君师恺
王萍,郑晨飞,王娇,胡璋健,邵淑君,师恺
番茄转录因子SlNAC29在调控植株衰老中的作用及机理
王萍,郑晨飞,王娇,胡璋健,邵淑君,师恺
浙江大学农业与生物技术学院,杭州 310058
【背景】番茄()作为连续发芽分化和坐果的重要园艺作物,早衰是限制其生长期长短、产量和品质的重要因素。NAC(NAM、ATAF1/2和CUC2)转录因子家族参与调控拟南芥、水稻等多种植物衰老进程,但在番茄中的研究尚不深入。目前已知,SlNAP2(NAC-like, activated by apetala3/pistillata)参与番茄植株衰老进程。【目的】为的同源基因,对其在番茄植株衰老中的功能及调控机制进行研究,以期为园艺栽培中番茄的衰老调控及种质创新提供科学依据。【方法】以野生型番茄(condine red,CR)为背景,采用qRT-PCR技术明确在不同衰老阶段叶片中的相对表达量,并分别利用CRISPR/Cas9基因编辑技术和基因过表达技术构建纯合突变体及OE:稳定过表达植株。在此基础上,在自然生长状态下和黑暗处理诱导衰老条件下,对野生型、突变体和OE:过表达植株的生长、叶绿素含量、光合作用、叶片衰老和叶绿素降解相关基因的相对表达量等参数进行分析,明确SlNAC29转录因子在调控番茄植株衰老中的生物学功能;进一步利用聚类热图分析过表达植株OE:中29个衰老相关基因、叶绿素降解基因以及ABA合成/信号转导相关基因的相对表达量。并选取在黑暗诱导衰老条件下不同株系植株中表达差异明显的4个基因进行凝胶迁移阻滞分析(electrophoretic mobility shift analysis,EMSA),以鉴定SlNAC29直接转录调控的靶标基因及其与衰老调控的关系。【结果】在初老叶和衰老叶片中的相对表达量较嫩叶和成熟叶显著上升。自然生长状态下,突变体材料与野生型长势以及光合速率无明显差异,而过表达材料OE:株高则显著低于野生型植株,叶绿素含量和光合速率分别是野生型植株的25%和50%。在黑暗诱导衰老的条件下,野生型植株叶片明显变黄,叶绿素含量显著下降。突变缓解了叶片衰老程度,叶片无明显变黄,叶绿素含量是野生型的3倍,衰老相关基因(senescence-associated genes,SAGs)和叶绿素降解基因的表达量均较低。OE:则相反,叶片衰老程度比野生型和突变体均明显严重。基因聚类分析表明多个衰老相关基因和叶绿素降解基因在OE:植株中显著上调表达。EMSA鉴定到SlNAC29能够直接与衰老相关基因家族SAGs成员()启动子绑定,且在OE:中的相对表达量较野生型和突变体显著增加。【结论】转录因子SlNAC29调控番茄植株的衰老,促进番茄叶片在黑暗诱导条件下的衰老进程。SlNAC29直接绑定衰老相关基因启动子区域调控其转录表达。
番茄;SlNAC29;转录调控;衰老;
0 引言
【研究意义】植物衰老是指植株在生长发育过程中由外部环境和内部遗传因素共同控制导致生理功能衰退,最终自然死亡的过程[1]。叶片衰老是植物叶片生长发育的最后阶段,是植物衰老的直接体现。提早衰老(即早衰)严重影响作物的光合作用、营养吸收和物质转运等过程。番茄是连续发育多叶片多节间植物,早衰在其长季节栽培中尤为突出,极大地限制了番茄产量和品质潜力的发挥[2]。植物衰老是基因控制的程序化衰亡,转录因子是其中重要的调控因子。相对于模式植物拟南芥而言,人们对早衰问题较为严重的番茄作物的衰老关键转录因子及其调控过程还知之甚少。对其开展研究,对于延长番茄生育期,提高作物产量和品质,具有重要的理论和现实意义。【前人研究进展】植物NAC(NAM、ATAF1/2和CUC2)家族是调控植株衰老的重要转录因子[3]。拟南芥自然衰老进程中,30余个NAC基因表达量增强[4]。过表达拟南芥NAC转录因子、、、、等会引起植株早衰,敲除这些基因则可以延缓衰老[5-8]。与此相似,过表达水稻显著加速了植株衰老进程,而敲除该基因可明显延缓衰老[9]。在棉花中,通过RNAi方法降低NAC转录因子亚家族成员的表达量可延缓植株衰老,提高棉花产量和纤维质量[10]。番茄早衰相关的研究也有一定报道,与拟南芥AtNAC2同源的NAC转录因子SlORE1s()的RNAi株系衰老延迟,碳同化增强,果实数量和可溶性固形物含量增加[11]。此外,在黑暗环境诱导衰老条件下,与同源的过表达番茄植株相较于野生型对照提早衰老。RNAi抑制则延缓其衰老进程,提高了果实产量和含糖量[12]。进一步探究发现,SlNAP2能够直接转录调控衰老相关基因()、叶绿素降解基因()和()、ABA合成基因()、转运基因()以及降解基因(ABA 8-hydroxylase)。【本研究切入点】多种植物NAC转录因子家族成员在调控植株衰老中发挥了重要的作用。番茄作为连续花芽分化和坐果的重要园艺作物,早衰是决定其生长期长短、产量高低和品质优劣的重要因素。目前,已有研究发现SlNAP2参与调控衰老及叶绿素降解等相关基因如、的表达。为的同源基因,在氨基酸水平有约72.7%的相似度,但其在调控番茄衰老中的功能及机制尚不明确,研究番茄SlNAC29在衰老中的功能及其靶标基因,有利于增进对番茄衰老及其调控机制的认识。【拟解决的关键问题】本研究拟在明确在番茄不同衰老阶段转录本变化及亚细胞定位的基础上,利用CRISPR/Cas9基因编辑技术构建突变体,利用基因过表达技术构建OE:稳定过表达植株;在自然状态及黑暗处理诱导衰老条件下,通过对植物叶绿素含量和光合作用相关参数的分析,明确SlNAC29在调控植株衰老中的生物学功能;利用基因转录分析和EMSA的方法,鉴定SlNAC29直接转录调控的靶标基因及其与衰老调控的关系,以期为园艺栽培中番茄的衰老调控及种质创新提供科学依据。
1 材料与方法
试验于2019—2020年在浙江大学农业与生物技术学院蔬菜研究所实验温室和实验室进行。
1.1 试验材料
供试番茄()材料Condine Red(CR)为野生型对照(wild-type,WT)。番茄种子置于28℃培养箱,2—3 d后,播种至装有草炭、蛭石混合基质(体积比为3﹕1)的72孔穴盘,待植物长出2片真叶,移苗至营养钵(外径10 cm,高8.5 cm)中继续生长。控制生长环境条件与番茄适宜的自然生长条件相一致[13]:光周期16 h/8 h(昼/夜),温度25℃/21℃(昼/夜),湿度80%左右,光照强度为200 μmol·m-2·s-1,其间观察植株长势、叶绿素含量和光合作用等指标。黑暗是一种被广泛用以诱导植株衰老的处理方式[14]。黑暗环境会引起叶片变黄,光合作用和叶绿素含量的下降,衰老相关基因的表达增强[15]。对于黑暗诱导叶片衰老试验,待播种后5周左右,摘取植株第二、三片完全展开叶于湿润滤纸上,覆以锡箔纸遮光诱导衰老,7 d后测定叶绿素含量及相关基因相对表达量。
1.2 SlNAC29突变体植株的构建
参照Pan等[16]方法构建的CRISPR/ Cas9载体。通过CRISPR-P 2.0网站设计靶序列(5′- TTCGATCCCTGGGTATTACC-3′)[17],将合成的靶序列退火并插入AtUb-sgRNA-AtUBQ-Cas9载体的Ⅰ位点,然后连接至双元表达载体pCAMBIA1301,转化至根癌农杆菌GV3101中,通过组织培养技术获得基因编辑材料[18]。采用DNA测序对CRISPR/Cas9诱导的突变进行测定,进一步筛选出2个纯合无Cas9的T2代突变体株系-1和-2,所用引物见表1。
1.3 SlNAC29过表达植株的构建
根据番茄基因组数据库(https://solgenomics.net/)获得(Solyc05g007770)的编码序列(coding sequence,CDS),利用Primer5软件设计过表达特异性引物(表1)。扩增并连接到载体pFGC1008-3×HA上,重组质粒构建成功后,电击转化至GV3101中,同样采用植物组织培养的方法将重组载体转化至番茄。组织培养获得的OE:T0代植株取嫩叶叶片提取蛋白后进行Western blot验证,具有目的条带的T0代株系用于连续繁种筛选,并获得2个纯合的T2代株系OE:-1和OE:-2。
1.4 SlNAC29的亚细胞定位
将的CDS区构建到C端含绿色荧光蛋白(green fluorescent protein,GFP)标签的载体上,引物序列见表1。将构建好的载体转化GV3101,在本氏烟草叶片(带有红色荧光蛋白RFP标签的核定位蛋白烟草)中进行瞬时过表达,2 d后进行激光共聚焦观察(Zeiss LSM 780)。GFP的激发光和发射光为488 nm/500—530 nm,RFP的激发光和发射光为561 nm/580—620 nm。
1.5 植物总RNA提取、反转录及qRT-PCR分析
参照TIANGEN RNA Simple Total RNA Kit的试剂盒说明书操作步骤提取植物总RNA。参照TOYOBO反转录试剂盒进行cDNA合成。qRT-PCR反应条件及体系参见SYBR Green TR-PCR Kit试剂盒(Takara,RR420A),在LightCycler®480Ⅱ Real-Time PCR detection system(Roche,Swiss)中进行。以作为内标,相对基因表达参照2-ΔΔCT法[19]计算。候选基因的特异性引物及引物见表1。
1.6 光合速率测定
选取5周龄左右植株的第二、三片完全展开叶,用LI-6400型光合荧光测量系统(美国LI-COR公司)测定番茄净光合作用速率(n)。测定条件:光强为500 μmol·m-2·s-1,CO2浓度为400 μL·L-1,叶面温度为(25±1.5)℃。
1.7 叶绿素含量测定
选取5周龄左右植株的第二、三片完全展开叶,采用英国Hansatech公司的手持式叶绿素含量测定仪CL-01 Chlorophyll Content Meter测量叶片叶绿素含量。
1.8 基因表达聚类热图分析
利用MeV(Multiexperiment viewer)聚类分析软件对OE:材料中衰老相关的29个基因表达进行分析。包括:1)()等衰老直接相关基因(SAGs);2)()等叶绿素降解相关基因;3)()等ABA合成和信号转导相关基因。
1.9 凝胶迁移阻滞分析(electrophoretic mobility shift analysis,EMSA)
参照Hellman等[20]方法构建原核表达载体pET32a-SlNAC29,将连接产物转化大肠杆菌BL21中。加入终浓度为0.1 mmol·L-1的异丙基硫代半乳糖苷(isopropyl β-D-thiogalactoside,IPTG),6 h后收集菌体,超声破碎后进行SDS-PAGE检测。利用Novagen公司的pET蛋白纯化体系进行蛋白纯化。探针的标记使用Thermo Fisher Scientific公司的Biotin 3´End DNA Labeling Kit试剂盒,退火为DNA双链。进一步利用LightShift™ Chemiluminescent EMSA Kit试剂盒进行EMSA反应,最后用Bio-Rad凝胶成像系统进行曝光显色。
1.10 数据分析
试验设3个重复,每个重复6棵植株。试验结果均为3次生物学重复的平均值。利用Microsoft Excel 2019整理数据,SAS 9.1 Tukey法进行差异显著性分析,Origin 2019进行图形绘制。
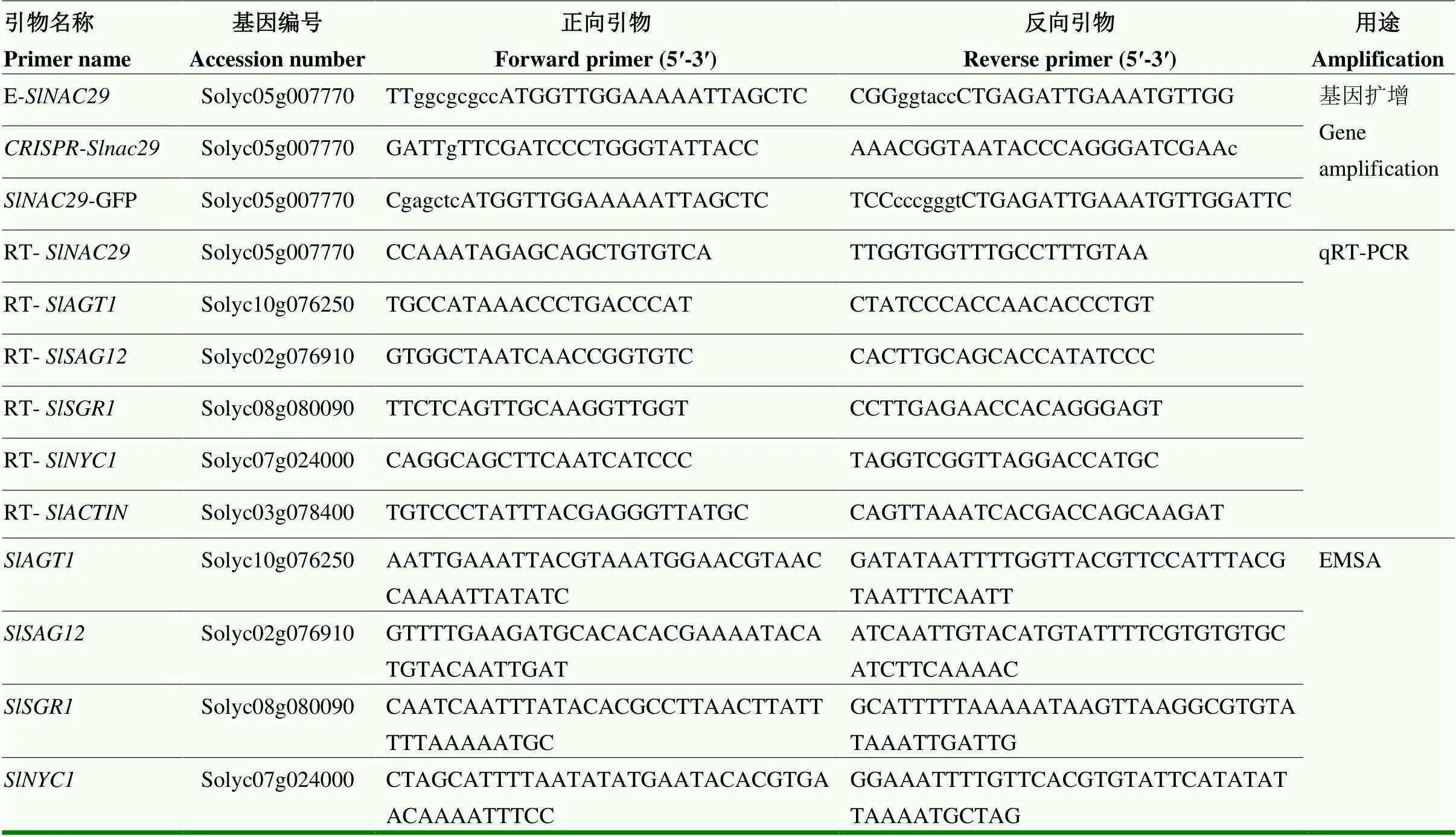
表1 引物
2 结果
2.1 不同衰老阶段叶片SlNAC29表达的差异
(Solyc05g007770)为(Solyc04g005610)的同源基因,在氨基酸水平约有72.7%的相似度(图1-A)。为了探究在植株衰老中的作用,参考Ma等[12]试验方法把叶片分为嫩叶(young leaves,YL)、成熟叶(mature leaves,ML)、初老叶(early senescent leaves,ES)和衰老叶(senescent leaves,SL)(图1-B),并采用qRT-PCR方法分析在不同衰老阶段叶片中的基因表达差异。与成熟叶相比,嫩叶中表达量无明显变化,而初老叶和衰老叶中的基因表达量均显著上升,分别是成熟叶的35倍和1 757倍(图1-B)。对SlNAC29进行亚细胞定位,发现SlNAC29-GFP定位在细胞膜和细胞核(图1-C)。
2.2 SlNAC29突变及过表达对自然生长状态下植株衰老的影响
为了探究SlNAC29是否参与植株衰老的调控,通过CRISPR/Cas9基因编辑技术和基因过表达技术分别获得2个纯合基因突变株系-1和-2以及2个纯合基因过表达株系OE:-1和OE:-2。其中,-1的靶序列处缺失7个碱基,-2的靶序列处缺失11个碱基,均导致翻译提前终止(图2-A),过表达株系在C端携带的HA蛋白标签能够利用Western Blot的方法予以鉴定(图2-B)。与WT相比,突变体植株的生长没有明显变化,而OE:植株叶片发黄、株型矮小(图2-C)。在第5周,对不同植株的第二、三片真叶进行叶绿素含量和气体交换参数测定,发现与WT均无显著差异,OE:的叶绿素含量和光合速率(n)分别是WT植株的25%和50%(图2-D和图2-E)。以上结果表明,在自然生长状态下,突变体和WT植株无明显差异,而过表达则加剧了植株的衰老进程。

A:SlNAC29和SlNAP2蛋白序列比对。*:2种蛋白中相同的氨基酸;B:qRT-PCR检测野生型植株不同衰老阶段叶片中SlNAC29的表达量。★:在P<0.05水平上差异显著(n=3)。YL:嫩叶,ML:成熟叶,ES:初老叶,SL:衰老叶;C:SlNAC29亚细胞定位。比例尺=50 μm

A:Slnac29突变体基因编辑位点示意图;B:Western Blot验证SlNAC29 2个纯合过表达株系中SlNAC29-HA融合蛋白;C:SlNAC29突变体及过表达植株5周龄生长表型;D:SlNAC29突变体及过表达植株叶绿素含量;E:SlNAC29突变体及过表达植株光合速率。叶绿素含量及光合速率均测定5周左右植株第二、三片成熟叶。误差线表示3次测量均值的标准差,不同小写字母表示不同处理组间在P<0.05水平显著性差异。下同
2.3 SlNAC29突变和过表达对黑暗条件下叶片衰老的影响
选取WT、和OE:植株叶片,参考Ma等[12]方法进行离体连续黑暗处理诱导叶片衰老,置于正常光周期的离体叶片作为对照。7 d后,突变植株叶片衰老程度明显较轻,OE:植株的叶片衰老较WT更为严重(图3-A)。植株叶片的叶绿素含量明显高于WT植株,而OE:植株则相反(图3-B)。进一步对叶片的基因表达进行分析,发现黑暗诱导衰老后叶片中衰老相关基因、以及叶绿素降解相关基因、表达量均明显低于WT植株,而OE:叶片中上述基因表达量均明显上升(图3-C)。此外,正常光周期对照条件下,中叶绿素含量和衰老相关基因转录本与WT没有显著性差异,而过表达植株较WT植株同样具有较低的叶绿素含量和较高的衰老相关基因转录本(图3-C)。以上数据表明,在黑暗诱导衰老的条件下,降低了衰老和叶绿素降解相关基因的表达量,减轻了叶片衰老程度,而OE:则提高了衰老和叶绿素降解相关基因的表达,加重了叶片衰老程度。
2.4 SlNAC29绑定及转录激活的靶标基因鉴定
为了进一步阐明SlNAC29在分子水平上调控衰老的机制,利用OE:材料对衰老相关的29个基因的相对表达量进行分析。如图4-A所示,大部分基因尤其是SAGs和叶绿素降解基因在OE:植株中显著上调表达。进一步选取黑暗诱导衰老条件下、OE:及WT植株中明显差异表达的、、和4个基因进行凝胶迁移阻滞分析。通常NAC转录因子的目标启动子核心结合位点为CACG[21],序列分析表明、、和的启动子均具有多个NAC核心结合位点(表2)。结果发现,特异性地与启动子绑定,但未与、和发生绑定结合(图4-B)。此外,在植株水平,株系中的表达量较WT植株无差异,但在OE:株系中其表达量显著增加(图4-C)。在分子水平上,SlNAC29直接绑定衰老相关基因启动子区域,并正调控其表达,表明SlAGT1可能在SlNAC29调控的植株衰老过程中发挥一定的作用。

A、B:黑暗诱导叶片衰老表型图及叶绿素含量;C:对照与黑暗诱导衰老条件下,叶片中衰老相关基因的相对表达

表2 目标基因启动子的SlNAC29靶标位点统计
3 讨论
番茄是中国栽培面积最大的重要蔬菜作物之一。近年来,随着长季节栽培等技术的规模化应用,植株早衰导致的产量不高和品质不良逐渐成为影响番茄产业健康发展的重要瓶颈[22]。因此,研究调控番茄衰老的关键基因及其调控机制对于延缓番茄衰老进程,充分发挥其生物潜能具有重要意义。
植株衰老由一系列的内部和外部信号触发,包括植株苗龄、植物激素、弱光/黑暗等环境压力和病原体感染等[23-24]。在这套错综复杂的调控网络机制中,转录因子是其中重要的调控因子[25]。NAC转录因子目前被广泛报道在植物植株衰老调控中发挥重要作用,但是作用机制尚不清楚[26-27]。已有研究发现番茄SlNAP2参与调控植株衰老,作为的同源基因,其在衰老中的作用及机制尚不清晰。本研究发现了一个新的NAC转录因子SlNAC29调控植株衰老及其机制。黑暗诱导条件下,过表达呈现早衰,然而突变体能够显著减轻植株衰老程度,维持碳同化的正常进行。本研究发现单突变体即可发挥作用,后续可以继续构建和的双突变体材料,以探究二者共同的作用机制。此外,同源基因拟南芥已被鉴定为植株衰老的中枢正向调节因子。近期有研究发现即可被多种胁迫强烈诱导,过表达对2种细菌性病害以及干旱的防御能力显著增强[28]。本研究发现突变体植株衰老程度显著减轻,是一种能够抑制番茄早衰的优良种质资源。
通常情况下,转录因子都是通过直接识别下游靶基因的启动子区域,转录激活或抑制靶基因表达,从而影响植株的生长发育[29]。探究转录因子下游的靶基因对明确转录因子的作用机制至关重要。据报道,拟南芥基因组中约有10%的基因或超过2 500个基因在植株衰老过程中上调[30-31]。许多衰老相关基因参与诸如基因调控、信号转导、大分子物质降解和营养物质再转化等衰老过程[32]。本研究中,黑暗诱导衰老条件下,OE:的(如)和叶绿素降解基因(如)显著上调表达,在植株中则相反。选取黑暗诱导衰老条件下突变体OE:及WT植株中明显差异表达的、、和4个基因进行凝胶迁移阻滞检测。其中,特异性地与启动子绑定,但未与、和发生绑定结合。在植株体内,株系中的表达较WT植株无差异,但OE:株系中其表达量显著增加。前人研究表明番茄SlNAP2能够激活衰老相关基因,叶绿素降解基因和等,从而直接调控叶片衰老。拟南芥中AtNAP能够直接绑定加速促进植株衰老[33]。然而,本研究发现OE:中衰老相关基因表达量与野生型相比无显著变化,而表达量则相对较高(图5-A),且其启动子区域能够被SlNAC29直接绑定。因此,推测SlNAC29与NAC家族其他转录因子的作用机制有所不同,是一条NAC转录因子调控植株衰老的新路径。作为衰老相关基因SAGs的成员,在植株衰老进程中能够显著上调表达,然而,关于其调控植株衰老的具体机理尚待进一步深入研究。
4 结论
在番茄作物中发现了一个重要的NAC家族转录因子SlNAC29,其在衰老叶片中显著上调表达。黑暗诱导衰老条件下,突变体能够延缓叶片衰老,而过表达OE:叶片衰老程度加剧,表明SlNAC29能够调控番茄植株的衰老,促进番茄叶片在黑暗诱导条件下的衰老进程。进一步发现SlNAC29直接绑定衰老相关基因启动子区域调控其转录表达。
[1] GAN S, AMASINO R M. Making sense of senescence: Molecular genetic regulation and manipulation of leaf senescence. Plant Physiology, 1997, 113(2): 313-319.
[2] 张金树.日光温室冬春茬番茄的早衰及预防. 中国蔬菜, 2001, 1(4): 42-43.
ZHANG J S. Premature senescence and its prevention of tomato in greenhouse at winter and spring. China Vegetables, 2001, 1(4): 42-43. (in Chinese)
[3] 张慧珍, 白雪芹, 曾幼玲. 植物NAC转录因子的生物学功能. 植物生理学报, 2019, 55(7): 915-924.
ZHANG H Z, BAI X Q, ZENG Y L. Biological functions of plant NAC transcription factors. Plant Physiology Journal, 2019, 55(7): 915-924. (in Chinese)
[4] BREEZE E, HARRISON E, MCHATTIE S, HUGHES L, HICKMAN R, HILL C, KIDDLE S, KIM Y S, PENFOLD C A, JENKINS D. High-resolution temporal profiling of transcripts duringleaf senescence reveals a distinct chronology of processes and regulation. The Plant Cell, 2011, 23: 873-894.
[5] KIM Y S, SAKURABA Y, Han S H, YOO S C, PAEK N C. Mutation of theNAC016 transcription factor delays leaf senescence. Plant Cell Physiology, 2013, 54: 1660-1672.
[6] BALAZADEH S, KWASNIEWSKI M, CALDANA C, MEHRNIA M, ZANOR M L, XUE G P, BERND M R. ORS1, an H2O2-responsive NAC transcription factor, controls senescence in. Molecular Plant, 2011, 4: 346-360.
[7] HIRONORI T, KYONOSHIN M, FUMINORI T, MIKI F, TAKUYA Y, KAZUO N, FUMIYOSHI M, KIMINORI T, KAZUKO Y S, KAZUO S. SNAC-As, stress-responsive NAC transcription factors, mediate ABA-inducible leaf senescence. The Plant Journal, 2015, 84: 1114-1123.
[8] BALAZADEH S, SIDDIQUI H, ALLU A D, MATALLANA- RAMIREZ L P, CALDANA C, MEHRNIA M, ZANOR M I, KOHLER B, MUELLER-ROEBER B. A gene regulatory network controlled by the NAC transcription factor ANAC092/AtNAC2/ORE1 during salt-promoted senescence. The Plant Journal, 2010, 62: 250-264.
[9] MAO C J, Lu S C, Lü B, ZHANG B, SHEN J B, HE J M, LUO L Q, XI D D, CHEN X, MING F. A rice NAC transcription factor promotes leaf senescence via ABA biosynthesis. Plant Physiology, 2017, 174(3): 1747-1763.
[10] FAN K, BIBI N, GAN S S, LI F, YUAN S N, NI M, WANG M, SHEN H, WANG X D. A novel NAP member GhNAP is involved in leaf senescence in. Journal of Experimental Botany, 2015, 66: 4669-4682.
[11] LIRA B S, GRAMEGNA G, TRENCH B A, ALVES F R R, SILVA E M, SILVA G F F, THIRUMALAIKUMAR V P, LUPI A C D, DEMARCO D, PURGATTO E, NOGUEIRA F T S, BALAZADEH S, FRESCHI L, ROSSI M. Manipulation of a senescence-associated gene improves fleshy fruit yield. Plant Physiology, 2017, 175(1): 452.
[12] MA X M, ZHANG Y J, Turečková V, XUE G P, FERNIE A R, BERND M R, BALAZADEH S. The NAC transcription factor SlNAP2 regulates leaf senescence and fruit yield in tomato. Plant Physiology, 2018, 177(3): 1286-1302.
[13] MULLER F, XU J M, KRISTENSEN L, WOLTERS-ARTS M, GROOT P, JANSMA S Y, MARIANI C, PARK S H, RIEU I. High-temperature-induced defects in tomato () anther and pollen development are associated with reduced expression of B-class floral patterning genes. Plos One, 2016, 11(12): e0167614.
[14] BUCHANAN-WOLLASTON V, PAGE T, HARRISON E, BREEZE E, LIM P O, NAM H G, LIN J F, WU S H, SWIDZINSKI J, ISHIZAKI K, LEAVER C J. Comparative transcriptome analysis reveals significant differences in gene expression and signalling pathways between developmental and dark/starvation-induced senescence in. The Plant Journal, 2005, 42: 567-585.
[15] KEECH O, PESQUET E, AHAD A, ASKNE A, NORDVALL D, VODNALA S M, TUOMINEN H, HURRY V, DIZENGREMEL P, GARDESTROM P. The different fates of mitochondria and chloroplasts during dark-induced senescence inleaves. Plant Cell & Environment, 2007, 30: 1523-1534.
[16] PAN C T, YE L, QIN L, LIU X, HE Y J, WANG J, CHEN L F, LU G. CRISPR/Cas9-mediated efficient and heritable targeted mutagenesis in tomato plants in the first and later generations. Scientific Reports, 2016, 7: 46916.
[17] LEI Y, LU L, LIU H Y, LI S, XING F, CHEN L L. CRISPR-P: a web tool for synthetic single-guide RNA design of CRISPR-system in plants. Molecular Plant, 2014, 7(9): 1494-1496.
[18] FILLATTI J J, KISER J, ROSE R, COMAI L. Efficient transfer of a glyphosate tolerance gene into tomato using a binarytumefacien vector. Nature Biotechnology, 1987, 5: 726-730.
[19] KENNETH J L, THOMAS D S. Analysis of relative gene expression data using real-time quantitative PCR and the 2-ΔΔCTmethod. Methods, 2002, 25: 402-408.
[20] HELLMAN L M, FRIED M G. Electrophoretic mobility shift assay (EMSA) for detecting protein–nucleic acid interactions. Nature Protocols, 2007, 2(8): 1849-1861.
[21] TRAN L.S, NAKASHIMA K, SAKUMA Y, SIMPSON S D, FUJITA Y, MATUYAMA K, FUJITA M, SEKI M, SHINOZAKI K, KAZUKO Y S. Isolation and functional analysis ofstress-inducible NAC transcription factors that bind to a drought-responsive cis-element in the early responsive to dehydration stress 1 promoter. The Plant Cell, 2004, 16: 2481-2498.
[22] GREGERSEN P L, CULETIC A, BOSCHIAN L, KRUPINSKA K. Plant senescence and crop productivity. Plant Molecular Biology, 2013, 82: 603-622.
[23] GUIBOILEAU A, SORMANI R, MEYER C, MASCLAUX- DAUBRESSE C. Senescence and death of plant organs: nutrient recycling and developmental regulation. Comptes Rendus Biologies, 2010, 333: 382-391.
[24] LIM P O, KIM H J, NAM H G. Leaf senescence. Annual Review of Plant Biology, 2007, 58: 115-136.
[25] BALAZADEH S, RIANO-PACHON D M, MUELLER-ROEBER B. Transcription factors regulating leaf senescence in. Plant Biology, 2008, 10 (s1): 63-75.
[26] 杨晓娜, 田云, 卢向阳. NAC转录因子在植物生长发育中的调控作用. 化学与生物工程, 2014, 31(1): 1.
YANG X N, TIAN Y, LU X Y. The regulation role of NAC transcription factors in plant growth and development. Chemistry and Bioengineering, 2014, 31(1):1. (in Chinese)
[27] KIM H J, NAM H G, LIM P O. Regulatory network of NAC transcription factors in leaf senescence. Current Opinion in Plant Biology, 2016, 33: 48-56.
[28] WANG J, ZHENG C F, SHAO X Q, HU Z J, LI J X, WANG P, WANG A R, YU J Q, SHI K. Transcriptomic and genetic approaches reveal an essential role of the NAC transcription factor SlNAP1 in the growth and defense response of tomato. Horticulture Research, 2020, 209: 1-11.
[29] 刘强, 张贵友, 陈受宜. 植物转录因子的结构与调控作用.科学通报, 2000(14): 1465-1474.
LIU Q, ZHANG G Y, CHEN S Y. Structure and regulatory function of plant transcription factors. Chinese Science Bulletin, 2000(14): 1465-1474. (in Chinese)
[30] GUO Y, CAI Z Y, GAN S S. Transcriptome ofleaf senescence. Plant Cell & Environment, 2004, 27: 521-549.
[31] BREEZE E, HARRISON E, MCHATTIE S, HUGHES L, HICKMAN R, HILL C, KIDDLE S, KIM Y S, PENFOLD C A, JENKINS D. High-resolution temporal profiling of transcripts duringleaf senescence reveals a distinct chronology of processes and regulation. The Plant Cell, 2011, 23: 873-894.
[32] HE Y H, TANG W N, SWAIN J D, GREEN A L, JACK T P, GAN S S. Networking senescence-regulating pathways by usingenhancer trap lines. Plant Physiology, 2001, 126: 707-716.
[33] ZHANG K W, XIA X Y, ZHANG Y Y, GAN S S. An ABA-regulated and Golgi-localized protein phosphatase controls water loss during leaf senescence in. The Plant Journal, 2012, 69(4): 667-678.
The Role and Mechanism of Tomato SlNAC29 Transcription Factor in Regulating Plant Senescence
WANG Ping, ZHENG ChenFei, WANG Jiao, HU ZhangJian, SHAO ShuJun, SHI Kai
College of Agriculture and Biotechnology, Zhejiang University, Hangzhou 310058
【Background】Tomato()is an important horticultural crop with continuous flower bud differentiation and fruiting. Premature senescence seriously limits tomato plants growth period, crop yield and fruit quality. NAC (NAM, ATAF1/2 and CUC2) transcription factor family regulates leaf senescence process in Arabidopsis, rice and other plants. Nevertheless, the roles of tomato NAC transcription factor in the regulation of leaf senescence have not been well understood. SlNAP2 (NAC-like, activated by apetala3/pistillata) is known to be involved in the regulation of tomato leaf senescence. 【Objective】SlNAC29 transcription factor is the homologous gene of SlNAP2 in tomato, while its function remains largely unclear. In this study, the role of SlNAC29 and its underlying mechanism in leaf senescence was investigated, which can provide some scientific basis for tomato senescence regulation and germplasm innovation.【Method】Condine Red (CR) was used as the wild-type background in this study. qRT-PCR was used to analyze the relative expression ofhomozygous mutant lines and the OE:stable overexpression lines were generated through CRISPR/Cas9 gene editing and over-expression approaches, respectively. Using these lines, plant growth phenotypes, chlorophyll content, leaf photosynthesis, transcription of senescence- and chlorophyll degradation- related genes were analyzed under both natural and dark-induced senescence conditions. The clustering heat map was used to analyze the relative expression of 29 genes, including senescence-, chlorophyll degradation- and ABA biosynthesis/signaling-associated genes. Based on gene expression profiles, four of them were selected to electrophoretic mobility shift analysis (EMSA) to identify the SlNAC29-target gene during senescence process.【Result】The relative expression ofwas significantly up-regulated in early senescent and senescent leaves, as compared with young and mature leaves. Under natural growth condition, themutant lines showed no differences with the wild-type in terms of plant growth phenotypes and photosynthetic rate. By contrast, the height of OE:plant was shorter than wild-type plants, OE:plants also showed lower chlorophyll content and photosynthetic rate, which were only 25% and 50% of the wild-type control, respectively. Under dark-induced senescence condition, the leaves of wild-type plants turned yellow and the chlorophyll content decreased significantly. The senescent phenotypes were alleviated inmutant lines, which not only have significant higher chlorophyll content, but also showed higher transcript level of senescence-associated genes (SAGs) and chlorophyll degradation-related genes. On the contrary, the dark-induced senescence effect was aggravated in OE:. Cluster analysis showed that several genes, especially SAGs and chlorophyll degradation-related genes,,and, were significantly up-regulated in OE:plants. The EMSA analysis showed that SlNAC29 could directly bind to the promoter of(), a member of. Moreover, the relative expression ofwas significantly higher than that of wild-type andplants. 【Conclusion】SlNAC29 transcription factor is involved in the regulation of leaf senescence in tomato plants, which promotes the senescence process under dark conditions. SlNAC29 may directly bind to the promoter region of the senescence-related geneto regulate its transcriptional expression.
tomato; SlNAC29; transcription regulation; senescence;

2021-02-06;
2021-05-03
国家自然科学基金优青项目(31822046)、浙江省重点研究发展计划(2021C02040)、国家重点基础研究发展计划(2019YFD1000300)
王萍,E-mail:11916061@zju.edu.cn。通信作者师恺,E-mail:kaishi@zju.edu.cn
(责任编辑 李莉)
