谷子株高及穗部性状主基因+多基因混合遗传模型分析
2022-01-17郭淑青宋慧杨清华高金锋高小丽冯佰利杨璞
郭淑青,宋慧,杨清华,高金锋,高小丽,冯佰利,杨璞
谷子株高及穗部性状主基因+多基因混合遗传模型分析

1西北农林科技大学农学院/旱区作物逆境生物学国家重点实验室,陕西杨凌 712100;2安阳市农业科学院谷子研究所,河南安阳 455000
【目的】株高和穗部性状是影响谷子产量的关键性状。探究谷子株高及穗部性状表型变异的遗传规律,为相关性状的遗传改良与基因定位提供参考依据。【方法】以谷子优质品种豫谷18为共同父本,分别与黄软谷和红酒谷杂交,构建2个分别包含250个家系的重组自交系F7群体(YYRIL和YRRIL)。采用主基因+多基因混合遗传模型,对YYRIL和YRRIL群体在2个环境下的株高、穗长、穗下节间长、穗码数、穗粒重等5个农艺性状的表型数据进行遗传分析。【结果】5个性状在所有环境中均表现连续变异且存在超亲分离现象,峰度和偏度绝对值小于1,近似正态分布,呈现数量性状的典型遗传特点。性状间相关性分析表明株高与穗长、穗下节间长在所有环境中均呈极显著正相关,穗码数与穗粒重呈极显著正相关。遗传模型分析显示YYRIL和YRRIL群体株高的最适遗传模型分别为PG-AI和PG-A多基因模型,多基因遗传率分别为95.15%和91.27%。2个群体穗码数的最适模型均为PG-AI,多基因遗传率为70.07%—71.58%。穗下节间长在2个群体的最适遗传模型分别为4MG-CEA和3MG-CEA,均为等加性主基因模型。穗下节间长在YYRIL群体的主基因遗传率为9.69%,4对主基因加性效应值相等,均为-0.34,具有负向效应;穗下节间长在YRRIL群体的主基因遗传率为45.78%,3对主基因加性效应值相等,均为1.17,具有正向效应。穗长在YYRIL群体的最适模型为MX2-ED-A,即2对显性上位主基因+加性多基因模型,主基因遗传率为43.56%,多基因遗传率为50.56%。控制穗长的2对主基因加性效应值分别为-1.21、1.68,多基因加性效应较小,为-0.0017;穗长在YRRIL群体的最适模型为MX2-AE-A,即2对累加作用主基因,加性多基因混合遗传模型;穗长的主基因遗传率为46.40%,多基因遗传率为46.91%。控制穗长的第1对主基因加性效应值为1.53,具有正向效应,第1对主基因加性×第2对主基因加性上位性互作效应值是0.60,多基因加性效应值为-0.47,表现为较低的负向遗传效应。穗粒重在YYRIL群体的最适遗传模型为MX2-ED-A;符合2对显性上位主基因+加性多基因模型,主基因遗传率为69.09%,多基因遗传率为12.08%;控制穗粒重的2对主基因加性效应值分别为0.58、5.82,以第2对主基因的加性效应为主,多基因加性效应值为-3.81。穗粒重在YRRIL群体的最适遗传模型为3MG-PEA,即3对部分等加性主基因遗传模型;穗粒重的主基因遗传率为81.10%,3对主基因加性效应值分别为-2.68、-2.68和2.66,前2对主基因的加性效应值相同,且均为负向效应。【结论】谷子株高、穗码数的最适遗传模型相似,均服从多基因遗传,遗传率较高,受环境影响较小;穗下节间长的遗传受主基因控制,主基因遗传率偏低,受环境影响较大,在栽培中应充分考虑环境因素;穗长遗传受主基因和多基因共同控制;穗粒重在2个群体均服从主基因遗传,主基因遗传率较高,可能存在主效QTL。
谷子;重组自交系;株高;穗部性状;主基因+多基因
0 引言
【研究意义】谷子(L.)抗旱耐贫瘠,是中国北方干旱半干旱地区重要的粮食作物。此外,谷子富含丰富的蛋白质、维生素、脂肪等营养物质,其食品保健价值受到广泛关注[1]。谷子株高及穗部农艺性状间相互联系,直接影响其产量[2]。然而谷子农艺性状多属数量性状,遗传基础复杂,易受环境条件影响,基因型与表型的对应关系不明确[3-4]。研究谷子株高及穗部性状的遗传规律,对提高和稳定谷子产量、维护区域粮食安全具有重要意义。【前人研究进展】主基因加多基因混合遗传模型是揭示株高及穗部性状遗传机制的重要途径[5]。泛主基因-多基因遗传理论认为,数量性状的遗传体系是由效应较大的主基因、效应较小的多基因或主基因和多基因共同组成,即主基因加多基因混和遗传体系[6-8]。基于该理论,章元明等[9-10]和盖钧镒等[8]提出了主基因加多基因混和遗传模型分析方法。该方法不仅可经济便捷地对表型性状主基因数目、基因效应做出初步判断,更能明晰基因间互作及其上位性以及基因与环境的相互作用,是分析植物数量性状遗传的重要途径[11-13]。大量的研究实践表明,基于主基因加多基因遗传模型对表型数据的分析与QTL定位的结果具有相对一致性,可相互验证[14-16]。主基因加多基因混和遗传分析方法在小麦[17]、水稻[18]、玉米[19]、油菜[20]、棉花[21]、大麦[22]、蚕豆[23]、番茄[24]、烟草[25]、白花丹[26]等植物的农艺性状、抗旱性、抗病性、化学成分等方面的遗传分析中得到了广泛应用。性状受基因和环境共同影响。相同性状在不同环境和不同群体中具有显著差异的遗传模型[27]。【本研究切入点】目前,已开始广泛研究谷子数量性状,但利用主基因加多基因混合遗传模型研究较少,对谷子重要农艺性状在多环境下的表型遗传分析鲜见报道。【拟解决的关键问题】本研究以豫谷18为共同亲本,分别与红酒谷、黄软谷杂交,并以单籽传法构建的2个重组自交系群体(YYRIL和YRRIL)为材料,在陕西榆林和河南安阳2个环境中开展表型鉴定,利用主基因+多基因混合遗传模型分析方法,鉴定谷子株高及穗部性状遗传模型和基因作用方式,以期为谷子重要农艺性状的QTL定位及性状改良提供依据。
1 材料与方法
1.1 试验材料
以高产、优质,适应性广的谷子品种豫谷18为共同父本,分别与地方品种红酒谷和黄软谷杂交,通过连续6代单籽传法自交,构建2个包含250个家系的重组自交系群体(RILs-F7),分别命名为YYRIL和YRRIL。试验材料均由西北农林科技大学小杂粮课题组提供。于2020年分别在陕西榆林(半干旱区,109°21′46″E,37°56′26″N,2020YL,E1)和河南安阳(半湿润区,114°23′32″E,36°58′32″N,2020AY,E2)种植这两个群体。每个环境的田间试验采取随机区组试验设计,3次重复,每个小区4行,行长2 m,行距30—40 cm,小区面积为4 m2(2 m×2 m)。试验点肥力均匀,田间管理参照常规农田统一管理。
1.2 性状调查
成熟期每个小区随机选取9株,利用钢尺测量亲本及群体株高(plant hight,PH)、穗长(panicle length,PL)、穗下节间长(internode length under panicle,PIL),并测量穗码数(spikelet number per panicle,SN)。成熟后单株收获脱粒,利用天平称量穗粒重(grain weight per panicle,GW)。性状调查方法及标准依据《谷子种质资源描述规范和数据标准》[28]。
1.3 数据分析
利用Excel 2010和SPSS 22.0软件对2个群体及3个亲本在2个环境下的表型数据进行初步分析处理,判断是否符合正态分布。利用R语言包绘制谷子重组自交系株高及穗部性状的频率分布直方图;利用章元明教授团队开发的R软件包SEA-G3DH(https://cran.r-project.org/web/packages/SEA/index.html)对亲本及群体的5个表型性状进行主基因+多基因混合遗传模型分析。通过极大似然法(maximum likelihood method,MLV)和迭代最大期望算法(iterated expectation and conditional maximization,IECM)对混合分布中的相关成分分布参数做出估计,采用最小赤池信息量准则(Akaike’s information criterion,AIC),选出AIC值最小的几个模型作为备选遗传模型,然后利用均匀性检验(12、22和32)、Smirnov检验(2)和Kolmogorov检验(D)对备选模型进行适合性检验,根据检验结果选择统计量达到显著性水平个数最少的模型为该性状的最适遗传模型,最后,用最小二乘法计算出最适遗传模型的一、二阶遗传参数。
2 结果
2.1 谷子株高及穗部性状的表型变异及频率分布
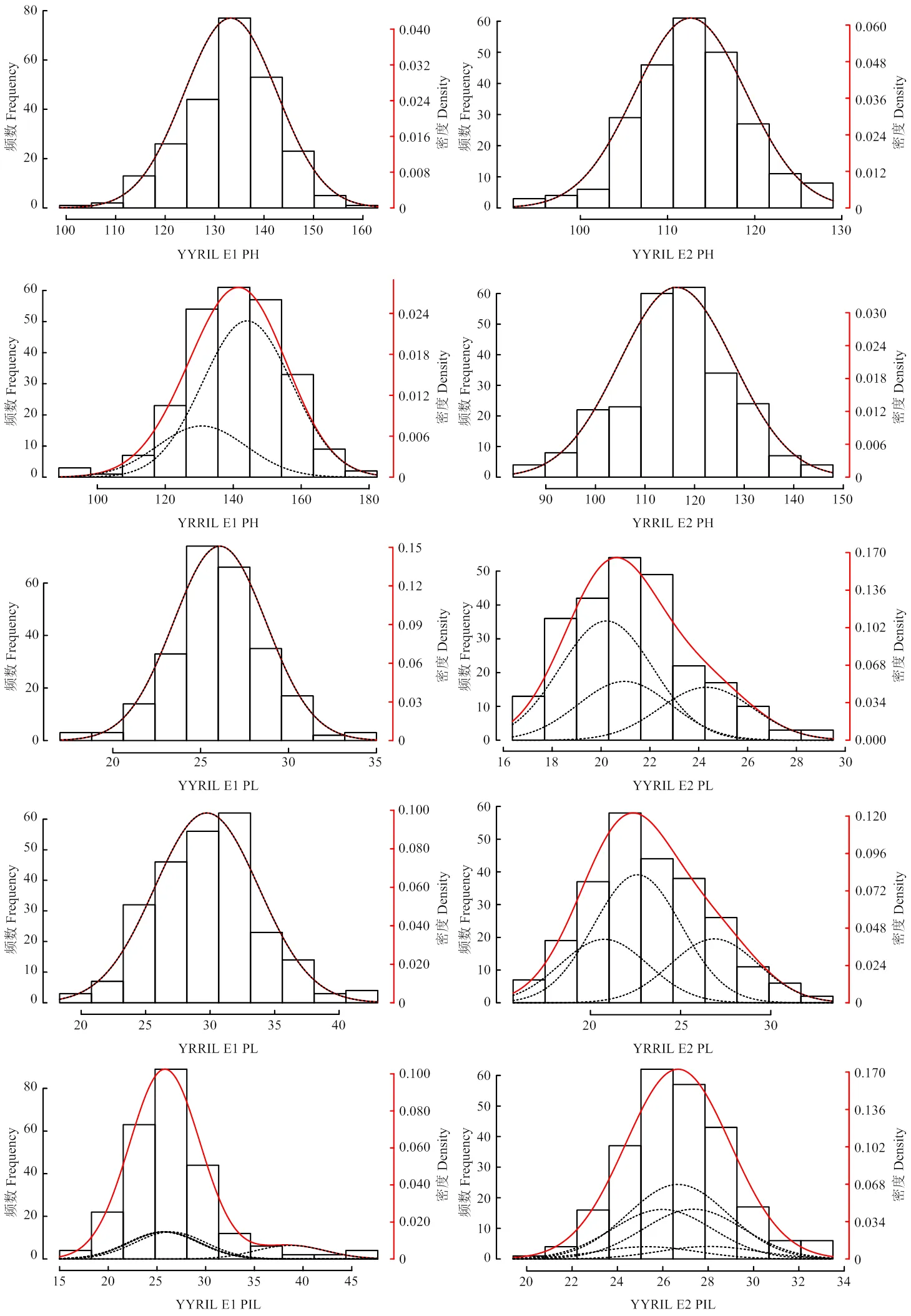
2.1.1 YYRIL群体株高及穗部性状的表型变异及频率分布 对YYRIL群体及亲本的株高及穗部性状进行分析可知(表1),亲本的性状差异在不同环境下表现不一致。双亲株高、穗码数在2个环境下均呈极显著差异,穗下节间长在榆林存在显著差异,而在安阳差异不显著。群体在2个不同环境下的表型性状存在不同程度的分离,变异系数从高到低依次为穗粒重(22.01%)、穗码数(14.07%)、穗下节间长(13.46%)、穗长(11.02%)、株高(6.37%)。在2个环境下,YYRIL群体的株高及穗部性状偏度为-0.72—1.29,峰度为-0.46—1.34。除E1环境下的穗长、穗下节间长外,各农艺性状的偏度和峰度绝对值均小于1,即表现为近似正态分布。农艺性状在2个环境的频率分布图存在一定程度差异(图1),YYRIL群体株高在2个环境均呈现单峰偏态分布,表明株高可能由多基因控制;穗下节间长在2个环境均表现多峰分布,可能存在主基因;穗长在榆林的频率分布为单峰分布,在安阳呈现双峰分布;穗码数在榆林表现为双峰分布,在安阳表现为单峰分布,穗粒重在榆林表现为单峰分布,在安阳表现为多峰分布。YYRIL群体株高及穗部性状在各个环境中呈连续分布且存在不同程度超亲分离现象,适宜进行遗传分析。利用Pearson相关系数对YYRIL群体在2个不同环境农艺性状进行相关性分析,发现株高与穗长、穗下节间长在2个环境中均呈极显著正相关,穗码数与穗粒重在E1呈极显著正相关(图2)。
2.1.2 YRRIL群体株高及穗部性状的表型变异及频率分布 YRRIL群体亲本间性状的差异在不同环境下表现不一致(表1),双亲株高、穗下节间长在2个环境的差异不显著,而穗码数、穗粒重在2个环境均呈现极显著差异。群体性状存在丰富的遗传变异,变异系数从高到低依次为穗粒重(30.79%)、穗码数(15.50%)、穗长(13.92%)、穗下节间长(13.54%)、株高(10.06%)。YRRIL群体株高、穗长、穗下节间长、穗码数、穗粒重在2个环境的偏度和峰度绝对值均小于1,表现为近似正态分布。群体性状表型频率分布图可以看出(图1),所有性状在不同环境均呈现连续性分布,分布特征存在差异,株高在榆林表现双峰分布,在安阳表现为单峰分布;穗长在榆林表现单峰分布,在安阳为多峰分布;穗下节间长在2个环境下均呈现多峰分布,表明存在主基因,穗码数在2个环境下均呈现单峰分布,穗粒重在榆林表现为单峰分布,在安阳表现多峰分布。YRRIL群体株高及穗部性状在各个环境中存在不同程度超亲分离现象,适宜进行遗传分析。YRRIL群体性状在各环境的相关性分析显示(图2),株高与穗长、穗下节间长在2个环境中均呈极显著正相关,穗码数与穗粒重在2个环境均表现极显著正相关。
2.2 株高及穗部性状主基因+多基因混合遗传分析
2.2.1 YYRIL群体株高及穗部性状主基因+多基因混合遗传分析 对YYRIL群体在2个环境下的株高及穗部性状分别进行主基因+多基因混合模型遗传分析,选取AIC值最小的一组模型为备选模型(表2),比较株高分别在2个环境下的AIC值,PG-AI模型的AIC值最低分别为1 874.259和1 643.336,为株高的备选模型;穗长在E1的最低AIC值为1 258.761,对应的备选模型是PG-AI,穗长在E2的最低AIC值为1 186.078,备选模型为MX2-ED-A;3MG-AI、4MG- CEA的AIC值较低,分别为1 520.849和1 164.932,为穗下节间长的备选模型;穗码数的备选模型分别为2MG-CE和PG-AI,AIC值分别为2 165.766和1 881.470,穗粒重在2个环境下最低的AIC值分别为1 686.369和1 485.489,对应的备选模型是PG-A和MX2-ED-A。
通过对YYRIL群体各个性状在2个环境下的备选模型采用均匀性检验、Smirnov检验和Kolmogorov检验,确定AIC值最小且统计量显著性水平个数最少的模型为最适模型(表3)。YYRIL群体株高的最适遗传模型均为PG-AI模型,即2对连锁主基因+加性-上位性多基因遗传模型。穗长在2个环境的备选模型的统计量达到显著水平的数量均为0;根据AIC准则进行筛选,穗长的最适模型为MX2-ED-A模型,即2对显性上位主基因+加性多基因模型。同理穗下节间长对应的最适遗传模型为4MG-CEA模型,即4对主基因模型,主基因加性效应相同,无多基因。穗码数对应的最适遗传模型为PG-AI模型。穗粒重对应的遗传模型为MX2-ED-A。

表1 谷子YYRIL和YRRIL群体株高及穗部性状描述性统计分析
PH:株高;PL:穗长;PIL:穗下节间长;SN:穗码数;GW:穗粒重。E1:陕西榆林;E2:河南安阳。*:差异达到显著水平(<0.05);**:差异达到极显著水平(<0.01)。表格中“-”表示空缺。下同
PH: Plant height; PL: Panicle length; PIL: Internode length under panicle; SN:Spikelet number per panicle; GW: Grain weight per panicle. E1: Yulin, Shaanxi; E2: Anyang, henan. *:Significant difference at the 0.05 level; **: Significant difference at the 0.01 level. “-”in the cells mean the value is absent. The same as below
PH:株高;PL:穗长;PIL:穗下节间长;SN:穗码数;GW:穗粒重。E1:陕西榆林;E2:河南安阳。下同
PH: Plant height; PL: Panicle length; PIL: Internode length under panicle; SN:Spikelet number per panicle ; GW: Grain weight per panicle. E1: Yulin, Shaanxi; E2: Anyang, henan. The same as below
图1 谷子YYRIL和YRRIL群体株高及穗部性状的频率分布(柱形)、拟混合分布(红线)与成分分布(黑线)
Fig. 1 Frequent (column), mixed (red line), and component (black line) distributions for plant height and panicle traits inYYRIL and YRRIL foxtail millet population
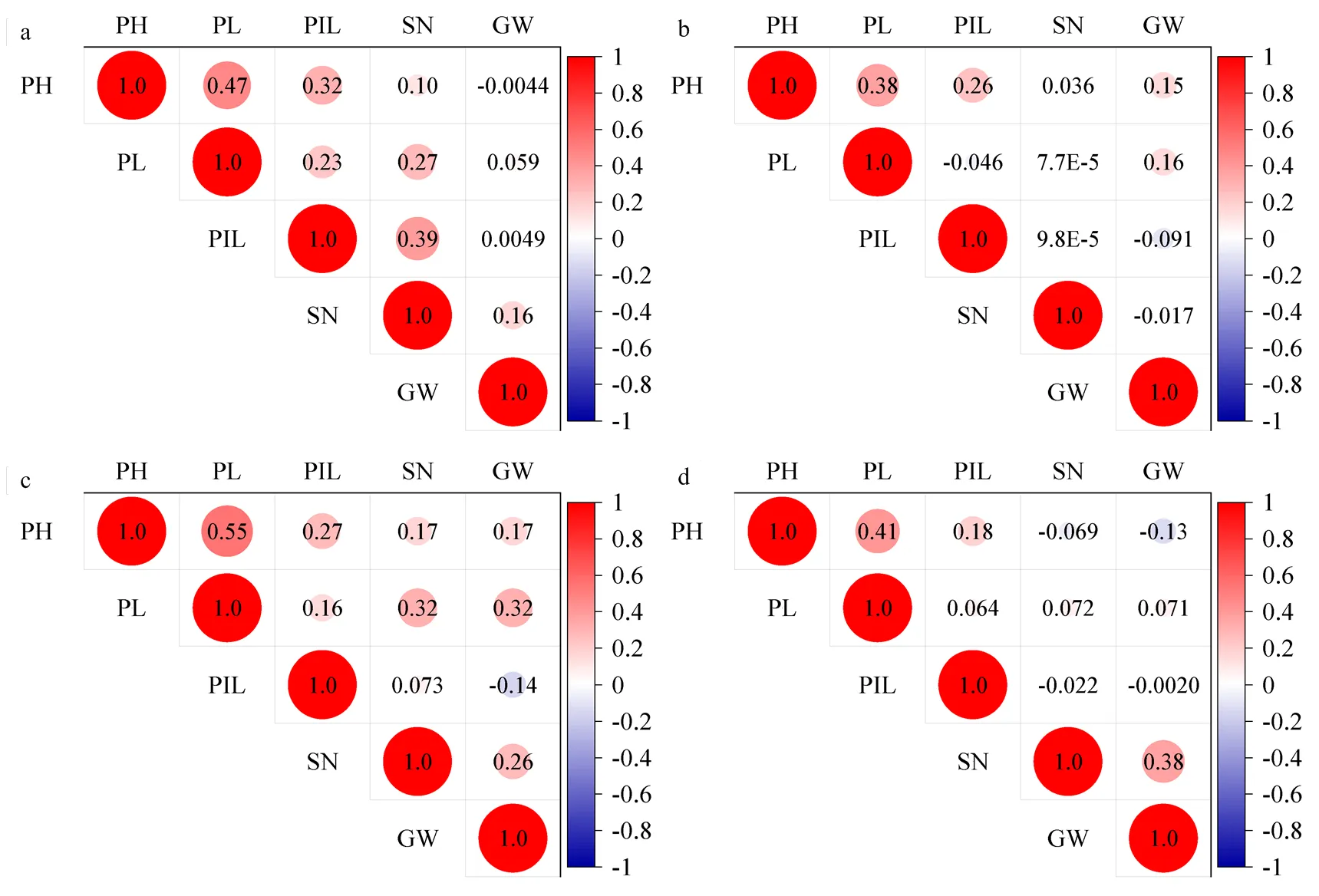
a:YYRIL群体株高及穗部性状在榆林的相关性;b:YYRIL群体株高及穗部性状在安阳的相关性;c:YRRIL群体株高及穗部性状在榆林的相关性;d:YRRIL群体株高及穗部性状在安阳的相关性
估算YYRIL群体株高及穗部性状最适遗传模型下的一阶参数和二阶参数可知(表4),2个环境中,株高的多基因遗传率介于74.68%—95.15%,株高以多基因遗传为主。穗长的主基因遗传率为43.56%,多基因遗传率为50.56%。控制穗长的第1对主基因加性效应值(da)为-1.21,具有负向效应,第2对主基因加性效应值(db)为1.68,具有正向效应,多基因显性效应较小为-0.0017。穗下节间长的主基因遗传率为9.69%,环境因素决定90.31%的变异,穗下节间长受环境因素影响较大。控制穗下节间长的4对主基因加性效应值(da、db、dc和dd)相同,均为-0.34,为负向遗传效应。穗码数的多基因遗传率是70.07%,穗码数受环境影响较小,一致性较好。穗粒重的主基因遗传率为69.09%,多基因遗传率为12.08%,穗粒重主要受主基因作用。穗粒重的2对主基因加性效应值(da和db)分别为0.58和5.82,其中第2对主基因的加性效应值较大,说明主基因加性效应以第2对主基因为主,且为正向遗传效应;穗粒重多基因加性效应值([d])为-3.81,为负向遗传效应。
2.2.2 YRRIL群体株高及穗部性状主基因+多基因混合遗传分析 根据AIC值最小原则,选取YRRIL群体株高及穗部性状在2个环境下的备选模型(表2)。株高在2个环境下PG-A和MX2-IE-A的AIC值最低,分别为1 966.339和2 126.700,为株高备选模型;PG-AI和MX2-AE-A的AIC值最低为1 463.860和1 324.700,为穗长的备选模型;穗下节间长在2个环境下的备选模型为3MG-CEA和4MG-EEEA,AIC值分别为1 285.900和1 546.820,穗码数的在2个环境下的备选模型均为PG-AI,穗粒重在2个环境下最低的AIC值分别为1 808.660和1 481.494,对应的备选模型是PG-AI和3MG-PEA。
通过对YRRIL群体各个性状在2个环境下的备选模型采用适应性检验,选出AIC值最小和统计显著性水平数目最少的模型作为最适模型(表3)。株高的2个备选模型的统计量达到显著水平的个数均为0,根据AIC准则进行筛选,株高对应的最适遗传模型为PG-A模型,即多基因模型,多基因以加性效应为主。穗长的最适模型为MX2-AE-A,即2对连锁主基因,累加作用多基因混合遗传模型。穗下节间长对应的最适遗传模型为3MG-CEA模型,即3对主基因模型,主基因的加性效应相同;穗码数在2个环境的最适遗传模型均为PG-AI模型,即2对连锁主基因+加性-上位多基因遗传模型;穗粒重对应的遗传模型为3MG-PEA,即3对主基因遗传模型。

表2 谷子YYRIL和YRRIL群体株高及穗部性状最适遗传模型分离分析的极大似然值函数MLV值和Akaike信息准则AIC值
MG:主基因模型;PG:多基因遗传模型;MX:主基因+多基因混合模型;A:加性效应;I:互作;E:相等;AI:加性上位性效应;AE:累加作用;ED:显性上位;CE:互补作用;IE:抑制作用;CEA:全等加性;PEA:部分等加性
MG: Major gene model; PG: Polygene model; MX: Mixed major gene and polygene model. A: Additive effect; I: Interaction; E: Equal; AI: Additive + epistasis effect; AE: Accumulative effect; ED: epistasis dominance; CE: complementary effect; IE: inhibition effect; CEA: congruent equal additive; PEA: Partial equal additive

表3 谷子YYRIL和YRRIL群体株高及穗部性状最佳遗传模型适应性检验
12、22、32:均匀性检验;nW:Smirnov检验;:Kolmogorov检验,括号内数值为概率值
12,22,32: the statistic of Uniformity test;nW: the statistic of Smirnov test;: the statistic of Kolmogorov test, the numbers in brackets are the distribution values in theory
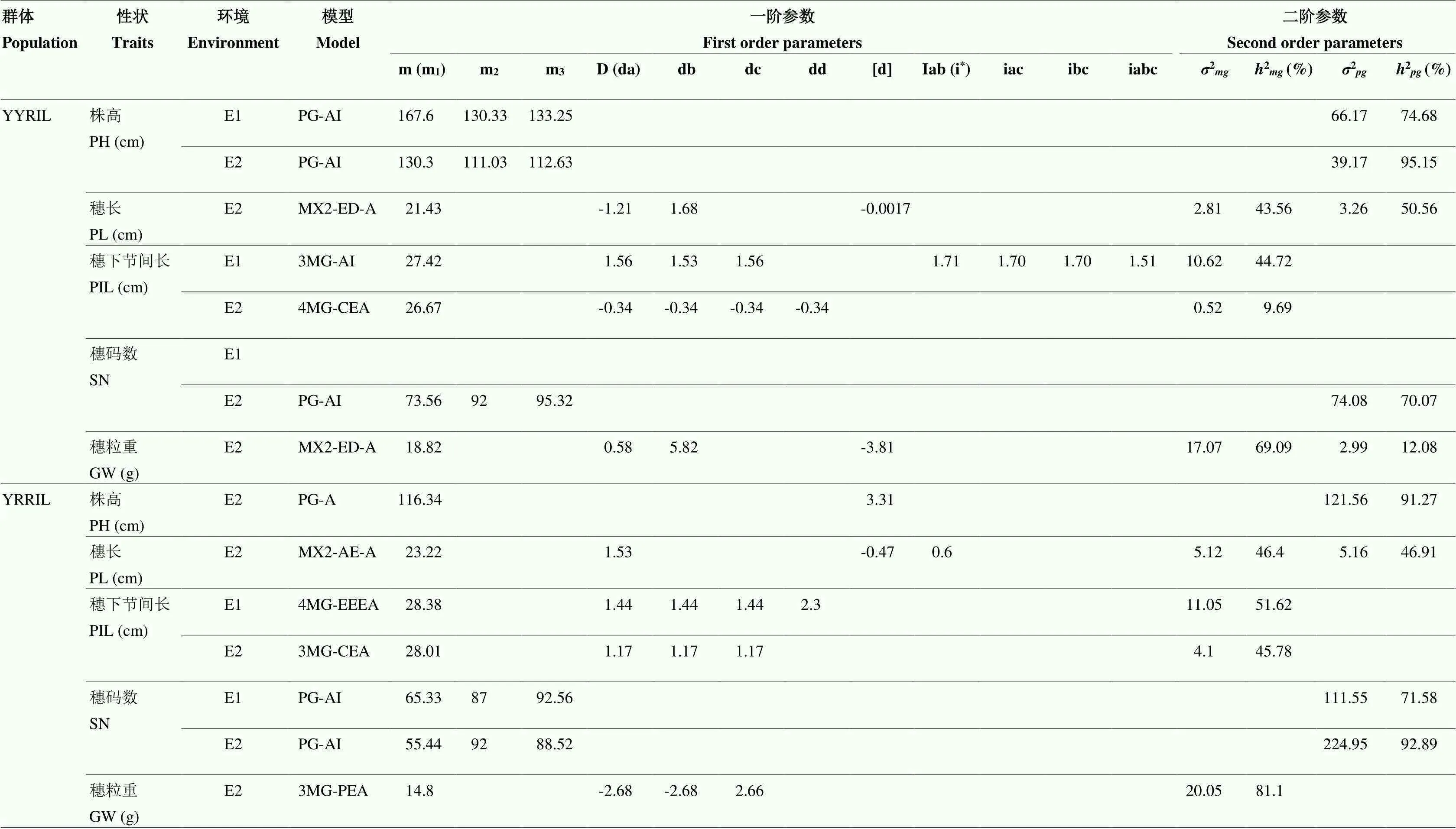
表4 谷子YYRIL和YRRIL群体株高及穗部性状最适模型遗传参数
m:群体均值;da:第1对主基因的加性效应;db:第2对主基因的加性效应;dc:第3对主基因的加性效应;dd:第4对主基因的加性效应;i:上位性效应;[d]:多基因加性效应;2:多基因方差;2:主基因方差;2(%):主基因遗传力;2(%):多基因遗传力
m: population mean; da: additive effect of the first major gene; db: additive effect of the second major gene; dc: additive effect of the third major gene; dd: additive effect of the fourth major gene; i: epistatic effect value; [d]: additive effect of polygene;2: polygene variance;2: major gene variance;2(%): heritability of major gene;2(%): heritability of polygene
由YRRIL群体株高及穗部性状的最适遗传模型估算一阶参数和二阶参数可知(表4),株高的多基因遗传率为91.27%,环境因素决定8.73%的变异,谷子YRRIL群体株高主要由多基因控制,受环境影响小。穗长的主基因遗传率为46.40%,多基因遗传率为46.91%。控制穗长的第1对主基因加性效应值(da)为1.53,具有正向效应,加性和第1对主基因×第2对主基因的加性上位性互作效应值(iab)是0.60,多基因加性效应值([d])为-0.47,表现为较低的负向遗传效应。穗下节间长的主基因遗传率为45.78%,环境因素决定54.22%的变异,穗下节间长受环境因素影响较大。控制穗下节间长的3对主基因加性效应值(da、db、dc和dd)相同,均为1.17,为正向遗传效应。穗码数的多基因遗传率介于71.58%—92.89%,穗码数受环境影响较小,一致性较好。穗粒重的主基因遗传率为81.10%,环境因素仅占18.90%,穗粒重主要受主基因作用。穗粒重的3对主基因加性效应值(da、db和dc)分别为-2.68、-2.68和2.66,其中前对2对主基因的加性效应值相同,均为负向效应,第3对主基因加性效应为正向遗传效应。
3 讨论
3.1 株高及穗部性状的最适遗传模型
谷子抗旱耐贫瘠,适应性广,是优化种植业区域布局、保证北方旱作区粮食作物生产长效发展的重要作物[29-30]。株高及穗部性状是影响谷子产量的重要因素,研究其遗传规律对提高和稳定谷子产量具有重要的意义。主基因+多基因模型是目前广泛用于分析作物数量性状遗传组成的方法,其不仅可通过表型数据对目标性状的遗传基础进行初步判断,更可校验QTL定位的结果,提高结果的准确性和可靠性,为数量性状QTL定位提供依据[31-32]。本研究以高产、优质、适应性广,综合抗性好的谷子品种豫谷18分别与黄软谷、红酒谷杂交构建的2个自交系群体(YYRIL-F7和YRRIL-F7)为材料,采用主基因+多基因混和遗传模型,研究谷子株高、穗长、穗下节间长、穗码数、穗粒重等5个重要农艺性状的遗传规律。研究发现5个性状在亲本间的差异在不同环境下表现不一致。5个性状在群体内变异丰富,且在两个群体的变异程度相似。穗粒重的变异系数最大,株高的变异系数最小,这表明穗粒重的离散程度大,改良潜力大,与方路斌等[33]、李晓宇等[34]研究结果一致。两群体株高、穗长、穗下节间长、穗码数、穗粒重均表现连续分布且存在超亲分离,峰度和偏度绝对值小于1,近似正态分布,符合多基因控制的数量性状的典型特征。农艺性状的相关性在2个群体内表现一致,株高与穗长、穗下节间长呈现极显著正相关,穗码数与穗粒重呈现极显著正相关。因此,育种过程应充分考虑性状间的相关性,提高对目标性状的选择效率[35]。
通过对2个群体在不同环境下的农艺性状的遗传模型进行筛选和适合性检验发现:YYRIL群体株高在2个环境下均符合PG-AI模型,即多基因遗传模型,多基因具有明显的加性上位性作用;株高的多基因遗传率介于74.68%—95.15%,环境因素决定4.85%—25.32%的变异。YRRIL群体株高的最适遗传模型为PG-A模型,即多基因模型,多基因以加性效应为主;株高的多基因遗传率为91.27%,环境因素决定8.73%的变异。尽管株高在两群体的最适模型不一致,但均符合多基因模型,且多基因遗传率较高,表明株高可能受稳定遗传的多基因控制。He等[36]利用高密度遗传图谱鉴定出26个与株高显著相关的QTL,并证明谷子矮秆/半矮秆表型是由多个QTL控制的,而不是由单个基因或者主效QTL控制,这也与前人关于高粱、玉米株高的研究相似[37-38]。但VANDANA等[39]利用全基因组关联分析鉴定了3个与株高相关的位点,可能是由材料类型差异较大所致。YYRIL群体穗码数的最适遗传模型为PG-AI模型,YRRIL群体穗码数在2个环境下的备选模型均为PG-AI,即2对连锁主基因+加性-上位多基因遗传模型。穗码数在2个群体的最适模型一致,多基因遗传率分别为70.07%和71.58%。Zhang等[40]在不同光照条件下鉴定了5个与穗码数相关的QTL,分别位于第2、4、9染色体上,与本研究有差异,可能由于定位到的QTL效应值较小,能解释的遗传变异小。杜希朋等[41]利用主基因+多基因模型对蚂蚱麦×碧玉麦杂交的F2小穗数等性状进行遗传分析,发现株高、小穗数都是多基因控制的数量性状,与本研究结果一致,两者可能具有遗传相似性。YYRIL群体穗下节间长在E1的最适模型是3MG-AI,在E2的最适模型为4MG-CEA,以穗下节间长在2个环境下的最佳模型作为备选模型,经适合性检验发现,穗下节间长的2个备选模型的统计量达到显著水平的数量均为0;根据AIC准则进行筛选,最适模型为4MG-CEA模型,即4对主基因模型,无多基因;穗下节间长的主基因遗传率较低,环境因素决定90.31%的变异,YYRIL群体穗下节间长受环境因素影响较大。YRRIL群体穗下节间长在2个环境下的备选模型分别为3MG-CEA和4MG-EEEA,经检验穗下节间长对应的最适遗传模型为3MG-CEA模型,即3对完全独立等加性主基因模型;主基因遗传率为45.78%,环境因素决定54.22%的变异。本研究中,同一群体穗下节间长在不同的环境条件下的遗传模型存在差异,而不同群体在同一环境下的穗下节间长的最适遗传模型相似,且本研究穗下节间长的最适模型与前人研究结果差异较大[42-44],可能由于穗下节间长的主基因遗传率偏低,极易受环境影响,导致分析结果差异大。
YYRIL群体穗长的最适模型为MX2-ED-A模型,即2对显性上位主基因-加性多基因模型;穗长的主基因遗传率为43.56%,多基因遗传率为50.56%;控制穗长的第2对主基因加性效应值(db)大于第1对主基因加性效应值(da),具有正向效应,即以第2对主基因的加性效应为主,多基因加性效应较小。YRRIL群体穗长的最适模型为MX2-AE-A,即2对累加主基因+加性多基因模型;穗长的主基因遗传率为46.40%,多基因遗传率为46.91%;控制穗长的第1对主基因加性效应值(da)为1.53,具有正向效应,多基因加性效应为-0.47,表现为较低的负向遗传效应。2个群体穗长均符合2对主基因加多基因模型,但主基因间的效应不同,这与群体遗传背景差异有关。杨坤[45]对谷子穗长进行初步定位,共检测到3个与穗长相关的位点,2个表现为微效,Wang等[46]检测到3个与谷子穗长相关的QTL,Zhi等[44]在13个环境中检测到35个与穗长相关的QTL,其中2个QTL分别在12和7个环境中检测到,Fang等[47]鉴定了2个穗长相关的QTL。与本研究结果不完全一致,可能是遗传模型分析简化了主基因间的连锁作用,也有可能是性状在不同群体和不同环境中的表达存在差异。YYRIL群体穗粒重在2个环境下的备选模型分别是PG-A和MX2-ED-A;经适合性检验,穗粒重的最适遗传模型为MX2-ED-A,即2对显性上位主基因+加性多基因模型。估算最适模型的一阶参数和二阶参数发现,穗粒重的第2对主基因的加性效应值大于第1对主基因的加性效应值,说明主基因加性效应以第2对主基因为主,且为正向遗传效应,穗粒重多基因加性效应为-3.81,负向遗传效应。穗粒重主基因遗传率为69.09%,多基因遗传率为12.08%,主基因效应显著大于多基因效应,YYRIL穗粒重主要受主基因作用。YRRIL群体穗粒重在2个环境下的最适模型为3MG-PEA,即3对主基因遗传模型。穗粒重的3对主基因加性效应值(da、db和dc)分别为-2.68、-2.68和2.66,前对2对主基因的加性效应值相同,均为负向效应,第3对主基因加性效应为正向遗传效应。穗粒重的主基因遗传率为81.10%,表明穗粒重主要受主基因作用,遗传因素决定了81.10%的变异,环境因素仅占18.90%。2个群体穗粒重均以主基因遗传为主。Zhi等[44]在9个环境中检测到16个与谷子穗粒重相关的QTL,其中一个位点在2个环境均检测到,其余仅在单一环境检测到。Liu等[48]将12个与穗粒重相关的QTL定位于第7染色体上,其中1个QTL在3个环境下均被检测到。Fang等[47]鉴定了1个与穗粒重相关的QTL。原因可能是试验材料和群体类型各不相同对分析结果造成影响。
3.2 遗传模型对育种的指导意义
谷子株高及穗部性状的遗传分析对于谷子后续遗传研究具有重要意义。从遗传率的角度,谷子穗长受主基因和多基因控制,主基因遗传率与多基因遗传率相似,环境对表型变异的影响小。穗粒重主要受主基因遗传控制,具有较高的遗传率,遗传因素对其影响较大,在育种过程中应重视利用其主基因特性、在早代进行定向选择。穗下节间长遗传率较低易受环境影响,且与株高有很强的相关性,因此,在栽培中充分考虑环境因素、通过控制水肥等缩短穗下节间长、降低株高[49-51]。主基因与多基因混合模型分析发现两群体在2个不同环境下的株高、穗码数的最适模型相似,可能存在基因连锁、一因多效,其结果还需要通过QTL验证分析。
4 结论
明确了2个谷子RIL群体株高及穗部性状最佳模型及遗传效应。2个群体的株高、穗码数最适遗传模型相似,均服从多基因遗传,遗传率较高,受环境影响较小,一致性较好,控制这两个性状的基因可能在相近的染色体区域。穗下节间长由完全等加性的主基因控制遗传,主基因遗传率低,受环境影响较大。穗长受主基因和多基因共同控制。穗粒重均由主基因控制,遗传率高,可能存在主效QTL。
References
[1] Jia G q, Huang X h, Zhi H, Zhao Y, Zhao Q, Li W j, Chai Y, Yang L F, Liu K Y, Lu H Y, Zhu C R, Lu Y Q, Zhou C C, Fan D L, Weng Q J, Guo Y L, Huang T, Zhang L, Lu T T, Feng Q, Hao H F, Liu H K, Lu P, Zhang N, Li Y H, Guo E H, Wang S J, Wang S Y, Liu J R, Zhang W F, Chen G Q, Zhang B J, Li W, Wang Y F, Li H Q, Zhao B H, Li J Y, Diao X M, Han B. A haplotype map of genomic variations and genome-wide association studies of agronomic traits in foxtail millet (). Nature Genetics, 2013, 45(8): 957-961.
[2] 贾小平, 张博, 董志平, 全建章, 王永芳, 张小梅, 袁玺垒, 李剑峰, 戴凌峰. 海南短日照条件下谷子穗部性状的全基因组关联分析. 河南农业科学, 2018, 47(9): 33-40.
Jia X P, Zhang B, Dong Z P, Quan J Z, Wang Y F, Zhang X M, Yuan X L, Li J F, Dai L F. Genome-wide association analysis of panicle traits of foxtail millet under hainan short-day condition. Journal of Henan Agricultural Sciences, 2018, 47(9): 33-40. (in Chinese)
[3] 盖钧镒. 植物数量性状遗传体系的分离分析方法研究. 遗传, 2005(1): 130-136.
Gai J Y. Segregation analysis of genetic system of quantitative traits in plants. Hereditas, 2005(1): 130-136. (in Chinese)
[4] 王小勤. 谷子高密度遗传图谱构建及产量和农艺性状QTL分析[D]. 重庆: 西南大学, 2017.
Wang X Q. Construction of high-density genetic map and QTL analysis of yield and agronomic traits in Foxtail millet[D]. Chongqing: Southwest University, 2017. (in Chinese)
[5] 章元明, 盖钧镒, 王永军. 利用P1、P2和DH或RIL群体联合分离分析的拓展. 遗传, 2001(5): 467-470.
Zhang Y M, Gai J Y, Wang Y J. An expansion of joint segregation analysis of quantitative trait for using P1, P2and DH or RIL populations. Hereditas, 2001(5): 467-470. (in Chinese)
[6] 张书芬, 傅廷栋, 朱家成, 王建平, 文雁成, 马朝芝. 甘蓝型油菜芥酸含量的基因分析. 中国农业科学, 2008, 41(10): 3343-3349.
Zhang S F, Fu T D, Zhu J C, Wang J P, Wen Y C, Ma C Z. Genetic analysis of erucic acid inL. using mixed major gene and polygene inheritance model. Scientia Agricultura Sinica, 2008, 41(10): 3343-3349. (in Chinese)
[7] 王金社, 李海旺, 赵团结, 盖钧镒. 重组自交家系群体4对主基因加多基因混合遗传模型分离分析方法的建立. 作物学报, 2010, 36(2): 191-201.
Wang J S, Li H W, Zhao T J, Gai J Y. Establishment of segregation analysis of mixed inheritance model with four major genes plus polygenes in recombinant inbred lines population. Acta Agronomica Sinica, 2010, 36(2): 191-201. (in Chinese)
[8] 盖钧镒, 章元明, 王建康. QTL混合遗传模型扩展至2对主基因+多基因时的多世代联合分析. 作物学报, 2000, 26(4): 385-391.
Gai J Y, Zhang Y M, Wang J K. A joint analysis of multiple generations for QTL models extended to mixed two major genes plus polygene. Acta Agronomica Sinica, 2000, 26(4): 385-391. (in Chinese)
[9] 章元明, 盖钧镒, 张孟臣. 利用P1F1P2和F2或F2:3世代联合的数量性状分离分析. 西南农业大学学报, 2000, 22(1): 6-9.
Zhang Y M, Gai J Y. Zhang M C. Jointly segregating analysis of P1P2F1and F2or F2∶3families. Journal of Southwest Agricultural University, 2000,22(1): 6-9. (in Chinese)
[10] 章元明, 盖钧镒. 数量性状分离分析中分布参数估计的IECM算法. 作物学报, 2000(6): 699-706.
Zhang Y M, Gai J Y. The IECM algorithm for estimation of component distribution parameters in segregating analysis of quantitative traits. Acta Agronomica Sinica, 2000(6): 699-706. (in Chinese)
[11] 咸丰, 张勇, 马建祥, 张显, 杨建强. 野生甜瓜‘云甜-930’抗白粉病主基因+多基因遗传分析. 中国农业科学, 2011, 44(7): 1425-1433.
Xian F, Zhang Y, Ma J X, Zhang X, Yang J Q. Genetic analysis of resistant to powdery mildew with mixed model of major gene plus polygene in wild melon material ‘Yuntian-930’. Scientia Agricultura Sinica, 2011, 44(7): 1425-1433. (in Chinese)
[12] 冯云超, 余志江, 霍仕平, 晏庆九, 向振凡, 张芳魁, 羊炼, 张兴端. 玉米雄穗抽雄至散粉间隔时间主基因+多基因遗传模型及遗传效应. 玉米科学, 2019, 27(4): 1-8.
Feng Y C, Yu Z J, Huo S P, Yan Q J, Xiang Z F, Zhang F K, Yang L, Zhang X D. Genetic effects of tassel-anthesis interval using mixture model of major gene plus polygene in maize. Journal of Maize Sciences, 2019, 27(4): 1-8. (in Chinese)
[13] Wang J, Podlich D W, Cooper M, DeLacy I H. Power of the joint segregation analysis method for testing mixed major-gene and polygene inheritance models of quantitative traits. Theoretical and Applied Genetics, 2001, 103(5): 804-816.
[14] 王培, 李晓林, 杨林, 吴青霞, 杨子博, 白志元, 李立群, 李学军. 小麦单株穗数的遗传分析及基于QTL定位的最优基因型预测. 麦类作物学报, 2012, 32(5): 820-827.
Wang P, Li X L, Yang L, Wu Q X, Yang Z B, Bai Z Y, Li L Q, Li X j. Genetic analysis of spike number per plant in wheat and prediction of superior genotype based on QTL information. Journal of Triticeae Crops, 2012, 32(5): 820-827. (in Chinese)
[15] 刘莹, 盖钧镒, 吕慧能, 王永军, 陈受宜. 大豆耐旱种质鉴定和相关根系性状的遗传与QTL定位. 遗传学报, 2005(8): 855-863.
Liu Y, Gai J Y, Lü H N, Wang Y J, Chen S Y. Identification of drought tolerant germplasm and inheritance and QTL mapping of related root traits in soybean ((L.) Merr). Acta Gentica Sinica, 2005(8): 855-863. (in Chinese)
[16] 王春娥, 盖钧镒, 傅三雄, 喻德跃, 陈受宜. 大豆豆腐和豆乳得率的遗传分析与QTL定位. 中国农业科学, 2008, 41(5): 1274-1282.
Wang C E, Gai J Y, Fu S X, Yu D Y, Chen S Y. Inheritance and QTL mapping of tofu and soymilk output in soybean. Scientia Agricultura Sinica, 2008, 41(5): 1274-1282. (in Chinese)
[17] 马斯霜, 白海波, 惠建, 吕雪莲, 陈晓军, 高颖银, 李树华. 旱胁迫下2个小麦RIL群体苗期性状主基因与多基因的遗传分析. 江苏农业科学, 2020, 48(14): 87-93.
Ma S S, Bai H B, Hui J, Lü X L, Chen X J, Gao Y Y, Li S H. Genetic analysis of main genes and polygenes in seedling traits of two wheat RIL populations under drought stress. Jiangsu Agricultural Sciences, 2020, 48(14): 87-93. (in Chinese)
[18] 苏展, 程海涛, 郭玉华, 曹宏, 张伟伟, 付飞. 水稻DH群体盐胁迫下苗高的主基因-多基因混合模型遗传分析. 华北农学报, 2011, 26(3): 210-213.
Su Z, Cheng H T, Guo Y H, Cao H, Zhang W W, Fu F. Genetic analysis of seedling height of rice DH population under salt stress by using major genes plus polygenes mixed model. Acta Agriculturae Boreali-sinica, 2011, 26(3): 210-213. (in Chinese)
[19] 刘鹏飞, 周富亮, 梁思维, 蒋锋. 甜玉米茎秆强度性状的主基因+多基因遗传分析. 西北农林科技大学学报(自然科学版), 2020, 48(9): 64-72+88.
Liu P F, Zhou F L, Liang S W, Jiang F. Mixed major genes and polygenes inheritance analyses for stem strength traits of sweet corn. Journal of Northwest A&F University (Natural Science Edition), 2020, 48(9): 64-72+88. (in Chinese)
[20] 刘霞, 张冰冰, 马兵, 赵娜, 田正书, 秦梦凡, 王阳, 郎丽娜, 刘亚萍, 黄镇, 徐爱遐. 甘蓝型油菜株高及其相关性状的主基因+多基因遗传分析. 西北农业学报, 2018, 27(4): 528-536.
Liu X, Zhang B B, Ma B, Zhao N, Tian Z S, Qin M F, Wang Y, Lang L N, Liu Y P, Huang Z, Xu A X. Mixed major gene plus poly-gene genetic analysis of plant height and its related traits inL.. Acta Agriculturae Boreali-Occidentalis Sinica, 2018, 27(4): 528-536. (in Chinese)
[21] 龚举武, 刘爱英, 李俊文, 姜骁, 段丽, 葛群, 邓晓英, 巩万奎, 石玉真, 商海红, 陈全家, 耿洪伟, 袁有禄. 陆地棉衣分性状的主基因-多基因遗传分析. 棉花学报, 2019, 31(3): 192-200.
Gong J W, Liu A Y, Li J W, Jiang X, Duan L, Ge Q, Deng X Y, Gong W K, Shi Y Z, Shang H H, Chen Q J, Geng H W, Yuan Y L. Major gene plus polygene genetic analysis of lint percent in upland cotton. Cotton Science, 2019, 31(3): 192-200. (in Chinese)
[22] 吕亮杰, 郭元世, 杜丽杰, 吕超, 张新忠, 郭宝健, 许如根. 大麦籽粒淀粉含量的主基因+多基因遗传模型分析. 麦类作物学报, 2014, 34(1): 13-22.
Lü L J, Guo Y S, Du L J, Lü C, Zhang X Z, Guo B J, Xu R G. Major genes plus polygenes mixed inheritance model for starch contents in barley seed. Journal of Triticeae Crops, 2014, 34(1): 13-22. (in Chinese)
[23] 杜成章, 龙珏臣, 龚万灼, 朱振东, 宗绪晓, 张继君. 蚕豆赤斑病抗性的主基因+多基因遗传分析. 植物保护, 2019, 45(6): 131-137.
Du C Z, Long J C, Gong W Z, Zhu Z D, Zong X X, Zhang J J. Analysis of major genes plus polygenes mixed inheritance for resistance to chocolate spot on faba bean. Plant Protection, 2019, 45(6): 131-137. (in Chinese)
[24] Sun X r, Liu L, Zhi X n, Bai J r, Cui Y n, Shu J h, Li J m. Genetic analysis of tomato internode length via mixed major gene plus polygene inheritance model. Scientia Horticulturae, 2019, 246: 759-764.
[25] 张兴伟, 王志德, 刘艳华, 任民, 杨洋. 植物数量性状“主基因+多基因”混合遗传模型及其在烟草上的应用. 中国烟草学报, 2013, 19(3): 41-44.
Zhang X W, Wang Z D, Liu Y H, Ren M, Yang Y. Major gene plus polygene mixed genetic model in quantitative characters and its application in tobacco. Acta Tabacaria Sinica, 2013, 19(3): 41-44. (in Chinese)
[26] Shen P, Gao S p, Chen X, Lei T, Li W j, Huang Y x, Li Y r, Jiang M y, Hu D, Duan Y f, Li M, Li J n. Genetic analysis of main flower characteristics in the F1generation derived from intraspecific hybridization betweenandf.. Scientia Horticulturae, 2020, 274: 109652.
[27] 龚举武, 刘爱英, 段丽, 姜骁, 李俊文, 邓晓英, 葛群, 巩万奎, 石玉真, 商海红, 陈全家,耿洪伟, 袁有禄. 不同环境下‘中棉所70’RIL群体棉铃重的主基因+多基因遗传分析. 中国农学通报, 2019, 35(15): 128-137.
Gong J W, Liu A Y, Duan L, Jiang X, Li J W, Deng X Y, Ge Q, Gong W K, Shi Y Z, Shang H H, Chen Q J, Geng H W, Yuan Y L. Major gene + polygene genetic analysis of boll weight in RIL population of 'CCRI70' under various environments. Chinese Agricultural Science Bulletin, 2019, 35(15): 128-137. (in Chinese)
[28] 陆平. 谷子种质资源描述规范和数据标准. 北京: 中国农业出版社, 2006.
Lu P. Descriptors and data standard for foxtail millet germplasm resources. Beijing: China Agriculture Press, 2006. (in Chinese)
[29] Hisato M, Hiroki T, Yohei M, Ryohei T, Kenji F. Genetic analysis ofgene involved in panicle branching of foxtail millet,(L.)P. Beauv., and mapping by using QTL-seq. Molecular Breeding, 2016, 36(5): 59.
[30] Sarika G, Kajal K, Mehanathan M, Swarup Kumar P, Manoj P. Population structure and association mapping of yield contributing agronomic traits in foxtail millet. Plant Cell Reports, 2014, 33(6): 881-893.
[31] 曹锡文, 刘兵, 章元明. 植物数量性状分离分析Windows软件包SEA的研制. 南京农业大学学报, 2013, 36(6): 1-6.
Cao X W, Liu B, Zhang Y M. SEA:a software package of segregation analysis of quantitative traits in plants. Journal of Nanjing Agricultural University, 2013, 36(6): 1-6. (in Chinese)
[32] 崔月, 陆建农, 施玉珍, 殷学贵, 张启好. 蓖麻株高性状主基因+多基因遗传分析. 作物学报, 2019, 45(7): 1111-1118.
Cui Y, Lu J N, Shi Y Z, Yin X G, Zhang Q H. Genetic analysis of plant height related traits inL. with major gene plus polygenes mixed model. Acta Agronomica Sinica, 2019, 45(7): 1111-1118. (in Chinese)
[33] 方路斌, 罗河月, 陈洁, 李平. 谷子主要农艺性状的相关和主成分分析. 天津农业科学, 2018, 24(11): 62-65.
Fang L B, Luo H Y, Chen J, Li P. Correlation analysis and principal component analysis on major agronomic characters of millet. Tianjin Agricultural Sciences, 2018, 24(11): 62-65. (in Chinese)
[34] 李晓宇, 王昆鹏, 刘迎春, 张一波. 谷子主要农艺性状分析. 内蒙古农业大学学报(自然科学版), 2015, 36(2): 26-30.
Li X Y, Wang K P, Liu Y C, Zhang Y B. Analysis on the agronomic traits of millet. Journal of Inner Mongolia Agricultural University(Natural Science Edition), 2015, 36(2): 26-30. (in Chinese)
[35] 王海岗, 贾冠清, 智慧, 温琪汾, 董俊丽, 陈凌, 王君杰, 曹晓宁, 刘思辰, 王纶, 乔治军, 刁现民. 谷子核心种质表型遗传多样性分析及综合评价. 作物学报, 2016, 42(1): 19-30.
Wang H G, Jia G Q, Zhi H, Wen Q F, Dong J L, Chen L, Wang J J, Cao X Y, Liu S C, Wang L, Qiao Z J, Diao X M. Phenotypic diversity evaluations of foxtail millet core collections. Acta Agronomica Sinica, 2016, 42(1): 19-30. (in Chinese)
[36] He Q, Zhi H, Tang S, Xing L, Wang S y, Wang H g, Zhang A y, Li Y h, Gao M, Zhang H j, Chen G q, Dai S t, Li J x, Yang J j, Liu H f, Zhang W, Jia Y c, Li S j, Liu J r, Qiao Z j, Guo E h, Jia G q, Liu J, Diao X m. QTL mapping for foxtail millet plant height in multi-environment using an ultra-high density bin map. Theoretical and applied genetics, 2020,134(2): 1-16.
[37] 白晓倩, 卢华雨, 于澎湃, 裴忠有, 罗峰, 孙守钧. 粒用高粱×苏丹草杂交F2代农艺性状的数量遗传分析. 江苏农业科学, 2019, 47(19): 188-193.
BAI X Q, LU H Y, YU P P, PEI Z Y, LUO F, SUN S J. Quantitative analysis of agronomic traits of Sorghum × Sudangrass F2generation. Jiangsu Agricultural Sciences, 2019, 47(19): 188-193. (in Chinese)
[38] 王铁固, 马娟, 张怀胜, 陈士林. 玉米株高主基因+多基因遗传模型分析. 玉米科学, 2012, 20(4): 45-49.
Wang T G, Ma J, ZHANG H S, CHEN S L. Genetic analysis on plant height by mixed inheritance model of major genes plus polygenes in maize. Journal of Maize Sciences, 2012, 20(4): 45-49. (in Chinese)
[39] Vandana J, Sarika G, Vijay G, Mehanathan M, Tirthankar B, Nirala R, Manoj P. Genome-wide association study of major agronomic traits in foxtail millet (L.) using ddRAD sequencing. Scientific Reports, 2019, 9: 5020.
[40] ZHANG K, FAN G y, ZHANG X x, ZHAO F, Wei W, Du G h, FENG X l,WANG X m, WANG F, SONG G l, ZOU H f, ZHANG X l, Li S d, NI X m, ZHANG G y, ZHAO Z h. Identification of QTLs for 14 agronomically important traits inbased on SNPs generated from high-throughput sequencing. G3-Genes Genomes Genetics, 2017, 7(5): 1587-1594.
[41] 杜希朋, 闫媛媛, 刘伟华, 高爱农, 张锦鹏, 李秀全, 杨欣明, 车永和, 郭小敏. 蚂蚱麦×碧玉麦杂交F2代部分重要农艺性状的遗传分析. 麦类作物学报, 2011, 31(4): 624-629.
DU X P, YAN Y Y, LIU W H, GAO A N, ZHANG J P, LI X Q, YANG X M, CHE Y H, GUO X M. Genetic analysis on several important agronomic traits in F2generation of Mazhamai×Quality. Journal of Triticeae Crops, 2011, 31(4): 624-629. (in Chinese)
[42] 解松峰, 吉万全, 张耀元, 张俊杰, 胡卫国, 李俊, 王长有, 张宏, 陈春环. 小麦重要产量性状的主基因+多基因混合遗传分析. 作物学报, 2020, 46(3): 365-384.
XIE S F, JI W Q, ZHANG Y Y, ZHANG J J, HU W G, LI J, WANG C Y, ZHANG H, CHEN C H. Genetic effects of important yield traits analysed by mixture model of major gene plus polygene in wheat. Acta Agronomica Sinica, 2020, 46(3): 365-384. (in Chinese)
[43] WANG Z l, Wang J, PENG J x, DU X f, Jiang M s, LI Y f, HAN F, DU G h, YANG H q, LIAN S c, YONG J p, CAI W, CUI J d, HAN K n, YUAN F, CHANG F, YUAN G b, ZHANG W n, ZHANG L y, PENG S z, Zou H f, Guo E h. QTL mapping for 11 agronomic traits based on a genome-wide Bin-map in a large F2population of foxtail millet ((L.). Springer Netherlands, 2019, 39: 18.
[44] ZHI H, HE Q, TANG S, YANG J j, ZHANG W, LIU H , JIA Y c, JIA G q, ZHANG A y, LI Y h, GUO E h, GAO M, LI S j, LI J x, QIN N, ZHU C c, MA C y, ZHANG H j, CHEN G q, ZHANG W f, WANG H g, QIAO Z j, LI S g, CHENG R h, XING L, WANG S y, LIU J r, LIU J, DIAO X m. Genetic control and phenotypic characterization of panicle architecture and grain yield-related traits in foxtail millet (). Theoretical and applied genetics, 2021, 134(9): 1-14.
[45] 杨坤. 谷子SSR标记连锁图谱构建及几个主要性状QTL分析[D]. 石家庄: 河北师范大学, 2008.
Yang K. Construction of SSR based linkage map and QTL analysis of several important traits in foxtail millet,[D]. Shijiazhuang: Hebei Normal University, 2008. (in Chinese)
[46] Wang J, Wang Z l, Du X f, Yang H q, Han F, Han Y h, Yuan F, Zhang L y, Peng S z, Guo E h. A high-density genetic map and QTL analysis of agronomic traits in foxtail millet [(L.) P. Beauv.] using RAD-seq. PloS one, 2017, 12: e0179717.
[47] Fang X m, Dong K j, Wang X q, Liu T p, He J h, Ren R y, Zhang L, Liu R, Liu X y, Li M, Huang M z, Zhang Z s, Yang T y. A high density genetic map and QTL for agronomic and yield traits in foxtail millet [(L.) P. Beauv.]. BMC Genomics, 2016, 17: 336.
[48] Liu T p, He J h, Dong K j, Wang X w, Wang W w, Yang P, Ren R y, Zhang L, Zhang Z s, Yang T y. QTL mapping of yield component traits on bin map generated from resequencing a RIL population of foxtail millet (). BMC genomics, 2020, 21: 141.
[49] 姚金保, 任丽娟, 张平平, 杨学明, 马鸿翔, 姚国才, 张鹏, 周淼平. 小麦株高及其构成因素的遗传及相关性分析. 麦类作物学报, 2011, 31(4): 604-610.
Yao J B, Ren L J, Zhang P P, Yang X M, Ma H X, Yao G C, Zhang P, Zhou M P. Genetic and correlation analysis of plant height and its components in wheat. Journal of Triticeae Crops, 2011, 31(4): 604-610. (in Chinese)
[50] 杨兆生, 阎素红, 王俊娟. 不同种植方式下小麦株高构成因素的相关研究. 麦类作物学报, 1999(4): 19-22.
Yang Z S, Yan S H, Wang J J. Study on components of wheat plant height under different planting patterns. Journal of Triticeae Crops, 1999(4): 19-22. (in Chinese)
[51] 赵万春, 王红. 小麦株高及其构成因素的遗传和相关性研究. 麦类作物学报, 2003(4): 28-31.
Zhao W C, Wang H. Genetic and correlation study on plant height and its components in wheat. Journal of Triticeae Crops, 2003(4): 28-31. (in Chinese)
Analyzing Genetic Effects for Plant Height and Panicle Traits by Means of the Mixed Inheritance Model of Major Gene Plus Polygene in Foxtail Millet
Guo Shuqing1, Song Hui2, Yang QingHua1, Gao Jinfeng1, Gao Xiaoli1, Feng Baili1, Yang Pu1
1College of Agriculture, Northwest A&F University/State Key Laboratory of Crop Stress Biology in Arid Areas, Yangling 712100, Shaanxi;2Institute of Millet Crops, Anyang Academy of Agricultural Sciences, Anyang 455000, Henan
【Objective】Plant height and panicle traits are key yield-dependent traits in foxtail millet. The objective was to probe into inheritance patterns of plant height and panicle traits and provide a reference basis for genetically improving related traits and mapping their genes.【Method】Yugu 18, a high performing foxtail millet variety, was arranged as the male parent to cross two foxtail millet varieties, Huangruangu and Hongjiugu, and thus two F7populations of which each was composed of recombinant inbred lines involving 250 family lines(YYRIL and YRRIL)were established. Phenotypic data of Five agronomic traits of the two populations, plant height, panicle length, internode length under panicle, spikelet number per panicle and grain weight per panicle, were genetically examined in two different environments using the mixed inheritance model of major gene plus polygene.【Result】In these two environments, all the five agronomic traits showed continuous variations with their kurtosis and skewness values standing at the absolute value of less than 1 and thus presenting a distribution close to a normal distribution, were characterized by typical inheritance of quantitative traits; some of these traits saw super-parent separation phenomena.The correlation analysis among the traits showed that the plant height appeared significantly and positively correlated with the panicle length, and an extremely significantly positive correlation between spikelet number per panicle and grain weight per panicle was also found in the two environments. The analysis by the inheritance model showed that the best inheritance models for the plant height of the YYRIL and YRRIL population were the PG-AI and PG-A polygene models, and the heritability of the polygenes standing at 95.15% and 91.27%, respectively. The best inheritance models for the spikelet number per panicle of the two populations were the PG-AI, with the heritability of the polygenes standing at 70.07%-71.58%. The best inheritance models for the internode length under panicle of the two populations were the 4MG-CEA and 3MG-CEA of which both were models for equally additive major genes. In YYRIL, the heritability of the major genes for the internode length under panicle stood at 9.69%, and the four pairs of major genes had an equal additive effect value of -0.34, taking negative effect; and in the YYRIL, the heritability of the major genes for the internode length under panicle stood at 45.78%, and the 3 major gene pairs in question had an equal additive effect value of 1.17, taking positive effect. In the YYRIL, the best inheritance model for the panicle length was the MX2-ED-A, a model for two pairs of dominant epistatic major genes and additive polygenes, with the heritability of the major genes and polygenes standing at 43.56% and 50.56%, respectively. the two pairs of panicle length-dependent major genes separately had the additive effect values of -1.21 and 1.68 and the polygenes had a lower additive effect value of -0.0017; in the YRRIL, the best inheritance model for the panicle length was the MX2-AE-A, a mixed inheritance model for two pairs of accumulative effect major genes and additive polygenes; the major genes and polygenes for the panicle length had heritability values standing at 46.40% and 46.91%, respectively. The first pair of panicle length-dependent major genes had an additive effect value of 1.53, taking positive effect; The additive and epistatic interactions effect value of the first×the second pairs of major genes were 0.60. The polygenes had an additive effect value of -0.47, taking the lower negative inheritance effect. In the YYRIL, the best inheritance model for the grain weight per panicle was the MX2-ED-A; the grain weight per panicle followed the inheritance model for two pairs of dominant epistatic major genes + additive polygenes with the heritability of the major genes and polygenes standing at 69.09% and 12.08%; the additive effect values of the two pairs of grain-weight per panicle-dependent major genes were separately 0.58 and 5.82, with the additive effect of the second pair of major genes dominating, and the additive effect value of polygenes stood at a value of -3.81. In the YRRIL, the best inheritance model for the grain weight per panicle was the 3MG-PEA, an inheritance model for three pairs of partially equal additive major genes; the heritability of the grain weight per panicle-dependent major genes stood at 81.10% and the additive effect values of the three pairs of major genes separately were -2.68, -2.68and 2.66, all taking negative effect.【Conclusion】In foxtail millet, the plant height and spikelet number per panicle had similar inheritance models, were all under polygenic control with a higher heritability and environmentally affected to a slight content; the inheritance of the internode length under panicle was genetically controlled by major genes, which had a lower heritability and were environmentally affected to a great extent, and thus environmental factors should be taken into full account in production; the panicle length was genetically controlled jointly by major genes and polygenes; the grain weight per panicle was genetically controlled by major genes with a high heritability in both of the two population and probably carried major QTL.
foxtail millet; RIL; plant height; panicle traits; major gene+polygene

2021-05-24;
2021-07-08
国家重点基础研究发展计划(2019YFD1000702,2019YFD1000702-4,2020YFD1000800,2020YFD1000803)、财政部和农业农村部:国家现代农业产业技术体系(CARS-06-A26)、陕西省小杂粮产业技术体系(NYKJ-2018-YL19)
郭淑青,E-mail:gsq055069@nwafu.edu.cn。宋慧,E-mail:837181622@qq.com。郭淑青和宋慧为同等贡献作者。
杨璞,E-mail:yangpu5532@hotmail.com
(责任编辑 李莉)
