基于计算机虚拟技术研究牦牛乳硬质干酪苦味肽的抑菌活性差异
2022-01-07杨保军宋雪梅
杨保军, 梁 琪*, 宋雪梅
(1. 甘肃农业大学食品科学与工程学院,甘肃兰州 730070;2. 甘肃省功能乳品工程实验室,甘肃兰州 730070)
抗菌肽主要来自乳源蛋白水解物[1-2],已证明乳铁蛋白和酪蛋白片段具有抑菌活性。 1930 年Jones等首次从牛乳中发现抑制链球菌的抗菌性物质,命名为乳铁蛋白[3]。 1974 年,Hill 等用凝乳酶水解牛乳αS1-酪蛋白,得到第一个对应于αS1-酪蛋白1~23 位残基的抗菌肽(isracidine)[4]。2005 年Mccann 等用胃蛋白酶酶解牛乳酪蛋白, 得到对应于αS1-酪蛋白99~109 位残基的新型抗菌肽(Cp1)和对应于αS2-酪蛋白183~207 位残基的抗菌肽(Cp2)[5]。 大多数抗菌肽在自然pH 下带正电荷, 正电荷数目的增加不仅可以提高抗菌肽与细菌细胞膜的结合能力,还能有效降低抗菌肽的杀菌浓度[6]。 Sedaghati 等研究发现肽的疏水性对抑菌活性有影响,抗菌肽的N 末端富含亲水性氨基酸残基,如赖氨酸、精氨酸等,C 末端富含疏水性氨基酸残基,如丙氨酸、甘氨酸,且通常被酰胺化[7]。 此外,抗菌肽结构的稳定性受肽链长度的影响, 从而抑菌活性也受到肽链长度的影响。Phambu 等通过傅里叶变换红外光谱(FTIR)、扫描电子显微镜(SEM)、热重分析(TGA)及示差扫描量热法(DSC)研究表明理想的肽链长度(肽段含有6个氨基酸)可以增加分子内氢键作用,从而保持肽链的稳定性, 进而与阴离子的脂质更好地结合,发挥更强的抑菌作用[8]。 Théolier 等研究了加拿大5 种市售干酪 (Mozzarella,Gouda,Swiss,old Cheddar,medium Cheddar)脱盐纯化的水溶性提取物后发现,只有Mozzarella 和Gouda 干酪对李斯特菌ivanovii和大肠杆菌MC4100 有明显抑制作用[9]。项目组前期研究牦牛乳硬质干酪水溶性肽粗提物的抑菌活性发现, 其对大肠杆菌的最大抑菌圈直径为15.91 mm,对金黄色葡萄球菌的最大抑菌圈直径为16.50 mm, 对沙门氏菌的最大抑菌圈直径为16.03 mm。Fontenele 等研究了Coalho 干酪酪蛋白衍生肽的生物活性后发现,β-酪蛋白肽(193~209 位)具有免疫调节、抑菌和降压活性,αS1-酪蛋白肽(1~23 位)是一种具有免疫调节活性和抑菌活性的肽片段,这些结果被确定为巴西干酪的权威性标志[10]。 当然牦牛乳硬质干酪中的肽粗提物也具有类似的抑菌、抗氧化和降压活性等。 Fialho 等结合MALDI-TOF/TOF、MASCOT Daemon 与UniProt 数据库对3 种市售Minas 干酪中提取的可溶性肽进行测序和鉴定,发现起源于αS1-酪蛋白的肽序列PKHPIKHQ、RPKHPIKHQG、RPKHPIKHQGLPQ 和RPKHPIKHQ GLPQE 是属于Isracidine 的片段, 另外发现了两种β-酪蛋白衍生的新抗菌肽序列HQPHQPLPPT 和MHQPHQPLPPT[11]。 宋雪梅等经葡聚糖凝胶(Sephadex G-25)色谱和液相色谱串联质谱(LC-MS/MS)从牦牛乳硬质干酪中鉴定出14 种苦味肽, 但肽与不同菌体蛋白之间相互作用的抑菌机理尚不清楚[12]。
基于结构的药物研究和设计主要采用的是分子对接技术。 分子对接就是把配体分子放在受体活性位点的位置,然后按照几何互补、能量互补和化学环境互补的原则实时评价配体与受体相互作用的好坏,并找到两个分子间的最佳结合模式。 目前对牦牛乳干酪中蛋白质降解肽抑菌活性的研究仍局限于其粗提物,关于净电荷、疏水性和疏水力矩等因素对肽段抑菌活性的影响鲜有报道。 因此,运用相关生物信息学软件计算牦牛乳硬质干酪中4种苦味肽(RPKHPIK、TPVVVPPFL、VYPFPGPIPN 和SLVYPFPGPIPN)的分子特性,运用分子对接技术从分子水平研究苦味肽抑菌活性、研究配体(苦味肽)和受体(不同菌体蛋白)间相互作用的分子机制,通过BIOPEP 数据库比对表征肽段抑菌活性。 为理论水平分析和分子水平解释干酪苦味肽的抑菌活性提供了有力支撑, 为完善BIOPEP 的抗菌肽数据库奠定了理论基础。
1 材料与方法
1.1 实验工具和材料
实验工具:ChemBioDraw14.0.0.117(化学结构绘图软件),Discovery Studio Client v16.1.0 (DS)(蛋白质结构分析软件);实验材料:以项目组前期从牦牛乳硬质干酪中鉴定出的4 种苦味肽为研究对象,4种苦味肽的基本信息见表1 (表中Q 为氨基酸自由能改变量与肽链中氨基酸残基摩尔数的比值)。

表1 牦牛乳硬质干酪中苦味肽氨基酸序列及其Q 值Table 1 Amino acid sequence and Q value of bitter peptide in yak milk hard cheese
1.2 实验方法
1.2.1 苦味肽分子特性计算用Peptide Property Calculator 软件计算4 种苦味肽的理论相对分子质量和净电荷[13];用HeliQuest 软件计算4 种苦味肽的疏水性指数和疏水力矩[14];用PEPTIDE 2.0 软件计算4 种苦味肽的疏水性氨基酸比例[13];用PEPTIDE 2.0 软件预测肽的二级结构[15]。

图1 RK7、TL9、VN10 和SN12 的分子结构Fig. 1 Chemical structure of RK7, TL9, VN10 and SN12
1.2.3 准备受体蛋白从Protein Data Bank(PDB)数据库(www.rcsb.org) 中下载大肠杆菌(PDB ID:5BNS,分辨率为2.2 Å)、金黄色葡萄球菌(PDB ID:4ALM, 分辨率为2.45 Å) 和沙门氏菌 (PDB ID:6CH3,分辨率为2.68 Å)蛋白晶体的X 射线衍射三维结构,并用DS 软件处理蛋白质,去除所有的水分子,去除蛋白质多构象,补充完整的氨基酸残基,为蛋白质加氢等(1Å=0.1 nm)。
1.2.4 分子对接基于结构的药物设计工具LibDock 适用于对大规模数据库进行快速精确地虚拟筛选[16],因此,选择DS 软件中的LibDock 程序进行分子对接研究。对接前用DS 软件中的Define 和Edit Binding Site 工具寻找菌体蛋白的可能结合部位,对接时将每个分子生成的对接构象数目设置为10,其他参数均为系统默认值,对接后根据对接得分和结合能,确定苦味肽在菌体蛋白活性部位的最佳对接位姿;DS 软件还用于识别肽与菌体蛋白结合位点上残基之间的氢键、盐桥和范德华力等。
1.2.5 肽数据库比对根据文献[17]对BIOPEP 数据库的描述,结合分子对接情况,将牦牛乳硬质干酪中4 种苦味肽与BIOPEP 数据库中的抗菌肽序列进行比对,根据苦味肽段中具有抑菌活性的肽序列来表征和预测苦味肽的抑菌活性。
2 结果与分析
2.1 苦味肽分子特性分析
有关研究表明,决定抗菌肽抑菌活性的因素包括肽的疏水性强弱、含正电荷数、疏水力矩和二级结构等[14],另外,具有α-螺旋构型的抗菌肽有较高的抑菌活性[15]。 4 种苦味肽分子特性的计算结果表明(见表2),RK7 含有3 个碱性氨基酸和3 个疏水性氨基酸,所带净电荷为3.1,疏水性氨基酸比例为42.86%,是4 种苦味肽中疏水性氨基酸含量最低的肽段;TL9 含有8 个疏水性氨基酸,疏水性氨基酸比例为88.89%,是4 种苦味肽中疏水性氨基酸含量最高的肽段;VN10 和SN12 分别含有7 个和8 个疏水性氨基酸,疏水性氨基酸比例为70.00%和66.67%,疏水力矩分别为0.189 和0.372;二级结构预测发现这4 种苦味肽均以无规则卷曲形式存在。 所以可排除RK7、TL9、VN10 和SN12 二级结构对其抑菌活性的影响,即这4 种苦味肽的抑菌活性很大程度上可能取决于其疏水性、正电荷数和疏水力矩。 将通过蛋白质结构分析软件和分子对接工具对RK7、TL9、VN10 和SN12 的抑菌活性进行研究。

表2 苦味肽的理化特性Table 2 Physicochemical properties of the bitter peptides
2.2 寻找受体结合区
利用DS 软件中的Define 和Edit Binding Site工具寻找受体中的空腔,以此来确定受体中可能的结合部位。 图2(a)为5BNS 的三维构象,经过处理后系统视图中自动添加9 个结合位点(Site1~9),即找到了9 个可能的结合区域;图2(b)为4ALM 的三维构象, 经过处理后系统视图中自动添加31 个结合位点 (Site1~31), 即找到了31 个可能的结合区域;图2(c)为6CH3 的三维结构,经过处理后系统视图中自动添加15 个结合位点(Site1~15),即找到了15 个可能的结合区域。

图2 从受体中寻找可能结合区域Fig. 2 Searching for possible binding regions from receptor
2.3 分子对接
2.3.1 大肠杆菌(5BNS)与配体的作用机制通过DS 软件中的LibDock 对接程序研究了RK7、TL9、VN10 和SN12 对5BNS 的抑制机制, 对接后RK7、TL9 与5BNS 成功形成配体-受体复合物构象,最佳对接构象见图3 和图4。
RK7 与5BNS 对接后得到26 个对接构象,这些构象均位于Site7 区域(X:6.28 Y:41.91 Z:28.88 R:9.00 Å),最佳对接构象的对接得分为163.023,结合能为5 622.91 kJ/mol。 图3 为RK7 与5BNS 对接形成的最佳构象图,结果显示RK7 与5BNS 的氨基酸残基Glu31(4.34 Å)、Thr35(3.90 Å)和Arg151(4.10 Å、6.48 Å)之间形成了4 个不同距离的氢键,其中RK7和氨基酸残基Arg151 之间含有2 个氢键,与Thr35之间形成的氢键最短, 结合最紧密;RK7 与氨基酸残基Trp32 (3.00 Å、3.89 Å)、Asp150 (4.69 Å)和Arg151(3.32 Å)形成4 个距离不同的碳氢键,与氨基酸残基Asp150(4.68 Å)和Arg151(3.41 Å)形成盐桥或电荷相互作用。
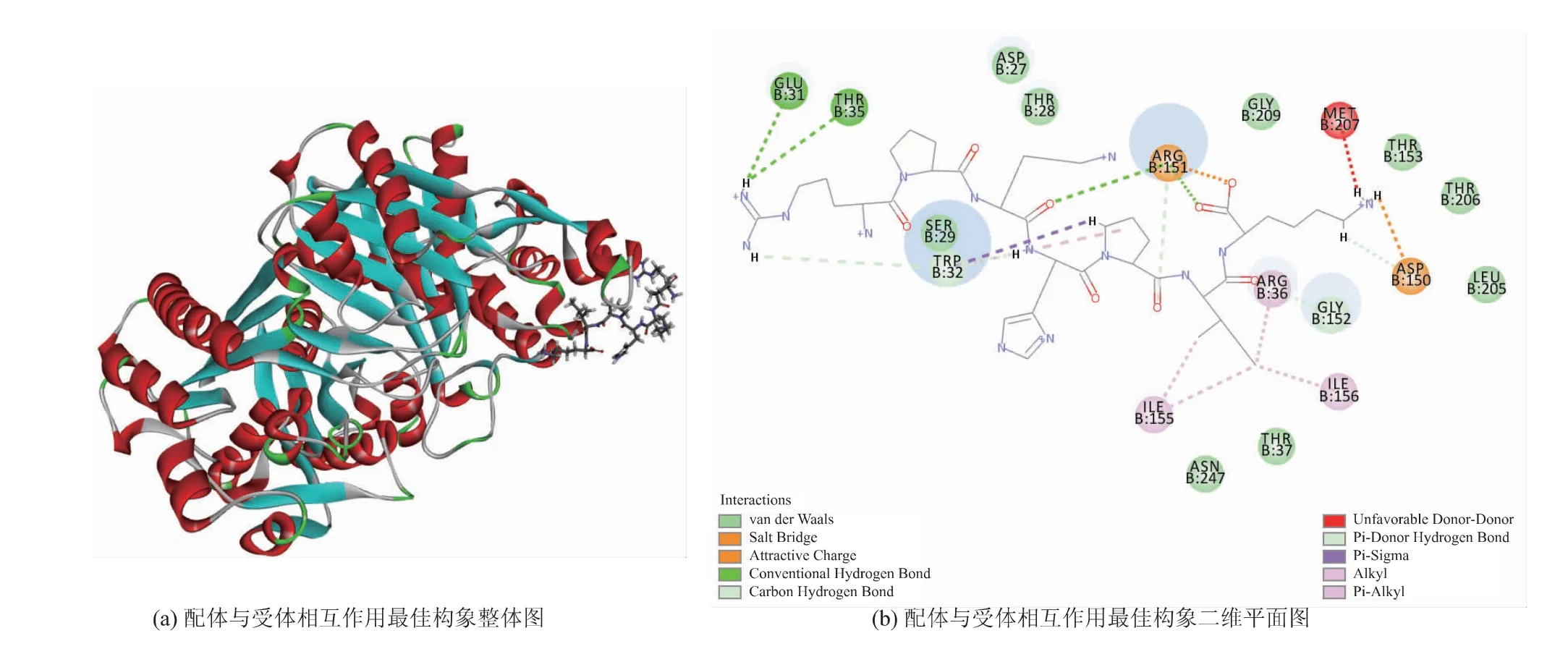
图3 RK7 与5BNS 的结合位点及相互作用Fig. 3 Binding site and interaction between RK7 and 5BNS
TL9 与5BNS 对接后得到2 个对接构象, 这2个构象均位于Site7 区域(X:6.28 Y:41.91 Z:28.88 R:9.00 Å),最佳对接构象的对接得分为148.819,结合能为8 391.90 kJ/mol。 图4 为TL9 与5BNS 对接形成的最佳构象图,结果表明,TL9 与5BNS 的氨基酸残基Asp27(4.31 Å)、Trp32(5.35 Å)、Arg36(4.05 Å)、Arg151(6.12 Å)和Asn210(5.06 Å)之间形成了5 个不同距离的氢键, 与Arg36 之间形成的氢键最短,结合最紧密;TL9 与氨基酸残基Asp27(4.45 Å、5.02 Å)、Thr28(4.18 Å)和Gly152(2.96 Å)形成4 个距离不同的碳氢键,与氨基酸残基Arg36(4.12 Å)形成电荷相互作用。
(2) 校对。由于WORD和WPS有区别或者WORD版本不同,PPT上会有很多字母、符号、上下标等编辑到视频中后不能显示或显示错误,所以需要仔细审核、修改。每一句配音到底应该从哪一个镜头到哪一个镜头,都需要认真校对、精确到秒。还有一些照片或者视频编辑出来后效果不理想,就得重新拍摄。

图4 TL9 与5BNS 的结合位点及相互作用Fig. 4 Binding site and interaction between TL9 and 5BNS
2.3.2 金黄色葡萄球菌(4ALM)与配体的作用机制RK7、TL9、VN10 和SN12 与4ALM 的对接构象见图5~8。RK7 与4ALM 对接后得到232 个对接构象,其中Site1 区域包含50 个对接构象,Site3 区域包含8个对接构象,Site6 区域包含39 个对接构象,Site8区域包含50 个对接构象,Site10 区域包含16 个对接构象,Site15 区域包含50 个对接构象,Site16 区域包含16 个对接构象,Site19 区域包含2 个对接构象,Site24 区域包含1 个对接构象,最佳对接构象位于Site8 区域 (X:-32.27 Y:13.97 Z:45.97 R:9.00 Å),对接得分为192.573,结合能为2 729.67 kJ/mol。图5 为RK7 与4ALM 对接形成的最佳构象图,结果表明,RK7 与4ALM 的氨基酸残基Leu176(4.44 Å)、Ser189(3.62 Å、4.59 Å)、Gly191(2.83 Å)、Lys209(6.03 Å)、Thr242(2.81 Å、3.90 Å)、Gly243(4.81 Å)和Asp249 (3.73 Å) 之间形成了9 个距离不同的氢键, 其中Ser189 和Thr242 分别参与了2 个氢键的形成, 在RK7 与4ALM 的相互作用中贡献较大;RK7 与氨基酸残基Thr242 (4.24 Å)、Glu244(4.19 Å)、Asp249(3.30 Å)和Ser250(4.27 Å)形成4 个距离不同的碳氢键, 与氨基酸残基Lys209 (5.05 Å)、Glu225(7.54 Å)、Glu244(5.28 Å)、His247(4.52 Å)和Asp249(4.35 Å)形成电荷或π-Cation 相互作用。
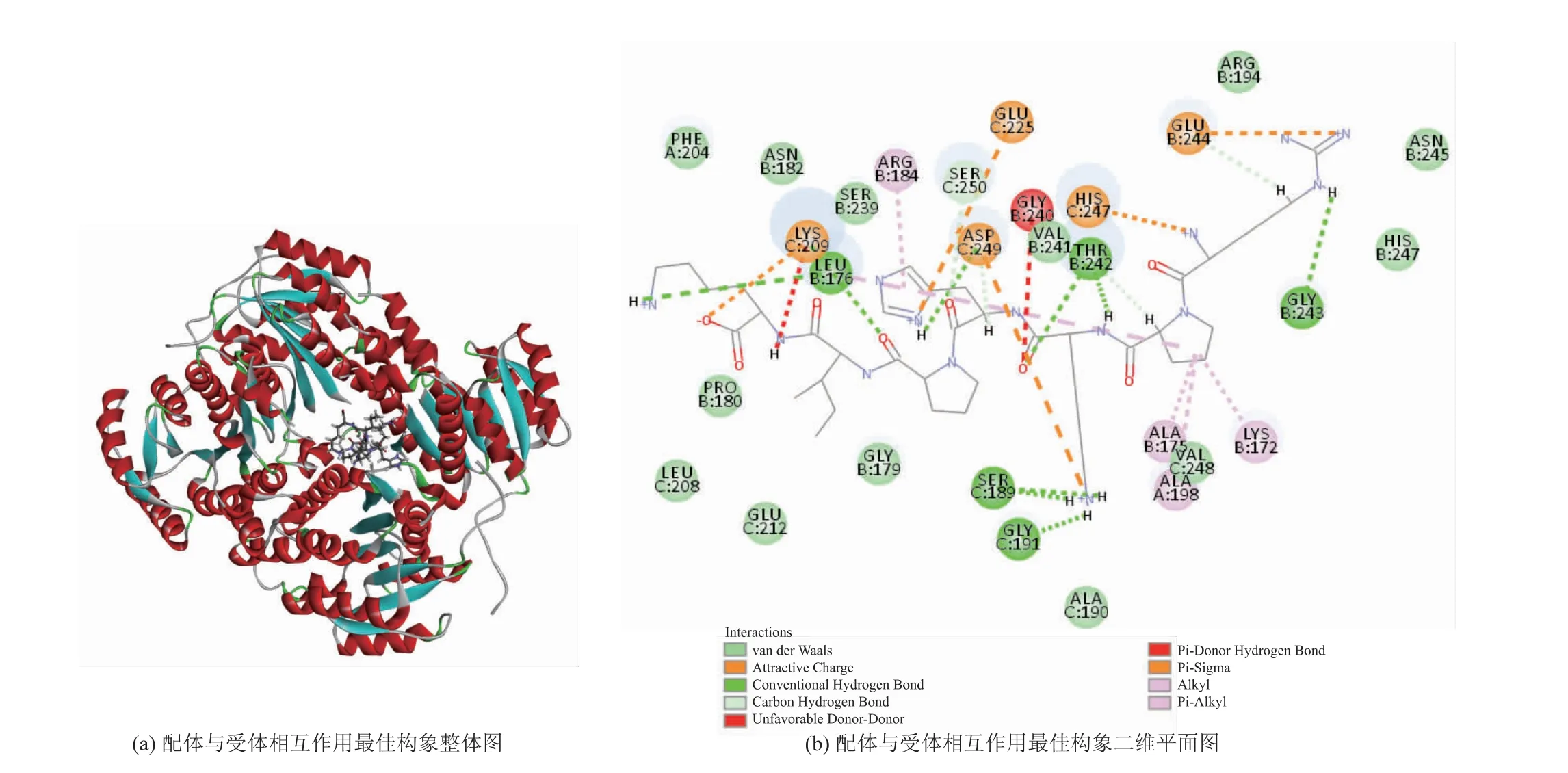
图5 RK7 与4ALM 的结合位点及相互作用Fig. 5 Binding site and interaction between RK7 and 4ALM
TL9 与4ALM 对接后得到47 个对接构象,其中Site1 区域包含20 个对接构象,Site6 区域包含5 个对接构象,Site8 区域包含12 个对接构象,Site15 区域包含10 个对接构象,最佳对接构象位于Site1 区域(X:-42.00 Y:-2.21 Z:32.23 R:9.00 Å),对接得分为158.314,结合能为3127.41 kJ/mol。图6 为TL9与4ALM 对接形成的最佳构象图,结果显示,TL9 与4ALM 的氨基酸残基Lys164 (5.05 Å)、Glu168(5.04 Å)和Lys256(5.57 Å)之间形成了3 个距离不同的氢键;TL9 与氨基酸残基His247(5.28 Å)形成1 个碳氢键,与Lys256(5.57 Å)形成电荷相互作用。

图6 TL9 与4ALM 的结合位点及相互作用Fig. 6 Binding site and interaction between TL9 and 4ALM
VN10 与4ALM 对接后得到21 个对接构象,其中Site1 区域包含20 个对接构象,Site8 区域包含1个对接构象, 最佳构象位于Site8 区域 (X:-32.27 Y:13.97 Z:45.97 R:9.00 Å), 对接得分为179.923,结合能为162 654.67 kJ/mol。图7 为VN10 与4ALM对接形成的最佳构象图, 结果表明,VN10 与4ALM的氨基酸残基Arg184 (4.19 Å)、Gly191 (4.50 Å)、Glu244(4.76 Å)和Asp249(3.55 Å)之间形成了4 个不同距离的氢键;VN10 与氨基酸残基Leu176(3.92 Å)、Gly179(3.36 Å)、Gly191(4.23 Å)、Thr242(2.90 Å)、Gly243(3.96 Å)、Glu244(5.09 Å)、His247(2.78 Å)、Asp249(4.06 Å、4.95 Å、5.34 Å)和Ser250(4.19 Å、4.25 Å) 形成碳氢键或π-Donor Hydrogen 键,与His247(4.73 Å)形成电荷相互作用。
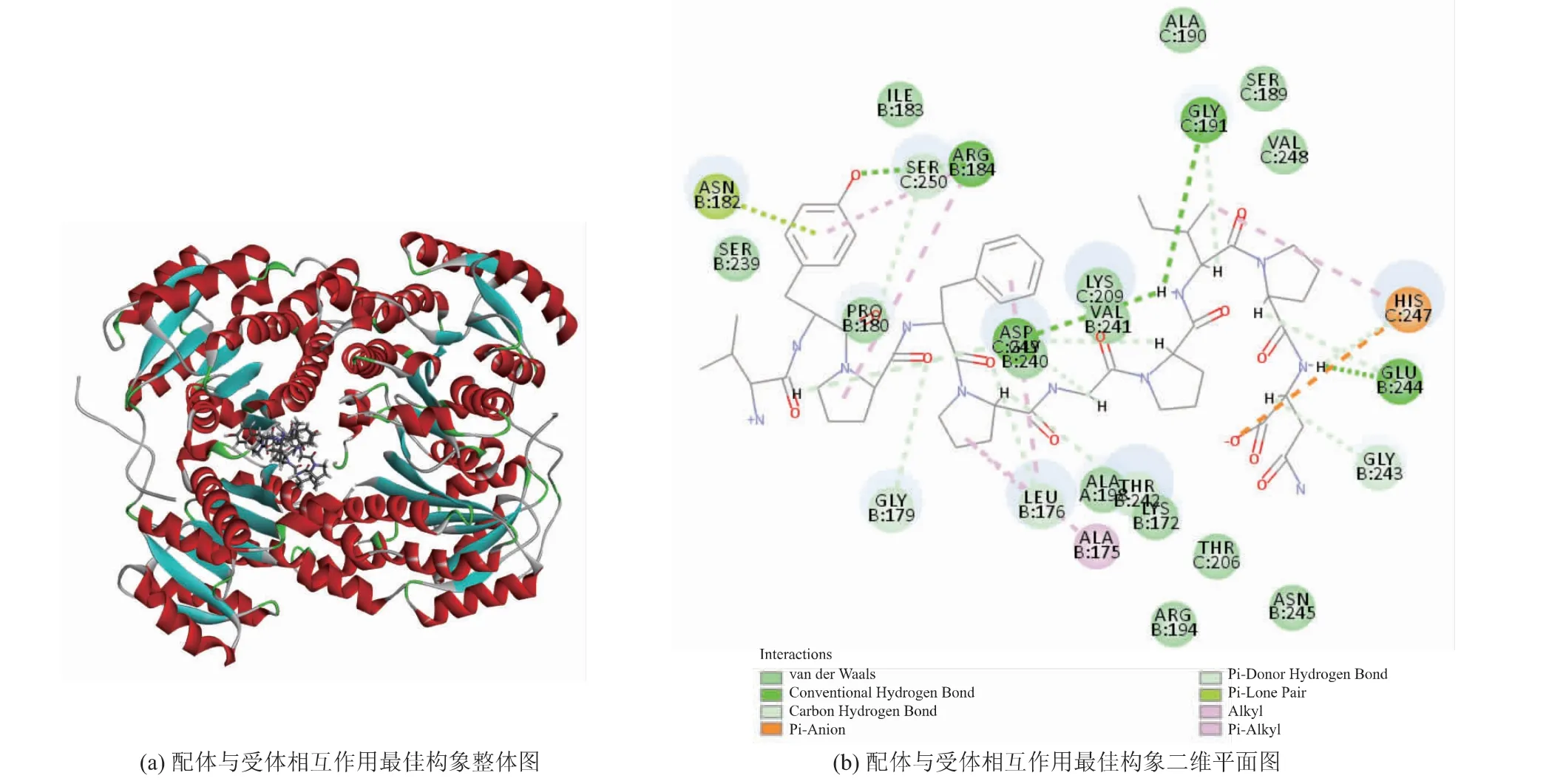
图7 VN10 与4ALM 的结合位点及相互作用Fig. 7 Binding site and interaction between VN10 and 4ALM
SN12 与4ALM 对接后得到22 个对接构象,其中Site1 区域包含10 个对接构象,Site8 区域包含9个对接构象,Site15 区域包含1 个对接构象,Site16区域包含2 个对接构象, 最佳构象位于Site1 区域(X:-42.00 Y:-2.21 Z:32.23 R:9.00 Å), 对接得分为31.892,结合能为5 703.22 kJ/mol。 图8 为SN12与4ALM 对接形成的最佳构象图, 结果显示,SN12与4ALM 的氨基酸残基Ala254(4.26 Å)和Lys256(3.89 Å、6.32 Å)之间形成了3 个距离不同的氢键;SN12 与氨基酸残基Ala165 (3.45 Å)、Glu168(3.78 Å)和His253(4.52 Å)形成3 个距离不同的碳氢键,与Lys164(5.51 Å、6.45 Å)、Arg194(5.89 Å)和Lys256(8.12 Å)形成电荷相互作用。

图8 SN12 与4ALM 的结合位点及相互作用Fig. 8 Binding site and interaction between SN12 and 4ALM
2.3.3 沙门氏菌(6CH3)与配体的作用机制 RK7、TL9、VN10 和SN12 与6CH3 的对接构象见图9~12。 RK7 与6CH3 对接后得到58 个对接构象,其中Site1 区域包含50 个对接构象,Site2 区域包含1 个对接构象,Site5 区域包含5 个对接构象,Site9 区域包含2 个对接构象, 最佳构象位于Site1 区域(X:4.21 Y:39.79 Z:4.74 R:9.00 Å), 对接得分为186.054,结合能为38 274.18 kJ/mol。图9 为RK7 和6CH3 对接形成的最佳构象图, 结果显示,RK7 与6CH3 的氨基酸残基Arg41(5.83 Å)、Lys60(6.15 Å)、Leu197(3.47 Å)、Arg198 (3.58 Å)、Gln475 (3.85 Å、5.05 Å)、Val481(4.53 Å)和Glu483(4.31 Å)之间形成了8 个距离不同的氢键,其中RK7 和氨基酸残基Gln475 之间含有2 个氢键, 与Leu197 之间形成的氢键最短, 结合最紧密;RK7 与氨基酸残基Met30(4.39 Å)、Gly34 (2.72 Å、3.38 Å)、Arg41 (3.44 Å)、Ala57 (3.41 Å、3.44 Å)、Leu197 (5.80 Å)、Gln475(6.56 Å)、Phe479(5.61 Å)和Thr480(4.23 Å)之间形成了10 个距离不同的碳氢键,与Asp33(6.10 Å)、Arg41(5.30 Å)和Glu483(6.36 Å)形成电荷相互作用。

图9 RK7 与6CH3 的结合位点及相互作用Fig. 9 Binding site and interaction between RK7 and 6CH3
TL9 与6CH3 对接后得到6 个对接构象, 这些构象均位于Site1 区域(X:4.21 Y:39.79 Z:4.74 R:9.00 Å),最佳构象的对接得分为144.172,结合能为4 198.23 kJ/mol。 图10 为TL9 与6CH3 对接形成的最佳构象图,结果表明,TL9 与6CH3 的氨基酸残基Ser14(4.18 Å)和Glu483(4.68 Å)之间形成了2 个距离不同的氢键;TL9 与氨基酸残基Gln475(5.14 Å)和Gly478(3.50 Å、3.58 Å)形成了3 个距离不同的碳氢键,与Glu483(7.27 Å)形成电荷相互作用。
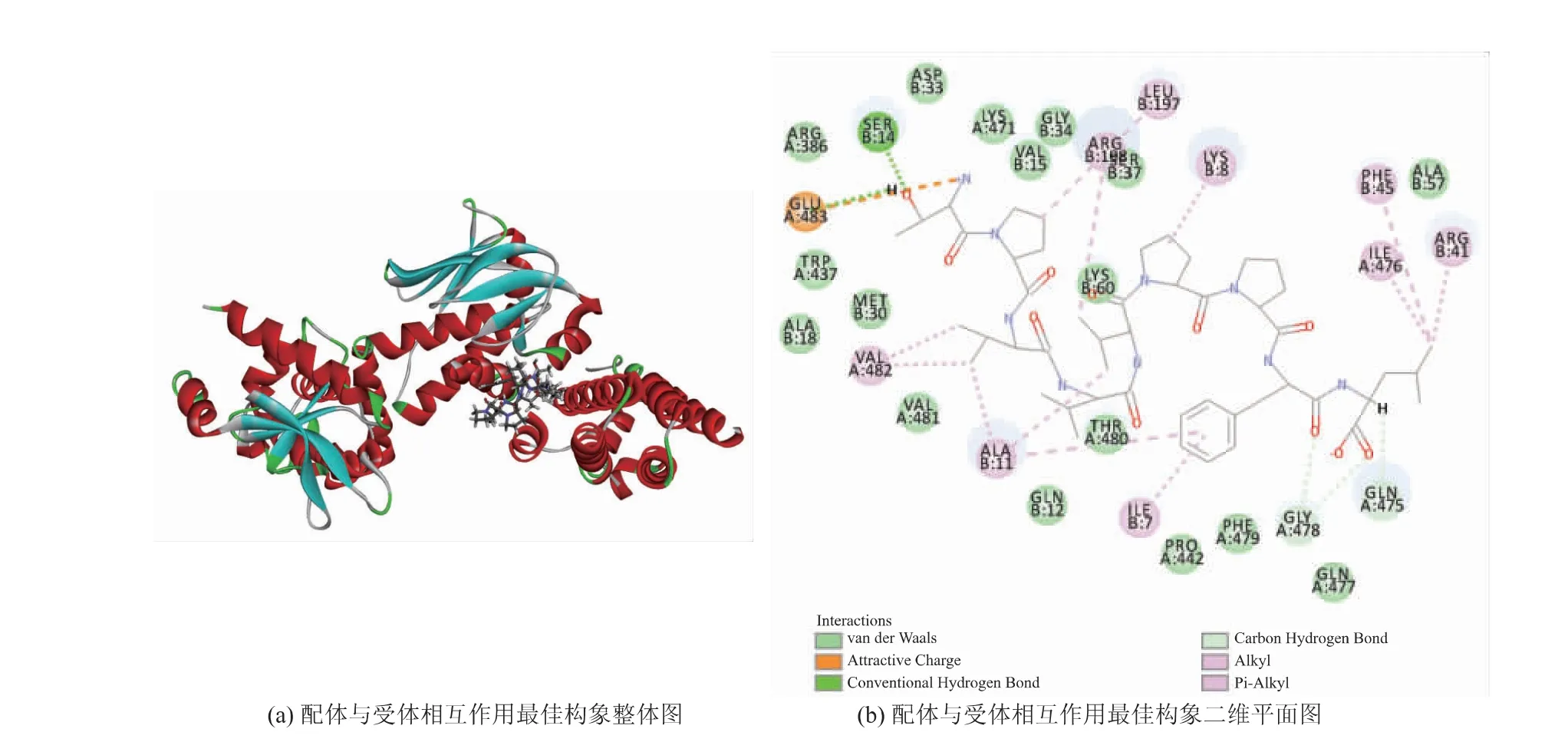
图10 TL9 与6CH3 的结合位点及相互作用Fig. 10 Binding site and interaction between TL9 and 6CH3
VN10 与6CH3 对接后得到1 个对接构象,该构象位于Site1 区域 (X:4.21 Y:39.79 Z:4.74 R:9.00 Å),对接得分为109.897,结合能为61 702.13 kJ/mol。图11 为VN10 与6CH3 对接形成的构象图,结果表明,VN10 与6CH3 的氨基酸残基Ser14(4.24 Å、4.39 Å)、Asp33(4.53 Å)、Arg198(3.92 Å)和Lys471(5.60 Å) 之间形成了5 个距离不同的氢键;VN10 与氨基酸残基Ala11(2.75 Å)、Met30(3.33 Å)、Arg41(6.44 Å)、Glu56(5.57 Å)和Lys471(5.60 Å)形成了5 个距离不同的碳氢键,与Glu56(6.60 Å)形成电荷相互作用。

图11 VN10 与6CH3 的结合位点及相互作用Fig. 11 Binding site and interaction between VN10 and 6CH3
SN12 与6CH3 对接后得到4 个对接构象,这4个构象全部位于Site1 区域(X:4.21 Y:39.79 Z:4.74 R:9.00 Å),最佳构象的对接得分为-272.228,结合能为252 021 549.25 kJ/mol。图12 为SN12 与6CH3对接形成的最佳构象图, 结果表明,SN12 与6CH3的氨基酸残基Lys60 (4.93 Å)、Arg198 (5.34 Å)和Val481 (4.90 Å) 之间形成3 个距离不同的氢键;SN12 与氨基酸残基Lys8(3.70 Å)、Ala11(5.43 Å)和Arg198(3.67 Å、5.87 Å)形成了4 个距离不同的碳氢键,与Arg41(5.82 Å)、Glu56(6.65 Å)和Lys60(7.40 Å)形成电荷相互作用。
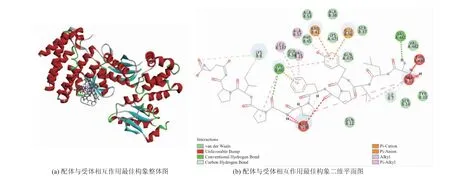
图12 SN12 与6CH3 的结合位点及相互作用Fig. 12 Binding site and interaction between SN12 and 6CH3
抗菌肽抑菌活性强弱与肽链中某些特殊氨基酸残基密切相关, 如含有Arg 和Val 的抗菌肽,Arg可以使阴离子膜上的磷脂形成环形孔[18],Val 可以促进β-折叠片段的延伸[19],因此这类抗菌肽具有更强的静电膜吸附作用和较高的抑菌活性。 Lee 等对人工合成抗菌肽P1(KWKLFK KIPKFLHLAKKF)C 端和N 端氨基酸逐一切除发现Pro 对该肽的抑菌活性发挥着重要作用[20]。 所有肽段当中均含有不同数量的Arg、Val 或Pro,这为肽段具有抑菌活性奠定了基础。
抗菌肽疏水性是影响其抑菌活性的重要因素之一[21]。 在一定范围内,抗菌肽疏水性越大,抑菌活性越强,但疏水性过高会导致抗菌肽自身聚集至沉淀析出,降低抑菌活性[22]。 Jiang 等研究表明,提高抗菌肽的疏水性,其抑菌活性得到提高,但其溶血活性也随之提高,且溶血活性提高幅度大于抑菌活性提高幅度,极大降低了抗菌肽的治疗指数[23]。溶血活性是抗菌肽的一个毒副作用,代表抗菌肽对血红细胞的破坏力度。 肽链长度与抗菌肽抑菌活性之间存在二次函数关系,且较短的抗菌肽往往拥有更低的细胞毒性和溶血活性[24]。 疏水力矩用于对多肽两亲性指标的定量描述[25],平均疏水力矩对抑菌活性的影响要大于疏水性对抑菌活性的影响[26]。Hawrani 等研究指出,增加抗菌肽的疏水力矩和疏水性使得抗菌肽的抑菌活性降低,溶血活性增加[27]。抗菌肽大部分为碱性阳性肽,可以与带负电荷的细菌细胞膜通过正负电荷之间的静电引力相结合,使细菌细胞膜破裂或形成孔洞,细胞内容物外泄,菌体不能维持正常生命活动所需的胞内渗透压而死亡[28-29]。
此外,抗菌肽与膜脂之间的氢键对抗菌肽维持活性结构至关重要[30],片层结构型抗菌肽主要通过肽与膜脂间的氢键或范德华力而不是残基间的氢键来发挥作用[31]。 经测定发现4 种苦味肽RK7、TL9、VN10 和SN12 的二级结构均以无规则卷曲形式存在,所以排除肽与脂膜间氢键对苦味肽抑菌活性的影响;氢键是分子间的较强作用力,对配体和受体形成稳定复合物构象发挥着重要作用。
2.4 苦味肽抑菌活性表征和预测
BIOPEP 数据库是目前最全面的生物活性肽数据库[18],截至2021 年1 月,BIOPEP 数据库记录了4 132 种功能活性已知的肽段, 其中510 种肽段具有抑菌活性, 将RK7、TL9、VN10 和SN12 与BIOPEP 中的抗菌肽数据库比对后发现RK7(ID:8164)来源于αS1-酪蛋白(1~7 位),为抑菌活性已知的肽段,最高相似度为100%,TL9、VN10 和SN12 当中均不含BIOPEP 数据库中抑菌活性已知的肽片段, 但分子对接显示这3 种肽仍能和5BNS、4ALM或6CH3 结合,所以推测TL9、VN10 和SN12 为新型抗菌肽。 综上所述,预测4 种苦味肽的抑菌活性,大肠杆菌抑制活性:RK7 > TL9,金黄色葡萄球菌抑制活性:RK7 > VN10 > TL9 > SN12,沙门氏菌抑制活性:RK7 > TL9 > VN10 > SN12。
3 结 语
通过对牦牛乳硬质干酪中4 种苦味肽RK7、TL9、VN10 和SN12 的分子特性进行计算和分析,对肽结构做能量最小化处理并将它们定义为配体,从PDB 数据库中获取来自不同菌体(大肠杆菌、金黄色葡萄球菌、沙门氏菌)蛋白质晶体的X 射线衍射三维结构(5BNS、4ALM、6CH3)并将它们定义为受体, 配体和受体经DS 软件优化处理后运用分子对接工具对苦味肽抑菌活性及其作用机制进行研究,结果表明RK7、TL9 均能和5BNS、4ALM、6CH3 形成配体-受体复合物构象,VN10 和SN12 仅能和4ALM、6CH3 形成配体-受体复合物构象。 将RK7、TL9、VN10 和SN12 与BIOPEP 数据库中的抗菌肽比对后发现RK7 为抑菌活性已知的完整肽段,TL9、VN10 和SN12 当中虽不含已知抑菌活性的肽片段,但仍能和5BNS、4ALM 或6CH3 结合, 所以推测TL9、VN10 和SN12 为新型抗菌肽。结合肽段分子特性分析、分子对接和数据库比对预测4 种苦味肽的抑菌活性,大肠杆菌抑制活性:RK7 > TL9,金黄色葡萄球菌抑制活性:RK7 > VN10 > TL9 > SN12,沙门氏菌抑制活性:RK7 > TL9 > VN10 > SN12。 下一步将通过体外实验对4 种肽段单体的抑菌活性进行验证。 分子对接等生物信息学方法有效揭示了牦牛乳硬质干酪中具有抑菌活性的苦味肽与不同微生物菌体蛋白间相互作用的分子机制,为抗菌肽的研究提供技术支持,此外,选取的3 种肽TL9、VN10和SN12 并不在BIOPEP 的抗菌肽数据库中, 因此有可能成为抗菌肽数据库的扩充片段。
