胃癌患者预后相关微RNA预测模型的构建及其应用价值探讨
2021-12-31王高明杨鹿笛郁丰荣
岳 犇,王高明,杨鹿笛,崔 然,郁丰荣
上海交通大学医学院附属仁济医院胃肠外科,上海 200127
胃癌发病率和病死率居全球第5位和第3位;尽管手术是目前主要的治疗手段,但半数以上行根治性切除手术的胃癌患者出现局部复发或远处转移,影响患者的预后[1-2]。异常表达的微RNA(microRNA,miRNA)是胃癌的特征之一[3]。miRNA是一类含有17~25个核苷酸的内源性非编码RNA,在转录后水平调控基因的表达[4]。越来越多的研究[5-6]证明,miRNA参与肿瘤的增殖、凋亡、侵袭/转移和血管生成等生物学过程,与肿瘤的发生、发展密切相关,可作为肿瘤诊断和预后的潜在标志物。近年来,随着基因检测技术的不断发展,高通量测序技术结合生物信息学分析,已经在临床应用中取得了重要的成果。本研究基于基因表达数据库(Gene Expression Omnibus,GEO)和癌症基因图谱(The Cancer Genome Atlas,TCGA),回顾性分析397例胃癌患者的临床病理资料,基于生物信息学构建胃癌患者预后相关miRNA预测模型并探讨其应用价值,为胃癌患者个体化诊疗方案的制定提供参考。
1 资料与方法
1.1 临床资料
采用回顾性队列研究方法。利用R 4.0.3 中的TCGAbiolinks包收集TCGA-STAD 中397例胃癌患者的临床资料[7],其中男258 例、女139 例,中位年龄67 岁(30~88 岁)。397 例患者中,356 例临床病理资料完整。将397 例患者采用随机抽样法按7∶3 比例分为训练集(278例)和测试集(119例);训练集用于构建预测模型,测试集用于验证模型效能。
纳入标准:①病理学确诊为胃癌。②具有miRNA 的样本测序数据。③具有预后信息及临床资料。排除标准:①正常胃组织样本信息。②转移性胃癌样本信息。③生存时间<10 d。
1.2 研究方法
1.2.1 差异miRNA 筛选 利用美国国立生物技术信息中心(National Center for Biotechnology Information,NCBI)平台下的GEO 数据库检索含有人源胃癌及癌旁组织样本的芯片GSE93415。该芯片共包含40 例组织样本,其中20 例为癌组织,20 例为癌旁正常组织。通过GEO Datasets 中GEO2R 在线分析癌组织和癌旁正常组织差异表达的miRNA,并筛选出候选差异表达miRNA[8]。
1.2.2 预后相关miRNA 模型的构建 使用R 4.0.3 中的DESeq2 包对TCGA 样本的基因表达数据进行标准化[9]。为了提高预测模型的准确性,首先在候选差异表达miRNA 中过滤掉低表达的miRNA,过滤条件为在397 个TCGA 胃癌样本中标准化后的miRNA 表达量之和>100。基于训练集患者信息,利用R 4.0.3 中的glmnet 包进行LASSO 回归分析,将候选差异表达miRNA 拟合成一个相对精简的预后相关miRNA模型[10-11]。
1.2.3 预后相关miRNA 模型的验证 分别在训练集和测试集中对构建的预后相关miRNA 模型的预测效能进行验证,用生存曲线和列线图展示[12-13]。
1.2.4 影响胃癌患者预后的相关因素分析 选取临床病理资料完整的356例胃癌患者的临床病理资料,分析年龄、性别、肿瘤病理分期、T分期、N分期、M分期以及预后相关miRNA模型评分与胃癌患者预后之间的相关性。
1.2.5 预后相关miRNA 模型与临床病理因素预测效能比较 在临床病理资料完整的数据集中比较构建的预后相关miRNA 模型与临床病理因素对胃癌患者5 年总体生存率的预测效能。
1.2.6 对化学治疗药物敏感度的预测 利用pRRophetic包中的算法和肿瘤样本的基因表达数据,估算患者对于不同化学治疗(化疗)药物的敏感度[14]。
1.2.7 预后相关恩诺图的构建和验证 使用多因素Cox回归的方法,将预后相关miRNA模型评分与患者年龄、性别以及肿瘤病理分期综合起来,构建一个相对准确的量化的恩诺图。使用Calibration曲线验证恩诺图的预测效果;其中45°虚线为参考线,表示预测的总体生存率与实际总体生存率完全一致;曲线越接近参考线,预测效果越好。
1.3 观察指标和评价标准
1.3.1 观察指标 ①训练集和测试集患者的临床资料和生存情况。②预后相关miRNA 模型的构建:筛选与生存相关的miRNA,构建预后相关miRNA 模型方程。③预后相关miRNA 模型的验证:在训练集和测试集中的预测效能。④影响胃癌患者预后的相关因素分析:年龄、性别、肿瘤病理分期、T 分期、N 分期、M 分期、预后相关miRNA 模型评分。⑤预后相关miRNA 模型与临床病理因素预测效能的比较:在356例临床病理资料完整的数据集中的预测效能。
1.3.2 评价标准 差异表达miRNA 筛选标准为在癌组织及癌旁组织中差异表达倍数>2 且矫正后的P值<0.05[15]。以Log-rank 检验进行生存分析,验证模型的可靠性;以受试者操作特征曲线(receiver operating characteristic,ROC)曲线下面积(area under curve,AUC)分析模型的准确性,AUC值越接近1,模型的预测效能越好[13]。
1.4 统计学分析
应用R 4.0.3 统计软件进行数据分析。正态分布的数据以xˉ±s 表示,2 组间比较采用Student-t检验。偏态分布的数据以M(P25~P75) 表示,组间比较采用Mann-WhitneyU检验。定性资料以绝对数或百分比表示,组间比较采用χ2检验。采用Kaplan-Meier法绘制生存曲线并计算生存率,采用Log-Rank 方法比较2 组患者的生存状况。P<0.05表示差异有统计学意义。
2 结果
2.1 患者基本资料及生存情况
2组患者性别、年龄、肿瘤分期、肿瘤位置、生存状态、生存时间、种族、是否接受化疗、是否接受放射治疗等一般资料比较,差异均无统计学意义(P>0.05),具有可比性,见表1。

表1 训练集与测试集患者一般资料比较Tab 1 Baseline of patients in the training and validation cohorts
278 例训练集患者随访时间为14~3 519 d,中位随访时间为495 d,1、3、5 年总体生存率分别为77.5%、49.1%、40.6%。119例测试集患者随访时间为16~3 720 d,中位随访时间为398 d,1、3、5 年总体生存率分别为73.9%、43.1%、29.7%。2 组患者总体生存率比较,差异无统计学意义(P=0.250),见图1。
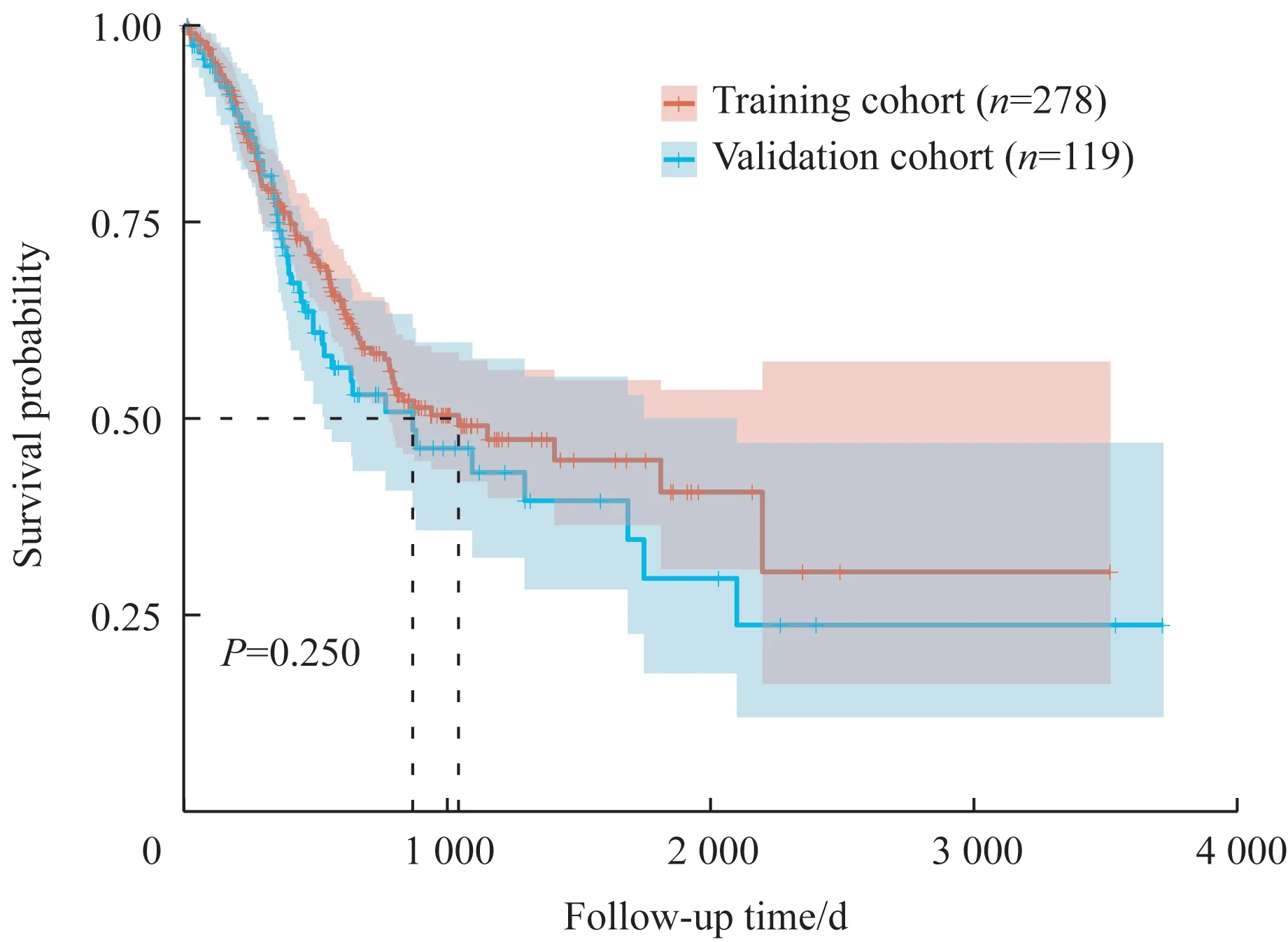
图1 278例训练集和119例测试集胃癌患者的生存曲线Fig 1 Survival curves of patients with gastric cancer in the training and validation cohorts
2.2 差异表达miRNA筛选结果
生物信息学分析结果显示:从GSE93415测序数据集中计算得到111 个候选差异表达miRNA,其中20 个在癌组织中上调,91个在癌组织中下调。对111个候选差异表达miRNA进行过滤后,得到59个候选差异表达miRNA。
2.3 预后相关miRNA模型的构建
对59个候选差异表达miRNA 和训练集样本数据进行LASSO 回归分析,采用10 折交叉验证法进行迭代分析。结果显示,当变量数为5时,模型均方根误差最小,其对应的λ=0.062。见图2A、B。筛选出的5个miRNA 分别为let-7i-5p、 let-7f-5p、 miR-708-5p、 miR-135b-5p、 miR-100-5p,差异表达模式(癌组织对比癌旁组织)均为降低,差异表达倍数分别2.55、2.78、2.17、3.08、3.26 倍。COX 回归分析结果显示miR-708-5p 为独立的危险因素,miR-135b-5p 为独立的保护因素,见图2C。通过分析这5个miRNA 的表达量和胃癌患者的生存数据,发现胃癌样本中高表达let-7f-5p、hsa-let-7i-5p 和hsa-miR-135b-5p 以及低表达hsa-miR-100-5p 和hsa-miR-708-5p 的患者的总体生存率较好,见图2D。由上述5 个miRNA 构建的预后表达方程:风险分数=(-0.049×let-7i-5p 表达量-0.033 2×let-7f-5p 表达量+0.202 9×miR-708-5p 表达量-0.088 9×miR-135b-5p表达量+0.016 3×miR-100-5p表达量)。

图2 LASSO-Cox回归分析筛选miRNAsFig 2 miRNAs screened by Lasso-Cox regression analysis
利用该预后表达方程对训练集278例样本数据计算模型评分,以风险评分的中位数为截断值,将患者分为高风险组(预后相关miRNA 模型评分>评分中位数)和低风险组(预后相关miRNA 模型评分≤评分中位数),见图3A、B。训练组患者的5 个miRNA 表达情况见图3C。对训练集中的高、低风险组患者进行Kaplan-Meier 生存分析,高风险组患者的总体生存率较低,差异有统计学意义(P=0.021),见图3D。利用该预后表达方程对训练集278 例样本数据进行时间依赖ROC 分析,结果显示,该预后相关miRNA 模型的1 年、3 年、5 年生存时间预测概率的AUC 分别为0.640、0.763、0.853,提示该模型对胃癌患者的预后具有一定的区分预测能力,见图3E。
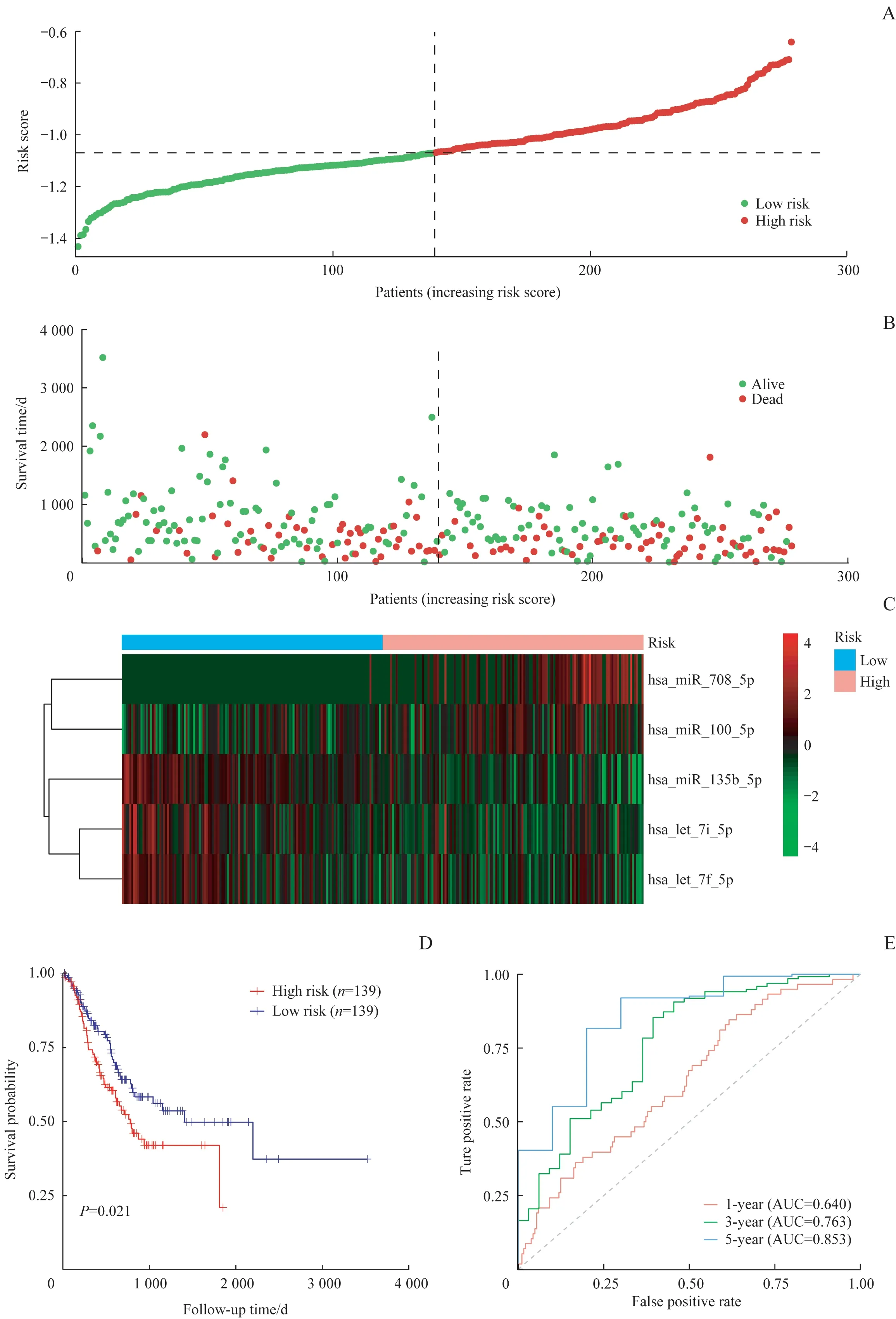
图3 训练集预后相关miRNA模型评分、时间依赖ROC分析以及Kaplan-Meier生存分析Fig 3 Prognostic miRNA model score,time-dependent ROC analysis,and Kaplan-Meier survival analysis of the training cohort
2.4 预后相关miRNA预测效能检验
为了进一步检验上述预后相关miRNA 模型的预测效能,利用测试集的119 例胃癌样本数据进行Kaplan-Meier生存分析,结果显示高风险组患者的总体生存率较低,差异有统计学意义(P=0.016)。利用测试集119例胃癌样本数据进行时间依赖ROC 分析,结果显示该预后相关miRNA 模型的1年、3年、5年生存时间预测概率AUC 分别为0.631、0.735、0.750,提示该模型对胃癌患者的生存状态和预后具有一定的区分预测能力(图4)。

图4 测试集预后相关miRNA模型评分、时间依赖ROC分析以及Kaplan-Meier生存分析Fig 4 Prognostic miRNA model score,time-dependent ROC analysis,and Kaplan-Meier survival analysis of the validation cohort
2.5 影响胃癌患者预后的相关因素分析
单因素COX 回归分析结果显示:年龄、肿瘤病理分期、T 分期、N 分期、M 分期和预后相关miRNA 模型评分是胃癌患者预后的相关因素(P<0.05),性别不是胃癌患者预后的相关因素(P>0.05),见图5A。多因素分析结果显示:年龄、M 分期和预后相关miRNA 模型评分是胃癌患者预后的独立危险因素(P<0.05),见图5B。在356 例临床资料完整的胃癌患者中,预后相关miRNA 模型对胃癌患者5 年生存时间预测的AUC 值为0.818,高于年龄(AUC=0.619)、性别(AUC=0.500)、肿瘤病理分期(AUC=0.574)、T 分期(AUC=0.726)、N 分期(AUC=0.602)、M分期(AUC=0.505)的预测效能,见图5C。
2.6 利用预后相关miRNA模型预测胃癌患者对于化疗药物的敏感度
利用pRRophetic 包和胃癌患者的基因表达数据计算胃癌患者对于一些常规化疗药物如阿霉素、丝裂霉素C、多西他赛、顺铂和紫杉醇的敏感度。结果显示,低风险组患者对于顺铂的敏感度明显高于高风险组患者(P=0.030),见图5D。

图5 影响胃癌患者预后的相关因素分析Fig 5 Analysis of prognostic factors in patients with gastric cancer
2.7 建立并评估与胃癌相关的预后恩诺图
综合预后相关miRNA 模型评分、年龄、性别和肿瘤病理分期等因素,建立预后相关恩诺图(图6A)。为了评估恩诺图预测胃癌患者生存率的准确性,采用calibration曲线进行验证(图6B)。图中45°虚线为理想曲线,表明恩诺图所预测的胃癌患者的总体生存率与患者实际的总体生存率一致。由图可知,恩诺图所预测的胃癌患者的1年和3 年总体生存率与实际总体生存率非常接近。此外,计算恩诺图、患者年龄、性别和肿瘤病理分期的一致性指数(consistency index,C-index)。通过比较,发现恩诺图的C-index 最高(图6C)。经过决策曲线分析(decision curve analysis, DCA)发现,与患者的年龄、性别和肿瘤病理分期相比,患者借助于恩诺图的辅助进行临床决策能够受益更多(图6D)。

图6 预后相关恩诺图的建立及评估Fig 6 Construction and evaluation of the prognostic nomogram
3 讨论
随着高通量测序技术的兴起,基因组、转录组及蛋白质代谢组等多组学联合分析逐渐成为时下热门的生物研究领域。在转录组学中,miRNA 调控网络在疾病的发生、发展中起到重要的作用。miRNA 是一类非编码内源性小RNA,可在转录后水平调节靶基因的表达。miRNA表达变化或miRNA 功能障碍可能影响肿瘤的发生和发展。Ke 等[16]研究表明miRNA-16 的低表达与肺癌的发生、发展有关,过表达miRNA-16可抑制非小细胞肺癌细胞的侵袭和迁移能力。另有研究[17-20]表明,miRNA-125家族与多种肿瘤的发生有密切关系。在结直肠癌、卵巢癌、肺癌和髓母细胞瘤等肿瘤中,miR-125表达较正常组织降低;而在前列腺癌、胰腺癌等肿瘤中,也发现其不同程度的上调。据报道[21],胃癌组织中miRNA 表达异常,提示miRNA 也参与了胃癌的发生。miR-21 通过作用于PTEN、PDCD4等基因,改变了细胞的增殖与侵袭能力[22];miR-130b通过靶向BIM和RUNX3,调控细胞的凋亡,介导胃癌的发生[23]。这些发现提示,miRNA 在胃癌的临床诊断、治疗选择、预后判断及复发预测等方面,有望成为新的分子标志物。
胃癌是一种上皮来源的恶性肿瘤,起源于胃壁最浅表的黏膜上皮细胞,发病率和病死率均较高。然而,胃癌的早期临床特征不明显,与胃溃疡等慢性疾病的症状相似;因此,在疾病早期易被忽略,往往很难得到有效、及时的治疗。目前,临床上广泛应用的胃癌相关的分子标志物有CEA、CA19-9、CA72-4等;然而,该肿瘤标志物的特异度较低,且不具备判断预后的能力。因此,临床上迫切需要开发新的胃癌生物标志物,有助于早期诊断和判断预后,辅助制定个性化的治疗方案。
本研究旨在通过高通量测序结合生物信息学的方法,筛选出与生存相关的miRNA,构建胃癌患者预后相关miRNA 预测模型。在本研究中,通过下载分析GEO 数据库的GSE93415 芯片,筛选出与预后生存显著相关的5 个miRNA,分别为let-7i-5p、let-7f-5p、miR-708-5p、miR-135b-5p、miR-100-5p。虽然这5 个miRNA 在肿瘤组织中的表达均低于正常水平,但通过生存分析发现,胃癌样本中高表达hsa-let-7f-5p、hsa-let-7i-5p、hsa-miR-135b-5p和低表达hsa-miR-100-5p、hsa-miR-708-5p 的患者总体生存状况较好。利用TCGA 数据库中的患者临床及生物学信息,最终构建并验证胃癌患者预后相关miRNA 预测模型。该模型的效能在测试集中得到了很好的验证,预后相关miRNA 模型的1 年、3 年、5 年生存时间预测概率AUC 值分别为0.631、0.735、0.750,提示该模型具较好的敏感度和特异度。为了更好地将构建的预后相关miRNA 模型应用于临床,本研究综合了预后相关miRNA模型评分以及患者年龄、性别和临床病理分期,建立了一个相对准确且量化的预后相关恩诺图。通过calibration曲线的验证和C-index 的比较,发现预后相关恩诺图相对于其他因素具有较高的预测准确度。通过DCA,发现借助于预后相关恩诺图进行临床决策,患者将会受益更多。因此,本研究所构建的预后相关恩诺图具有良好的应用价值。综合以上结果,提示该模型具有一定优越性。并且,相较于TNM 分期,该模型可从上游分子生物学的角度,从多维度更精准地判断预后。
本研究构建的胃癌预后预测模型对于胃癌患者的预后判断有良好的预测价值,但仍存在一定的局限性。首先,该模型的训练集及测试集均来自TCGA 数据库中的同一队列。若设置独立的外部验证,尤其是中国胃癌患者的验证,可提升该模型的可信度。其次,TCGA作为常用的公共数据库,其患者以白种人为主,缺少亚洲人,尤其是中国人的临床数据。有待在今后的研究中加入中国胃癌患者的生存预后数据,对该模型进行进一步的预测验证。
综上,本研究通过筛选出的5 个胃癌相关miRNA,构建了可用于胃癌患者生存预测的miRNA 模型。该模型具良好的敏感度和特异度,一定程度上弥补了形态学判断预后的不足,有助于指导临床上制定个体化治疗方案。
