基于阿霉素治疗的胃癌中lncRNA表达谱的微阵列分析
2021-12-30刘子倩李哲王胤曾焕玉李培峰
刘子倩 李哲 王胤 曾焕玉 李培峰
摘要:
为了研究药物阿霉素作用前后胃癌细胞中lncRNA表达的变化,采用微阵列技术分析AGS细胞中lncRNA和mRNA的基因表达谱,运用基因本体(GO)分析和KEGG分析探究基因表达功能,通过实时定量PCR实验对添加DOX前后AGS细胞中具有功能的lncRNA表达水平进行分析。研究结果显示,添加DOX后的AGS细胞lncRNA和mRNA的表达出现显著异常,这与多种生物过程、细胞成分和分子功能有关。
关键词:
胃癌,长链非编码RNA,阿霉素,微阵列分析,实时定量PCR
中图分类号: R735.2
文献标志码:A
收稿日期:2021-04-19
基金項目:
国家自然科学基金海外青年基金(批准号:82050410450、81850410551)资助;山东省自然科学基金(批准号ZR2019BH014)资助。
通信作者:
曾焕玉(AUNG Lynn Htet Htet),女,博士,主要研究方向为利用高通量测序,生物信息学,细胞生物学方法研究衰老及心脑血管疾病的机理机制;李培峰,男,博士,教授,主要研究方向为神经元衰老、自由基生物医学、细胞凋亡与心血管疾病及其分子机制的研究。E-mail:peifli@qdu.edu.cn
癌症的发病率和死亡率逐年上升[1],已严重影响到人们的身体健康,目前还没有行之有效的治愈方式[2]。胃癌常发生于胃肠道系统中,是世界上最常见的六种恶性肿瘤之一,死亡率位列第二,仅排在肺癌之后[3-4]。胃癌组织中发现了许多异常表达的长链非编码RNA(long non-coding RNA,lncRNA)[5],并与癌细胞的增殖和转移密切相关[6]。人类基因组中有关蛋白质编码的基因约20 000 个,但仅占总基因组的2%[7],而大多数其他基因被转录生成非编码RNA(ncRNA)[8]。lncRNA是ncRNA中一个独立的分支,是一种非蛋白质编码RNA[9],长度超过200 个核苷酸[10]。随着高通量测序等生物信息学技术的发展,lncRNA被发现在细胞周期调控、X染色体失活、细胞分化等包括胃癌在内的许多疾病的生物学过程中发挥着重要作用[11-12]。越来越多的研究发现lncRNA在某些疾病中可能是一个新的生物标志物或潜在的治疗位点[13]。阿霉素(doxorubicin,DOX)是一种广谱抗癌药,属于蒽环类药物[14],于20世纪60年代从链霉菌[15]中分离出来,主要用于急性白血病、恶性淋巴瘤、胃癌、乳腺癌等多种癌症的化疗[16]。DOX可以插入DNA并抑制拓扑异构酶II,最终破坏双链DNA结构,阻碍蛋白的合成[17-18]。lncRNA在胃癌中的研究虽已有报道,但机制尚不清楚。本研究主要运用生物信息学分析方法,通过对测序数据的分析得到胃癌中差异表达的lncRNA图谱,总结lncRNA的作用通路图,最终筛选出上调差异最大的lncRNA。
1 材料与方法
1.1 细胞培养与收集处理
(1)实验中所用到的AGS细胞来自中国科学院细胞研究所。在进行微阵列芯片分析实验时,实验组AGS细胞用2 μmol DOX处理6 h后收集;(2)AGS细胞培养基成分为:10 %胎牛血清(FBS)和1 %双抗的RPMI-1640培养基;(3)培养箱环境为37 ℃,95 %空气和5 %二氧化碳;(4)细胞的传代采用胰酶法,用磷酸盐缓冲盐水(PBS)洗涤细胞后,用025 %含EDTA的胰酶消化处理,随后用新的培养基重悬细胞分盘培养。
1.2 RNA提取、逆转录及qRT-PCR
1.2.1 RNA提取 在RNA提取实验中,为避免RNA发生降解,整个实验耗材需使用无RNA酶,过程需在冰上进行。实验前提前将低温离心机冷却至4 ℃。使用 Trizol 法提取 RNA:(1)在每盘细胞样品中加入1 mL Trizol试剂,冰上静置5 min,收集细胞至15 mL EP管中,冰浴5~10 min;(2)向步骤(1)的EP管中加入200 μL氯仿,用力上下震荡15 s后,在冰上静置10 min;(3)4 ℃,12 000 rpm对步骤(2)中样品离心15 min,轻轻取出,上层无色透明水相为RNA;(4)用移液枪将上层水样吸取到新的EP管中,加入等体积的异丙醇,轻微震荡混匀,冰上静置10 min,4 ℃,12 000 rpm离心10 min;(5)加入预冷过的75 %乙醇清洗RNA 2次,4 ℃,12 000 rpm离心5 min,去除上清留底部沉淀;(6)干燥2 min,加入20 μL DEPC水溶解,-80 ℃冷冻保存。采用Nanodrop浓度检测仪测量并记录RNA样品A260 nm/A280 nm的比值,当A260 nm/A280 nm在18~20范围内时,提取的RNA纯度合格,否则RNA可能有污染。
1.2.2 RNA逆转录实验 (1)在无RNA酶的200 μL tube中加入2 μL的5×DNA Eraser buffer、1 μL的DNA Eraser、RNA总量不超过1 μg的样本,若总体系不够10 μL用DEPC水补齐,混合均匀后在PCR仪中运行42 ℃,2 min程序;(2)在步骤(1)管中加入Primer mix和Enzyme mix各1 μL、4 μL的Buffer 2,4 μL DEPC水使得管内总体系为20 μL,涡旋混匀后瞬离;(3)在PCR仪中运行程序:37 ℃反应15 min,85 ℃反应5 s,降温至4 ℃停止反应;(5)反应产物-20 ℃保存。
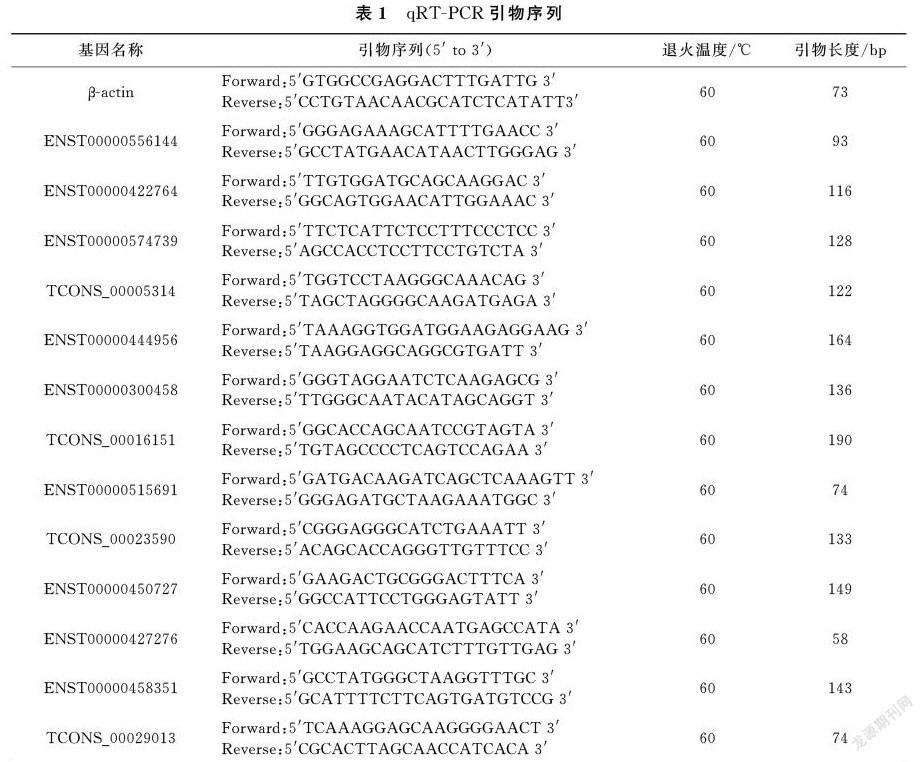
1.2.3 qRT-PCR实验 (1)qRT-PCR引物设计:qRT-PCR实验中所用到的引物信息如表1所示,β-actin基因用作对照。
(2)反应成分:每个反应孔内加2×SYBR 10 μL,Forward/Reverse引物各04 μL,cDNA样本1 μL,水82 μL,总体系20 μL。每个样品设置三个重复孔实验。
(3)qRT-PCR反应条件共分三步:95 ℃预变性30 s;95 ℃反应5 s,60 ℃反应1 min,循环35次;95 ℃反应15 s,60 ℃保温1 min。
(4)相对表达量分析:扩增实验结束后,仪器Bio-Rad CFX manager 31自动生成扩增曲线,随后进行数据处理分析。
1.3 微阵列分析
采用安捷伦阵列平台对AGS细胞和DOX处理6 h的AGS细胞进行微阵列分析,从中提取RNA后再纯化出mRNA,用随机引物法扩增每个样本,沿转录本全长转录成无3′端偏差的荧光cRNA。然后与人类lncRNA阵列v30杂交(8×60K, Arraystar),清洗载玻片后用安捷伦扫描仪扫描阵列。对所得原始数据进行分位數归一化,标记出对照组和DOX组的lncRNA和mRNA。筛选二者之间的改变倍数,找出有统计学意义的差异表达lncRNA和mRNA。至少发生2倍倍数变化的lncRNA才能被筛选和鉴定出来(P≤005)。
1.4 编码基因功能分析
对筛选出的lncRNA和mRNA进行GO和KEGG通路分析。GO分析将lncRNA在AGS细胞中从生物学过程(Biological Process,BP)、细胞成分(Cellular Component,CC)和分子功能(Molecular Function,MF)三个相关方面进行解释。KEGG通路分析对lncRNA生物学路径进行富集,使得lncRNA的生物学功能图像化。
1.5 统计学方法
运用GraphPad 8统计软件进行数据分析,单因素变量两组之间的比较用t检验统计,多组间均数的比较则用方差分析。*P<005被认为差异具有统计学意义。
2 实验结果
2.1 胃癌细胞中lncRNA和mRNA表达谱
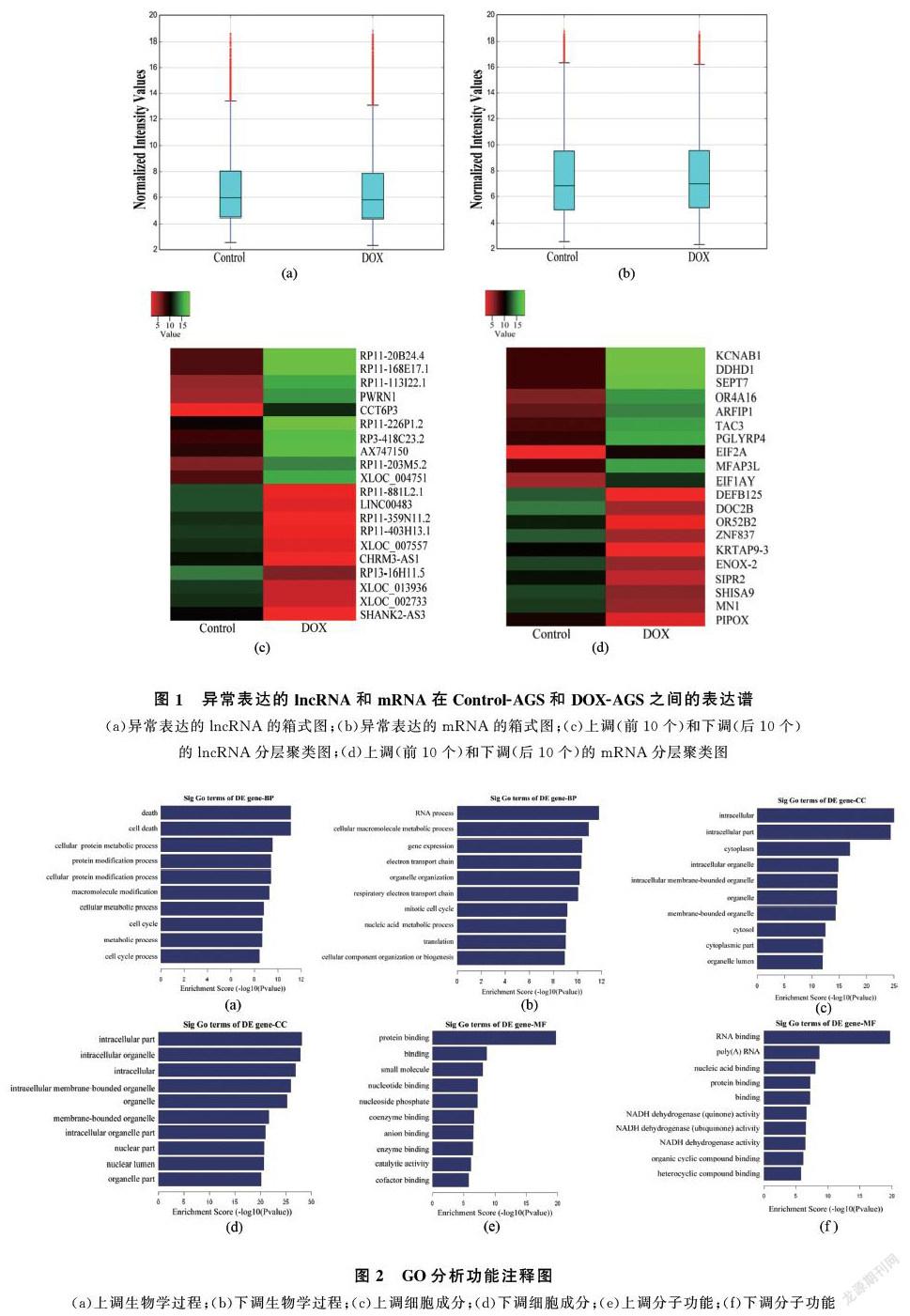
采用对照组AGS细胞和实验组DOX处理6 h后的AGS细胞,提取RNA,进行微阵列芯片分析。结果发现与AGS细胞相比,DOX处理6 h后的AGS细胞中出现异常表达的lncRNA共有10 981个,mRNA共有8 294个(fold change≥2,P≤005),如图1所示。这表明DOX的加入使得胃癌细胞中lncRNA和mRNA发生了异常变化,其中,上调差异最大的是RP11-20B244(上调了1 462689 266倍),而下调最明显的lncRNA是RP11-881L21(下调了1 131864 075倍)。
2.2 GO分析
本实验的GO分析体现了AGS细胞中生物学过程、蛋白的细胞定位和分子功能方面的情况(P≤005)。由图2可知,上调的lncRNA主要在细胞死亡、细胞质成分、蛋白质结合方面发挥作用。下调的lncRNA主要在RNA加工、胞内成分、RNA结合方面发挥作用。
2.3 KEGG通路分析
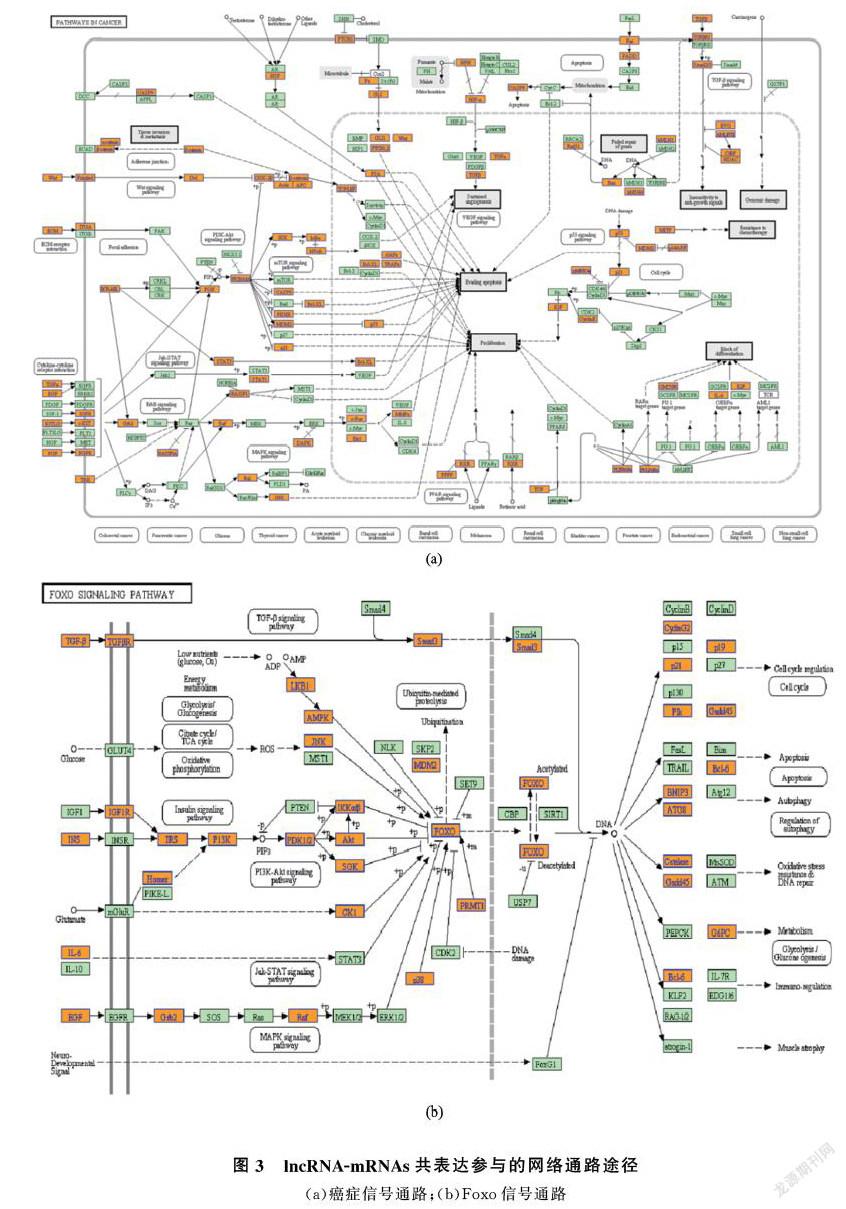
分子间相互作用、反应和关系网络的通路图如图3所示。可知,与lncRNA共表达的mRNA参与了肿瘤通路的调控包括:细胞周期、细胞凋亡、转化生长因子-β信号通路、p53信号通路、VEGF信号通路、PPAR信号通路、Wnt信号通路、PI3K-Akt信号通路、cytokine-cytokine受体相互作用、MAPK信号通路、mTOR信号通路、Jak-STAT信号通路、ErbB信号通路和ECM受体相互作用等。图3(b)提示lncRNA很有可能通过下游蛋白发挥作用。
2.4 qRT-PCR验证筛选差异表达最大的lncRNA
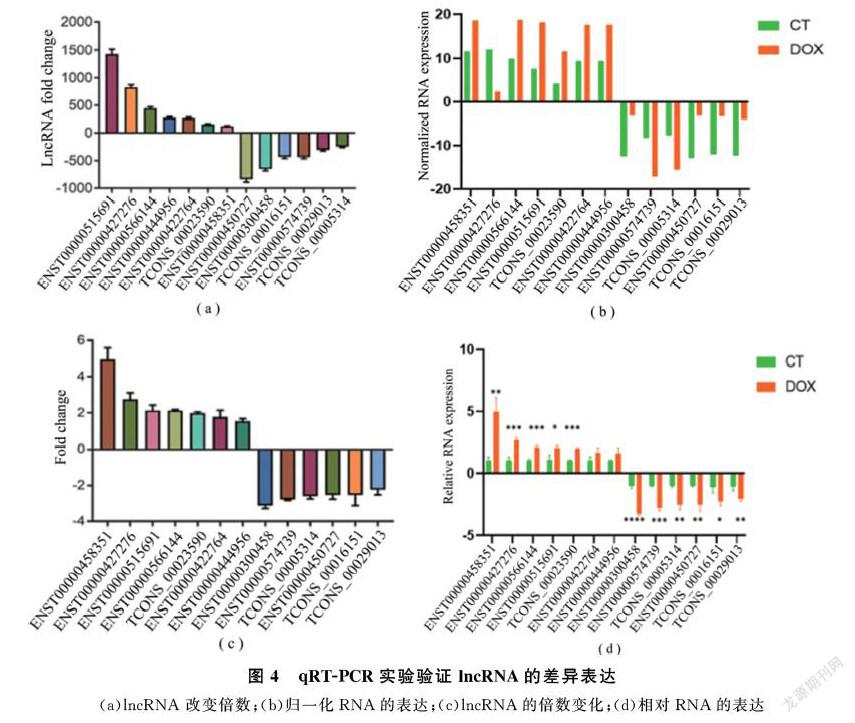
通过qRT-PCR实验验证lncRNA结果在微阵列分析中的准确性,筛选差异表达最大的lncRNA。基于前期对所有测序所得lncRNA数据的功能性分析,选取13个在功能上上调和下调变化较大的lncRNA进行qRT-PCR实验验证。由图4可知,lncRNA ENST00000515691、ENST00000427276、ENST00000556144、ENST000000444956、ENST000000422764、TCONS_00023590和ENST00000458351表达上调,而lncRNA ENST00000450727、ENST00000300458、TCONS_00016151、ENST00000574739、TCONS_00029013和TCONS_00005314的表达下调,这与测序所得数据一致。图4(d)表明所有验证的异常表达的lncRNA在DOX组与对照组之间的差异,证实了微阵列分析的准确性和重复性,实验数据由三次以上实验的平均值计算所得。
3 讨论
胃癌具有高度的异质性和恶性[19],早在2012 年就有数据记载,70% 以上的新增胃癌病例发生在亚洲、中欧、东欧和拉丁美洲等地的发展中国家[20-22]。近年来,lncRNA已由最初的“无功能副产物”[23],逐渐转变为有可研究意义的“明星分子”[24]。因此,研究胃癌与lncRNA之间调控关系显得格外重要。
本研究利用lncRNA能参与许多病理过程[25]和DOX能致使相关的癌细胞凋亡增加的特点[26-27],来分析DOX作用前后的AGS细胞lncRNA和mRNA的表达差异,鉴定出10 981个异常表达的lncRNA,其中5 673个lncRNA表达上调,5 308个lncRNA表达下调。还有8 294个异常表达的mRNA,其中有4 778个mRNA表达上调,3 516个mRNA表达下调。GO 和 KEGG 富集分析显示失调的lncRNA可能在细胞后续的生命过程中发挥作用,提示胃癌的发生发展可能与lncRNA异常表达有关[28]。有研究表明lncRNA TUG1在乳腺癌组织中高表达,构建敲除TUG1的siRNA表达载体可以调节乳腺癌细胞对DOX的敏感性[29-30]。对比本研究筛选出的差异表达最明显的lncRNA ENST00000458351,DOX处理后lncRNA ENST00000458351表达升高且与对照组之间差异明显,提示可以从lncRNA ENST00000458351入手展开对胃癌中lncRNA调控变化的研究,构建敲除lncRNA ENST00000458351的siRNA表达载体,以期发现新的胃癌诊断生物标志物。
4 结论
本研究通过微阵列技术分析,运用生物信息学方法对DOX处理的胃癌细胞进行全面系统分析,筛选出13个具有功能差异的lncRNA。这些lncRNA可能是胃癌潜在的治疗靶点,其中上调最明显的是lncRNA ENST00000458351,今后将重点研究lncRNA的调控机制。
参考文献
[1]TORRE L A, SIEGEL R L, WARD E M, et al. Global cancer incidence and mortality rates and trends-an update[J]. Cancer Epidemiology Biomarkers & Prevention, 2016, 25(1): 16-27.
[2]KENTSIS A. Why do young people get cancer?[J]. Pediatric Blood & Cancer, 2020, 67(7): e28335.
[3]KOBAYASHI D, KODERA Y. Intraperitoneal chemotherapy for gastric cancer with peritoneal metastasis[J]. Gastric Cancer, 2017, 20: 111-121.
[4]LI B F, HU X C. Time-varying effects of marital status on gastric cancer: A population-based study[J]. Cancer Management and Research, 2019, 11: 10949-10955.
[5]CHEN Y, ZHANG K D, YU Z L, et al. Long intergenic non-coding RNA (lincRNA)-01317 suppresses human gastric cancer growth by inhibiting migration and invasion of cancer cells[J]. American Journal of Translational Research, 2021, 13(2): 770-780.
[6]ZHANG S, YANG Y, HUANG S L, et al. SIRT1 inhibits gastric cancer proliferation and metastasis via STAT3/MMP-13 signaling[J]. Journal of Cellular Physiology, 2019, 234(9): 15395-15406.
[7]何增, 魏缨霁, 张久聪. 长非编码RNA在胃癌发病机制中相关作用的研究进展[J]. 生物技术通讯, 2016, 27(4): 586-591.
[8]XU Z Y, JI G H, CUI Y, et al. The impacts of non-coding RNAs and N6-methyladenosine on cancer: Past, present, and future[J/OL]. Current Cancer Drug Targets, 2021. [2021-01-20]. https://pubmed.ncbi.nlm.nih.gov/ 33475064/. DOI: 10.2174/1568009621999210120193636.
[9]惠毓坤. RNA研究的新進展[J]. 青岛大学学报(自然科学版), 2006, 19(2): 89-94.
[10] YAMASHITA A, SHICHINO Y, YAMAMOTO M. The long non-coding RNA world in yeasts[J]. Biochimica Et Biophysica Acta-Gene Regulatory Mechanisms, 2016, 1859(1): 147-154.
[11] NAGANO T, FRASER P. No-nonsense functions for long noncoding RNAs[J]. Cell, 2011, 145(2): 178-181.
[12] LI T W, MO X Y, FU L Y, et al. Molecular mechanisms of long noncoding RNAs on gastric cancer[J]. Oncotarget, 2016, 7(8): 8601-8612.
[13] GUTSCHNER T, DIEDERICHS S. The hallmarks of cancer: A long non-coding RNA point of view[J]. RNA Biology, 2012, 9(6): 703-719.
[14] CAPPETTA D, DE ANGELIS A, SAPIO L, et al. Oxidative stress and cellular response to doxorubicin: A common factor in the complex milieu of anthracycline cardiotoxicity[J]. Oxidative Medicine and Cellular Longevity, 2017, 2017: 1521020.
[15] ARCAMONE F, CASSINELLI G, FANTINI G, et al. Adriamycin, 14-hydroxydaunomycin, a new antitumor antibiotic from S. peucetius var. caesius[J]. Biotechnology and Bioengineering, 1969, 11(6): 1101-1110.
[16] SIMU。NEK T, STERBA M, POPELOV O, et al. Anthracycline-induced cardiotoxicity: Overview of studies examining the roles of oxidative stress and free cellular iron[J]. Pharmacological Reports, 2009, 61(1): 154-171.
[17] FERREIRA A, CUNHA-OLIVEIRA T, SIMOES R F, et al. Altered mitochondrial epigenetics associated with subchronic doxorubicin cardiotoxicity[J]. Toxicology, 2017, 390: 63-73.
[18] CHEN C, LU L, YAN S C, et al. Autophagy and doxorubicin resistance in cancer[J]. Anti-cancer Drugs, 2018, 29(1): 1-9.
[19] CAI J, NIU X, CHEN Y Y, et al. Emodin-induced generation of reactive oxygen species inhibits RhoA activation to sensitize gastric carcinoma cells to anoikis[J]. Neoplasia, 2008, 10(1): 41-51.
[20] FERLAY J, SOERJOMATARAM I, DIKSHIT R, et al. Cancer incidence and mortality worldwide: Sources, methods and major patterns in GLOBOCAN 2012[J]. International Journal of Cancer, 2015, 136(5): E359-E386.
[21] CHENG J, KAPRANOV P, DRENKOW J, et al. Transcriptional maps of 10 human chromosomes at 5-nucleotide resolution[J]. Science, 2005, 308(5725): 1149-1154.
[22] SANYAL A, LAJOIE B, JAIN G, et al. The long-range interaction landscape of gene promoters[J]. Nature, 2012, 489(7414): 109-113.
[23] STRUHL K. Transcriptional noise and the fidelity of initiation by RNA polymerase II[J]. Nature Structural & Molecular Biology, 2007, 14(2): 103-105.
[24] SUNITHA M C, DHANYAKRISHNAN R, PRAKASHKUMAR B, et al. p-Coumaric acid mediated protection of h9c2 cells from doxorubicin-induced cardiotoxicity: Involvement of augmented Nrf2 and autophagy[J]. Biomedicine & Pharmacotherapy, 2018, 102: 823-832.
[25] ZHOU Z X, LIN Z J, HE Y Q, et al. The long noncoding RNA D63785 regulates chemotherapy sensitivity in human gastric cancer by targeting miR-422a[J]. Molecular Therapy Nucleic Acids, 2018, 12: 405-419.
[26] BHAN A, SOLEIMANI M, MANDAL S S. Long noncoding RNA and cancer: A new paradigm[J]. Cancer Research, 2017, 77(15): 3965-3981.
[27] HAN T, HU H, ZHUO M, et al. Long non-coding RNA: An emerging paradigm of pancreatic cancer[J]. Current Molecular Medicine, 2016, 16(8): 702-709.
[28] KHANDELWAL A, BACOLLA A, VASQUEZ K M, et al. Long non-coding RNA: A new paradigm for lung cancer[J]. Molecular Carcinogenesis, 2015, 54(11): 1235-1251.
[29] 趙丹风, 钱冬萌, 王斌. 下调ATF5基因表达对神经胶质瘤细胞增殖及巨细胞病毒复制的影响[J]. 青岛大学学报(自然科学版), 2017, 30(1):51-55.
[30] WANG S Q, CHENG M J, ZHENG X X, et al. Interactions between lncRNA TUG1 and miR-9-5p modulate the resistance of breast cancer cells to doxorubicin by regulating eIF5A2[J]. Oncotargets and Therapy, 2020, 13: 13159-13170.
Abstract:
In order to study the changes of lncRNA expression in gastric cancer cells before and after DOX treatment, microarray analysis was used to analyze the gene expression profiles of lncRNA and mRNA in AGS cells. GO analysis and KEGG analysis were used to explore gene expression function. Quantitative real-time PCR (qRT-PCR) was used to analyze the expression levels of functional lncRNA in AGS cells before and after DOX addition. The results show that the expressions of lncRNA and mRNA in AGS cells are significantly abnormal after DOX addition. The occurrence of such abnormalities is associated with a variety of biological processes, cellular components, and molecular functions.
Keywords:
gastric cancer; long non-coding RNA; doxorubicin; microarray analysis; qRT-PCR
