重组DNA分子模型的再设计
2021-12-29张艳娟
张艳娟

摘要 丰富重组DNA分子模型的内涵,突破目的基因正连和倒连、单酶切和双酶切差异等教学难点,引导深度学习。
关键词 重组DNA分子 模型 深度学习
中图分类号 G633.91
文献标志码 B
文献标志码 B
“DNA重组技术的基本工具”是人教版高中生物选修3专题1第一节的内容,教材给出了重组DNA分子的模拟制作活动建议,也有教师尝试过展板模型。模型直观模拟了分子水平的操作,带给学生“边做边学”的生动体验。然而,由于模型设计的局限性还有一些难点有待突破,如限制酶如何选择、目的基因正连和倒连、双酶切与单酶切的区别等。为此,笔者尝试对模型进行再设计,丰富模型内涵,引导深度学习。
1模型设计要点
1.1含目的基因的DNA片段模型
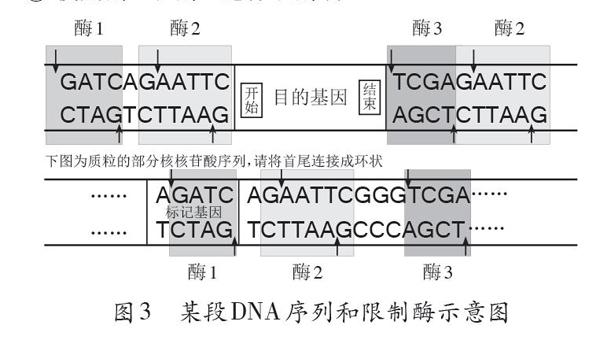
该模型有两个设计要点:1目的基因有方向性,反向连接会导致错误表达,所以用开始和结束表明方向(目的基因不一定带启动子和终止子,所以未标启动子和终止子);2获取目的基因时要既可用单酶切,又可用双酶切。所以,目的基因两侧各设计了一个G↓AATTC的识别序列,并在目的基因左右两侧分别增加↓GATC和↓TCGA的识别序列。值得注意的是目的基因左侧↓GATC和G↓AATTC之间要增加一个无关碱基对,否则两个酶切序列连起来中间会出现一个↓TCGA的识别序列,增加不必要的干扰;目的基因右侧↓TCGA要设计在G↓AATTC的左侧。这样,可以在G↓AATTC酶切后模型的基础上再切割↓TCGA位点。另外,如果↓TCGA在G↓AATTC的右侧,即使添加两种限制酶(酶2和酶3),目的基因两侧也只有酶2切割后产生的黏性末端。
1.2运载体模型
该模型模拟了最常用的运载体——质粒,模拟目标是既要体现单酶切和双酶切的区别,又要至少体现运载体具备的两个条件:具有1个至多个限制酶切割位点便于与外源基因连接、具有某些标记基因便于重组DNA的检测与鉴定。因此,质粒上设计了三种限制酶的识别序列G↓AATTC、↓GATC和↓TCGA,其中↓GATC的识别序列位于标记基因中。
2模型制作方法
根据教学需要设计了两种模型:分组活动模型和黑板展示模型。
分组活动模型(图1)以学案的形式印发给学生,确保每一位学生都能亲自动手操作。为了使目的基因与质粒的连接美观规范,两种DNA模型的宽度要相同、碱基大小和间距要一致。
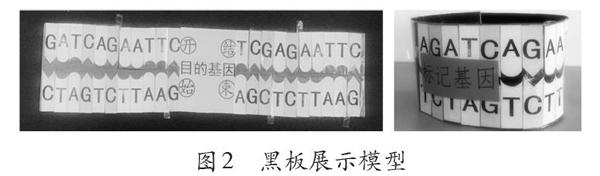
黑板展示模型(图2)的要求是字大醒目,可在黑板上吸附和移动,并能多次重复使用。该模型利用“不织布”制作,方便折叠,经久耐用。不织布的正面粘贴印有“A、T、C、G”四个字母的彩纸,代表四种脱氧核苷酸;背面粘贴软磁片,可吸附在黑板上。为了延长模型使用时间,彩纸上贴一层宽胶带。各限制酶的切口提前剪开,断开处用透明胶带粘起来,把透明胶带的一头对折,使其失去粘性,方便手持操作。因透明胶带粘在光滑的宽胶带表面,所以将透明胶带撕开、粘上的操作可多次重复。为了展示目的基因与目的基因相连和质粒与质粒相连,目的基因和质粒各做2个。
3模型使用策略
该模型构建活动适合在学生初步了解相关知识的基础上,进行深入拓展探究,所以安排在“DNA重组技术的基本工具”第1课时后。
3.1目的基因和质粒的再认识
为提高课堂效率,教师在课前下发印有模型的学案,请学生将目的基因所在DNA和质粒DNA沿实线剪下来,并将质粒首尾相连成环状备用。课上,在学生观察模型后,教师提出问题:1什么是目的基因?2目的基因的方向会不会影响其表达?3从DNA中分离出目的基因需具备几个限制酶酶切位点,水解几个磷酸二酯键?4目的基因所在DNA被切割后会断成几部分?5质粒的化学本质是什么?6质粒作为运载体必须具备哪些条件?7质粒被一种限制酶切割后会形成几个DNA片段,出现几个游离的磷酸基团?被两种限制酶切割呢?8如何区分不同长度的DNA片段以便找到所需的DNA片段?接着,补充介绍凝胶电泳技术,帮助学生理解限制酶切割后应对DNA片段进行分離和鉴定,以防止切割后的DNA片段再度连接。
3.2体验重组质粒的构建
教师展示图3,提出问题:如何选择获取目的基因和切割质粒的限制酶,切割后的末端应具备什么特点?学生分析可知,不能选用酶1,因为会破坏标记基因,从而确定两种可行方案:1使用酶2进行单酶切;2使用酶2和酶3进行双酶切。
为了更好地对比体验单酶切和双酶切的区别,学生先尝试用酶2切割目的基因所在DNA片段和质粒。教师提出问题,引导学生思考:假如借助凝胶电泳技术分离和纯化了目的基因片段,排除了目的基因两侧的DNA片段。现将很多目的基因和切割后的质粒放进一个容器,加入DNA连接酶后会产生多少种连接方式?学生两人一组将各自切割后得到的目的基因和质粒放在一起,模拟可能的连接方式。学生代表利用黑板展示模型汇报,可能的连接方式有6种:目的基因自身环化、质粒自身环化、目的基因和目的基因连接、运载体和运载体连接、目的基因和运载体连接(其中包括目的基因正连和倒连两种情况)。接着,启发学生思考:如何减少错误连接,提高转基因的成功率呢?学生自然会想到双酶切,接着分组模拟体验酶2和酶3切割后的DNA片段连接方式,分析双酶切(图4)比单酶切(图5)的优越性。双酶切排除了目的基因和质粒的自身环化现象,也避免了目的基因倒连,但仍存在目的基因和目的基因、运载体和运载体相连的情况,所以转基因后需进行目的基因的检测与鉴定。教师追问,启发学生思考:如果标记基因为氨苄青霉素抗性基因,如何鉴定目的基因是否导入?如果标记基因为氨苄青霉素抗性基因,酶3位于抗四环素基因中,如何鉴定目的基因是否导入?
4教学反思
该重组DNA分子模型补充设计了目的基因的方向性、从一种限制酶识别序列增加到三种限制酶识别序列,深入浅出地破解了目的基因正连和倒连、单酶切和双酶切的差异等难点问题,并将限制酶与凝胶电泳技术有机衔接,丰富模型内涵。教师创设了驱动深度学习的真实情境,促使学生主动参与中解决有挑战性的问题发展了科学思维。手脑并用的真实体验有利于模型建构内化为自觉的思维习惯和能力。
参考文献:
汪林静.设计展板模型突破基因工程的教学难点[J].生物学教学,2014,39(9):33.
