基于外部知识的药物相互作用关系抽取方法
2021-12-29刘宁宁
王 芳, 龙 欣, 周 刚, 刘宁宁
(1.四川省医学科学院四川省人民医院辅助生殖医学中心, 成都 610110; 2.四川大学计算机学院, 成都 610065)
1 引 言
药物和药物的相互作用(Drug-Drug Interactions,DDI)指的是多种药物被病人在同一时间服用时,不同药物之间产生的协同或拮抗等作用[1]. 这些作用所带来的副作用会导致治疗费用增加,严重的话,甚至会对患者的身体健康造成极大的威胁.因此了解药物和药物之间的相互作用知识对于患者的诊治和医学的发展有着非常重要的意义与价值. 现阶段已经提出的药物相互作用关系提取研究,在系统生物学、个性化医学等[2]许多生物医学领域具有重要的价值.
现阶段的学者们通常从DrugBank[3],Phar-mGKB[4]等医学数据库中获取药物相关知识.然而,调研发现,医学数据库只存储了少量DDI.相比而言,相关医学文献中记录的药物之间的相互作用信息更加丰富. 且现有医学数据库主要采用手动的方式更新数据库,即依靠医学专业人员从文献中手动抽取出DDI,这种方法需要的成本高且效率较低.因此,医学数据库的更新速度远远落后于文献的增长速度,导致无法充分利用生物医学文献中海量的医学资源. 药物相互作用关系抽取旨在自动从海量的医学文献中高效、准确地抽取出关系来更新医学数据库. 此外,药物相互作用知识在药物研发、辅助医生开药、构建医学知识图谱、更新医学数据库、预防药物的不良反应等应用中也发挥着巨大的作用. 自2011年和2013年先后发布的两个(DDIExtraction2011[5],DDIExtraction2013[6])对药物相互作用进行关系抽取的任务起,如何从文本中自动提取DDI逐渐成为研究热点. 目前在DDI抽取任务上,已经有许多成功应用的方法.
早期对于DDI的抽取通常使用基于规则的方法,其中规则的制定一般需要医学领域中专业人员的辅助.这种方法的召回率偏低,这是因为语言表达形式具有多样性,部分药物之间相互作用关系可能不能被制定的规则所覆盖. 随着机器学习技术的日益进步,药物相互作用关系开始更多地利用机器学习方法来进行抽取. 这类方法的抽取性能很大一部分取决于外部自然语言处理工具的效果.这是因为该方法需要用到大量由词性标注器、句法分析器等自然语言处理工具生成的如词性,句法,语法等人工定义的特征. 为了减少人工设计特征所耗费的成本且取得比传统方法更好的抽取效果,通过利用深度学习技术来自动学习特征并抽取DDI逐渐成为趋势.
本文提出了一种结合外部知识的药物关系抽取模型,针对药物相互作用数据集中存在的样本数目较少以及不同关系类别样本数目差异较大的问题,提出将外部药物知识融入关系抽取模型中. 首先需要对DrugBank药物数据库中存在的药物知识进行抽取,从中抽取出带有药物描述信息的药物相互作用对,以此构建带有药物描述信息的数据集. 然后通过在此数据集上进行训练,得到训练好的药物描述信息模型. 最后将该模型与基于注意力机制的药物相互作用关系抽取模型相结合,即为融入外部知识的药物相互作用关系抽取模型. 该模型可以有效利用药物数据库DrugBank中已有的知识,从而提高模型的抽取效果、缓解不同关系类别之间抽取结果差异过大的问题. 最后,本文模型的有效性也在数据集DDIExtraction2013上得到了验证.
本文的章节结构安排如下:第2节介绍关于药物相互作用关系抽取的相关工作;第3节中详细地描述并展示了本文提出的模型;第4节中通过利用DDIExtraction2013数据集对本文模型的有效性进行了综合验证,并与其他模型进行对比;第5节总结全文并提出了未来的发展方向.
2 相关工作
目前,该领域主要有三类方法被应用于提取药物和药物的相互作用关系,即基于规则的方法,基于传统机器学习的方法和基于深度学习的方法.
基于规则方法的重点在于如何从医学文本中找到合适的规则. 如Segura-Bedmar等[7]提出了一种混合方法来从生物医学文本中提取药物之间的关系,该方法结合了浅层分析和模式匹配,通过药剂师根据专业经验以及对语料库中描述DDI语法结构的分析观察,制定出一组特定的DDI抽取规则,最后在生成的简单句上应用该规则进行关系抽取. Blasco等[8]认为在医学文本中存在特定的描述DDI的语句形式,故采用Apriori算法来提取医学文本中最大频繁序列,然后利用该序列进行DDI的抽取. Santiago等[9]通过使用临床资料、科学文献和社交媒体的数据挖掘研究检测药物相互作用. 数据挖掘在DDI分析中具有重要应用:发现药物之间的不利影响,建立知识数据库、黄金标准以及抽取新的DDI.
基于传统机器学习方法的重心在于对各种特征的使用以及核函数的设计. 如Chowdhury等[10]通过从文本中提取出一组触发词特征、否定词特征、句法特征和词汇特征,然后使用支持向量机完成DDI的抽取. Zheng等[11]提出了一种新型的基于图的内核函数,该内核函数不仅计算顶点本身的属性,还计算了顶点的相邻属性,从而可以充分捕获语句的结构信息,提高了抽取效果. Zhang等[12]针对句子结构中存在的噪声问题,提出了一种图修剪方法,可以从原始句子结构中修剪明显的噪声信息,并强调相关的句法信息. 该方法可以更准确的计算和表示语法结构信息,在当时取得了最佳结果. Chowdhury等[13]提出了一种复合内核的方法,可以充分利用依赖特征、上下文特征以及全局特征等特征,效果比单个的核函数好.
近年来,利用深度神经网络来对药物关系进行抽取取得了较好的发展. Liu等[14]通过将单词嵌入和位置嵌入信息输入到卷积网络中,首次提出利用CNN模型来进行药物关系抽取.实验结果表明,该方法即使在不提取任何词性,句法等特征的基础上,也能取得较好的结果,不仅证明在解决药物关系抽取问题上深度学习方法是适用的,而且显示该方法相对于基规则和传统机器学习方法能取得更好的结果. 为了更好地对句子语义的表示进行获取,Kavuluru等[15]考虑到相比词级别的嵌入,字符级别的嵌入更适用于表示形态较为丰富的自然语言,首次将字符级别的循环神经网络应用到DDI抽取上. Zhou等[16]认为判断药物之间的关系,单词的位置信息有非常重要的作用,为了能更好地利用位置信息,他们提出将BiLSTM层产生的隐层状态与位置嵌入结合,从而生成位置感知注意力. Zhao等[17]首次使用图卷积网络编码语法图进行医学关系的提取,通过将句法知识建模为图卷积网络中的边,将单词作为节点,利用双向门控循环单元网络学习句子的序列特征,图卷积网络学习句法图表示的特征. 实验结果表明,双向门控循环单元网络和图卷积网络的结合能够进一步提高模型性能. Asada等[18]采用卷积神经网络和图卷积神经网络相结合的方法进行DDI抽取,该模型首先利用卷积神经网络对文本语句进行编码,然后利用图卷积神经网络对药物的分子结构信息进行编码,然后将二者的池化层相结合,生成最终的预测结果. 该方法能够有效利用药物分子结构信息. Zhang等[19]通过将卷积神经网络与循环神经网络相结合,取得了比单个模型更好的结果. Feng等[20]用图卷积网络和深度神经网络结合,提出了一种有效且鲁棒的方法,通过利用DDI网络信息而不是考虑药物特性来预测潜在的DDI. 该方法在其他与DDI相关的场景中也有用,例如药物组合指导、检测药物副作用等.
3 本文方法
本文提出了一种结合外部知识的药物关系抽取模型,模型整体流程图如图1所示.
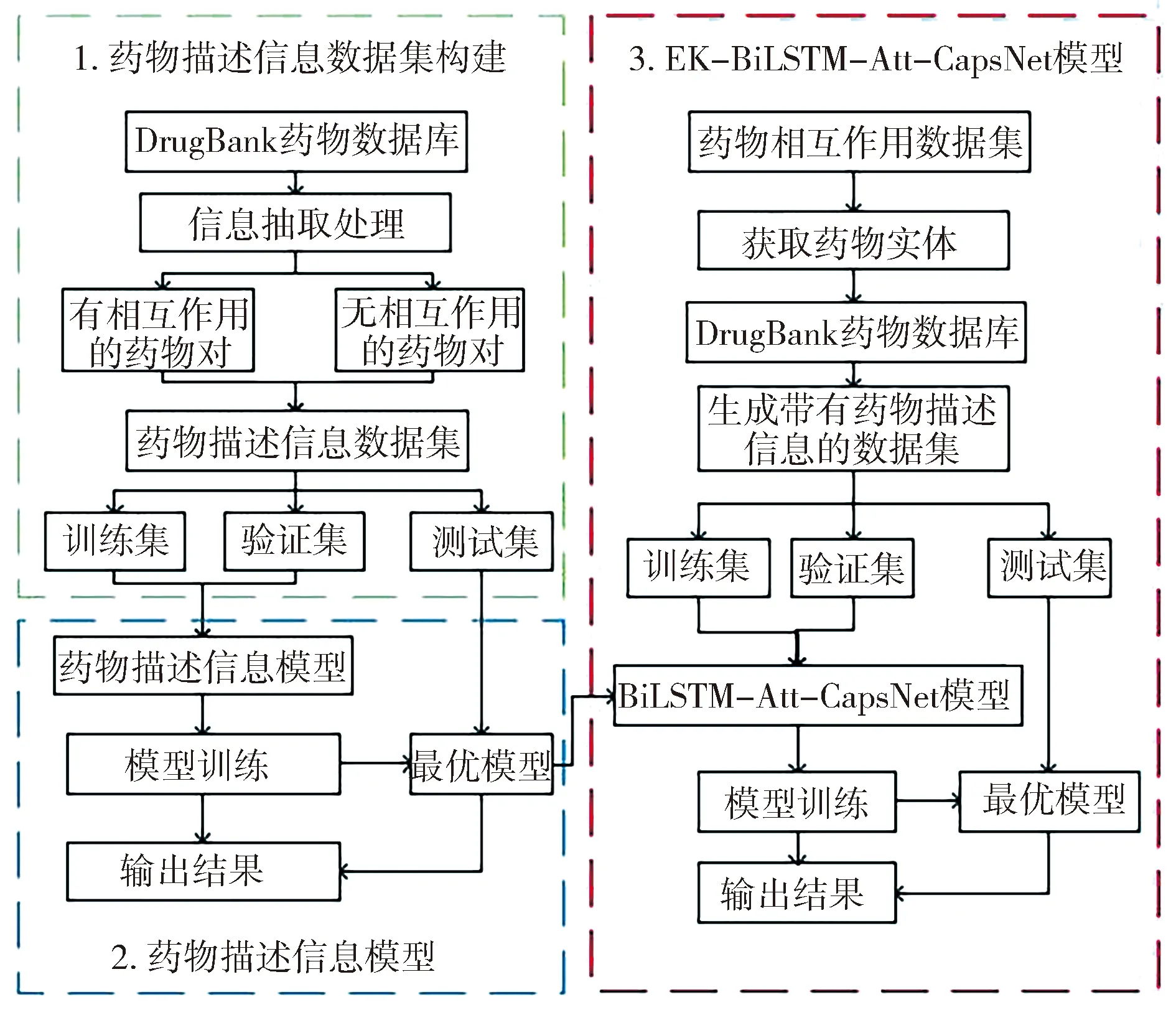
图1 模型整体流程图Fig.1 Flow chart of model architecture
图1主要包括三个部分,分别是药物描述信息数据集构建、药物描述信息模型以及EK-BiLSTM-Att-CapsNet模型. 各部分的主要内容如下.
第1部分,药物描述信息数据集构建. 为了更好地利用外部知识来提高模型的抽取效果,在该部分中通过对药物数据库DrugBank中的知识进行分析和处理,从中抽取出有相互作用的药物对,并生成无相互作用的药物对,同时保留每个药物的描述信息,以此构建带有药物描述信息的药物相互作用数据集.
第2部分,药物描述信息模型. 为了更好地利用药物数据库中已有的药物相互作用知识以及药物描述的相关信息,在本部分中构建基于BiLSTM的药物描述信息训练模型,并在第一部分构建的药物描述信息数据集上进行训练,保存最优模型.
第3部分,EK-BiLSTM-Att-CapsNet模型. 在本部分中,首先需要对药物相互作用数据集DDIExtraction2013进行处理,从中识别出药物实体,再在药物数据库DrugBank中找到对应的药物描述信息,并将该信息保存. 然后将第二部分中保存的最优模型与BiLSTM-Att-CapsNet模型相结合,即为结合外部知识的药物相互作用关系抽取模型EK-BiLSTM-Att-CapsNet. 最后通过对该模型进行训练,得到最优的模型,用于药物相互作用关系抽取.
3.1 药物描述信息数据集构建
DrugBank是较为知名的药物数据库,通过该数据库可以查询到药物的各种信息,如药物类型、药物结构、药物描述信息、药物别名以及与该药物之间有相互作用的其他药物等相关信息. 例如在DrugBank数据库的查询框中输入“Amoxicillin”(阿莫西林)时,页面就会显示出所有与Amoxicillin相关的信息,如图2所示.

图2 DrugBank数据库药物查询示例Fig.2 Example of DrugBank database inquiry
通过对药物数据库中药物的各种信息进行分析,我们发现药物的描述信息是对该药物的详细介绍.在描述信息中一般包括该药物适用的疾病类型、组成成分、作用效果等信息. 两个药物的描述信息中,有可能隐含着这两个药物之间的潜在关系,有助于判断二者是否存在相互作用关系. 因此本文从药物的各种信息中,选用药物的描述信息作为本文模型使用的外部知识之一. 在对DrugBank中的信息进行处理后,从中共计获取到11 930个药物以及对应的描述信息.
在药物数据库DrugBank中,每个药物的相关信息中包含与该药物有相互作用的药物信息,通过对该信息进行处理,可以从中抽取出有相互作用的药物对,以此利用DrugBank中已经存在的大量的药物相互作用的相关知识. 由图2可以看出,与Amoxicillin有相互作用关系的有Tienilic acid、Vecuronium、Ulipristal以及Trestolone等药物,即可产生如下几组有相互作用的药物对:(Amoxicillin,Tienilic acid)、(Amoxicillin,Vecuronium)、(Amoxicillin,Ulipristal)、(Amoxicillin,Trestolone).
由于模型训练用到的数据集不仅需要有相互作用的药物对,还需要无相互作用的药物对,即数据集中需要正样本和负样本同时存在.而药物数据库中不存在现成的无相互作用的药物对,故本文为了得到无相互作用的药物对,首先根据药物编号随机组合两个药物实体为一组药物对,然后从中把有相互作用的药物对过滤掉,即可得到无相互作用的药物对. 为了不对实验结果造成干扰,我们将DDIExtraction2011和DDIExtraction2013数据集中出现过的药物对过滤掉.
经过上述数据处理操作,即可构建成带有药物描述信息的药物相互作用数据集.该数据集中的两个药物之间的关系有两种,分别是有相互作用关系和无相互作用关系. 本文通过对DrugBank中的药物信息进行处理,获取了总计60万组有相互作用的药物对,并随机生成了60万组无相互作用的药物对.将其按照一定的比例划分为训练集、验证集、测试集,数据集的详细信息如表1所示.

表1 药物描述信息数据集
3.2 药物信息描述模型
为了更好地利用药物的描述信息,我们设计了药物描述信息模型.该模型共由五层构成,分别是输入层、嵌入层、BiLSTM层、全连接层、输出层,如图3所示.
由图3可以看出,该模型首先将两个药物实体的描述信息作为模型的输入,并在嵌入层中将输入层的文本转换为向量表示. 然后利用BiLSTM层分别获取到两个药物描述信息的长序列依赖信息,并对该信息进行处理. 最后经过softmax等操作,得到最终的结果. 模型各层详细的内容介绍如下.

图3 药物描述信息模型结构Fig.3 Structure of drug description information model
(1) 输入层:在该层中,将两个药物实体对应的描述语句进行输入.药物实体1的描述语句用p表示,p={p1,p2,…,pi,…,pn},药物实体2的描述语句用q表示,q=(q1,q2,…,qi,…,qn).其中pi和qi分别表示药物实体1和药物实体2的描述语句中的第i个单词.
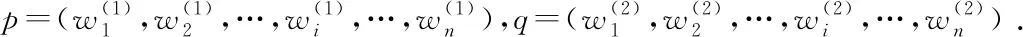
(1)
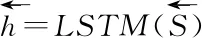
(2)
将药物实体1的描述信息p经过BiLSTM层之后,可以表示为h1∈RN,药物实体2的描述信息q经过BiLSTM层之后,可以表示为h2∈RN.将二者拼接,即h*=[h1;h2]∈R2N送入全连接层中,其中N表示BiLSTM隐藏层单元数目.
(4) 全连接层:由于BiLSTM的输出维度为2N,而输出层的节点为关系类别数目m,故需要使用全连接层进行线性变换.如式(3)所示,其中W(fc)和b(fc)为全连接层的参数.
output=W(fc)·h*+b(fc)
(3)
为了提高模型的泛化能力,降低模型过拟合的风险,我们在全连接层应用Dropout机制.其思想是在训练过程中,从神经网络中随机丢弃神经元(以及它们的连接),以此缓解神经元之间过度协同适应[21].
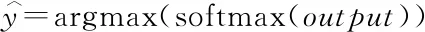
(4)

(5)
该模型使用3.1节构建的带有药物描述信息的药物相互用数据集进行训练,并将取得最优结果的模型进行保存.
3.3 EK-BiLSTM-Att-CapsNet模型
为了充分利用外部药物描述信息和药物相互作用知识,本文将3.2节中保存的最优的药物描述信息模型与基于注意力的药物关系抽取模型相结合,构成EK-BiLSTM-Att-CapsNet模型. 该模型将药物的描述信息作为低层胶囊网络的一部分,然后将其动态的传输到高层胶囊网络中,从而更好地使用药物描述信息,此外,药物描述信息模型是在构建的药物相互作用数据集上进行训练得到,该数据集是对药物数据库中已有的信息进行处理得到,故可以充分利用外部已有的药物相互作用知识. EK-BiLSTM-Att-CapsNet的模型结构如图4所示.

图4 EK-BiLSTM-Att-CapsNet模型结构Fig.4 Structure of EK-BiLSTM-Att-CapsNet model
假设输入原语句Soriginal,该语句中两个药物实体之间的最短依存路径为Ssdp,原语句中的两个药物实体在DrugBank中的描述信息分别des1用和des2表示.
如图4上部所示,将以文本表示的Soriginal和Ssdp经过嵌入层,可得到以向量形式进行表示的Soriginal和Ssdp,再将二者分别送入BiLSTM层,即可得到原语句的长序列依赖信息Horiginal和最短依存路径的长序列依赖信息表示Hsdp.语句中不同单词的重要性不同,因此将单词级别的注意力机制应用在原语句Horiginal上,即可得到加权后的Horiginal. 为了缓解使用最短依存路径可能造成的噪声干扰问题,在Horiginal和Hsdp应用句子级别的注意力机制,可以有效地将原语句信息和最短依存路径信息相融合,用Hall表示.
如图4下部所示,两个药物的描述信息des1和des2经过嵌入层之后,我们将其分别送入BiLSTM层,即可得到相对应的BiLSTM隐藏层输出Hdes1和Hdes2,将二者结合,即可得到Hdes=[Hdes1;Hdes2].
将Hall以及Hdes分别经过卷积操作,得到低层胶囊表示,uall=(u1,u2,…,um)和udes=(u1,u2,…,un),其中m和n分别表示Hall产生的低层胶囊个数,以及药物描述信息Hdes产生的胶囊个数. 二者共同构成胶囊网络层的低层胶囊u=[uall;udes].低层胶囊向高层胶囊传输的信息量可以利用胶囊网络层的动态路由机制来动态地控制,这样就可以动态地利用药物描述信息这一外部知识.
本模型采用的损失函数,如式(6)所示.
Lk=Tkmax(0,m+-‖vk‖)2+
λ(1-Tk)max(0,‖vk‖-m-)2
(6)
4 实 验
4.1 数据集介绍以及评价指标
本文采用DDIExtraction2013数据集来进行实验,该数据集是由792篇DrugBank中的医学文本和MedLine中的233篇摘要组成,并事先对药物实体进行了标注.因此,识别出药物实体不需要经过命名实体识别等操作. 该数据集中共有如下5种药物相互作用类型.
(1) Advice:文本中描述了同时使用两种药物时的建议.
(2) Effect:文本中清楚地表明了两种药物相互作用的结果.
(3) Mechanism:文本中明确讨论了药物动力学机制.
(4) Int:文本中表明两种药物有一定关系,但具体关系类型没有被定义
(5) Negative:相互作用关系并没有存在于两种药物之间.
数据集的详细信息见表2. 本文采用精准率,召回率和F1值这三个指标来对实验结果进行评价.
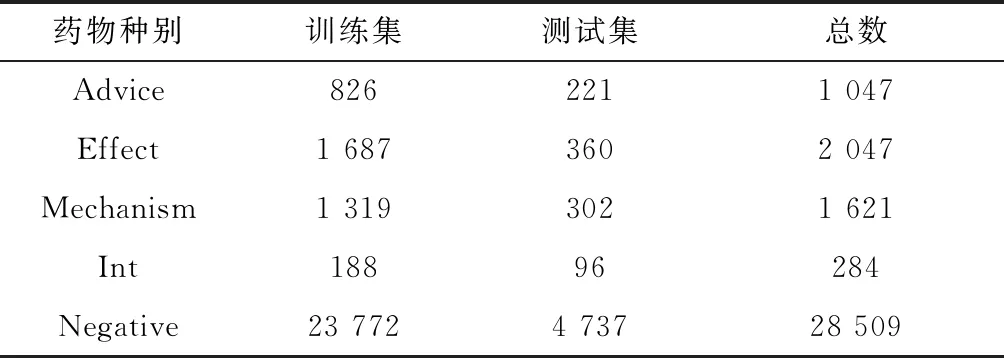
表2 DDIExtraction2013 数据描述
4.2 参数设置
我们将药物描述信息的长度设置为50,并将单词转换为300维的Glove词向量.对于未在Glove词表中出现的单词,我们采用随机初始化的方式表示该单词的词向量.胶囊网络的迭代次数设置为3,Batchsize设置为64,dropout取值为0.5,学习率设置为0.001,药物描述信息模型的迭代次数设置为50,EK-BiLSTM-Att-CapsNet模型的迭代次数设置为35,模型损失函数中的m+为0.9,m-为0.1,λ为0.25.
4.3 实验结果
为了验证模型的有效性,我们从以下三个方面进行实验,分别是消融实验、不同关系类别上的结果对比以及与现有方法的对比实验.
(1) 消融实验.为了验证本章模型的有效性,即验证本章模型中利用的药物描述信息以及药物数据库DrugBank中已有的相互作用知识是否有用,设计消融实验,在DDIExtraction2013数据集上的实验结果,如表3所示.
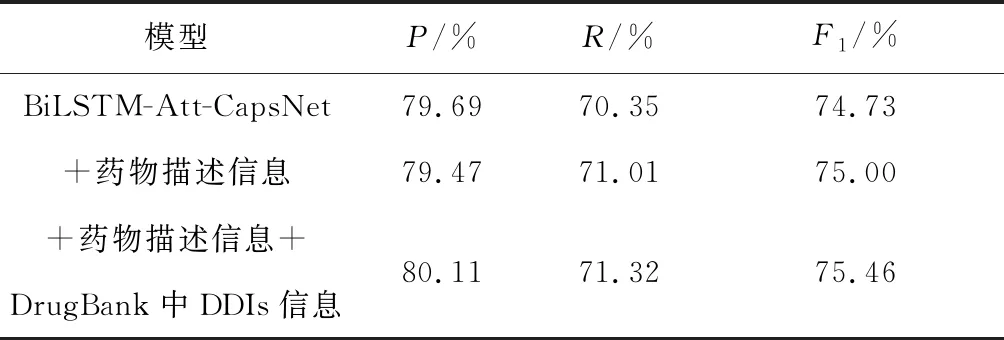
表3 消融实验结果
由消融实验结果可以看出,在直接添加外部的药物描述信息后,模型的F1值提高了0.27%,说明药物描述信息中存在对药物相互作用关系判断的信息.通过使用该信息,可以提高模型的抽取效果. 在使用DrugBank中的带有药物描述信息的药物相互作用数据集进行训练后,模型的效果提升了0.73%.这证明了外部药物数据库中已有的知识对于模型抽取效果的提升有很大的帮助. 消融实验的结果表明,我们提出的模型可以有效利用外部已有的药物描述信息,同时可以利用DrugBank中已有的大量的药物相互作用知识,有助于提高模型的抽取结果.
(2) 不同关系类别上的结果对比.通过对数据集的分析可知,数据集中不同类别的数目相差较大,因此可能造成不同类别的预测结果相差较大.为了验证利用药物描述信息能够缓解该问题,将本文模型与相关模型在不同类别上的实验结果进行对比,具体信息如表4所示.其中Xu等[22]提出的BR-LSTM和Sahu等[23]提出的Joint AB-LSTM都是基于LSTM的模型.
由表4可以看出,对比模型的不同类别之间的最大F1值差值在31.35%到36.15%之间浮动,与他人模型相比,使用本文模型不同类别之间的F1值最大相差仅为25.34%,较大降低了不同类别的F1值之间的差距. 由此可见,与其他模型相比,本章提出的模型能够有效缓解因不同类型样本数目差异较大造成的抽取结果差异较大的问题.更直观的结果如图5所示.
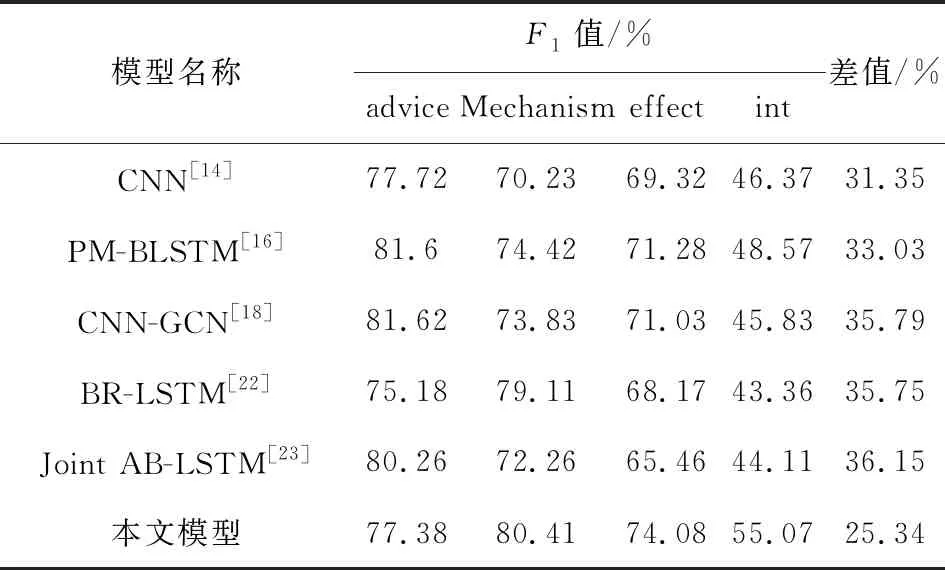
表4 不同关系类别上的实验结果
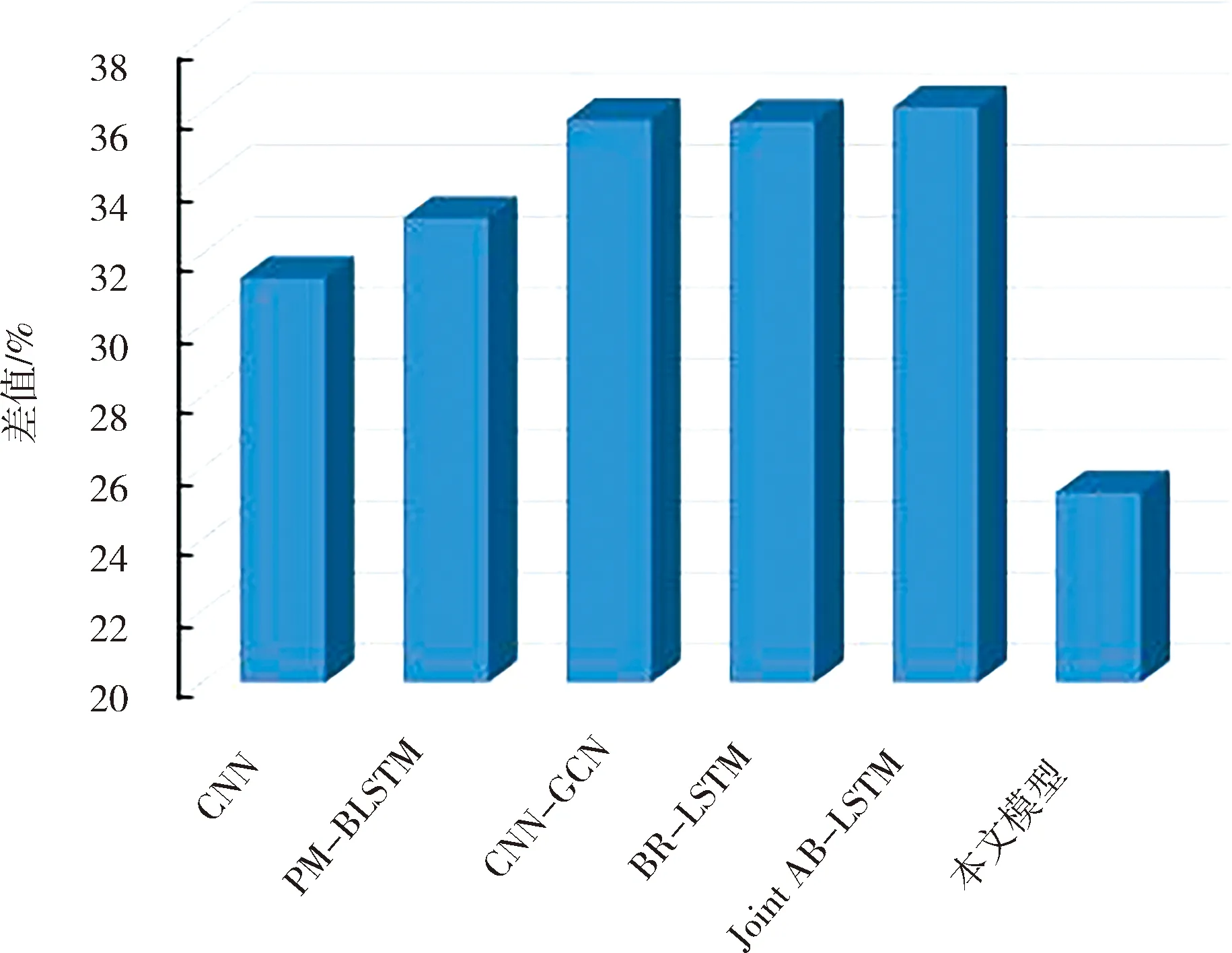
图5 不同类别间的最大F1差值Fig.5 Maximum F1 score difference between different categories
由图5可以明显看出,与其他模型相比,本章提出的EK-BiLSTM-Att-CapsNet模型的不同类别之间的F1值差距较小,能够较好地缓解数据集中因不同类别的数目差异较大造成的不同类别的F1值差异较大的问题.
(3) 与现有方法的对比实验.目前在药物相互作用关系抽取任务上,已有大量的研究人员设计出了多种关系抽取模型,并进行了大量实验.为了验证本文模型的有效性,将本文模型与现有模型在DDIExtraction2013数据集上的实验结果进行对比.
本文对比了不同模型在DDIExtraction 2013数据集上的实验结果.其中Quan等[24]提出的MCCNN基于卷积神经网络进行自动特征提取;Wang等[25]的模型通过将基于依赖的技术引入Bi-LSTM,建模药物之间的依赖关系;Yi等[26]的模型通过将一个循环神经网络结合多个注意力层从生物医学中提取 DDI文本.实验结果如表5所示.
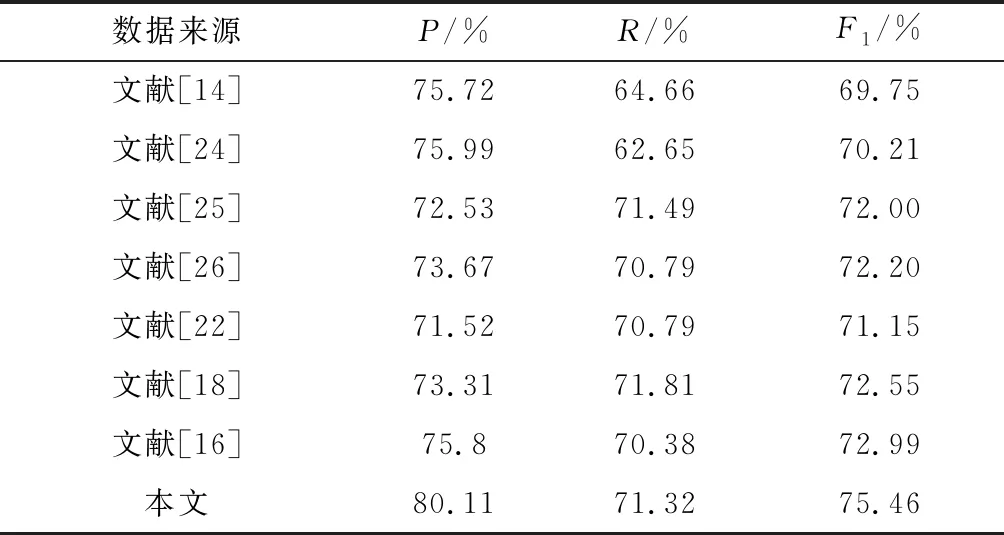
表5 不同模型在DDIExtraction2013数据集上的实验结果
由表5可以看出,本文模型的F1值比其他文献中最佳模型高2.47%.实验结果表明,与现有模型相比[27-28],本文提出的模型能够更好地自动抽取药物之间的相互作用关系.更直观的结果如图6所示.
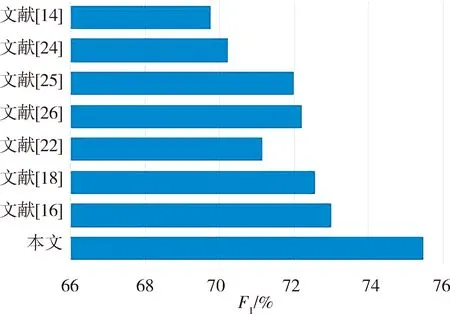
图6 不同模型在DDIExtraction2013数据集上的F1值
5 结 论
本文充分利用了外部药物数据库中的相关信息,使用胶囊网络的结构进行药物关系抽取. 实验结果表明,本文模型不仅提升了抽取效果,还降低了不同类别抽取结果差异较大的问题. 在未来的工作中可以尝试借助药物的其他信息进行辅助.此外,还可以考虑使用远程监督的方法,用于解决数据集中样本数目过少以及不同类型样本数目差异较大的问题.
