产胞外多糖嗜热链球菌eps基因簇的表达及生物信息学分析
2021-12-17田佳乐李嘉雯乔少婷胡海敏
田佳乐,刘 洋,李嘉雯,乔少婷,胡海敏,丹 彤
(内蒙古农业大学食品科学与工程学院 呼和浩特010018)
嗜热链球菌(Streptococcus thermophilus)是一种低GC 含量、兼性厌氧、过氧化氢酶阴性、同型发酵的乳酸菌[1]。嗜热链球菌在发酵牛乳时具有生长繁殖快、发酵酸化活力高、产香等特性。此外,部分菌株能代谢培养基中的碳源生产胞外多糖(Exopolysaccharides,EPS)[2]。EPS 是乳酸菌重要的次生代谢产物,依附于微生物细胞壁形成荚膜多糖或进入培养基形成黏液多糖[3]。EPS 在提高乳制品黏度、质地和口感等方面发挥着重要作用[4],其中一些EPS 还具有抗肿瘤[5]、降低胆固醇[6]、调节免疫系统[7]、抗氧化[8-9]、调节肠道菌群生长因子[10]、抑制病原微生物[11]等功能。
乳酸菌的EPS 生物合成由eps基因簇调控,决定EPS 重复单元的合成、聚合、输出等过程[12-13]。Shan 等[14]、Stingele 等[15]和Luo 等[16]发现嗜热链球菌ASCC1275 在培养基中能同时产生荚膜多糖和黏液多糖,这主要是因为eps基因簇中有2 对控制链长基因,即eps1C-eps1D和eps2C-eps2D;Delcher 等[17]将转座子插入到嗜热链球菌eps基因簇上,发现含有转座子的突变株失去了合成EPS的能力,说明eps基因簇在EPS 生物合成中发挥着重要作用。
嗜热链球菌IMAU20756 分离自蒙古国乌兰巴托市海日玛特苏木酸牛奶中,在M17 液体培养基中EPS 产量为197.02 mg/L。本试验采用二代测序技术(Illumina Hiseq 4000)对嗜热链球菌IMAU20756 基因组进行测序,找出与EPS 生物合成相关的基因簇,同时结合实时荧光定量PCR 技术(Real-time fluorescent quantitative PCR,RTqPCR)分析eps基因在菌体生长不同时间点的表达量,为解析乳酸菌EPS 生物合成途径和调控机制奠定理论基础。
1 材料与方法
1.1 菌株
嗜热链球菌IMAU20756 由内蒙古农业大学乳品生物技术与工程教育部重点实验室提供。
1.2 主要试剂
cDNA 合成试剂盒、qPCR 合成试剂盒(SYBR Green),天根生化科技(北京)有限公司;通用型RNA 提取试剂盒,集思慧远生物公司;M17 肉汤培养基,海博生物公司。
1.3 主要试验仪器
AR2202CN 型电子天平,奥豪斯仪器上海有限公司;HA-300M 型全自动高压蒸汽灭菌器,日本HIRAYAMA 公司;HWS28 型电热恒温水浴锅、LRH-250 型电热恒温培养箱,上海一恒科技有限公司;ZHJH-C1214C 型超净台,上海智城分析仪器制造有限公司;BX50 型光学显微镜,日本奥林巴斯(OLYMPUS);Vortex-genie2 型漩涡振荡器,美国Scientific Industries 公司;5810R 型高速控温离心机、移液器,德国Eppendorf 公司;Nano-Drop2000 型微量分光光度计,美国Thermo Scientific 公司。
1.4 试验方法
1.4.1 嗜热链球菌IMAU20756 基因组测序
1.4.1.1 菌株DNA 提取 将真空冷冻干燥保藏的菌株在脱脂乳培养基中活化,然后在M17 液体培养基中继续活化2 代并进行扩大培养,37 ℃培养24 h,接种量为2%。其中每次传代均需进行革兰氏染色和显微镜观察,在确保无污染的前提下,进行传代培养。最后将第3 代M17 液体培养液离心(3 000×g,5 min),并收集菌体,收集的菌体采用OMEGA-D3392 基因组DNA 提取试剂盒提取样品中基因组DNA,置于4 ℃备用。
1.4.1.2 DNA 质控及基因组重测序 完成基因组DNA 抽提后,采用微量分光光度计测定DNA 的浓度,然后通过1%琼脂糖凝胶电泳判断基因组DNA 的纯度和完整度,并对质量合格的DNA 液采用Illumina Hiseq 4000 进行二代基因组重测序。
1.4.1.3 基因组拼接组装 首先,将Illumina Hiseq 4000 高通量测序平台获得的嗜热链球菌IMAU20756 基因组重测序数据过滤;其次,利用SOAPdenovo 程序包[18]选取合适的kmer 值,对过滤后的数据进行拼接组装和单碱基校正。以基因组大小、脚手架数量、N50 长度、N90 长度、GC 含量值为指标,对组装结果进行评估,最后选取脚手架数量较小、N50 和N90 较长的序列进行后续soap验证及校验。
1.4.1.4 功能基因的预测及注释 采用Glimmer v3.02 软件[19](http://www.cbcb.umd.edu/software/glimmer/)进行基因预测,获得开放阅读框信息。结合RAST(Rapid annotation using subsystem technology)Server-RAST Annotation Server(http://rast.nmpdr.org/)网上在线工具和COG(http://www.ncbi.nlm.nih.gov /COG/)数据库,完成对嗜热链球菌基因组的预测与注释。
1.4.1.5 生物信息学分析 核酸序列相似性比较采用NCBI 的BLAST 软件在线分析(http:// www.ncbi.nlm.nih.gov),多序列比对采用ClustalW 程序在线分析(http://www.ebi.ac.uk/Tools/clustalw/)。
1.4.2 嗜热链球菌IMAU20756 实时荧光定量PCR
1.4.2.1 实时荧光定量PCR 扩增引物的设计 本试验所用引物均采用Primer premier 5 软件设计,由上海桑尼生物科技有限公司合成,引物信息如表1所示。

表1 本试验所用引物Table 1 The primers used in this experiment
1.4.2.2 嗜热链球菌IMAU20756 菌株培养及菌体收集 将嗜热链球菌IMAU20756 按2%接种量接种于M17 液体培养基中,37 ℃培养24 h,分别于6,12,18,24 h 收集菌体,可直接提取总RNA 或于-80 ℃冻存。
1.4.2.3 总RNA 提取及反转录cDNA 合成 取5 mL 菌液离心去上清(5 000×g,6 min),加入RNAiso Plus 试剂提取样品总RNA,使用1%琼脂糖凝胶电泳检测RNA 的质量。基因组DNA 的去除和cDNA 的合成使用PrimeScriptTMRT 试剂盒。基因组DNA 的去除反应条件为42 ℃,2 min;反转录cDNA 合成的反应条件为:50 ℃,15 min,85 ℃,5 s。
1.4.2.4 实时荧光定量PCR 根据eps基因簇5’端deoD到3’端orf14.9之间的保守基因序列设计引物,再以从嗜热链球菌IMAU20756 菌体细胞中提取的DNA 为初始模板进行实时荧光定量PCR反应,即用Taq 酶扩增eps基因簇并对所得PCR产物进行鉴定及测序。本试验参照基因使用的是gapdh,miRNA 反应体系的配置(总体积20 μL)如表2所示。1.4.2.5 反应程序 两步法荧光定量PCR 扩增反应程序设置为95 ℃预变性15 min;94 ℃变性20 s;60 ℃复性并延伸34 s,其中变性、复性、延伸进行40 个循环;反应程序结束后95 ℃,15 s;60 ℃,60 s;95 ℃,15 s 连续测定样品的荧光强度。

表2 miRNA 反应体系Table 2 The reaction system of miRNA
2 结果与分析
2.1 嗜热链球菌IMAU20756 基因组基本特征
经检测,嗜热链球菌IMAU20756 基因组全长为1 838 440 bp,GC 含量为38.8%,共编码了2 120 个基因,包含1 962 个功能基因、36 个tRNA、515 个假基因、1 个tmRNA 和2 个重复单位。其中和EPS 生产相关的基因有12 个,分别为epsA(1 460 bp)、epsB(732 bp)、epsC(692 bp)、epsD(692 bp)、eps1E(1 367 bp)、eps2E(251 bp)、epsG(491 bp)、epsF(917 bp)、epsH(1 112 bp)、epsI(896 bp)、epsJ(977 bp)、epsK(1 442 bp)。
2.2 嗜热链球菌IMAU20756 胞外多糖基因簇
通过比较基因组分析发现嗜热链球菌IMAU20756 基因组中与产EPS 相关的基因有12个(图1)。在基因簇5' 端是deoD,为嘌呤核苷磷酸化酶,主要参与核苷酸的生物合成和分解代谢[20],基因簇3'端是orf14.9,主要参与嗜热链球菌细胞的生长[21],epsA、epsB是调控基因,epsC、epsD决定多糖链长,eps1E、eps2E、epsG、epsF、epsH、epsI、epsJ是糖基转移酶基因,负责将单糖转运到脂载体上构成重复单位,其中eps1E、eps2E编码半乳糖基转移酶基因,epsF编码鼠李糖基转移酶基因,epsK编码多糖聚合及输出。

图1 嗜热链球菌IMAU20756 eps基因簇Fig.1 The eps gene cluster of S.thermophilus IMAU20756
2.3 嗜热链球菌IMAU20756 胞外多糖基因簇分析
利用NCBI Blast 在线搜索嗜热链球菌IMAU20756 产EPS 生物合成的相关基因并预测基因功能,所得部分结果如表3所示。由表可知,位于基因簇5' 端deoD编码嘌呤核苷磷酸化酶,该酶与嗜热链球菌NWC1 菌株的嘌呤核苷磷酸化酶(ID:CP02925.1)及嗜热链球菌CS8 菌株的嘌呤核苷磷酸化酶(ID:CP016439.1)均具有较高同源性,达到98.73%,与嗜热链球菌S9 菌株(ID:CP013939.1)的同源性达到98.59%。epsA与嗜热链球菌ASCC1275 菌株epsA(ID:CP006819.1)具有极高同源性,达到100%,该基因是LytR家族的转录调节因子,包含短的信号锚定区域,由N 末端的胞浆区和跨膜区组成,与荚膜多糖的产量密切相关[22]。epsB为酪氨酸蛋白磷酸酯酶基因,epsD为酪氨酸蛋白激酶基因,它们与嗜热链球菌ASCC1275 菌株的epsB及eps1D同源性分别达到100%和93%;另外,epsA还与嗜热链球菌STCH41 菌株的epsA(ID:MK483531.1)同源性达到98.97%,epsB与该菌株的epsB同源性达到99.04%。epsC负责调控EPS 链长度,与嗜热链球菌STCH19 和STCH38 中的epsB(ID:MK483538.1)有较高的同源性(同源性分别为99.86%与99.42%)。eps1E、eps2E、epsF是编码糖基转移酶的基因,负责EPS 寡糖重复单元的合成,其中,eps1E编码半乳糖糖基转移酶基因,与嗜热链球菌LMG18311(ID:CP000023.1)菌株半乳糖糖基转移酶及嗜热链球菌STCH39(ID:MK483550.1)菌株的半乳糖糖基转移酶基因同源性都达到99.85%,eps2E与嗜热链球菌STCH13(ID:MK483559.1)及嗜热链球菌STCH38 的半乳糖糖基转移酶基因的同源性都达到85.84%。epsF基因是鼠李糖基转移酶,与嗜热链球菌LMG18311 鼠李糖糖基转移酶基因同源性达到96.89%,与嗜热链球菌STCH13菌株的鼠李糖糖基转移酶基因同源性达到96.67%。epsG是嗜热链球菌IMAU20756 特有的糖基转移酶基因,与其它乳酸菌eps基因簇的同源性较低,如与嗜热链球菌LMG18311 全基因组同源性为82%,与嗜热链球菌STCH13 的eps基因簇同源性仅为66%。
表3 嗜热链球菌IMAU20756 胞外多糖合成基因簇基因功能预测Table 3 Gene function prediction of extracellular polysaccharide synthesis gene cluster of S.thermophilus IMAU20756
2.4 嗜热链球菌IMAU20756 总RNA 质量评定
RNA 的质量是影响基因表达结果的重要因素。表4 是总RNA 的OD 值(260nm/280nm,260nm/230nm),图2 是RNA 在1%琼脂糖凝胶电泳的结果。样品RNA 的OD260nm/OD280nm比值在2.20~2.23 之间,琼脂糖凝胶电泳结果清晰地显示23S 亚基(上)和16S 亚基(下)条带,这些结果说明RNA 降解程度较小,质量良好,可用于后续试验。
图2 琼脂糖凝胶电泳结果图Fig.2 The result of agarose gel electrophoresis

表4 总RNA 提取结果Table 4 The extraction results of total RNA
2.5 实时荧光定量PCR 结果分析
嗜热链球菌IMAU20756 和EPS 生产相关的12 个基因不同时间点(6,12,18,24 h)的表达结果如图3所示。epsA、epsB基因表达量在6 h 达到峰值,12 h 开始出现下调表达趋势,24 h 其表达量显著降低,epsC、epsD基因表达量也有相同的增减趋势,说明嗜热链球菌IMAU20756 菌株在6 h 时EPS 合成量较多,随后呈现逐渐减少的趋势;eps1E、eps2E编码半乳糖基转移酶基因,在发酵6 h 时其表达量达到峰值,12 h 呈现下调表达趋势,24 h 时表达量降至最低;epsF是编码鼠李糖糖基转移酶基因,其表达量也在6 h 时达到峰值,24 h降至最低;epsG、epsH、epsI、epsJ均为调控糖基转移酶基因,整体呈现逐渐减少趋势,而epsJ基因在6 h 至12 h 有增多趋势,随后开始下降。另外epsK基因调控EPS 的聚合及输出,其在6 h 至18 h 呈现较为平稳的增长趋势,说明这一阶段EPS 在不断的延伸、聚合,并分泌至培养基中。
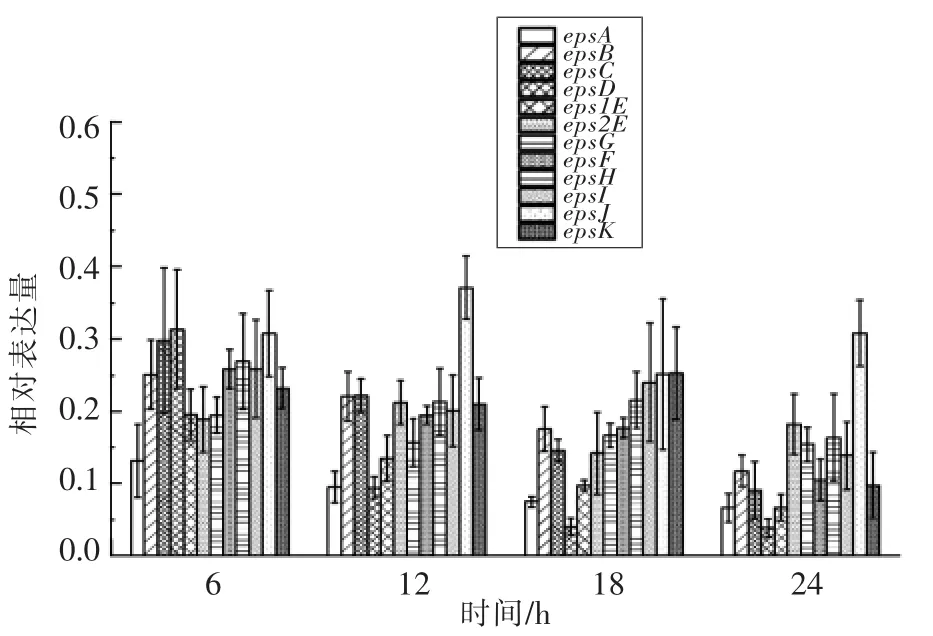
图3 嗜热链球菌IMAU20756 eps基因簇在菌体生长不同阶段相对表达量Fig.3 Relative expression level of eps gene cluster of S.thermophilus IMAU20756 in different time
2.6 嗜热链球菌IMAU20756 菌株eps1E 多序列比对
乳酸菌EPS 的生物合成是由eps基因簇调控。这些基因簇一般含有多个基因,分别控制着EPS 的合成、聚合以及向细胞外的输出。重复单位的合成是在糖基转移酶的催化作用下将糖核苷酸顺序性转移到异戊二烯醇磷酸酯(脂载体)上形成的[23]。在eps基因簇中一般含有多个糖基转移酶,其中,负责将第一个糖基供体转运到脂质携带体上的糖基转移酶被称为引导糖基转移酶[24]。引导糖基转移酶是合成 EPS 重复单元的第一步,在EPS 合成过程中发挥着重要的作用。
嗜热链球菌IMAU20756 菌株eps基因簇长度为1 838 440 bp,与EPS 生产相关的基因有12个,分别控制着EPS 的合成、聚合及向细胞外的输出。其中,eps1E、eps2E、epsG、epsF、epsH、epsI、epsJ编码糖基转移酶基因,负责将单糖转运到脂载体上构成重复单位。应用ClustalW 在线多序列比对将eps1E、eps2E、epsG、epsF、epsH、epsI、epsJ等糖基转移酶基因分别与乳酸乳球菌(Lactococcus lactis)NIZO B40、肺炎链球菌血清型(Streptococcus pneumoniaeserotype)14、嗜热链球菌Sfi6、乳酸乳球菌NIZO B35 的引导糖基转移酶进行氨基酸多序列比对。结果发现在eps1E的氨基酸序列中,含有引导糖基转移酶所共有A、B 和C 保守区存在(如图4所示)。其中A、B 区被认为是催化磷酸糖向脂载体转移的功能区,C 区的功能是决定所转移糖基的特异性[25]。根据这个结果,可以得出eps1E可能是引导糖基转移酶的基因,还需后续实验验证。嗜热链球菌IMAU20756 生产的EPS 主要由EPS-1a 和EPS-3a 两种多糖混合而成,EPS-1a主要由葡萄糖、甘露糖和半乳糖构成,三者的摩尔比为3.62∶1∶2.99;EPS-3a 主要由甘露糖、半乳糖和阿拉伯糖构成,其摩尔比为1.19∶1∶1.08(数据未发表)。eps1E编码半乳糖糖基转移酶基因,说明在重复单位的合成过程中,eps1E将UDP-半乳糖转运到脂载体上,然后其它糖基转移酶陆续将葡萄糖、甘露糖、阿拉伯糖转运到和脂载体相连的糖基上,构成EPS 的主链。

图4 嗜热链球菌IMAU20756 菌株eps1E 多序列比对Fig.4 eps1E multisequence alignment of S.thermophilus IMAU20756 strain
引导糖基转移酶将单糖转运到脂载体上,是构成EPS 生物合成的第一步,在基因工程中具有重要意义。例如Dabour 等[26]报道了当乳酸乳球菌乳脂亚种(Lactococcus lactissubsp.cremoris)SMQ- 461 的起始糖基转移酶基因发生突变时,该菌株失去了生产EPS 的能力,证明该酶是调控EPS 生产的关键酶。类似的结果发生在Lebeer等[22]报道的鼠李糖乳杆菌(Lactobacillus rhamnosus)GG 中的welE基因中,将引导糖基转移酶基因敲除后,和野生菌株对比突变株分泌到环境中的EPS 减少甚至是终止了EPS 的产生。利用基因工程方法在产EPS 的菌株中导入糖基转移酶基因,可产生特定新型的EPS,这些方法的实施有助于提高EPS 在食品工业中的应用。
3 结论
本实验采用二代测序技术及实时荧光定量PCR 技术对高产EPS 的嗜热链球菌IMAU20756进行基因组测序并对不同时间点eps基因表达量定量分析发现该菌株基因组全长为1 838 440 bp,GC 含量为38.8%,共编码2 120 个基因,包含1 962 个功能基因,36 个tRNA、515 个假基因、1个tmRNA 和2 个重复单位。其中和EPS 生产相关的基因有12 个,epsA、epsB是调控基因,epsC、epsD决定多糖链长,eps1E、eps2E、epsG、epsF、epsH、epsI、epsJ是编码糖基转移酶基因,epsK调控多糖聚合及输出。所有和EPS 生物合成相关的基因在发酵过程中均能表达,特别是糖基转移酶的基因表达量在发酵6 h 时达最高。
