不同地区奶酪中乳酸菌多样性研究
2021-12-17吕瑞瑞杨成聪李伟程赵飞燕孙志宏张和平
吕瑞瑞,杨成聪,李伟程,赵飞燕,马 腾,孙志宏,张和平
(内蒙古农业大学 乳品生物技术与工程教育部重点实验室 农业农村部奶制品加工重点实验室内蒙古乳品生物技术与工程重点实验室 呼和浩特010018)
奶酪是一种具有悠久历史的发酵乳制品,常被称为干酪或奶豆腐,因营养丰富、风味独特而广受人们的喜爱。奶酪通常是由动物乳发酵后,排出乳清再经成熟得到的一种发酵乳制品[1],其含有丰富的蛋白质、脂质、矿物质及维生素等成分,具有很高的营养价值[2],可以预防龋齿和降低慢性疾病的发生[3-4]。
奶酪中含有丰富的微生物资源,其中乳酸菌(Lactic acid bacteria,LAB)对于改善奶酪感官品质及风味具有重要作用[5]。LAB 是一类可发酵碳水化合物产生乳酸的革兰氏阳性细菌总称[6],在发酵过程中可促进奶酪成熟,提高营养价值及强化风味物质的产生[7],在改善奶酪质地、拉伸性、气孔形成和抑制病原微生物生长等方面发挥着重要作用[8-9]。摄入一定量的LAB 可改善人体肠道菌群,提高人体免疫力,抗肿瘤,降低血液胆固醇及血压[10],并且奶酪中的乳酸菌及其代谢产物可在一定程度防止便秘和腹泻,有利于人体健康[11]。因此发掘奶酪中LAB 的物种多样性有利于乳酸菌资源开发。
第三代测序技术具有超长读长、测序时间短、高通量等优点,可在种水平对样品中的微生物进行分析[12]。LAB 作为发酵食品中最主要的一类细菌,在多种发酵食品中均有参与。本研究对14 份奶酪样品进行乳酸菌特异性测序和后续生物信息学分析,探究不同地区奶酪中LAB 的物种多样性,为挖掘乳酸菌资源提供参考。
1 材料与方法
1.1 材料与试剂
1.1.1 样品收集 14 份奶酪样品采自不同地区,其中12 份与Yang 等[13]所研究的奶酪样品相同。现将样品命名为CA1~CA14,具体信息见表1。将采集的奶酪样品速冻后,迅速运输至内蒙古农业大学乳品生物技术与工程教育部重点实验室-80℃保藏备用。

表1 样品信息Table 1 Sample information
全自动高压蒸汽灭菌器,日本三洋公司;HWS24 型恒温水浴锅,上海一恒科学仪器有限公司;5810R 高速冷冻离心机,德国Eppendorf 公司;Applied Biosystems PCR 仪,美国Applied Biosystems 系统公司;微量紫外分光光度计,美国Thermo Fisher Scientific 公司;CDS8000 型UPV 凝胶成像分析系统,上海赛智创业科学公司;Qubit 2.0 核酸蛋白定量仪,美国Life Technologies 公司;SMRT RS II 测序平台,美国Pacific Biosciences公司。
1.2 试验方法
1.2.1 DNA 提取方法 取0.5 g 奶酪样品充分研磨,采用试剂盒按照说明书提取奶酪样品宏基因组DNA,随后检测DNA的片段完整度、纯度及浓度,将符合要求的宏基因组DNA 贮存到-80 ℃冰箱进行后续试验。
1.2.2 乳酸菌目的片段扩增、建库及测序 本文参照徐海燕等[14]研究中乳酸菌引物、建库及测序方法进行试验。乳酸菌引物(表2)两端添加Barcode 用于区别同一文库中不同样品。PCR 扩增程序设置为:95 ℃预变性1 min;95 ℃变性30 s,60℃退火40 s,72 ℃延伸1 min,共循环28 次;72 ℃终端延伸7 min,4 ℃终止。各样品PCR 产物纯化后,用PacBio SMRT RS II 测序平台配套试剂盒构建文库,根据说明书进行后续试验操作。

表2 乳酸菌特异性引物Table 2 The specific primers of lactic acid bacteria
1.2.4 生物信息学分析 下机数据的生物信息学处理具体分析流程参考Yang 等[13]的方法。其中乳酸菌注释使用Greengenes(v13.8)、RDP(v11.5)和Silva(v128)3 个数据库对分类地位进行确定。
1.2.5 数据处理 采用香农曲线(Shannon index curve)和稀疏曲线(Observed species curve)评估每个样本测序的多样性,并判断当前测序量是否能够满足后续生信分析的需要。计算α 多样性指标,其中包括香农指数(Shannon index)、发现物种数(Observed species)、超1 指数(Chao1 index)和辛普森指数(Simpson index)。基于Unifrac 距离对14 份奶酪样品进行主坐标分析(Principal coordinate analysis,PCoA),利用Venn 图在OTU 水平上分析不同分组间OTU 组成差异。采用Lefse 分析(Linear discriminant analysis effect size)比较差异菌种。用斯皮尔曼(Spearman)相关系数分析样品中乳酸菌菌种的相关关系并进行显著性检验,检验水平为95%。不同地区14 份奶酪样品测序数据已上传至SRA 数据库(PRJNA643871)。
2 结果与分析
2.1 乳酸菌测序丰度及多样性
为评估测序深度是否覆盖了全部样品的绝大部分乳酸菌,绘制稀疏曲线(图1a)和香农曲线(图1b)。结果显示,稀疏曲线虽未达到最终状态,但有趋于平缓的趋势,香农曲线显示全部样品进入或接近平台期,由此可见,当前测序量可满足后续生物信息学分析的要求。

图1 稀疏曲线(a)和香农曲线(b)Fig.1 Observed species curve(a)and Shannon index curve(b)
14 份奶酪样品的序列信息和α 多样性指数如表3所示,在物种丰度上,采自西班牙地区的CA6 样品Chao1 指数最大,乳酸菌物种数最丰富;而采自意大利地区的样品CA7 Chao1 指数最小,乳酸菌物种数最少。在物种多样性上,采自西班牙地区的CA6 和中国西藏自治区的CA14 样品对应的香农指数和辛普森指数最高,说明物种多样性程度最高;而意大利地区的CA7 香农指数和辛普森指数均最小,反映出二者物种多样性程度最低。
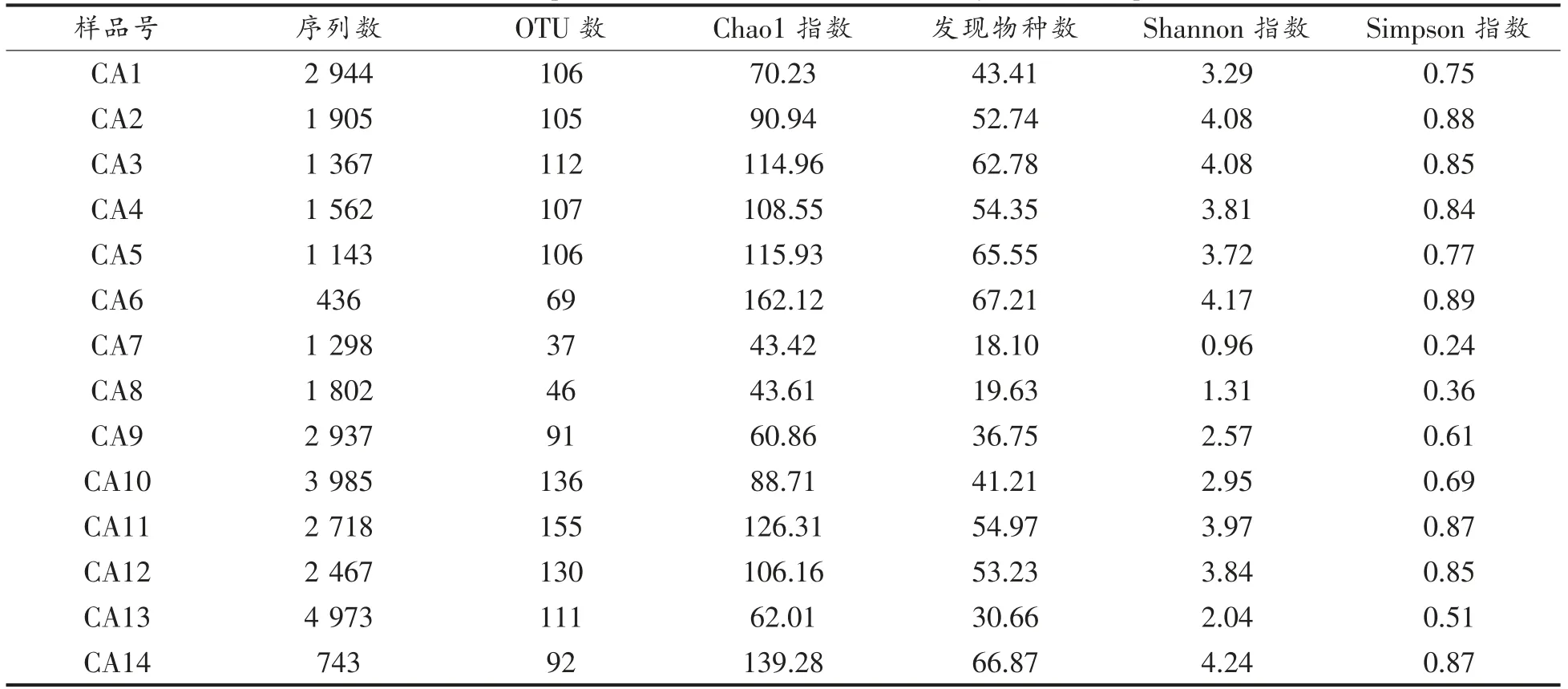
表3 样品的测序序列信息和α 多样性指数Table 3 Sequence abundances and α-diversity in all samples
2.2 奶酪中乳酸菌组成分析
在属水平上,14 份奶酪样品中乳酸菌菌群构成如图2所示,14 份奶酪样品共鉴定到14 个乳酸菌属,其中平均相对含量大于1.0%有4 个菌属,分别是乳杆菌属(Lactobacillus,30.28%)、乳球菌属(Lactococcus,28.31%)、链球菌属(Streptococcus,24.75%)和葡萄球菌属(Staphylococcus,15.16%),检出率全部为100%(14/14)。平均相对含量大于0.1%且小于1.0%有4 个菌属,分别为肠球菌属(Enterococcus,0.68%)、片球菌属(Pediococcus,0.36%)、明串珠菌属(Leuconostoc,0.17%)、魏斯氏菌属(Weissella,0.14%),其中除魏斯氏菌属检出率为57%(8/14),其余3 个菌属检出率均小于50%。

图2 奶酪在属水平的乳酸菌菌群构成Fig.2 Genus-level composition of LAB in the cheese
Yang 等[13]使用细菌通用16S rRNA 引物27F/1495R 与PacBio SMRT 测序技术相结合对其中12 份样品进行分析,检测到的优势菌属为乳杆菌属、链球菌属、乳球菌属、葡萄球菌属和明串珠菌属三代测序技术结合细菌通用引物27F/1492R 一般情况下捕获乳酸菌序列占总序列数不到3%[14]。本研究中采用PacBio SMRT 三代测序与乳酸菌特异性引物相结合的方法还鉴定到肠球菌属、片球菌属和魏斯氏菌属。赵顺先等[15]采用Illumina 高通量测序技术对新疆干酪中乳酸菌多样性和环境关联性分析发现,乳杆菌属、双歧杆菌属、魏斯氏菌属和片球菌属是优势菌属,奶酪营养成分也会对其中的乳酸菌菌群组成造成影响。段宇珩等[16]从青海曲拉中检测到的优势菌属为明串珠菌属、肠球菌属和乳杆菌属。上述相同菌属作为奶酪中的常见乳酸菌菌属,然而在不同地区样品中的优势菌种会存在显著差异[17]。由于奶酪发酵中无外源发酵剂的添加,产生这种情况的原因可能是采样地区,气候、环境及制作方式等多种因素对奶酪中乳酸菌菌群结构产生影响。
在种水平上,所有奶酪样品共发现65 个乳酸菌种,其中平均相对含量大于1%的有12 个菌种,主要包括乳酸乳球菌(Lactococcus lactis,27.10% )、嗜热链球菌(Streptococcus thermophilus,14.35%)、副干酪乳杆菌(Lactobacillus paracasei,9.92%)、马胃葡萄球菌(Staphylococcus equorum,8.93%)、乳房链球菌(Streptococcus uberis,7.29% ),鼠李糖乳杆菌(Lactobacillus rhamnosus,7.15%)、德氏乳杆菌(Lactobacillus delbrueckii,5.10%)、腐生葡萄球菌(Staphylococcus saprophyticus,4.02%)、植物乳杆菌(Lactobacillus plantarum,2.01% )、副乳房链球菌(Streptococcus parauberis,1.95%)、鸡乳杆菌(Lactobacillus gallinarum,1.24%)和Lactobacilluskisonensis(1.05%)。
其中,乳酸乳球菌、嗜热链球菌、马胃葡萄球菌和腐生葡萄球菌检出率为100%(14/14)。其中在CA6、CA9、CA10、CA12 和CA13 中乳酸乳球菌相对含量最高,在CA3 和CA1 中嗜热链球菌相对含量最高。在CA4 和CA14 中检测到相对含量较高的马胃葡萄球菌和腐生葡萄球菌。只在CA2 中检测到相对含量较高的副乳房链球菌和Lactobacillus kisonensis。德氏乳杆菌检出率为93%(13/14)。鸡乳杆菌检出率为78%(11/14)。副干酪乳杆菌、鼠李糖乳杆菌、植物乳杆菌检出率为71%(10/14)。不同样品的乳酸菌组成及其相对含量差异较大。乳房链球菌和副干酪乳杆菌平均相对含量分别7.29%和1.95%,但二者检出率均为36%(5/14),值得注意的是乳房链球菌在CA8 平均相对含量为98.22%,副乳房链球菌在CA2 中平均相对含量为26.27%。仅在西藏样品CA14 中检测相对含量较高的马乳酒样乳杆菌(Lactobacillus kefiranofaciens),该菌常在西藏自然发酵乳制品中检出。刘飞翔等[18]发现可做益生菌的微生物有乳酸乳球菌、嗜热链球菌、副干酪乳杆菌、鼠李糖乳杆菌、德氏乳杆菌、植物乳杆菌、肠膜明串珠菌、屎肠球菌。然而屎肠球菌仅在CA6 中检出,肠膜明串珠菌仅在CA6 和CA14 中检出。乳酸乳球菌和肠膜明串珠菌的协同作用会使乳制品香气更浓烈[19]。在工厂生产中,适当添加肠膜明串珠菌作为辅助发酵剂,明显缩短发酵周期且改善产品风味[13]。

图3 奶酪乳酸菌在种水平的组成Fig.3 Species-level composition of LAB in the cheese
2.3 乳酸菌群落结构分析
为探究不同样品中乳酸菌结构的组成差异,基于非加权的Unifrac 距离进行主坐标分析(图4a),样品CA10、CA12 和CA13 物种组成可能较为相似,故聚为一簇,然而其它样品之间并没有显著聚类,这可能由于乳酸菌菌种组成差异较大所致。在基于加权的Unifrac 距离进行主坐标分析图中(图4b),奶酪样品呈现明显趋势聚为3 组,然而3 组之间乳酸菌多样性存在较大差异,每组样品乳酸菌群落结构更为相似。根据乳酸菌群落组成及丰度,将14 份奶酪分为3 组,第1 组包含CA6、CA9、CA10、CA12 和CA13,第2 组包含CA1、CA2、CA3 和CA8,第3 组包含CA4、CA5、CA7、CA11 和CA14,各类型代表性菌属分别为乳球菌属、链球菌属和乳杆菌属,然而第3 组样品中CA4 和CA14 的绝对优势菌属为葡萄球菌属,相对含量分别为83.04%和66.26%。
根据不同分组进行属水平LEfSe 分析(图4c和4d)发现,乳球菌属在第1 组中显著富集,链球菌属在第2 组中丰度更高,然而图中第3 组并未发现显著差异的菌种,该结果与PCoA 结果基本一致;在种水平进行LEfSe 分析,仅在第1 组中发现乳酸乳球菌含量显著高于其它类型。
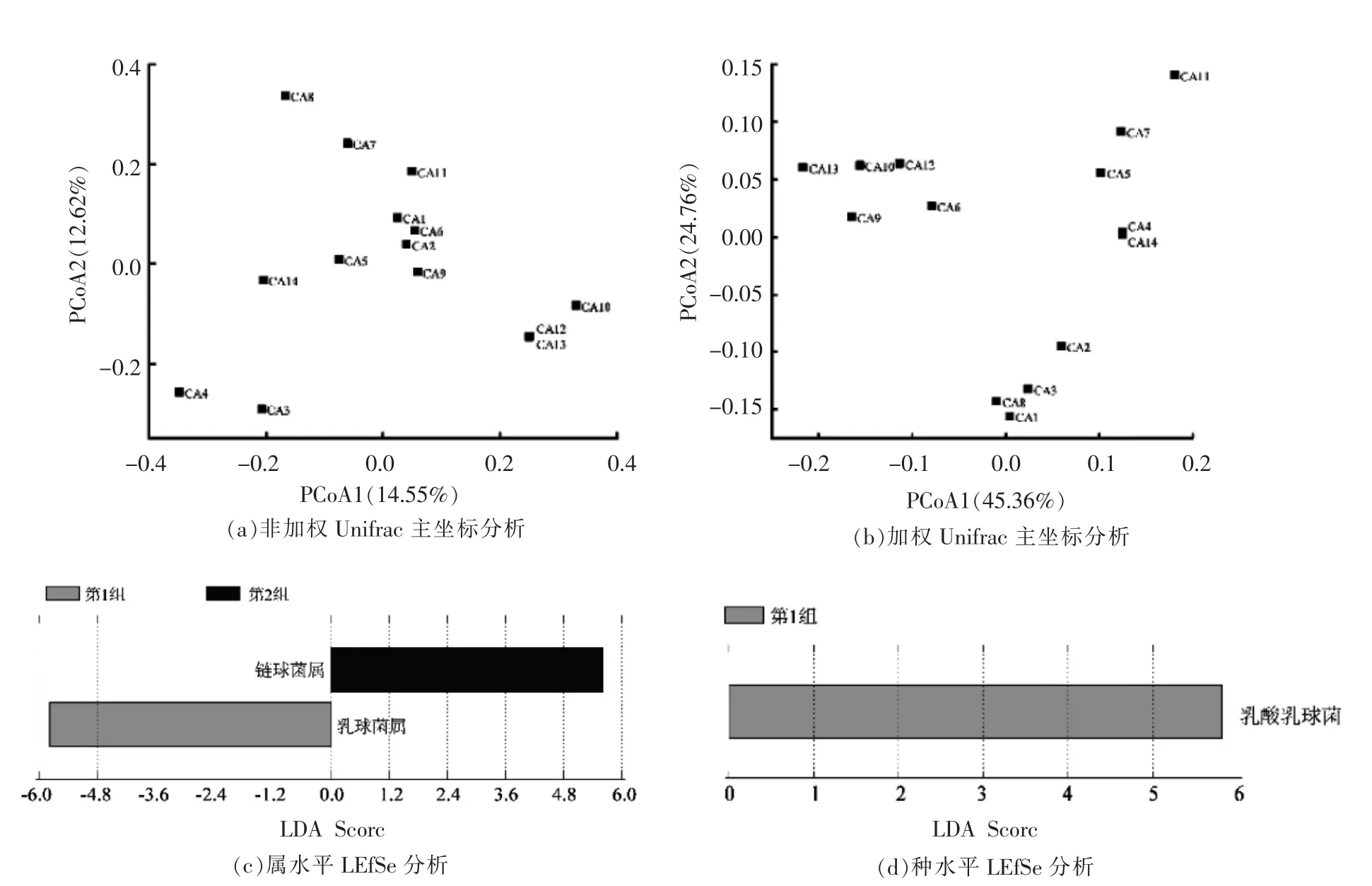
图4 奶酪乳酸菌菌群多样性分析Fig.4 Diversity analysis of lactic acid bacteria in cheese
在奶酪的3 组样品中,第1 组CA6 和CA13采自西班牙,其它分别采自意大利、法国和希腊,以上地区气候类型全部属于地中海气候,表明地理位置和环境会对奶酪中乳酸菌菌群结构及丰度产生影响。第2 组中CA1、CA3 和CA8 分别采自荷兰、德国和意大利,以上3 个地区临海空气湿度大、冬温夏凉;第3 组中CA7 和CA11 采自意大利,其余分别采自法国、瑞士和中国西藏自治区。除中国西藏自治区外,其余3 个国家毗邻,研究发现海拔、空气湿度、培养和生产技术也会影响奶酪微生物菌群结构[13]。值得注意的是CA7、CA8、CA9和CA11 均采自意大利,但并没有显著聚类,这可能是由于同一地区,不同的原料、生产环境、制作手法及发酵方式也会对奶酪的细菌菌群结构产生影响。赵顺先等[15]研究发现制作工艺、生产温度及奶源本身微生物的差异,可能导致自然发酵奶酪样品细菌群落组成产生差异。
奶酪样品中微生物菌群结构会因采样地区、地理环境和样品制作手法的不同而有所差异[20]。Venn 图可反映不同组之间共有和独有物种情况,本研究利用Venn 图对各组样品中乳酸菌在OTU水平上进行比较分析,结果显示各组之间乳酸菌差异比较明显。在OTU 水平上(图5),14 份样品共有646 个OTU,其中每组特有OTU 分别为179,112 和171 种,核心OTU 为86 种,相对含量占总OTU 数目的13.3%。由图可知,不同地区的奶酪样品,因气候、地理环境等影响会形成其特有OTU,Dugat-Bony 等[21]发现采样地不同,干酪中乳酸菌菌群结构表现出较大差异。Kamimura 等[22]研究表明不同地区的样品会因生产环境的不同而导致细菌菌群结构会有显著的差异。

图5 不同地区样品在OTU 水平下乳酸菌物种的Venn 图Fig.5 Venn figure of LAB OUT of all cheese samples from different region
微生物互作在发酵食品中发挥着重要作用,通过代谢营养物质产生细菌素、乳酸和其它代谢物,对其它细菌产生抑制、促进等作用[23]。为揭示奶酪中乳酸菌之间的相关关系,对每组样品中平均相对含量大于1%的核心菌群进行Spearman 相关性分析(图6)。结果发现植物乳杆菌和副乳房链球菌、鸡乳杆菌、德氏乳杆菌呈显著正相关(r=0.62,r=0.54,r=0.55;P<0.05),德氏乳杆菌和鸡乳杆菌、鼠李糖乳杆菌呈显著正相关(r=0.56,r=0.54;P<0.05),腐生葡萄球菌和马胃葡萄球菌呈显著正相关(r=0.66;P<0.01);鸡乳杆菌和乳酸乳球菌呈显著负相关(r=-0.55;P<0.05)。有研究表明乳杆菌产酸较快,且其酸耐受能力较乳球菌强,在发酵过程产酸会对部分乳球菌产生抑制作用,且乳酸乳球菌会产生对革兰氏阳性细菌有抑制作用的尼生素[24-25],这可能会导致鸡乳杆菌和乳酸乳球菌呈显著负相关。在奶酪发酵过程中,发酵剂乳酸菌菌群(如德氏乳杆菌)可为非发酵剂菌群(如植物乳杆菌、鼠李糖乳杆菌)提供重要的代谢前体物和适宜的生长环境[26],该结论与本研究发现具有一致性。

图6 奶酪中乳酸菌的相关性Fig.6 Spearman’s rank correlation of LAB in the cheese
3 结论
本研究采用乳酸菌特异性引物与PacBio SMRT 测序技术相结合的方法,对14 份不同地区的奶酪样品进行乳酸菌的物种多样性分析,共鉴定到14 个乳酸菌属,65 个乳酸菌种。由于地理环境、生产方式及原料等因素影响,不同地区的奶酪样品其乳酸菌菌群结构会存在显著差异,且奶酪中部分低丰度乳酸菌间互作作用有利于奶酪成熟及贮藏。奶酪作为益生乳酸菌分离重要来源,通过研究乳酸菌多样性及相关性分析可为今后挖掘有商业价值的乳酸菌、乳酸菌益生特性开发和改良新型奶酪提供一定的理论基础。
