基于MMR缺陷的高频突变食源性沙门氏菌环丙沙星耐药机制
2021-12-03杨保伟盛焕精李怡澜施春雷史贤明肖英平
王 银,杨保伟,盛焕精,李怡澜,施春雷,史贤明,肖英平,杨 华
(1.西北大学食品科学与工程学院,陕西 西安 710069;2.上海交通大学农业与生物学院,中美食品安全联合研究中心,上海 200240;3.西北农林科技大学食品科学与工程学院,陕西 杨凌 712100;4.浙江省农业科学院农产品质量标准研究所,浙江省植物有害生物防治省部共建国家重点实验室培育基地,浙江 杭州 310021)
沙门氏菌(Salmonella)是一类典型的人畜共患病的食源性致病菌,约有94%的沙门氏菌感染通过食品传播[1-2]。畜牧养殖及临床治疗中抗生素的滥用会加速多重耐药沙门氏菌的出现,这些多重耐药沙门氏菌同时也会通过食物链及动物性产品的消费进一步在人群中传播。有研究推测,若按现今发展趋势,预计到2050年全球范围内因多重耐药细菌感染会引起1 000万 人死亡,经济损失将高达100万亿 美元[1]。因此,多重耐药沙门氏菌的广泛传播将给全球范围内公众健康及食品安全带来严重危害[3]。
细菌中抗生素作用位点的编码基因突变以及抗性基因水平转移是引起多重耐药的最主要原因[4]。在自然状态下,细菌的自发突变频率约在1×10-9~1×10-8之间,凡是突变频率超过自然状态下自发突变频率1 000 倍的菌株就被认为是高频突变子[5-6]。相较于野生型菌株,高频突变子明显具有更高的突变频率、基因水平转移能力与多重耐药及高水平耐药能力[4,6]。研究表明,引起细菌发生高频突变的原因有很多,例如环境压力、抗生素胁迫、SOS应答系统等,但细菌高频突变子的形成主要与其甲基导向错配修复(methyl-directed mismatch repair,MMR)系统中的任一主要功能基因(mutS、mutH、mutL和uvrD)突变有关[7-9]。MMR系统是一种常见的DNA错配修复系统,其主要功能是通过识别错配DNA,并对在DNA合成过程中错配的位点进行剪切与替换,修复两个碱基以上的缺失或插入,保障细菌在DNA合成过程中的忠实性[10]。MutS、MutL、MutH和UvrD是MMR系统中最主要的4 种功能蛋白,任一蛋白突变则会增加细菌突变及重组频率。针对MMR突变/缺失与细菌高突变频率与耐药能力间关系的报道表明,MMR缺陷型高频突变子细菌完整性和稳定性被破坏,其突变频率及同源基因间片段相互交换、转移的几率大幅升高,易出现新的耐药表型及强耐药能力[11]。相关研究发现大肠杆菌(Escherichia coli)、结核杆菌(Mycobacterium tuberculosis)、阴沟肠杆菌(Enterobacter cloacae)以及金黄色葡萄球菌(Staphylococcus aureu)中高频突变子的突变频率均与MMR系统功能的缺失呈正相关[12-16]。除此之外,MMR缺陷也会对细菌耐药相关基因(例如抗生素结合靶点、外排泵系统和膜蛋白编码基因)的表达产生影响[7,16-21]。
氟喹诺酮类抗生素广泛应用于沙门氏菌感染的临床治疗。细菌对氟喹诺酮类抗生素耐药性主要与喹诺酮耐药决定区(quinolone resistance determining region,QRDR)中氨基酸突变及acrAB-tolC、oqxAB外排泵基因表达水平有关[13,22-23]。MMR系统缺陷与细菌突变频率、药敏性以及基因突变能力密切相关,但其对沙门氏菌高频突变子突变频率及环丙沙星(ciprofloxacin,CIP)敏感性的影响及作用机制仍尚未可知。
因此,本研究通过探究MMR缺陷的沙门氏菌高频突变子突变频率、CIP最小抑菌浓度(minimum inhibitory concentration,MIC)与耐药基因表达差异间的关系,揭示MMR缺陷对食源性沙门氏菌高频突变子CIP耐药性的影响及调控机制,旨在为降低沙门氏菌高频突变子的传播,保障食品及公共卫生安全提供新思路及理论依据。
1 材料与方法
1.1 材料与试剂
1.1.1 菌株
90 株沙门氏菌高频突变子,为前期研究中Wang Yin等[24]筛选自本实验室保存的2007—2010年在我国北京、上海、福建、河南、四川、广东、广西、陕西等地各类零售食品(鸡肉、猪肉、羊肉、牛肉、鱼、凉拌菜、速冻水饺)中分离出的1 264 株沙门氏菌[24]。E.coliATCC 25922及E.faecalisATCC 29212为本实验室保存。互补实验中携带野生型mutH、mutL、mutS及uvrD基因的供体质粒NR9931、NR9932、NR9933及NR9934由美国马里兰大学(College Park, MD, USA)提供。
1.1.2 培养基
LB(Luria-Bertani)琼脂、LB肉汤、MHA(Mueller Hinton)琼脂 美国BD公司。
1.1.3 抗生素
萘啶酮酸(nalidixic acid,NAL)、CIP、利福平(rifampicin,RIF)、氨苄西林(ampicillin,AMP)(均为分析纯) 美国Sigma公司。
1.1.4 引物
MMR关键蛋白编码基因(mutS、mutH、mutL、uvrD)、喹诺酮及氟喹诺酮耐药相关基因(gyrA、gyrB、acrA、acrB、ompF、parC、parE、tolC、marA、marR、acrE、ompR、soxS、hilA、dnaQ)聚合酶链式反应(polymerase chain reaction,PCR)及实时PCR(realtime PCR)扩增引物均由北京奥科鼎盛生物科技有限公司合成(表1)。

续表1
1.1.5 试剂
CCMB80 buffer、30%甘油、75%乙醇、rTaq(TaKaRa)、十二烷基硫酸钠(sodium dodecyl sulfonate,SDS)、5×TBE缓冲液、TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit Ver.3.0、PrimeScript RT reagent Kit、SYBR Premix ExTaqTMII、LATaqTMDNA Polymerase、rTaqTMDNA Polymerase、DL2000 DNA Marker 宝生物工程(大连)有限公司;细菌总RNA提取试剂盒RNA Purification Bacteria Kit 天根生化科技有限公司。
1.2 仪器与设备
Mycycler PCR仪、DNA电泳和凝胶成像系统、Gene Pulser Xcell电转仪、电击杯、iQ5 real-time PCR仪 美国Bio-Rad公司;GF16RX高速低温冷冻离心机 日本Hitachi公司;核酸蛋白测定仪 杭州奥盛仪器有限公司;Milli-Q纯水仪 法国Millpore公司;磁力加热搅拌器美国Fisher公司;-80 ℃超低温冰箱 日本Sanyo公司;百分之一天平、万分之一天平 德国赛多利斯公司;立式压力蒸汽灭菌器 上海申安高压仪器设备有限公司;隔水式恒温培养箱 北京科伟实验仪器有限公司;移液器、高速离心机 德国Eppendorf公司;生物安全柜美国Labgard公司;超净工作台 苏州净化设备有限公司。
1.3 方法
1.3.1 突变频率测定
高频突变子突变频率测定依据Wang Yin等[24]方法进行。具体如下:将待测菌株活化后挑取单菌落接种于5 mL灭菌LB肉汤培养基中,37 ℃、100 r/min过夜培养。取100 μL菌悬液按103~106进行梯度稀释,分别取100 μL稀释液涂布于LBrif(RIF 100 μg/mL)、LBnal(NAL 20 μg /mL)及LB平板上,37 ℃培养18~24 h。计数后根据浓度梯度换算原液中可生长菌落数,每株菌3 个平行。突变频率(f)计算公式如下:

式中:fR为RIF筛选下的突变频率;fN为NAL筛选下的突变频率;CR为换算后原液中可在RIF平板上生长菌落数;CN为换算后原液中可在NAL平板上生长菌落数;CB为换算后原液中菌落数。
当fR或/和fN大于10-6则为高频突变子。
1.3.2 CIP的MIC测定
CIP对高频突变子的MIC采用美国国家临床实验室标准化委员会(Clinical and Laboratory Standards Institution,CLSI)推荐使用的琼脂稀释法测定[25]。CIP质量浓度为2、4、8、16、32、64 μg/mL和128 μg/mL。E.coliATCC 25922和E.faecalisATCC 29212为标准质控菌。
1.3.3 MMR关键蛋白编码基因及耐药相关编码基因突变检测
采用普通PCR检测MMR关键蛋白编码基因及耐药相关编码基因,引物如表1所示。使用TaKaRa MiniBEST Bacteria Genomic DNA Extraction Kit制备DNA模板,PCR体系为50 μL:上、下游引物各0.6 μmol/L,rTaq或LATaq酶2.5 U,DNA模板500 ng。反应条件:94 ℃预变性10 min;94 ℃变性1 min,相应退火温度下退火1 min,72 ℃延伸1 min,35 个循环;72 ℃延伸10 min,退火温度由相应基因的引物序列决定。取5 μL PCR产物经1%凝胶电泳后于凝胶成像检测。将PCR产物送至上海桑尼生物科技有限公司测序,测序结果采用在线比对软件BLAST进行分析比对(http://www.ncbi.nlm.nih.gov/BLAST/)。
1.3.4 互补实验
依据前期研究(血清型:Salmonellaenteritidis;采样类型:冰鲜鸡翅;采样地点:陕西西安杨凌;采样时间:2010年;耐药谱:AMP-AMC-TCY-SXT-FIS-TMPNAL)筛选103D2作为受体菌[24]。103D2为UvrD(Val 440 Ala)突变及MutS沉默突变型高频突变子。采用电转化法将携带野生型mutH、mutL、mutS及uvrD基因的质粒转入感受态103D2中,通过含有100 μg/mL AMP的LB平板筛选转化后阳性克隆[7],并分别命名为103D2:P-mutH、103D2:P-mutL、103D2:P-mutS和103D2:P-uvrD。
1.3.5 real-time PCR分析
互补菌株耐药相关基因表达水平采用real-time PCR法进行测定,引物如表1所示。方法如下:分别取5 µL 103D2、103D2:P-mutL、103D2:P-mutS、103D2:P-mutH和103D2:P-uvrD过夜培养菌悬液,转接至20 mL含有CIP(2 μg/mL)的LB液体培养基中,37 ℃、150 r/min培养8 h。采用试剂盒对总RNA提取及反转录,通过Nano 200核酸蛋白测定仪测定cDNA含量及质量。反应体系为25 µL:12.5 μL 1×SYBR Premix ExTaqII,上、下游引物各0.4 μmol/L,cDNA 100 ng,ddH2O 8.5 μL。反应在Bio-Rad iQ5 Multicolor Real-time PCR System进行,运行条件为:95 ℃预变性30 s;95 ℃变性5 s,相应退火温度退火30 s,40 个循环;60 ℃延伸30 s。每个基因每个菌株做3 次平行,以gyrB基因为内参基因[26],以等量无核酸酶水替代RNA为无模板对照进行质控,采用2-ΔΔCt方法,进行不同基因在mRNA水平相对表达量差异分析[26-27]。
1.4 数据统计分析
利用SPSS ver.19.0软件进行数据处理,mut基因缺陷与QRDR突变及CIP MIC间相关性分析采用χ2检验或Fisher精确检验,组内变化采用单因素方差分析,103D2与4 种互补菌株中耐药相关基因表达差异采用GLM程序进行方差分析,P<0.05,差异显著。互补菌株与耐药相关基因表达差异、突变频率间的关系则采用R软件包进行分析[28]。
2 结果与分析
2.1 沙门氏菌高频突变子CIP药敏性及耐药相关基因突变
90 株沙门氏菌高频突变子中有89 株(98.9%)对CIP耐药,其中CIP MIC为4 μg/mL共4 株(4.4%),MutS突变1 株(33.3%);16 μg/mL共16 株(17.8%),其中MutS突变11 株(68.8%);32 μg/mL共32 株(35.5%),其中MutS突变11 株(34.4%);64 μg/mL 29 株(32.2%),其中MutS突变17 株(58.6%);128 μg/mL 9 株(10.0%),其中MutS突变9 株(100.0%)。通过CIP对高频突变子MIC及MutS突变关联分析发现,除CIP MIC为16 μg/mL以外,MutS突变检出率随着CIP MIC升高而增加(P<0.05),表明沙门氏菌高频突变子MutS缺陷与CIP耐药性具有潜在相关性(图1)。

图1 不同CIP MIC下超级突变子MutS突变情况Fig.1 Distribution of MutS mutation among different CIP MICs
测序结果表明,沙门氏菌高频突变子中GyrA及ParC蛋白发生突变的菌株分别为53 株(58.9%)及69 株(76.7%),所有GyrA突变均含有87位氨基酸突变(Asp 87 Asn或Asp 87 Gly),ParC突变均含有80位氨基酸突变(Ser 80 Arg),少数菌株还含有ParC 57位氨基酸突变(Thr 57 Ser)及GyrA 83(Ser 83 Phe)、89位氨基酸突变(IIe 89 Val)(表2)。

表2 90 株沙门氏菌高频突变子QRDRs氨基酸突变Table 2 Amino acid substitutions in QRDRs of 90 Salmonella hypermutators
2.2 突变频率与MMR突变及QRDR突变关联分析
90 株高频突变子中,49 株(54.4%)MMR系统中主要功能蛋白MutS突变(MutS-),且这49 株突变位点均为Val 246 Ala突变。在49 株MutS突变株中,GyrA及ParC突变检出率分别为67.3%(33 株)及87.6%(43 株);在MutS未突变菌株中,GyrA及ParC突变检出率分别为48.8%(20 株)及63.4%(26 株),其中ParC在MutS突变和未突变株中突变检出率具有显著性差异(图2、表3)。
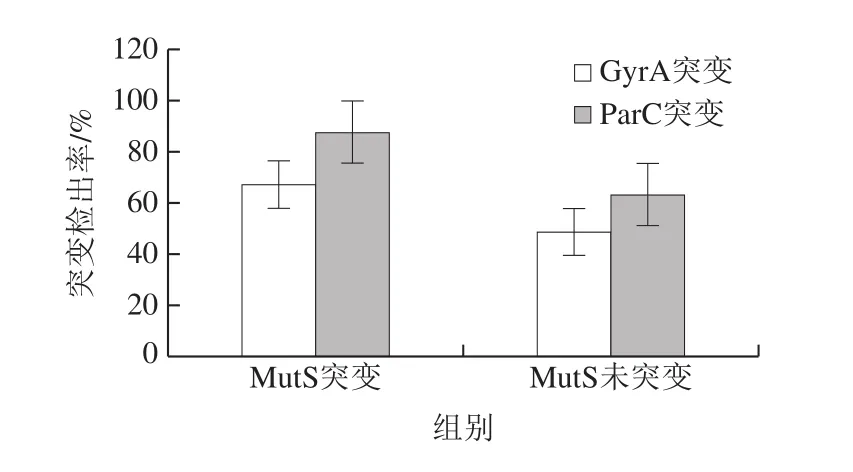
图2 GyrA、ParC突变检出率与MutS突变间的关系Fig.2 Relationship between MutS mutation and GyrA and ParC mutation
使用RIF筛选时,沙门氏菌高频突变子突变频率分布在4.05×10-8~2.37×10-1之间,使用NAL筛选时突变频率分布在1.34×10-1~9.71×10-1之间,绝大多数菌株分布在10-4~10-1之间。MutS突变检出率随着突变频率升高而增加,且具有显著差异(P=0.018)(图3、表3)。不同突变频率下gyrA(P=0.986)与parC(P=0.417)突变检出率,以及不同CIP MIC下,gyrA(P=0.865)与parC(P=0.164)突变检出率并未发现具有显著差异。
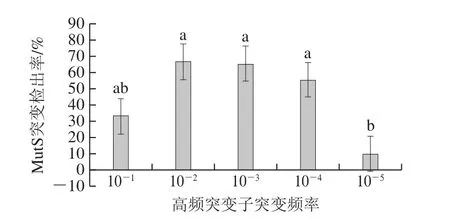
图3 不同突变频率下高频突变子MutS突变情况Fig.3 Distribution of MutS mutation among different mutation frequencies
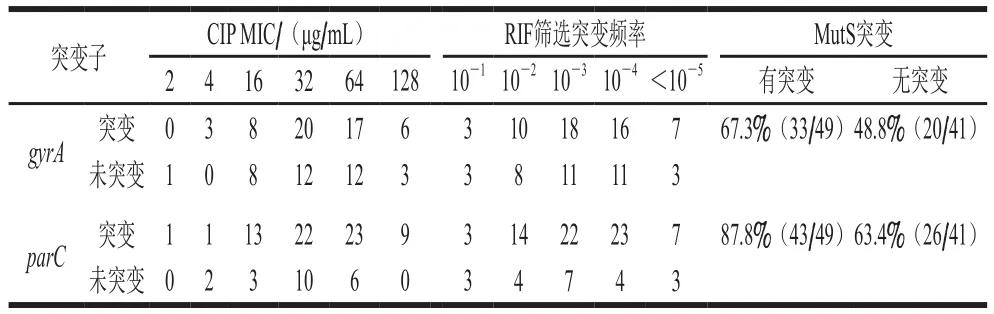
表390 株沙门氏菌高频突变子CIP MIC、QRDR突变、RIF筛选突变频率及MutS突变分布Table 3 CIP MICs against and QRDR mutation, rifampin mutation frequency, and MutS mutation of 90 Salmonella hypermutators
2.3 互补实验
携带野生型mutH、mutL、mutS及uvrD基因的质粒转入103D2,互补前后耐药相关基因对应的氨基酸突变情况并未发现变化(数据未显示)。RIF筛选下103D2突变频率((4.05±0.589)×10-8)与4 株互补菌株间突变频率((3.87±0.387)×10-8~(4.13±0.150)×10-8)并没有显著变化(P>0.05)。而NAL筛选下103D2:P-mutL突变频率((3.99±0.189)×10-1)、103D2:P-uvrD突变频率((3.97±0.27)×10-1)及103D2:P-mutS突变频率((5.34±0.378)×10-1)均显著下降(表4)。虽然CIP MIC由5 μg/mL(103D2)降至3.75 μg/mL(103D2:P-mutS、103D2:P-uvrD),但统计学分析并无显著差异(表4)。

表4103 D2和互补菌株突变频率及CIP MIC变化Table 4 Mutation frequencies of and CIP MICs against 103D2 and its four transformants
2.4 real-time PCR分析
除gyrA、hilA、acrA、dnaQ及soxS以外,互补菌株在其他耐药相关基因的表达与103D2具有显著差异(P<0.05)(表5、图4)。其中tolC(外排泵基因)与marA(多重耐药相关转录激活因子)在互补前后表达量具有极显著差异(P<0.01)。103D2:P-mutH中acrE、parC、tolC、marR及mutT基因表达显著上调,acrB、marA、ompF及ompR基因显著下调,其中parC、tolC及hilA基因表达主要受mutH互补影响(表5、图4)。103D2:P-mutL中acrB、ompF、tolC及mutT基因表达显著上调,parC及marA基因表达显著下调,其中parC、hilA及soxS表达主要受mutL互补影响(表5、图4)。103D2:P-mutS中acrE、tolC、marR及ompR基因表达显著上调,acrB、parC及marA基因表达显著下调,其中ompF、acrB及acrE表达主要受mutS互补影响(表5、图4)。103D2:P-uvrD中parE及marR基因表达显著上调,tolC、marA、ompR及ompF基因表达显著下调,其中marA主要受uvrD互补影响(表5、图4)。总体来看,对103D2进行互补后,marA基因表达量显著下调。除103D2:P-uvrD外,其余互补株在互补后tolC基因表达显著上调。
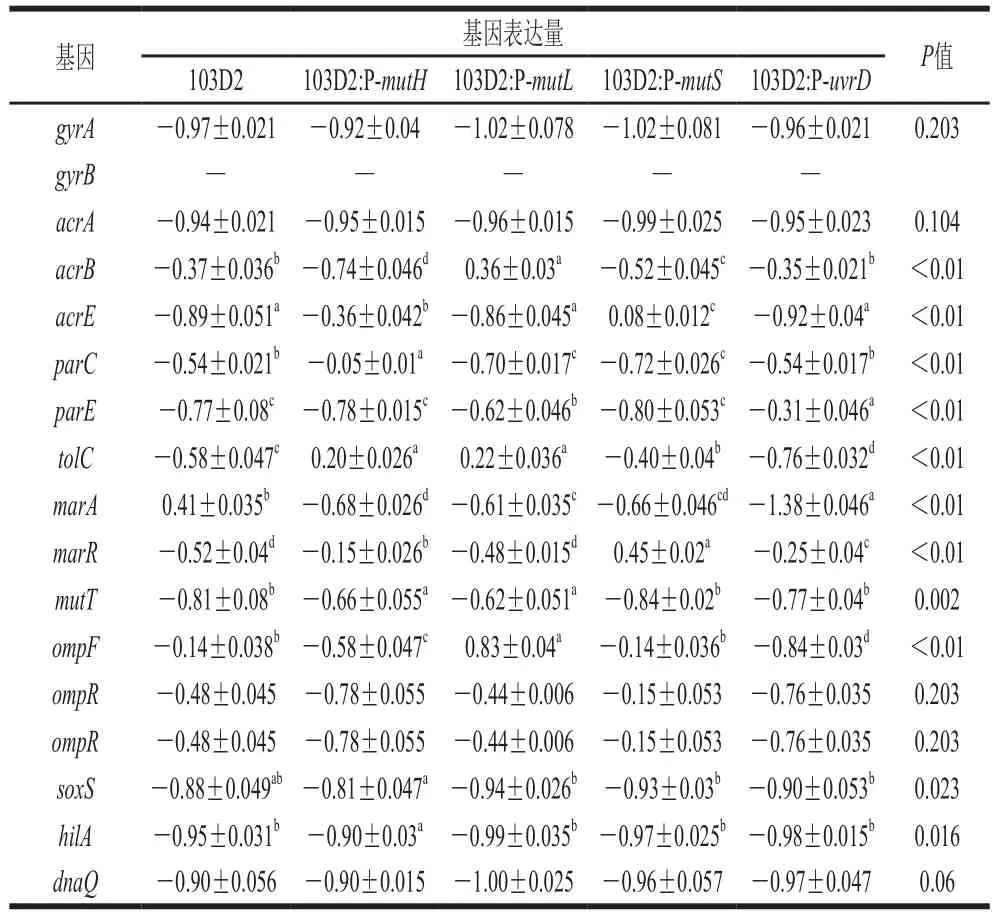
表5 103D2及互补菌株耐药相关基因表达差异Table 5 Differential expression levels of antibiotic resistant genes in 103D2 and its four transformants
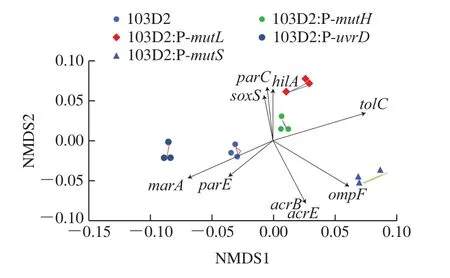
图4 mut基因转入对耐药相关基因表达的影响Fig.4 Effect of exogenous mut genes on the expression of antibiotic resistance genes
在103D2及其互补株中,tolC及acrA表达量与CIP MIC显著正相关,而tolC表达与突变频率(NAL、RIF)间并无明显相关性。acrA表达与NAL筛选下突变频率正相关,与RIF筛选下突变频率负相关。ompF、ompR与RIF筛选下突变频率显著正相关。marR表达与CIP MIC负相关,但与突变频率(NAL、RIF)间并无明显相关性。不同基因间,marR与acrA、marA、acrB及parE均负相关,与其他基因间未有明显正相关(图5)。
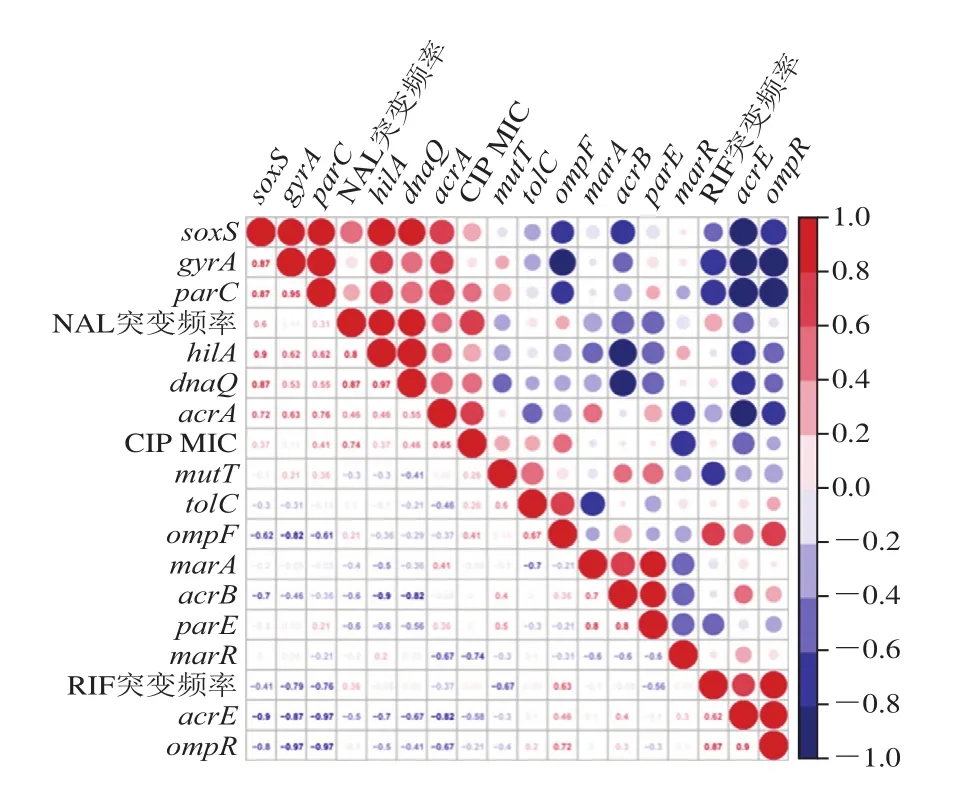
图5 103D2及其互补株中耐药相关基因表达、突变频率及CIP MIC相关性分析Fig.5 Correlation between antibiotic resistance genes and mutation frequency of and CIP MIC against 103D2 and its four transformants
3 讨论与结论
NAL或RIF筛选下,细菌的突变频率在4×10-8~4×10-7之间为弱突变子,突变频率≥4×10-7为强突变子[29-30]。本研究中90 株食源性沙门氏菌中除1 株在RIF筛选下为弱突变子,其余为强突变子,在NAL筛选下均为强突变子。而国内外有关沙门氏菌[5]、结核杆菌(M.tuberculosis)[28]、肺炎克雷伯菌(Klebsiella pneumoniae)[17]、E.coli[16,21,29]、阴沟肠杆菌(E.cloacae)[14]及金黄色葡萄球菌(S.aureus)[28]类似研究中,强突变子检出率均低于弱突变子。导致结果差异的主要原因可能是:1)菌株种属不同;2)菌株来源地不同,菌株来源国家、地点及环境不同会导致其生理特性产生差异;3)菌株采样样品不同,相似研究中绝大多数供试菌株分离自人类临床感染,而本研究供试菌株均分离自食品。因此,尽管突变频率筛选方法一样或者类似,突变频率分布也有差异[19,31-32]。
研究并未发现沙门氏菌高频突变子的突变频率与CIP MIC间显著相关,早前研究也有类似结论[17]。然而也有相关研究发现突变频率的小幅增加便可显著提高氟喹诺酮类抗生素对细菌的MIC[14,16,28,33]。除MMR系统外,细菌细胞内还有很多自修复系统及应答系统(例如SOS系统、直接修复、切除修复、碱基切除修复等),均可以阻止细菌细胞自发突变频率及耐药性增加[31]。虽然有研究表明SOS修复系统对降低细菌对喹诺酮类药物敏感性及突变频率影响非常有限,但其他修复系统仍可以一定程度上降低由MMR系统缺陷引起的突变频率及氟喹诺酮类抗生素敏感性升高[9]。
前期Salmonella、E.coli及M.tuberculosis高频突变子相关研究发现MMR系统缺陷可显著提高细菌突变频率,增加抗生素作用靶位点基因突变或影响耐药相关基因表达(例如:外排泵激活、外膜蛋白改变),从而使突变子耐药表型增加耐药能力增强[12,17,33-34]。细菌氟喹诺酮类抗生素耐药性主要与喹诺酮耐药决定区中GyrA、GyrB、ParC及ParE蛋白突变有关[28,35-36]。尽管本研究在突变频率与CIP MIC间、突变频率与QRDR突变检出率间和CIP MIC与QRDR突变检出率间并没有发现显著的相关性,但MutS缺陷型高频突变子中QRDR突变检出率显著高于MutS正常的突变子(表3)。由此可以发现MMR缺陷的确能提高耐药相关蛋白突变频率,从而影响细菌耐药性。
国内外大量研究表明,mutS、mutH、mutL及uvrD缺陷是细菌产生高频突变表型及高水平耐药的最主要因素[14,17-18,20,28,37-40]。本研究中野生mut基因转入高频突变子103D2后,NAL筛选下突变频率显著降低(表4)。Sheng Huanjing[13]及Yang Baowei[8]等研究发现,向MMR缺陷型Salmonella和E.coli高频突变子中转入野生mut基因后,其突变频率显著降低,这与本研究结果相似。然而RIF筛选条件下103D2互补菌株突变频率并没有显著降低,主要原因可能是:1)前期类似研究中菌株RIF筛选下突变频率在10-7~10-5之间,远高于本研究中103D2 RIF筛选下突变频率10-8。因此尽管互补野生mut基因后103D2突变频率有所降低,但由于数值较小,统计学计算并没有显著差异;2)由于103D2并非工程菌,而是自然条件下从食品样品中分离的高频突变子,虽然与类似研究中研究对象都是Salmonella,但本研究中103D2是否有其他基因突变影响突变频率尚未可知。
从实验结果可发现沙门氏菌高频突变子CIP MIC与MutS突变呈正相关。然而在CIP MIC为16 μg/mL时,MutS突变株高于MIC为32 μg/mL及64 μg/mL(图1),该差异产生的主要原因可能与其他耐药因素有关。虽然细菌CIP耐药性与MMR缺陷呈正相关,但同时也受其他因素影响,例如,抗生素结合位点突变(例如GyrA、ParC),外排泵系统非正常表达(例如AcrAB-TolC),以及染色体基因外编码的抗生素水解酶等[12-13,23]。
转入野生mut基因后,103D2 CIP MIC从5 μg/mL降至3.75~4.0 μg/mL。为探究差异原因,研究采用realtime PCR对耐药相关基因表达差异进行测定。与103D2相比,103D2:P-mutS及103D2:P-uvrD中marR基因表达上调,marA基因表达下调。尽管103D2:P-mutS中acrA没有明显变化,103D2:P-uvrD中acrA及acrB没有明显变化,但是103D2:P-mutS中acrB及103D2:P-uvrD中tolC均显著下调(表5、图4)。有研究表明marR的钝化可以增加marA的表达,外排泵编码基因acrAB及tolC转录会增加,从而提高CIP抗药能力[38-39]。本研究4 株互补株中marR基因表达与其CIP MI、acrA、marA、acrB表达变化均呈负相关,该结果也进一步证实该结论(图5)。因此,推测103D2:P-mutS及103D2:P-uvrD有可能是通过影响外排泵基因表达降低对CIP耐药能力。然而,由于耐药相关基因不同系统间相互作用、相互补偿、相互影响,因此MutS及UvrD具体是通过哪种路径,以及103D2 CIP MIC的减少是否直接由外排泵基因表达异常所引起,仍需要更进一步的研究。
尽管本研究中高频突变子突变频率与QRDR突变和CIP MIC间并没有显著的相关性,但MutS缺陷型高频突变子中QRDR突变比例显著高于MutS正常的突变子。同时研究结果发现MMR缺陷可影响外排泵和膜编码基因marA、marR和tolC的表达,进一步影响沙门氏菌高频突变子的耐药性及突变频率。总之,MMR系统缺陷可通过影响食源性沙门氏菌高频突变子突变频率与耐药相关基因表达影响突变子对抗生素耐药能力。研究其影响机制对控制高水平耐药沙门氏菌的传播,保障食品安全具有重要意义。
