利用全基因组关联分析挖掘粳稻直链淀粉含量调控基因
2021-11-16郑洪亮孙世臣丁国华王彤彤赵宏伟王敬国刘化龙邹德堂来永才
郑洪亮,孙世臣,丁国华,王彤彤,赵宏伟,王敬国,刘化龙,韩 笑,邹德堂,来永才
(1.黑龙江省农业科学院博士后科研工作站,哈尔滨 150086;2.东北农业大学寒地粮食作物种质创新与生理生态教育部重点实验室,哈尔滨 150030;3.黑龙江省农业科学院,哈尔滨 150086)
淀粉作为稻米主要成分,占稻米干重90%左右,其组分主要包括直链淀粉和支链淀粉,其中直链淀粉含量是影响水稻蒸煮食味品质重要因素,研究认为稻米直链淀粉含量、结构和组成影响胶稠度、糊化温度[1],且稻米蒸煮食味与直链淀粉含量呈显著负相关,与胶稠度呈显著正相关[2]。因此,挖掘稻米直链淀粉含量QTLs/基因,对于改良稻米品质具有重要意义。
稻米直链淀粉含量是由多基因控制的数量性状,利用正向遗传学和反向遗传学方法挖掘到与直链淀粉含量相关QTLs/基因。目前,已利用传统QTL分析方法从32个遗传群体中定位直链淀粉含量相关QTL 141个[3],但由于不同研究人员采用试验材料遗传背景差异较大,且选用分子标记不同,导致定位结果差异较大。李修平等进一步采用元分析方法对141个QTL构建一致性图谱,最终映射得到30个“一致性”QTL[3]。目前,已克隆水稻直链淀粉相关基因11个,包括位于第6染色体上的颗粒结合型淀粉合成酶基因Wx,也是公认的水稻直链淀粉含量主效基因[4],位于第6和第8染色体上与可溶性淀粉合成酶相关的3个基因SSSI[5]、SSIIa[6]和SSIIIa[7],位于第1、3、5、7、8、9染色体上与ADP葡萄糖焦磷酸化酶相关的6个基因[8]:AGPL1、AGPL2、AGPL3、AGPL4、AGPS1、AGPS2a,以及位于第2染色体上与淀粉分支酶相关的基因SBEIIb[9]。但以上研究结果对于解析稻米直链淀粉含量复杂的遗传基础仍不充分。
除传统QTL分析外,全基因组关联分析是另外一种解析复杂性状遗传基础的重要方法,具有群体构建简单,表型变异丰富,且可在同一位点上分析多个等位基因,定位精度高等优势。目前稻米品质相关研究应用广泛,如Chen等利用收集世界各地527份水稻品种为试验材料,结合3 916 415个SNP对稻谷籽粒4种贮藏蛋白作全基因组关联分析,共检测到34个位点与4种贮藏蛋白显著关联,其中有28个位点与已知QTLs/基因处于相同或相近位置[10]。Qiu等以272份籼稻品种构成的自然群体为试验材料,利用18 824个高质量SNP对粒长、粒宽、长宽比、粒厚、千粒重、垩白度、垩白率、糙米率等10个性状作全基因组关联分析,共检测到38个显著关联QTL,其中有5个QTL为已知QTLs/基因[11]。但利用全基因组关联分析研究粳稻直链淀粉含量遗传基础及基因挖掘的报道较少。
本研究以收集的295份温带粳稻种质组成的自然群体为试验材料,于2019~2020年测定稻米直链淀粉含量,结合高通量测序获得788 396个多态性SNP作GWAS分析,并针对重要QTL区间挖掘候选基因,以期为解析粳稻直链淀粉含量遗传机制及利用分子育种手段改良稻米品质奠定基础。
1 材料与方法
1.1 试验材料
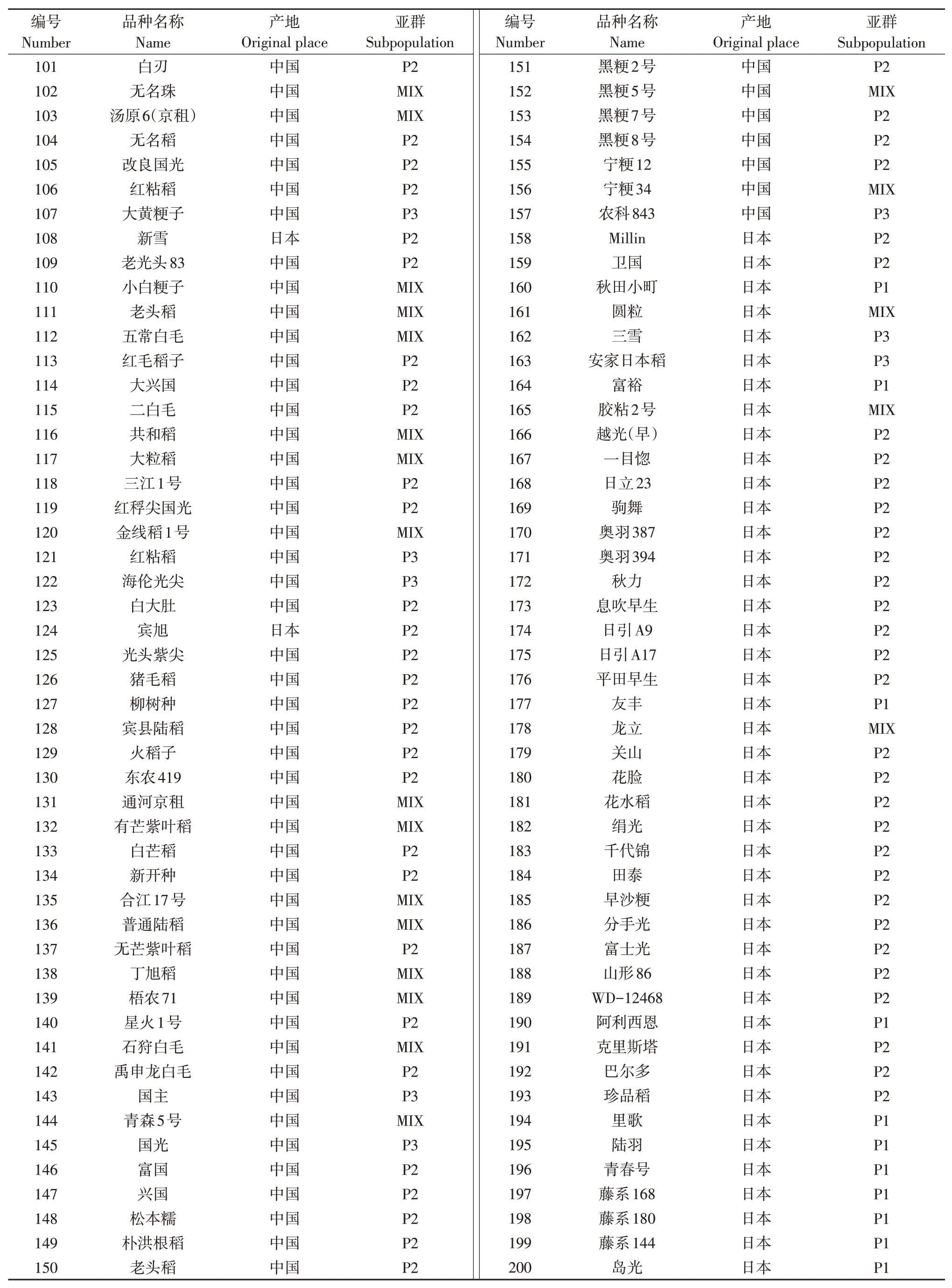
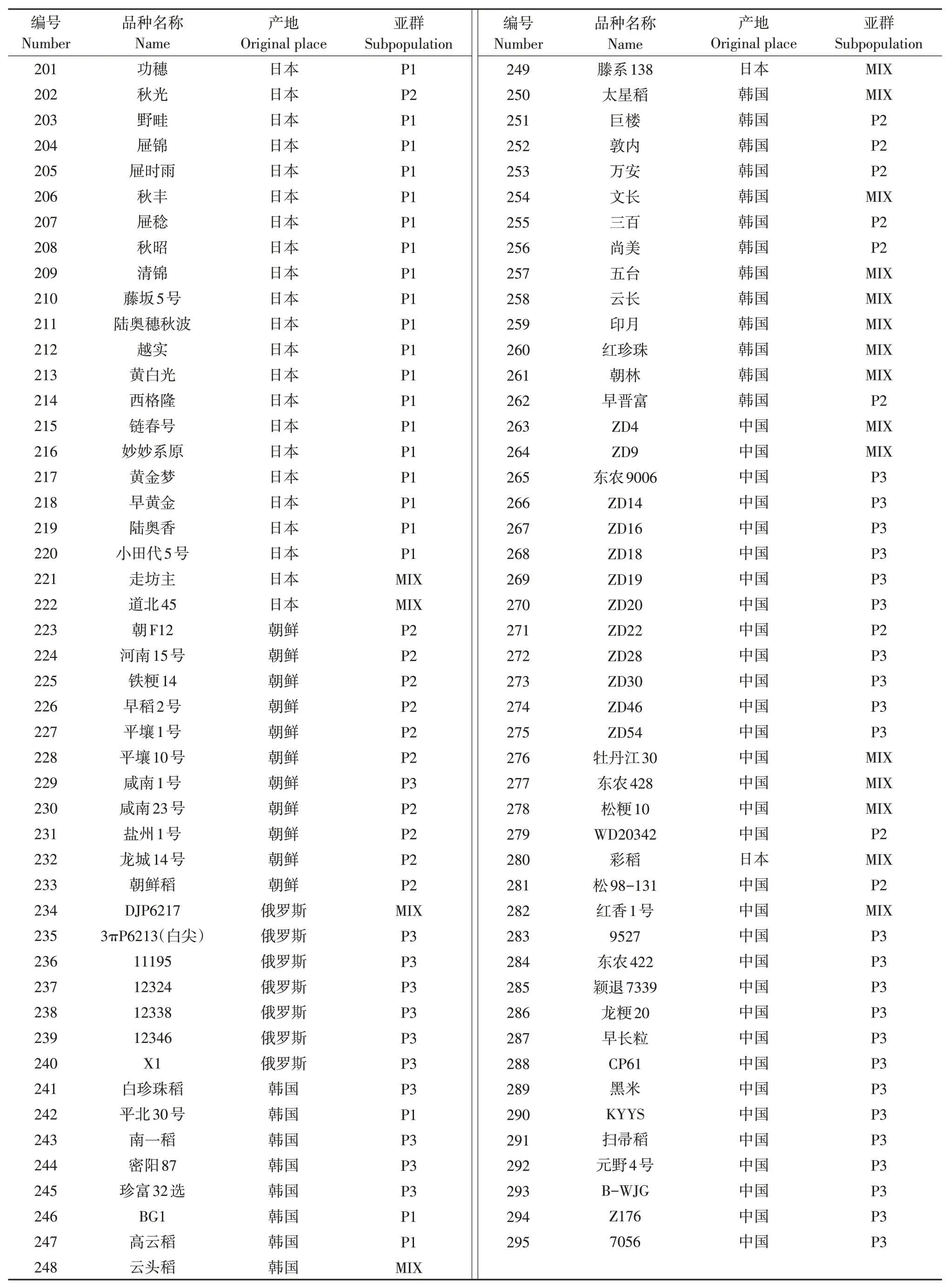
以国内外收集的295份温带粳稻种质构成的自然群体为试验材料,国内材料主要来自于黑龙江、吉林、辽宁和宁夏,国外材料主要来自于日本、韩国、朝鲜和俄罗斯。具体材料明细见表1。

表1 295个粳稻品种具体信息Table 1 Detailed information of 295 japonica rice varieties
1.2 田间种植及稻米直链淀粉含量测定
2019~2020年将所有供试材料种植于东北农业大学阿城实验实习基地水稻试验基地,4月15日播种,5月20日插秧,插秧密度为30 cm×16.7 cm,4行区,每行40株,田间采用随机区组设计,3次重复,田间管理同当地大田生产。于水稻成熟后收获籽粒,置于40℃烘箱中烘干48 h至水分含量为14%,经糙米机、精米机及磨粉机加工后,用于稻米直链淀粉含量测定,测定方法参照国家标准(GB/T17891-2017)[12]。
续表
续表
1.3 全基因组重测序、群体结构、亲缘关系及连锁不平衡分析
本研究所用自然群体基因型数据获取及群体结构、亲缘关系、连锁不平衡分析等工作前期实验室已完成,具体分析过程及结果如下:①利用Illumina HiSeq XTen平台作高通量测序,平均测序深度14.62×,以粳稻品种日本晴(IRGSP-1.0)为参考基因组,采用BWA软件序列比对,采用GATK软件“Best Practice”作群体SNP检测,共获得3 437 749个多态性SNP标记,进一步利用Plink软件作数据质控,最终筛选出最小等位基因频率(MAF)>5%,且缺失率(Missing data)<20%的788 396个SNP用于后续分析;②利用ADMIXTURE软件分析群体结构[13],结果表明,当K=3时,群体CV值最小,因此将群体划分为3个亚群,并将其对应Q矩阵用于后续关联分析;③利用Tassel 5.0软件评估群体材料间亲缘关系[14],结果表明,亲缘关系系数小于0.1材料占78.8%,>0.5材料仅有0.4%,因此本研究所用群体材料间亲缘关系较远,对后续GWAS分析结果影响较小;④利用PopLDdecay软件计算得到水稻全基因组r2值[15],依据Huang等方法[16],将r2衰减到最大值一半时对应的物理距离作为LD衰减距离,经计算,位点间最大r2值为0.84,群体LD衰减距离为109.7 kb。
1.4 稻米直链淀粉含量全基因组关联分析
利用Tassel 5.0软件混合线性模型(Q+K)对稻米直链淀粉含量作GWAS分析[14],采用Li等方法通过GEC软件计算有效独立SNP数目,最终将P<5.46×10-6作为显著性关联阈值[17]。如果在LD区间内有多个显著SNP存在,则将这些SNP视为同一个QTL,结果中仅列出P值最小的SNP作为峰值SNP,且峰值SNP贡献率代表QTL贡献率,以峰值SNP位置上下游分别增加109.7 kb(LD衰减距离)作为QTL区间范围。GWAS分析结果的曼哈顿图和Q-Q图使用R语言中“qqman”软件包绘制。
1.5 候选基因单倍型分析
将两年共同检测且不含已知基因的QTL作为重要QTL,根据区间内基因注释结果,分析区间内所有基因单倍型。具体操作过程参考文献[18]:①根据水稻注释数据库(http://rapdb.dna.affrc.go.jp/)获取QTL区间内全部基因;②利用水稻3K RGP的Rice SNP-Seek Database网站提取所有基因非同义突变SNP[19],结合QTL区间内所有SNP确定本研究自然群体最终非同义突变SNP;③对具有非同义突变SNP的所有候选基因作单倍型分析;④对不同单倍型(≥10份材料)直链淀粉含量作方差分析,筛选出具有显著性差异基因,并结合基因功能注释和前人研究结果确定候选基因。
2 结果与分析
2.1 粳稻品种稻米直链淀粉含量表型分析
通过测定2019~2020年295份粳稻材料直链淀粉含量。结果表明,直链淀粉含量在两年内均表现出丰富的表型变异且趋势一致,总体上,2020年各品种直链淀粉含量略高于2019年,两年平均值分别为19.96%和20.37%,变异范围分别为15.21%~25.34%和15.55%~25.68%,变异系数分别为12.12%和11.59%(见表2)。自然群体偏度值和峰度值绝对值均小于1,表明直链淀粉含量两年表型分布呈近似正态分布,符合典型数量性状遗传特征(见图2)。

图2 粳稻群体中直链淀粉含量频率分布Fig.2 Frequency distribution of the amylose content in 295 japonica rice

表2 295份粳稻种质直链淀粉含量表型值统计分析Table 2 Phenotypic analysis of amylose content in 295 japonica rice germplasms
2.2 稻米直链淀粉含量全基因组关联分析
利用Tassel 5.0软件混合线性模型(MLM)对295份粳稻品种直链淀粉含量作全基因组关联分析,曼哈顿图和QQ散点图(见图3)。在显著性阈值P<5.46×10-6条件下,2019~2020两年共检测到与直链淀粉含量相关QTL 12个,分布在水稻第3、4、11和12染色体上,贡献率范围为8.78%~11.62%。2019年和2020年均检测到7个QTL,其中qAAC4-2和qAAC12-2在两年中重复检测到,qAAC4-2表型贡献率两年中分别为11.12%和9.15%,qAAC12-2表型贡献率两年中分别为10.62%和10.30%。根据全基因组LD衰减距离,最终将qAAC4-2和qAAC12-2分别定位于水稻第4和12染色体20.27~20.49 Mb和19.14~19.36 Mb(见表3)。

表3 粳稻淀粉相关性状显著相关位点Table 3 Significant correlation loci of starch related traits in japonica rice

图3 粳稻直链淀粉含量全基因组关联分析结果曼哈顿图和QQ散点图Fig.3 Manhattan plot and quantile-quantile(Q-Q)plots of genome-wide association studies for the amylose content in 295 japonica rice
2.3 稻米直链淀粉含量候选基因单倍型分析
针对两年中同时检测到的2个QTL(qAAC4-2和qAAC12-2)区间内所有基因分析单倍型。qAAC4-2位于水稻第4染色体20.27~20.49 Mb区间内,该区间包含32个基因,单倍型分析结果表明,共有6个基因不同单倍型直链淀粉含量存在显著差异(见图4a~f)。

图4 候选基因不同单倍型之间直链淀粉含量箱线图Fig.4 Boxplots of amylose content between different haplotypes of candidate genes
LOC_Os04g33520非同义突变SNP分为两种单倍型,Hap2(CA)显著大于Hap1(TG)(见图4a);LOC_Os04g33590被非同义突变SNP分为两种单倍型,Hap2(A)显 著 大 于Hap1(G)(见 图4b);LOC_Os04g33640非同义突变SNP分为两种单倍型,Hap2(T)极显著大于Hap1(G)(见图4c);LOC_Os04g33660被非同义突变SNP分为两种单倍型,Hap2(G)显著大于Hap1(A);LOC_Os04g33700被非同义突变SNP分为两种单倍型,Hap2(CCTACC)显著大于Hap1(TACGTG);LOC_Os04g33710非同义突变SNP共分为两种单倍型,Hap2(CT)显著大于Hap1(AG)(见表4)。

表4 候选基因单倍型分组及每种单倍型SNP组成Table 4 Candidate gene haplotype group and the composition of each haplotype SNP
根据基因功能注释(见表5),LOC_Os04g33640编码糖苷水解酶,即一种水解糖苷键的酶[22],该酶对糖和糖缀合物水解与合成具有调节作用[23],推测其可能影响淀粉链长度和分支[24],进而影响直链淀粉及支链淀粉含量。因此,推测LOC_Os04g33640最可能为qAAC4-2候选基因。另外一个QTL,qAAC12-2位于水稻第12染色体19.14~19.36 Mb区间内,该区间包含27个基因,单倍型分析结果表明,该QTL区间内所有基因不同单倍型直链淀粉含量差异不显著。

表5 候选基因基因注释Table 5 Candidate gene of gene annotation
3 讨论与结论
稻米直链淀粉含量除受遗传因素控制外,温度、光照、海拔等环境因素及施肥、收获时期、贮藏时间、碾磨精度等农艺措施均对直链淀粉含量有影响[25]。本研究以295份粳稻种质为试验材料,于2019年和2020年种植于阿城基地,从测定的稻米直链淀粉含量看,2019年直链淀粉含量平均值为19.96%,变异范围为15.21%~25.34%,2020年直链淀粉含量平均值为20.37%,变异范围为15.55%~25.68%,其海拔、施肥、收获时期、贮藏时间、碾磨精度等均不存在差异,而温度和光照在年际间有较大差异,根据试验地气象数据统计,2019年和2020年4~9月份≥10℃活动积温和日照时数分别为2 764℃、1 272.3 h和2 922℃、1 301.3 h,可知2020年活动积温和日照时数比2019年分别增加158℃和29 h。说明温度和光照对稻米直链淀粉含量略有影响,且稻米直链淀粉含量随活动积温和日照时数增加而有所提高。
长期以来针对稻米直链淀粉含量遗传基础解析开展大量研究,除已克隆主效基因Wx外,近年来通过遗传群体和自然群体鉴定到众多QTLs/基因。根据国家水稻数据中心基因数据库(http://www.ricedata.cn/gene/)和水稻QTL数据库(https://archive.gramene.org/qtl/)公布信息,将本研究检测到的12个与直链淀粉相关QTL与前人结果作比较,发现部分QTL与前人已定位QTL位于相同、相近区间或包含已克隆淀粉相关基因。如Fan等通过籼稻品种珍汕97和H94杂交得到的F1构建双倍单倍体(DH)群体为试验材料[20],利用水稻218个SSR标记位点,检测到两个与直链淀粉含量相关QTL(AQGA007,AQGA017),分别与本研究2019年检测到的qAAC3和2020年检测到的qAAC12-3位于相同区间。另外,本研究2020年检测到与直链淀粉含量相关QTL(qAAC4-1)区间内包含可溶性淀粉合酶Ⅲ基因OsSSIIIb,与Zhao等研究结果一致[21]。表明本研究全基因组关联分析的检测结果具有较高准确性,同时也缩小已定位QTL区间。
由于传统基因图位克隆需耗费大量人力和时间,因此克隆影响复杂性状QTL一直是植物遗传学家和分子生物学家面临的重大挑战。采用GWAS方法作QTL分析,并针对QTL区间内所有基因作单倍型分析,筛选出不同单倍型表型值之间存在显著性差异候选基因,再结合基因注释和前人研究结果挖掘候选基因,可提高候选基因筛选效率及准确性[26]。本研究针对两年共同检测到且不含已知基因的两个重要QTL(qAAC4-2、qAAC12-2)分析候选基因单倍型,结果发现qAAC12-2区间内所有基因不同单倍型直链淀粉含量差异不显著,而qAAC4-2区间内共有6个基因不同单倍型直链淀粉含量存在显著差异,其中编码糖苷水解酶的LOC_Os04g33640,对糖和糖缀合物水解与合成具有调节作用。研究表明,α-淀粉酶和β-葡萄糖苷酶均含有这种糖基水解酶结构域,可水解淀粉产生糊精、低聚糖和单糖,从而降低淀粉积累[23-24]。因此,推测LOC_Os04g33640可能具有类似α-淀粉酶功能。下一步将重点针对LOC_Os04g33640开展转基因功能验证和分子育种利用,为粳稻品质改良提供理论依据。
