野生动物源肠杆菌科细菌鉴定及同源性分析
2021-10-28刘龙海黄淑芳应志豪张秀秀陈玎玎龚利洋郑应婕
刘龙海,黄淑芳,应志豪,张秀秀,陈玎玎,龚利洋,郑应婕
(杭州动物园,浙江 杭州 310008)
肠杆菌科细菌广泛存在于人、动物和自然环境中,肠杆菌科细菌的致病性差异很大,大多数种类为条件性致病菌,可附殖于健康动物的体表和消化道,也有部分细菌为致病性菌,如沙门杆菌属、志贺杆菌属[1]。革兰氏阴性杆菌,特别是大肠埃希氏属是引起园内感染最常见细菌之一,常引发泌尿道感染、肠炎、脑膜炎、败血症等[2]。在野生动物上,肠杆菌科细菌极易感染哺乳动物和禽类,特别是幼龄动物,且死亡率较高[3]。
基于生化反应和抗原构造的分类方法,可将肠杆菌科细菌分为5个族12个属,而通过生化反应和DNA数据分析,分类更加准确细化,可将其分为25个属[4]。16S rDNA聚合酶链式反应是目前细菌鉴定中最为准确、高效的手段,通过与NCBI基因数据库中的已知菌株数据作对比,可快速确定细菌种类[5-6]。同种肠杆菌的亚型分类对肠杆菌遗传进化和溯源有重要意义,主要的方法有血清型分型法、基因重复序列聚合酶链式反应(ERIC-PCR)、脉冲场凝胶电泳(PFGE)、多位点序列分型(MLST)、基质辅助激光解吸飞行时间质谱(MALDI-TOF-MS)等[7]。其中,ERIC-PCR 技术以其操作简单、试验重复性高和分辨力强等优点而被广泛利用,常用于肠道菌群和细菌的同源性分析[8]。Huiyong Duan 等对猪场空气和粪便分离的120株大肠埃希菌进行ERIC-PCR分析,结果发现空气分离菌来自于粪便[9];Soltani等通过凝胶电泳从95个样本中识别出65个不同的禽大肠杆菌菌株,其中有232~2690 bp条带,并在廉价和简单的分子工具中证实ERIC-PCR是一种很好的细菌基因分型技术[10]。在其他细菌的基因分型中,也有研究验证了ERIC-PCR的可行性,李鹏等根据副猪嗜血杆菌 15 种血清型的特异性 ERIC 指纹图谱,对中国东南部不同猪场的111株副猪嗜血杆菌样品进行鉴定,验证了ERIC-PCR在其他细菌基因型分型的应用[11]。
1 材料与方法
1.1材料
1.1.1菌株 从患病野生动物的患处脓液、死亡动物内脏、野生动物生活环境中经分离的细菌,经生化鉴定的肠杆菌科细菌38株。
1.1.2培养基及试剂 生化鉴定试剂盒、营养肉汤培养基、麦康凯培养基、营养琼脂培养基、细菌DNA提取试剂盒、细菌16S rDNA扩增试剂盒(大连宝生物工程有限公司)、超纯水、50×TAE缓冲液(北京索莱宝科技有限公司)、Premix Taq(大连宝生物有限公司)。
1.1.3仪器设备 恒温培养箱(上海一恒科学仪器有限公司)、PCR仪(赛默飞世尔科技)、电泳仪(北京六一有限公司)凝胶成像仪(北京科锐创新科技有限公司)、微量移液枪(Eppendorf)。
1.2方法
1.2.1细菌DNA的提取 使用Takara公司,细菌提取试剂盒提取保存细菌的DNA。
1.2.216S rDNA和ERIC-PCR扩增 使用Takara公司的细菌16S rDNA的鉴定试剂盒,扩增保存肠杆菌DNA的16S rDNA序列,对扩增PCR产物使用1%的琼脂糖凝胶进行电泳,检验是否扩增成功。将扩增产物送华大基因(北京)测序,并将结果在NCBI数据库中比对。
用于ERIC-PCR扩增的引物序列参照文献设计,上游引物为:5′-ATGTAAGCTCCTGGGGATTCAC-3′;下游引物为:5′-AAGTAAGTGACTGGGGTGAG CG-3′。反应条件为94 ℃预变性5 min;94 ℃变性45 s,55 ℃复性45 s,72 ℃延伸3 min,30个循环,72 ℃延伸10 min。对扩增产物使用1%的琼脂糖凝胶进行电泳。
2 结果
2.1肠杆菌科细菌16S rDNA鉴定结果 38株肠杆菌科细菌的16S rDNA扩增片段(部分)琼脂糖凝胶电泳图如图1所示。
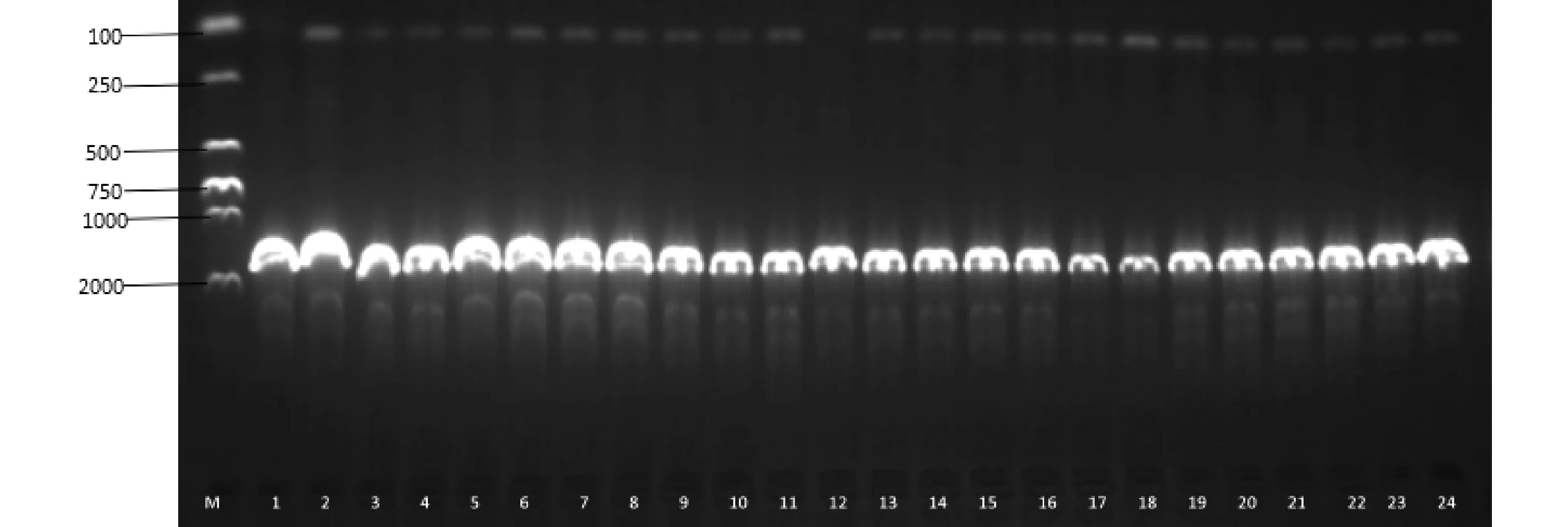
图1 38株肠杆菌科细菌的16S rDNA扩增片段(部分)琼脂糖凝胶电泳图
扩增片段经华大基因公司(杭州)测序后,将测序结果与NCBI数据库已有序列比对,结果发现克雷伯氏菌4株,埃希氏菌27 株,摩氏摩根菌2株,沙门氏菌1株,弗氏柠檬酸杆菌1株,哈夫尼氏菌2株,勒克氏菌1株,如表1所示。

表1 肠杆菌科细菌16S rDNA鉴定结果
使用MEGA 6.0软件,对27株埃希氏菌16S rDNA序列使用临近法构建系统发生树,结果如图2所示。
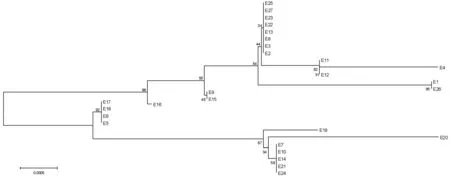
图2 27株埃希氏菌16S rDNA序列系统发生树
从构建的系统发生树中发现,置信度在90%可分为23个亚群,其中E1和E26,E5、E6、E17和E18,进化关系较近。
2.2埃希氏菌ERIC-PCR指纹图谱 对ERIC-PCR扩增产物,使用1%的琼脂糖凝胶进行电泳,部分结果如图3所示。
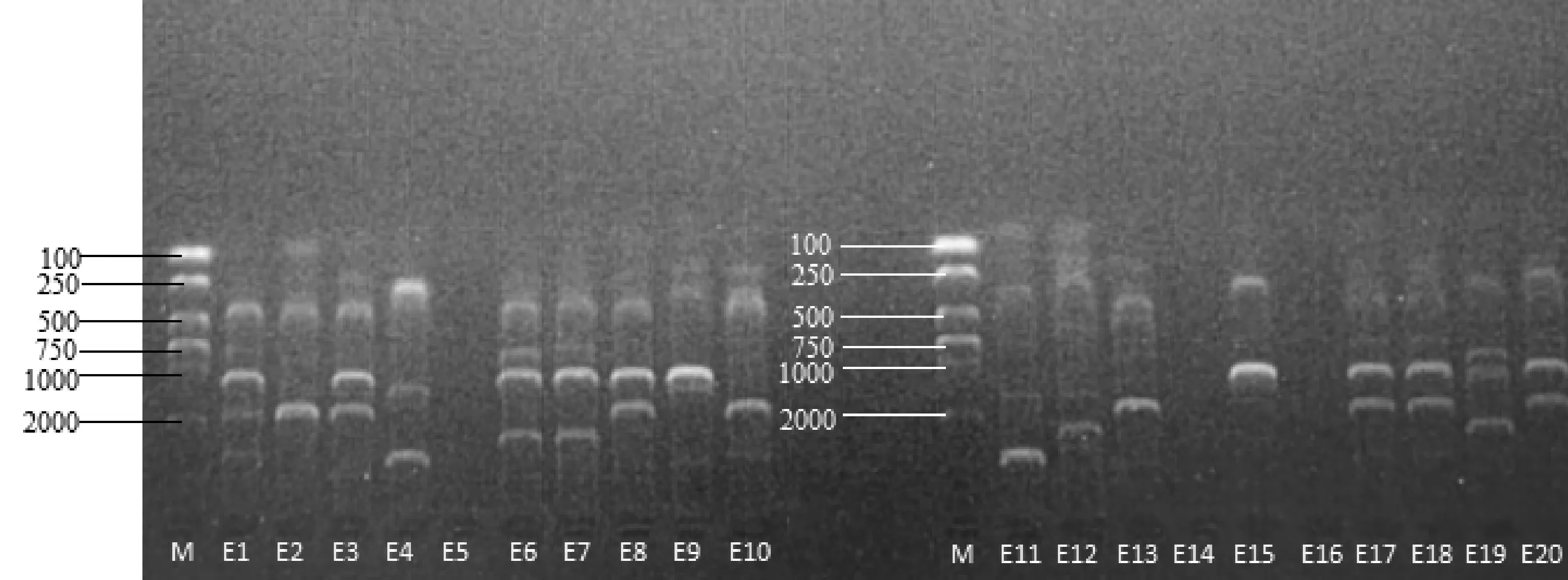
图3 埃希氏菌ERIC-PCR扩增产物(部分)
通过对电泳条带统计,每株埃希氏杆菌扩增出0-8条条带,共扩增出不同条带11种,其中E5、E14、E16未扩增出条带,使用NTSYSpc2.0软件,对扩增条带进行遗传相似度分析,结果如图4所示。
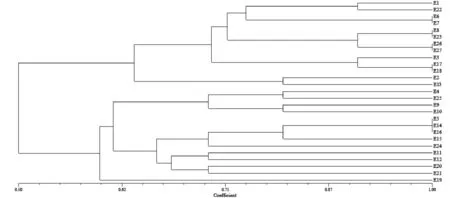
图4 埃希氏菌ERIC-PCR指纹图谱遗传相似度分析
通过对ERIC-PCR 指纹图谱分析,在相似系数为0.87时,埃希氏菌属可分为18个亚群,其中E1和E22,E6和E7,E8和E23,E26和E27,E17和E18遗传相似度较高。
3 讨论
16S rDNA基因是原核生物核糖体小亚基rDNA(16S rDNA)的基因,是细菌分类学研究中最常用、最有用的“分子钟”,全长序列包含10个可变区,可精确指示细菌之间的亲缘关系,所含信息能反应生物界进化关系,易操作,适用于各级分类单元。本研究,通过扩增肠杆菌的16S rDNA的全长序列、测序和比对,确定了临床分离细菌7个属38株肠杆菌科细菌。利用16S rDNA序列信息,进一步通过MEGA6.0软件构建27株埃希氏菌属细菌系统发生树,若把置信度定位在90%时,则可以分为23个亚群。在确定遗传关系上,ERIC-PCR指纹图谱技术比16S rDNA基因分析更具有鉴别性[12]。本文中77.8%(n=21)埃希氏菌属细菌ERIC-PCR电泳结果显示出独特条带,表明菌株之间非相同菌株传播。经遗传聚类分析,在相似系数为0.87时,埃希氏菌属菌可分为18个亚群,显示其传播情况为非同株菌的爆发式传播。
在本研究中,通过16S rDNA序列信息构建的系统发生树中,置信度在90%时,可分为23个亚群,其中E1和E26,E5、E6、E17和E18,进化关系较近;而在通过对ERIC-PCR 指纹图谱分析,在相似系数为0.87时,埃希氏菌属菌可分为18个亚群,其中E1和E22,E6和E7,E8和E23,E26和E27,E17和E18遗传相似度较高。从两种分析结果来看有所差异。然而,在ERIC-PCR 指纹图谱分析时,在相似系数为0.75时,可分为12个亚群,排除未出现条带的E5、E14、E16,有1个亚群包含16S rDNA序列信息构建的系统发生树中置信度大于90%的所有菌株。
本研究中,通过细菌的16S rDNA快速鉴定细菌种类,并结合ERIC-PCR 指纹图谱分析快速分析出埃希氏菌属细菌的遗传关系,在今后肠杆菌科细菌感染的快速鉴定、分类和遗传溯源上有一定的指导意义。然而,在ERIC-PCR 指纹图谱分析中,E5、E14、E16未出现有效条带,需进一步研究,分析原因。
