CsgRNA减少APOBECs在Cas9-D10A介导的基因编辑过程中引入的突变
2021-10-16任蓉蓉陈红全
任蓉蓉,陈红全
1.浙江医院 医学检验科,浙江 杭州 310013;2.浙江大学医学院附属邵逸夫医院 检验科,浙江 杭州 310016
CRISPR/Cas9系统是强大的基因编辑工具,近几年得到广泛的应用[1-4]。近期,研究者将APOBECs(members of the apolipoprotein B mRNA-editing enzyme,catalytic polypeptide-like)蛋白和CRISPR系统紧密的联系在了一起[5-7]。我们的前期研究发现应用Cas9-D10A/sgRNA进行基因编辑时,APOBEC-3B蛋白能够引入非目的突变[5],因此有必要优化Cas9-D10A/sgRNA系统的保真性。应用Cas9-D10A/sgRNA进行基因编辑时,D10A能够切割双链DNA的单链后形成切口样结构,从而暴露出DNA单链,而APOBECs蛋白具有胞苷脱氨酶活性,能够攻击单链DNA的胞嘧啶脱氨使其变为尿嘧啶,使单链DNA发生碱基突变。本研究根据上述现象提出以下理论设想:对Cas9-D10A/sgRNA系统设计csgRNA(chaperone sgRNA),使D10A进行基因编辑时产生的DNA单链结构能够与csgRNA形成碱基互补配对,从而形成双链样结构,降低APOBECs的攻击,以减少APOBECs对Cas9-D10A进行的基因操作形成的单链DNA引入突变,从而降低突变率。
1 材料和方法
1.1 质粒构建与引物设计 Cas9(pST1374-N-NLSFlag-linker-Cas9)、D10A(pST1374-N-NLS-flaglinker-D10A)、H840A(pST1374-N-NLS-flag-linker-H840A)和dCas9(pST1374-N-NLS-flag-linker-dead Cas9)等表达载体由上海科技大学黄行许教授馈赠,sgRNA表达载体由pGL3-U6-sgRNA-PGK-Puro(Addgene 51133)构建完成;sgRNA4和FANCF打靶位点的sgRNAs序列和其各自的上下游PCR引物序列见表1。

表1 sgRNAs序列和引物序列
1.2 细胞培养和转染 293FT、293FT-A3B和293FTA3Bm细胞系由上海科技大学陈佳教授馈赠,所有细胞均在DMEM(10566,美国Gibco公司)+10% FBS(16000-044,美国Gibco公司)、37 ℃条件下培养;细胞以2×105/孔,接种于12 孔细胞培养板,8 h后用LipofectamineTM2000(美国Thermo Fisher Scientific 公司)转染,转染试剂包括125 μL Opti-MEM+2 μg Cas9表达载体+1 μg sgRNA表达载体+3 μL Lipofectamine,24 h后加终质量浓度为 1 μg/mL的嘌呤霉素(ant-pr-1,美国InvivoGen公司)药筛,48 h后用酚氯仿法抽提基因组DNA。
1.3 T7EN1酶切实验 首先用PCR方法扩增sgRNA打靶位点上下游DNA序列,PCR反应程序如下:94 ℃,5 min;(98 ℃,10 s,72~62 ℃,每循环降低1 ℃,15 s;72 ℃,30 s)10个循环,(98 ℃,10 s;62 ℃,15 s;72 ℃,30 s)25个循环;72 ℃,5 min;4 ℃ 保持,PCR产物用PCR清洁试剂盒(美国Axygen公司,AP-PCR-50)纯化,纯化后的PCR产物进行退火(体系为:NEB Buffer2,2 μL,PCR产物,200 μg,纯水补齐至20 μL。退火程序:95 ℃ 5 min;95~85 ℃,每秒降低2 ℃;85~25 ℃,每秒降0.1 ℃;4 ℃保持)。退火产物放于冰上,每个体系加入0.3 μL T7核酸内切酶I,37 ℃孵育25 min,之后用2.5%琼脂糖凝胶电泳(100 V,25 min)。
1.4 统计学处理方法 采用GraphPad Prism5统计学软件进行分析,2组间比较采用独立样本t检验。P<0.05为差异有统计学意义。
2 结果
2.1 APOBEC-3B显著增强Cas9-nickase基因编辑的突变率 有研究表明APOBEC-3B过表达可显著增强Cas9-nickase基因编辑的突变率[5]。本研究首先在293FT-A3B过表达细胞系上检测Cas9-nickase的基因编辑效率。选取sgRNA4和sgFANCF打靶位点,构建载体,脂质体法转染293FT、293FT-A3B和293FTA3Bm细胞系,通过T7EN1酶切实验检测其编辑效率,结果如图1所示,Cas9在三种细胞系上对四个位点均产生了非常明显的切割条带,在293FT-A3B过表达细胞系中,Cas9-D10A对两个位点均产生了明显的切割,Cas9-H840A的切割条带较弱,而在293FT和293FT-A3Bm细胞系则检测不到肉眼可见的切割条带,这表明APOBEC-3B过表达显著增强了Cas9-D10A进行基因编辑时产生的突变。
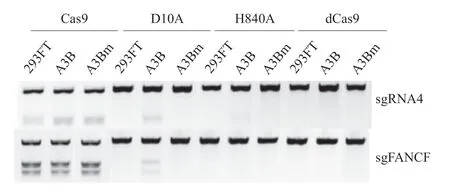
图1 T7EN1酶切凝胶图显示APOBEC-3B过表达显著增加了Cas9-nickase/sgRNA的突变率
2.2 csgRNA降低由APOBEC-3B引起的Cas9-nickase基因编辑突变率 为降低APOBEC-3B对Cas9-D10A进行基因编辑时引入的突变,我们拟通过csgRNA策略予以解决,其原理示意图见图2A。根据以上理论原理,本研究在sgRNA4和sgFANCF位点上设计csgRNA,其长度为20 bp,构建双U6(sgRNA+csgRNA)载体、转染293FT-A3B细胞系,T7EN1酶切实验检测结果见图2B,结果显示在A3B过表达细胞系中,csgRNA降低了D10A在sgRNA4和sgFANCF打靶位点所产生的切割条带,这表明csgRNA策略能够降低D10A进行基因编辑时APOBEC3B蛋白对单链DNA的攻击,从而减少突变的发生。
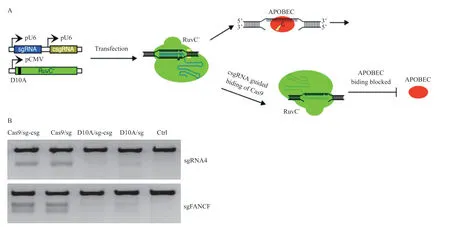
图2 CsgRNA策略对Cas9-D10A基因编辑效率分析
2.3 csgRNA条件的优化 对csgRNA的长短、作用位置进行优化。选取sgRNA4打靶位点,设计了24个csgRNA,其长度和作用位置见图3A,构建双U6载体,脂质体法转染293FT-A3B细胞系,T7EN1酶切实验检测基因编辑效率,结果见图3B。1、2、8、9、10、13号csgRNA其D10A切割条带显著降低了,其csgRNA对Cas9的切割条带无影响。对Cas9和D10A产生的切割条带进行灰度扫描,D10A/Cas9切割条带灰度值比值见图3C,其中1、11、15、21号csgRNA和S4比差异无统计学意义(P>0.05),其他csgRNA差异均具有统计学意义(P<0.05)。8号(csgRNA长度为16 bp、向PAM端延伸2个碱基)和9号(csgRNA长度为20 bp、向PAM端延伸4个碱基)csgRNA既能确保Cas9/sgRNA4的基因编辑效率,对降低D10A/sgRNA4进行基因编辑时APOBEC-3B蛋白引入的突变其效果相对更好一些。
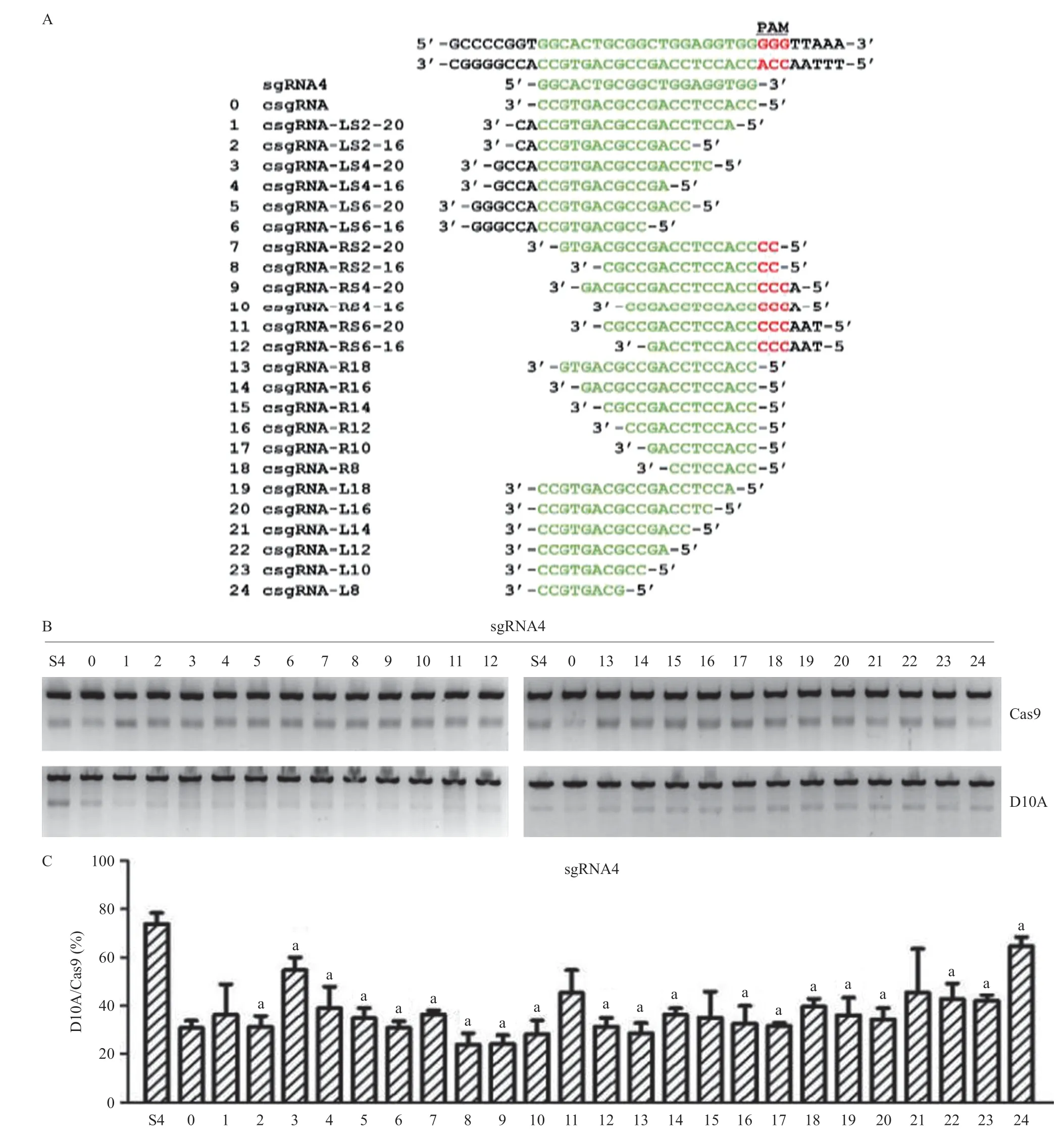
图3 csgRNA长度和位置的优化
2.4 8 号csgRNA在Cas9 nickase上的作用效果 选取8号csgRNA,以sgRNA4和sgFANCF为打靶位点,在293FT-A3B细胞系上进一步验证8号csgRNA的保护性效果,并看其在Cas9-D10A和Cas9-H840A上的作用效果。转染293FT-A3B细胞系,T7EN1酶切结果和D10A/Cas9切割条带灰度值比值见图4,8号csgRNA在sgRNA4和sgFANCF位点上均显著降低Cas9-D10A的表达,差异有统计学意义(P<0.05)。以上结果表明在sgRNA4和sgFANCF打靶位点上,8号csgRNA可明显降低APOBEC-3B对D10A进行基因编辑时产生的突变。

图4 csgRNA在Cas9 nickase上的作用效果
3 讨论
自从CRISPR/Cas9 系统被研究者成功开发出来,其广泛应用于生命科学研究[8-11],但其临床应用(如基因治疗)一直受限于严重的脱靶效应[12-14],因此进一步提高特异性是其临床应用的前提。本研究结果显示,在sgRNA4和sgFANCF位点上,csgRNA 策略对D10A/sgRNA系统有明显保护作用,而对H840A/sgRNA保护性无效果,这是因为H840A蛋白只有RuvC切割活性结构域,RuvC切割DNA非互补链,随后的缺口过程暴露的DNA互补链和sgRNA结合的双链结构占据主要部分,因此csgRNA策略主要降低APOBECs对D10A/sgRNA系统进行基因编辑时产生的突变;在对csgRNA条件进行优化时,发现较短的csgRNA保护性效果不佳,可能是因为D10A蛋白切割DNA单链后暴露的缺口过大造成的,这需要进一步验证。
科学家们在提高CRISPR/Cas9系统保真性方面付出了巨大的努力,如通过优化sgRNA的设计来提高CRISPR系统的保真性[15]、高保真性Cas9蛋白的开发[16]、利用Cas9-D10A结合双sgRNAs策略降低的脱靶效应的研究[17]等,但这些方法均不能有效抑制APOBECs对Cas9-D10A/sgRNA系统进行基因操作时引入的突变[5]。最近研究者将CRISPR-Cas9系统融合ArIIA4-Cdt1 表达元件,其在确保基因编辑效率的同时,可降低脱靶效应[18],这是学者们在提高CRISPR系统保真性上的又一大进步。我们设想,将融合ArIIA4-Cdt1的CRISPR系统与csgRNA策略相结合,对APOBECs高表达的细胞进行基因编辑时,其保真性会进一步提高。
免疫细胞基因编辑在治疗人类各种疾病方面具有广阔的潜力[12,19-21],然而免疫细胞中APOBECs高水平表达并且在细胞激活过程中APOBECs的表达持续上调[5,22],这就使人们对免疫细胞进行基因编辑时更需谨慎。本研究为降低APOBECs对Cas9-D10A/sgRNA系统进行基因编辑时产生的突变提供了csgRNA策略,为其进一步向临床应用,尤其是对APOBECs高表达的初始T细胞进行基因编辑时,做出了一定的贡献。
综上,本研究通过csgRNA策略成功降低了 APOBECs对Cas9-D10A/sgRNA进行基因编辑时引入的突变,为提高CRISPR系统保真性提供参考。
