产茸相关SNP位点在7个梅花鹿群体中的验证
2021-10-14田雪琪李洋刘汇涛王天骄邢秀梅
田雪琪,李洋,刘汇涛,王天骄,邢秀梅
(中国农业科学院特产研究所,特种经济动物分子生物重点实验室,吉林 长春130112)
2020年5月29日,以梅花鹿等16种特种畜禽列入《国家畜禽遗传资源目录》[1]。鹿茸作为传统的中药材在中国已有两千多年的应用历史[2],产茸性能一直是评估梅花鹿优劣的重要指标。梅花鹿的选种育种一直停留在常规表型选种阶段,茸重、茸型、茸色和对称性是梅花鹿茸用品种培育的主要选种依据。分子标记辅助选种仍处于起步阶段,田万年等[3]对双阳梅花鹿胰岛素样生长因子-1(IGF-1)基因的研究,发现在824 bp处存在T→C突变,所产生的AB基因型个体的产茸性能显著高于其他两种基因型个体;杜智恒等[4]对东北梅花鹿生长激素基因(GH)进行研究,发现在第2内含子中存在G→A突变,这个SNP位点不同基因型可能对梅花鹿产茸性状有影响,尤其是对第5锯的产茸量有显著影响趋势;熊家军[5]以两个地区梅花鹿种群作为研究对象,对褪黑素基因(MTNR1A)、生长激素基因(GH)以及雄激素受体基因(AR)的SNPs与产茸量相关性进行了分析,发现在MTNR1A第2外显子中存在G→C的位点突变,对湖北荆门五三梅花鹿种群产茸量具有显著影响;GH基因139 bp处存在C→T突变对吉林东丰地区梅花鹿种群产茸量具有显著影响;AR第3外显子存在的C→T突变对两个种群的产茸量均有显著影响;陈斌[6]对梅花鹿转化生长因子-1基因(TGF-1)的研究,发现在371 bp处的SNP位点存在3种基因型,其中AA基因型与其他两种基因型的产茸量存在显著差异。由此可见,在IGF-1、GH、MTNR1A、AR、TGF-1基因上均存在着与产茸量相关的SNP位点,且不同群体中与产茸相关的SNP位点存在一定差异,见表1。前期实验室在四平梅花鹿、东大梅花鹿两个群体筛选得到8个与产茸性能相关的SNP位点。其中,SNP1427_255为极显著相关,其余SNP位点为显著相关。本试验应用KASP分型技术在7个梅花鹿群体中进行基因分型,并对不同基因型与产茸量做关联分析,以探究东北梅花鹿产茸量相关基因型或SNP组合,为早期选种提供依据。

表1 各研究中与产茸相关SNP位点信息Table 1 The information of SNP locus in studies
1 试验材料和方法
1.1 试验材料
试验用雄性梅花鹿个体共计190头,分别采集于吉林省辽源市东丰县文福鹿场、吉林省通化市通化县通化山宝鹿业、吉林省长春市双阳区东鳌鹿业、吉林省辽源市东丰县鑫利达鹿业、吉林省梅河口市大成加隆畜禽有限公司、吉林省四平市吉梅花鹿种鹿场和吉林省长春市双阳区鹿业良种繁育有限公司。所有试验个体使用抗凝真空采血管采集颈静脉血约10 mL,带回实验室后﹣20℃保存,用于后续基因组DNA提取。同时记录所有试验个体的原始产茸记录,用于后续产茸性能关联分析。
1.2 试验方法
1.2.1 基因组DNA的提取使用天根生化科技(北京)有限公司所生产的血液/细胞/组织基因组DNA提取试剂盒(离心柱型)提取试验样品的基因组DNA,相关操作参照试剂盒说明书。用1.0%的琼脂糖凝胶电泳检测DNA的完整性;用NanoDrop分光光度计测DNA的浓度,确定基因组DNA质量合格后,置于﹣20℃保存备用。

表2 试验样品Table 2 The samples submitted to this study
1.2.2 竞争性等位基因特异性PCR分型技术本实验室提供8个SNP位点的序列信息,后续KASP分型实验中引物设计、合成及位点检测均由北京康普森公司完成。该技术是根据对终端荧光信号的读取对位点进行判断,每孔反应都是采用双色荧光检测1个SNP位点的2种基因型,不同的SNP对应着不同的荧光信号。通过扫描试验用多重PCR板,采集不同的荧光信号得出检测位点的具体信息。
1.2.3 产茸性能估测在实际生产中,由于鹿场的管理模式、养殖人员更换等因素,可能会造成某些个体的产茸记录部分缺失。李和平等[7]建立了根据整个群体和个体产茸记录来准确估测公鹿产茸能力的数学模型,这个模型中假设存在一个包含若干头公鹿的群体,其中第i头公鹿有Ki次产茸记录,则这头公鹿i最有可能的产茸能力(Pi)为

其中P指鹿群锯平均产茸量;t指同一公鹿不同锯别产茸记录之间的相关(重复力);Xi指公鹿的锯平均产茸量。本试验根据这个模型对试验个体进行产茸能力估测。
1.2.4 SNP位点统计分析根据群体遗传学的研究方法,运用Cervus软件对群体的遗传参数进行计算,其中包括基因频率、基因型频率、观测杂合度(Ho)、期望杂合度(He)、有效等位基因数(Ne)、多态信息含量(PIC)和哈迪-温伯格(H-W)平衡检验等[8],同时运用SPSS19.0软件,采用一般线性模型对遗传信息与产茸性能的关联性进行最小二乘法分析。
2 结果
2.1 基因组DNA提取
将所提取的基因组DNA经1%琼脂糖凝胶电泳和NanoDrop2000进行基因组DNA完整性、纯度和浓度检测,结果见图1,电泳条带单一且清晰明亮,大小位于15 000 kb Marker顶端条带之上;Nano-Drop2000检测结果显示,所提取的DNA浓度在50~100ng/L,OD值介于1.8~2.0之间,紫外吸收峰峰型正常。

图1 基因组DNA琼脂糖凝胶电泳图Fig.1 Genomic DNA agarose gel electrophoresis
2.2 引物设计结果及相关信息
应用Primer 5软件,对8个位点所在序列进行分析,经过筛选得出最适引物。本试验中所用8个SNP位点的引物序列及相关信息见表3。

表3 SNP位点引物信息Table 3 The information of SNP’s primer
2.3 SNP位点分型结果
运用KASP技术,对190个样本的8个SNP位点进行分型。研究表明,用于表型关联分析的位点,基因型检出率不低于94%[9]。本研究对8个SNP位点检出率情况进行统计。结果显示,位点的检出率介于95.26%~98.42%,平均检出率为97.43%,均大于94%,全部分型成功。同时还对这些位点进行最小等位基因频率(MAF)计算分析,平均MAF值为0.188,结果见表4。
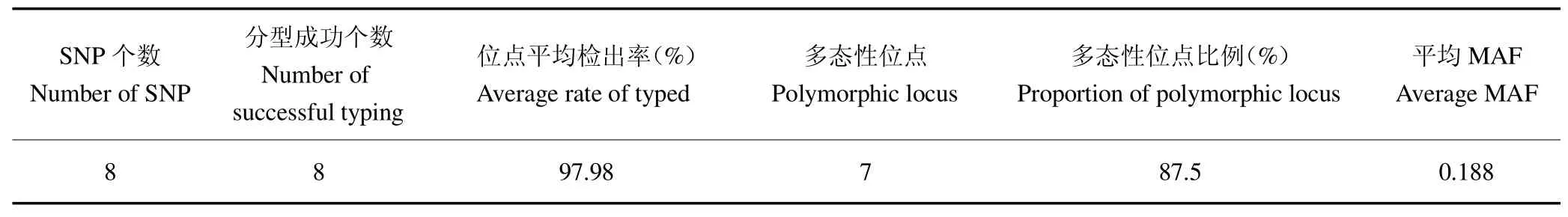
表4 SNP位点MAF值计算结果Table 4 The calculated results of MAF value of SNP locus
2.4 群体遗传多样性分析
运用KASP对8个SNP位点在验证群体进行基因分型,所得到的基因型信息使用Cervus软件计算。通过得到的基因型信息对该群体的遗传多样性进行分析,结果见表5。8个SNP位点中,有7个位点具有多态性,其多态信息含量(PIC)介于0.078~0.369,平均值为0.231 6;最小基因等位基因频率(MAF)介于0.044~0.396之间,平均值为0.188;各位点的观测杂合度(Ho)介于0.090~0.426,平均值为0.265;期望杂合度(He)为0.082~0.490,平均值为0.282;有效等位基因数(Ne)介于1.2~2.8,平均值为1.9。观测杂合度与期望杂合度不存在统计学上的显著差异(P>0.05)。Hardy-Weinberg平衡分析结果显示,SNP614_2585、SNP1881_130和SNP596_3774这3个SNP位点显著偏离平衡(P<0.05)。

表5 群体中9个SNP位点的遗传参数信息Table 5 The genetic parameter information of 9 SNP locus in the population
2.5 产茸性能估测结果
运用茸重估测模型,计算验证群体中每个个体的产茸估测值。计算结果显示,试验群体的产茸估测值介于1.12~8.55 kg。对所有个体的产茸估测值进行统计分析,根据相关研究,将产茸估测值分大于3.5 kg的高产组和小于2.5 kg的低产组[10]。对两组数据进行单因素方差分析,结果显示两组产茸估测数据存在极显著差异(P<0.01),证明本群体中存在高、低产个体。部分样本的产茸记录和估测值,见表6。
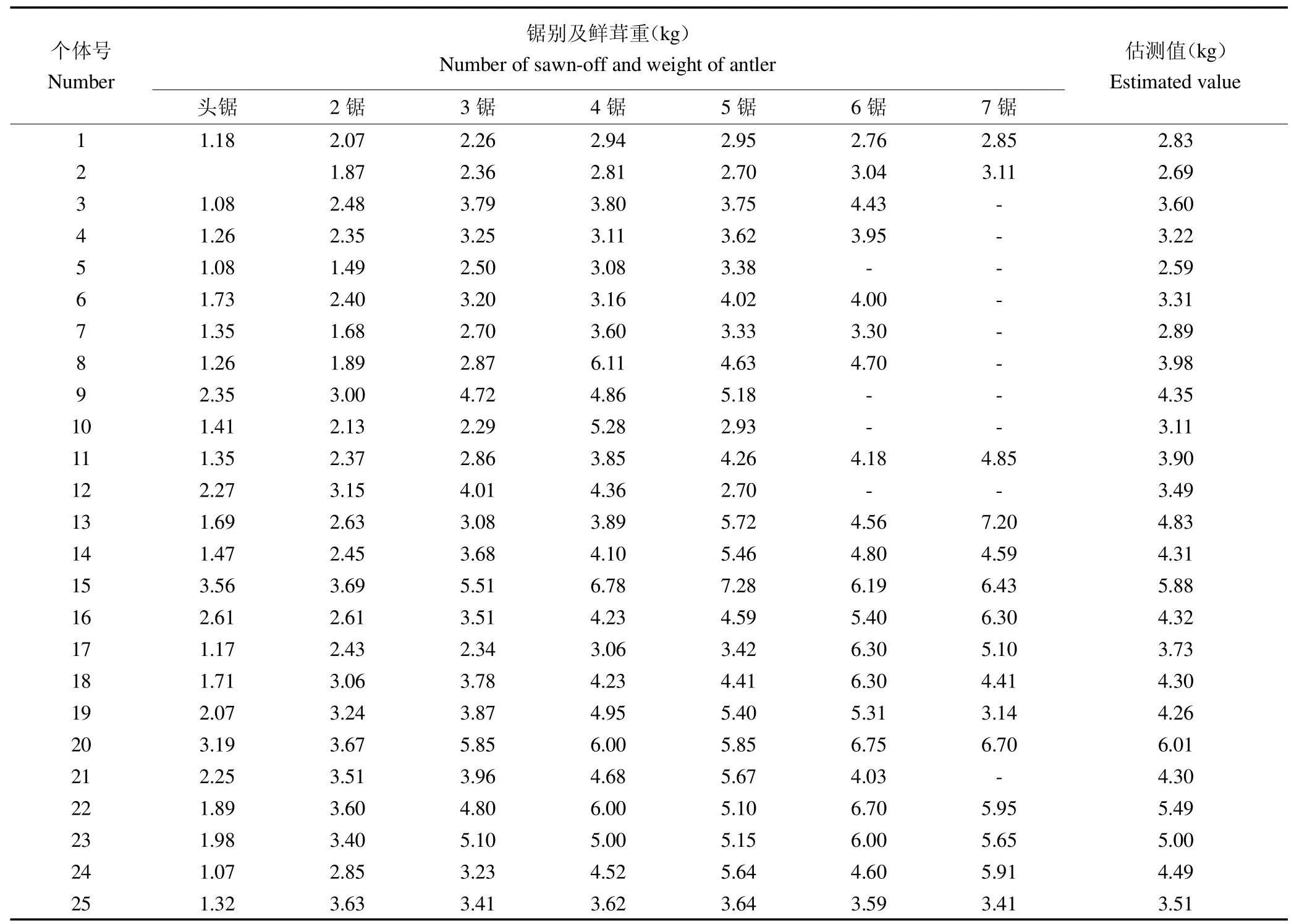
表6 部分梅花鹿产茸性能估测Table 6 The estimation of the antler performance
2.6 SNP位点与产茸性状的关联分析
对190头梅花鹿进行产茸性状与SNP标记相关性分析。经过计算,7个SNP位点的相关性P值介于0.264~0.849之间,均大于0.05,显示所有位点SNP位点与产茸性状均不存在显著相关性(P>0.05),结果见表7。

表7 SNP位点关联性分析结果Table 7 The phenotypic interpretation rate of SNP Locus
根据检测结果显示,位点SNP219_9123所有个体基因型均为CC,无多态性。SNP1427_255、SNP307_1564、SNP123_7847、SNP2027_423、SNP614_2585和SNP596_3774均存在3种基因型,但各基因型所占比例不同。SNP1881_130中仅存在两种基因型,CC和CT,其中CC为优势基因型,基因型频率为0.91。SNP1427_255中,纯合CC为优势基因型,基因型频率为0.56。SNP307_1564中,纯合GG为优势基因型,基因型频率为0.63。SNP123_7847中,纯合TT为优势基因型,基因型频率为0.56。SNP2027_423中,杂合AG为优势基因型,基因型频率为0.43。SNP614_2585中,纯合AA为优势基因型,基因型频率为0.87。SNP596_3774中,纯合TT为优势基因型,基因型频率为0.82。对每个SNP位点不同基因型的产茸性估测值均值进行单因素方差分析。结果显示,各位点的不同基因型间产茸估测值均值不存在显著差异。进一步验证了8个SNP位点在本验证群中与产茸性能不存在显著相关性。但在SNP307_156、SNP614_258和SNP596_377位点具有明显的高产基因型。在SNP307_156中,存在与已知研究相同的高产CC基因型[11],分别高于CG基因型和GG基因型14.36%和17.85%。在该位点中,对具有CC基因型个体的产茸值与CG基因型和GG基因型个体进行比较,发现两组产茸量具有显著差异性(P<0.05)。还发现SNP614_258位点高产基因型为GG,SNP596_377位点高产基因型为CC。具体位点详细信息见表8。

表8 多态性位点的基因型信息Table 8 The genotype information of the polymorphic locus
3 讨论
根据群体遗传学的研究方法,运用Cervus软件计算多态信息含量(PIC)、平均期望杂合度(He)、观测杂合度(Ho)和Hardy-Weinberg群体平衡,以分析群体遗传多样性。多态信息含量这一指标是由Botsticn等[12]在1980年提出,该参数的高低用以描述家系连锁分析标志基因(或标志序列)的多态性大小。Botsticn认为,当多态信息含量大于0.5时,该标志基因具有高度信息含量,小于0.5而大于0.25则为中度信息含量,小于0.25时具有低度信息含量。本试验中所验证的7个SNP位点的多态信息含量(PIC)介于0.078~0.369,其中SNP614_2585、SNP1881_130和SNP596_3774为低度多态性位点,SNP1427_255、SNP307_1564、SNP123_7847和SNP2027_423为中度多态性位点,没有高度多态性位点。相关研究表明,与微卫星标记相比,SNP的多态性较低,但因SNP位点为全基因组层面内分布,可在一定程度上弥补其相较于微卫星等其他分子标记方法中多态性较低的问题,使得SNP位点可用于相关性状分析[13]。
杂合度用来评价群体在检测位点上出现杂合的频率,是评价群体遗传变异的重要参数。通过对比观测杂合度(Ho)和期望杂合度(He)的相对大小来判定群体近交程度。当观测杂合度小于期望杂合度时,群体遗传多样性低,为近亲繁殖;当两者相等时,群体内个体为随机交配;当观测杂合度大于期望杂合度,群体遗传多样性较高,为远亲繁殖。本试验群体的平均观测杂合度(Ho)为0.265,平均期望杂合度(He)为0.282,呈观测杂合度略小于期望杂合度,表明群体杂合度低,群体遗传多样性低。
Hardy-Weinberg平衡是以完全随机交配所组成的自然种群为基础而做出的假设。因此,通过Hardy-Weinberg群体平衡卡方检测来衡量群体遗传平衡状态。本研究群体经过Hardy-Weinberg群体平衡检测发现,在具有多态性的7个位点中,有3个位点显著偏离平衡状态。现有研究结果显示,在小规模种群中,近亲交配和无效等位基因等因素会导致杂合度不足,造成位点偏离Hardy-Weinberg平衡[14]。所以这3个显著偏离Hardy-Weinberg平衡的位点可能与群体遗传多样性低、人工选育干预强度大的情况有关。东北地区的家养梅花鹿均为野生东北梅花鹿的后裔,结合本研究群体的多个遗传信息结果可以看出,东北地区各梅花鹿种群间的遗传背景十分相似。
本试验是对已知与产茸量相关的SNP位点进行验证。在试验群体中,这些SNP位点与本群体中的产茸量不存在显著相关性。推测其原因,可能是由于在筛选群体中,主要是以四平梅花鹿和东大梅花鹿为主。这两个品种的梅花鹿在已知品种中为高产品种。特别是东大梅花鹿群体,在产茸性能方面极显著高于四平梅花鹿和其他群体。据统计,东大梅花鹿和四平梅花鹿的上锯三杈鲜茸平均单产为4 kg和3.42 kg,明显高于其他品种,如双阳梅花鹿上锯三杈鲜茸单产为2.9 kg[15],敖东梅花鹿上锯三杈鲜茸单产为3.34kg[16]。究其原因可能与东大梅花鹿在育种期间引入马鹿血源及高强度人工选择有关[11]。本研究中的190头梅花鹿群体经全基因组SNP鉴别芯片检测,判定为纯种梅花鹿,纯种梅花鹿在产茸量方面较杂交个体相差甚远。因此,我们推测群体遗传背景差异可能是造成与产茸相关性SNP位点在本试验中不相关的主要原因。后续对梅花鹿产茸性能相关的SNP位点的筛选,建议扩大筛选群体,增加群体数量。在表型数据中,除依据产茸估测之外,还可以通过相同锯龄的实际产茸量进行关联分析,为吉林梅花鹿的产茸性能SNP位点提供有效参考。
4 结论
本研究运用竞争性等位基因特异性PCR分型技术对190个个体的8个SNP位点进行基因分型检测,得出7个具有多态性的SNP位点。使用一般线性模型对这7个SNP位点遗传信息与产茸量进行关联分析,发现在本试验中所建立的随机验证群体中,所有SNP位点与产茸量均不存在显著关联,但存在SNP307_156、SNP614_258和SNP596_377位点具有明显的高产基因型。本研究表明,前期基于部分群体筛选出的与产茸量相关SNP位点在本试验群体的适用性不理想。
