利用微卫星分子标记分析渤海湾的口虾蛄遗传多样性
2021-09-22谷德贤许玉甫李文雯刘国山
谷德贤,王 婷,许玉甫,吴 宁,王 娜,郝 郁,王 刚,李文雯,刘国山
( 1.天津市水产研究所,天津 300221; 2.河北省海洋与水产科学研究院,河北 秦皇岛 066200 )
口虾蛄(Oratosqillaoratoria),属节肢动物门、甲壳纲、口足目、虾蛄科、口虾蛄属[1],从俄罗斯的彼得大帝湾到日本、中国沿海、菲律宾、马来半岛、夏威夷群岛均有分布,我国以黄海、渤海产量最大[2]。作为中国沿海重要的近海渔业资源种类,随着捕捞强度的增加,资源量不断减少,因此近年来国内外有关口虾蛄的研究日益增多,研究内容主要集中在口虾蛄的基础生物学[3-6]、形态学[6]、发育生物学[7-8]、资源学[9-12]等方面。口虾蛄遗传学方面的研究有:张代臻等[13-14]基于线粒体COⅠ基因研究了黄海、渤海口虾蛄遗传多样性;隋宥珍等[15]基于线粒体Cytb基因研究了黄海、东海、南海口虾蛄遗传多样性;刘海映等[16]基于RAPD研究了大连海域口虾蛄资源遗传多样性的分析;张阳等[17-18]开发了部分口虾蛄的微卫星标记。
微卫星即简单重复序列(SSR)技术是常用的分子标记技术之一,它符合孟德尔遗传规律,多态性高且为共显性遗传,是一种极具应用价值的遗传分子标记。笔者采用微卫星标记技术,利用已知的微卫星标记对渤海湾5个自然种群的口虾蛄进行种群结构和遗传多样性分析,旨在为渤海湾海域口虾蛄的种质资源保护及合理有效地开发利用提供理论依据。
1 材料与方法
1.1 试验材料
2019年11月在渤海湾的秦皇岛、唐山、昌黎、天津、黄骅5个海域,通过单拖网调查取样,每个海域采集口虾蛄鲜活样本40尾,取背部肌肉组织,无水乙醇固定后保存备用。
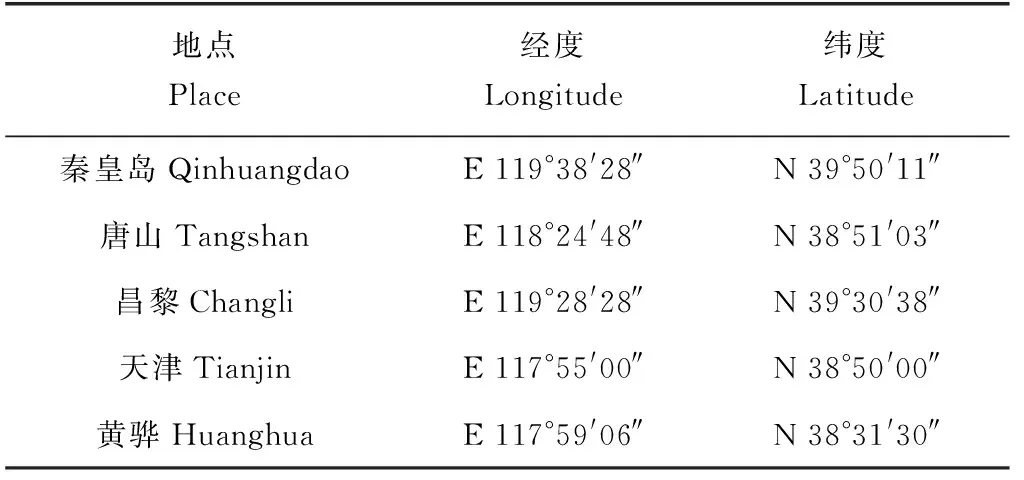
表1 取样站位经纬度Tab.1 The latitude and longitude of sampling stations
1.2 基因组DNA提取
采用醋酸铵法提取基因组DNA,用核酸蛋白仪测量DNA质量浓度,并根据DNA质量浓度将DNA稀释到100 μg/mL作为工作液。
1.3 引物合成及PCR扩增
本试验所用的16对微卫星引物为张阳等[17-18]已发表的微卫星序列(表2),由生工生物工程(上海)股份有限公司合成。

表2 16对微卫星引物及其退火温度Tab.2 Sequences and annealing temperature of sixteen pairs of microsatellite maker
PCR反应体系为25 μL:引物F(10 μmol/L) 1 μL,引物R(10 μmol/L) 1 μL,dNTP(10 μmol/L)1 μL,Taq Buffer(10×)2.5 μL,Taq酶(5 U/μL )0.5 μL,无菌水补齐到25 μL。PCR反应条件:95 ℃预变性 3 min;94 ℃变性30 s,60 ℃退火30 S,72 ℃延伸30 s,10个循环;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸30 s,35个循环;72 ℃修复延伸5~8 min。PCR 扩增在PE9700型PCR仪上进行。将甲酰胺与分子质量内标按100∶1的体积比混匀后,取10 μL加入上样板中,再加入1 μL PCR产物。然后使用3730XL测序仪进行毛细管电泳,使用Genemapper软件得到扩增片段大小。
1.4 数据统计
应用Popgen 32软件进行数据处理,分别完成等位基因数、有效等位基因数、观测杂合度、期望杂合度、群体间遗传距离、相似指数计算以及聚类分析、哈迪温伯格平衡检验。采用软件MEGA 6.0绘制系统进化树。最后根据等位基因频率计算多态信息含量(PIC)[19]:
式中,pi和pj分别是第i和第j个等位基因数的频率,n为等位基因数。
2 结果与分析
2.1 群体遗传多样性分析
根据16对引物的PCR预试验结果,筛选出12对引物用于遗传多样性分析,12个位点在5个群体中的等位基因数、有效等位基因数、观测杂合度、期望杂合度、平均多态信息含量和哈迪温伯格平衡数据见表3。
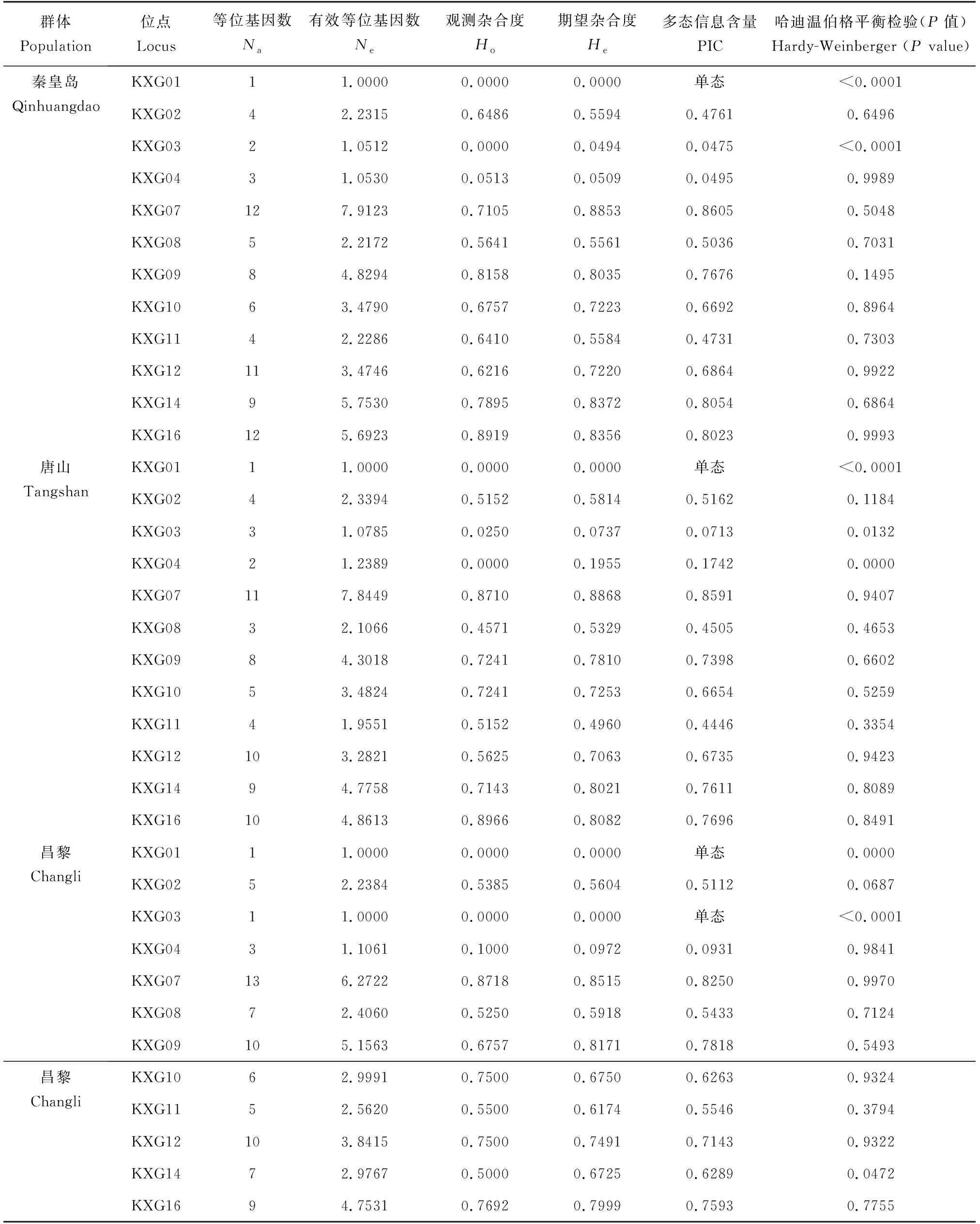
表3 5个口虾蛄群体在12个微卫星位点的多样性指数Tab.3 The polymorphic information at 12 microsatellite loci of five populations of mantis shrimp O. oratoria
利用12对微卫星引物对5个群体进行PCR扩增检测,共检测到等位基因375个,其中秦皇岛群体77个、唐山群体70个、昌黎77个、天津群体74个、黄骅群体77个。5个群体共获得99个不同等位基因,其中每个位点等位基因数3~16个,KXG01和KXG04检测到等位基因最少(均为3个),KXG07检测到等位基因最多(17个),其他位点检测到等位基因数为4~13个。
各群体平均有效等位基因数为2.9063~3.4102,平均观测杂合度为0.5004~0.5342,平均期望杂合度为0.5310~0.5491。其中除期望杂合度唐山群体最高外,其他上述指标数值均为秦皇岛群体最高。通过基因频率计算多态信息含量,检测的60个位点中,KXG01在秦皇岛、唐山、昌黎、黄骅群体中及KXG03在昌黎、天津群体中为单态,5个群体平均多态信息含量为0.5422~0.6038,其中,昌黎群体的多态信息含量最高,黄骅群体的多态信息含量最低。12个位点中,KXG01、KXG03、KXG04为低多态性位点(平均多态信息含量<0.25),KXG08为中度多态(0.25<平均多态信息含量<0.5),其他8个位点均为高度多态(平均多态信息含量>0.5)。哈迪温伯格平衡检测,49个位点符合哈迪温伯格平衡(P>0.05),11个位点偏离哈迪温伯格平衡(P<0.05),偏离哈迪温伯格平衡的现象在5个群体中均存在。本研究中8个位点(KXG02、KXG07、KXG08、KXG09、KXG10、KXG11、KXG12、KXG16)在5个群体中均符合平衡。
2.2 群体间遗传结构
应用Popgen 32软件计算群体间遗传距离和相似指数,5个群体相互之间的遗传距离为0.0143~0.0365,遗传相似性为0.9775~0.9858,表明黄骅群体与昌黎、秦皇岛群体遗传距离最小,亲缘关系最近。天津群体与唐山群体的遗传距离最大,亲缘关系较远。聚类树分析显示,秦皇岛种群与唐山群体聚在一起,昌黎群体与黄骅群体聚在一起,而后又与天津群体聚在一起。
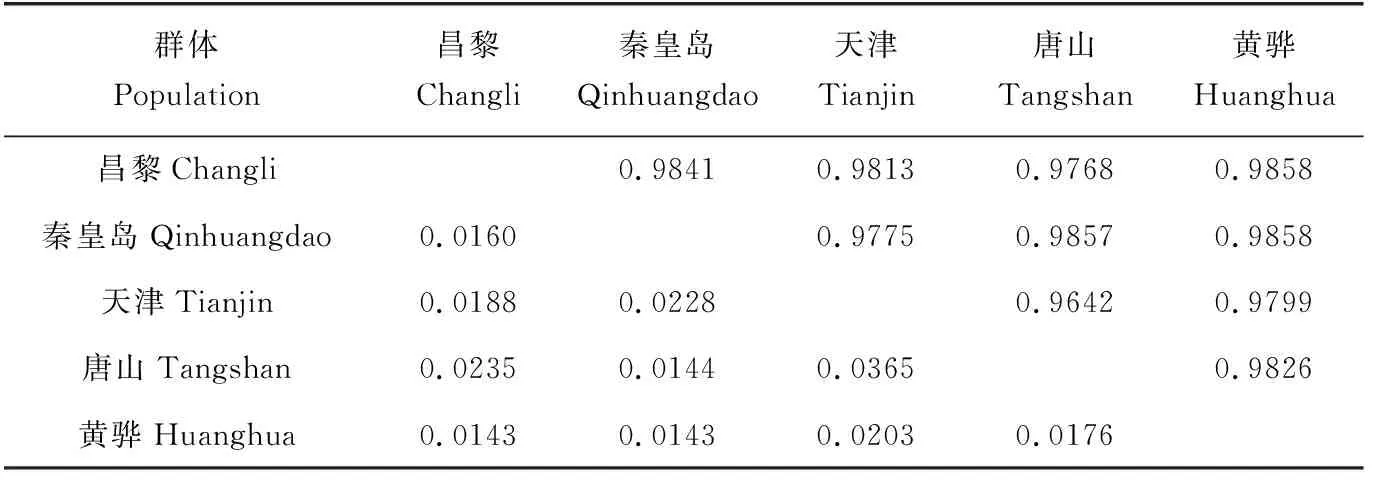
表4 5个群体的遗传距离和相似性指数Tab.4 The genetic distances and genetic identity of the five populations

图1 5个群体的UPGMA聚类树Fig.1 The UPGMA dendrogram of the five populations
3 讨 论
3.1 遗传多样性
依据微卫星选择标准[20],当等位基因数超过4时能较好地反映群体遗传变异水平,本研究中5个群体中检测的平均等位基因数为5.8333~6.4167,平均有效等位基因数为2.9063~3.4102,该数据能够有效地用于5个群体的遗传多样性分析。杂合度越高,群体的遗传多样性丰度越高,群体对环境的适应能力越强[21]。5个研究群体中,秦皇岛群体的平均观测杂合度与期望杂合度相对于其他4个群体较高,其对环境适应能力较强,具有较丰富的育种和遗传改良潜力。
多态信息含量是衡量基因位点群体多态性的重要指标之一,可以反映出群体的遗传变异程度[22]。12个微卫星位点,8个高度多态性位点,1个中度多态性位点,3个低度多态性位点。总体来说,位点多态性较高,在群体遗传多样性分析中,得出的可靠性较高[23]。对5个群体的多态信息含量计算表明,5个群体的多态信息含量均为高度多态性,其中昌黎群体多态信息含量最高,其次为秦皇岛群体,黄骅群体最低。哈迪温伯格平衡检测表明,81.67%符合哈迪温伯格平衡,偏离平衡的现象不明显。其中,天津群体哈迪温伯格平衡P值最低,可能由于天津群体处于渤海湾最西部,与其他群体杂交较少,导致群体中杂合子缺失。
综合分析,渤海湾5个海域的口虾蛄群体遗传多样性处于高度多态性,其中秦皇岛、昌黎口虾蛄群体相对较高,天津、黄骅口虾蛄群体相对较低。
3.2 群体间遗传结构
遗传距离越远,分化时间越长,杂种优势越大,即遗传距离与分化关系呈正相关。Shaklee等[24]研究认为,0.05≤遗传距离<0.3为同种不同群体;0.3≤遗传距离<0.9为同属不同种群体;遗传距离≥0.9为不同属群体。本试验5个群体间遗传距离均小于0.05,认为5个群体为同种群,未发生明显遗传分化。张代臻等[13]基于线粒体COⅠ基因对东营、葫芦岛、天津海域口虾蛄的遗传分化进行研究,发现3个不同海域没有明显的分化。由此可见,渤海湾各海域的口虾蛄可能起源于一个祖先种群,而后不断形成不同的种群。
遗传距离和聚类树分析表明,天津群体与其他4个群体关系较远,这可能是因为天津地处渤海湾的最西侧,与其他海域口虾蛄交流较少。其他4个群体中,秦皇岛、唐山群体聚在一起,昌黎、黄骅群体聚在一起。这些海域的口虾蛄种群没有以海域为单位各自聚集,而是交错聚集,说明渤海湾内口虾蛄的种群遗传分化不显著。
4 结 论
研究结果表明,渤海湾5个海域的口虾蛄群体遗传多样性处于高度多态性,同时符合哈迪温伯格平衡的程度较高,5个海域的口虾蛄遗传分化程度较低。秦皇岛群体的杂合度、多态信息含量较高,天津群体与其他4个群体关系较远。建议加强口虾蛄群体的保护工作,防止过度捕捞,避免出现种质遗传资源的衰退,并适度开展人工繁育。在进行人工繁殖研究时选取遗传距离较远的个体作为亲本,同时对同一繁殖群体可进行不同地理区域放流,以增加群体遗传多样性,保证种质资源永续利用。
