肝癌靶点的筛选与验证
2021-09-20王丽萍田振波唐旭清
王丽萍,田振波,唐旭清
(江南大学理学院,无锡 214122)
引 言
肝癌是临床上一种常见的消化道恶性肿瘤,具有较高的发病率和死亡率,在恶性肿瘤中分别处于第6 位和第3 位[1‑2]。肝癌的发生和发展是多个分子(基因或蛋白质)协同作用的结果[3],是一个复杂的生物过程,一般可以分为4 个阶段:早期亚临床期、亚临床期、中期和晚期。早期患者没有明显的症状,随着病情的逐渐加重,会出现肝区疼痛、消瘦、乏力等症状,中晚期可能会出现恶液质,全身多脏器发生转移。肝癌总的病程大约为两年半,其中有两年时间可能都是出于没有症状的早期阶段,所以大部分肝癌患者确诊时已是晚期,从而导致治疗效果欠佳[4]。近年来索拉菲尼开辟了一种新的靶向药物治疗方法,但是长期使用药物治疗容易产生耐药性[5],所以需要发现新的靶点、开发新药物。高通量基因芯片技术的快速发展可以从基因组或转录组水平探索疾病的发生、发展过程,使用GEO(Gene expression omnibus)数据库提供的大量和疾病相关的基因表达谱信息[6],可以寻找疾病关键基因[7]以及研究特异的分子标志物。
本文首先对肝癌组织和癌旁组织的基因表达谱数据做差异表达分析,筛选差异表达基因;其次对差异表达基因做GO(Gene ontology)功能分析和KEGG(The Kyoto encyclopedia of genes and genomes)通路分析,取同时显著富集的基因作为目标基因;然后通过网络分析寻找目标基因的相互作用关系,寻找高相互作用的关键基因;最后对关键基因进行生存分析验证和文献验证。
1 数据与方法
1.1 数 据
分析数据来自国家生物技术信息中心(National center for biotechnology information,NCBI)的公共数据平台GEO 数据库(https://www.ncbi.nlm.nih.gov/geo/)。根据以下标准进行数据筛选:(1)来源于人体组织而非动物;(2)具备肝癌组织和正常癌旁组织的基因表达谱数据,从而得到GSE60502 和GSE41804 两组数据集。其中GSE41804 数据集包含20 个肝癌患者样本和20 个正常样本,GSE60502数据集包含18 个肝癌患者样本和18 个正常样本。
1.2 方 法
(1)筛选差异表达基因
通过R 语言软件包分别对两个数据集的表达谱数据进行背景矫正、标准化及表达值计算;利用lim‑ma 程序包[8]筛选出两个数据集中的差异表达基因(Differential expressed genes,DEGs);采用union 包合并两组数据中筛选出的DEGs 作为此次研究分析的对象。
(2)差异表达基因的生物功能分析
本文使用在线分析网站David(Functional annotation bioinformatics microarray analysis,https://da‑vid.ncifcrf.gov/)对DEGs 进行GO 分析和KEGG 分析,并选取两者筛选出来的共同基因作为后续分析的目标基因。GO 分析是指使用标准表达的术语对基因和蛋白的功能进行生物学功能描述,包括3 个部分:生物学过程(Biology process,BP)、细胞结构(Cellular components,CC)和分子功能(Molecular function,MF)。KEGG 分析根据挑选出的差异基因,计算它们与每个Pathway 之间的关系,然后对每个DEG 存在的通路返回一个p‑value 和错误发现率(False discovery rate,FDR)值,进而可以定位每个DEG 最可能相关的通路。GO 分析从单个基因角度分析其可能的功能,KEGG 分析从集团的角度分析多个基因相互作用的功能,将两者结合更能展现所选基因的重要性。
(3)基因调控网络及核心基因筛选
肝癌的发生和发展涉及多基因、多因子的协同作用,通过蛋白质互作(Protein‑protein interaction,PPI)网络可以了解疾病分子间的相互作用和调控关系来挖掘致病基因[9]。String 数据库(Search tool for the retrieval of interacting genes)是一个研究蛋白质相互作用模式的在线分析工具,可以构建PPI 网络并提供相关通路及功能信息[10]。本文使用String 数据库对目标基因进行网络分析,找出基因调控关系,通过信息分析学软件Cytoscape[11]得到基因调控网络(Gene regulatory network),并使用CytoHubba插件[12]中的最大团中心性(Maximal clique centrality,MCC)算法筛选出最大团中心度排名前10 的关键基因。
(4)生存分析
生存分析可以检验所选基因是否具有预后价值。本文使用KM‑Plotter 数据库及Oncolnc(http://www.oncolnc.org/)生存分析[13]网站分别对10 个关键基因进行生存分析,研究它们对肝癌患者总生存期的预后价值。其中KM‑Plotter 数据库中包含364个肝癌患者信息,Oncolnc 生存分析网站中包含360个不同的肝癌患者临床数据。
以上4 部分从生物功能和相互作用关系两个角度综合考虑了分子间的作用机理,通过生存分析检验其预后价值,为筛选肝癌相关分子标志物及药物靶点提供了理论基础,具体的方法流程见图1。

图1 实验方法流程图Fig.1 Flow chart of the experimental method
2 结果与验证
2.1 结 果
设置参数| log2FC| > 1 且FDR < 0.05 为筛选条件,从GSE41804 和GSE60502 两个数据集分别得到571 和445 个差异表达基因,其中上调基因分别为373 和280 个,下调基因分别为198 和165 个(见图2,红色表示上调基因,绿色表示下调基因)。取并集得到807 个DEGs,其中上调基因496 个,下调基因311 个。

图2 差异基因的火山图Fig.2 Volcano map of differential genes
对DEGs 进行GO 功能分析,以基因数量≥15 和FDR<0.01 为筛选条件,得到21 条相关富集功能,具体结果如表1 所示,通过R 软件包对筛选结果可视化如图3 所示。由GO 分析可知:在生物学过程中,差异表达基因主要在氧化还原过程、异型生物质的代谢过程、类固醇代谢过程、细胞分裂、内肽酶活性的负调控、血小板脱粒、有丝分裂核分裂方面显著富集;在细胞结构中,差异表达基因主要在细胞外的外来体、细胞外区域、细胞外空间、血液微粒、细胞器膜、中体方面显著富集;在分子功能中,差异基因主要在氧化还原酶活性、单氧酶活动、铁离子结合、氧气结合、血红素结合、氧化还原酶活性、丝氨酸型内肽酶抑制剂活性、电子载体活动方面显著富集。整理显著富集功能上的差异基因共有429 个。

图3 肝癌相关差异基因的GO 功能分析结果可视化Fig.3 Visualization of GO function analysis results of different genes related to liver cancer
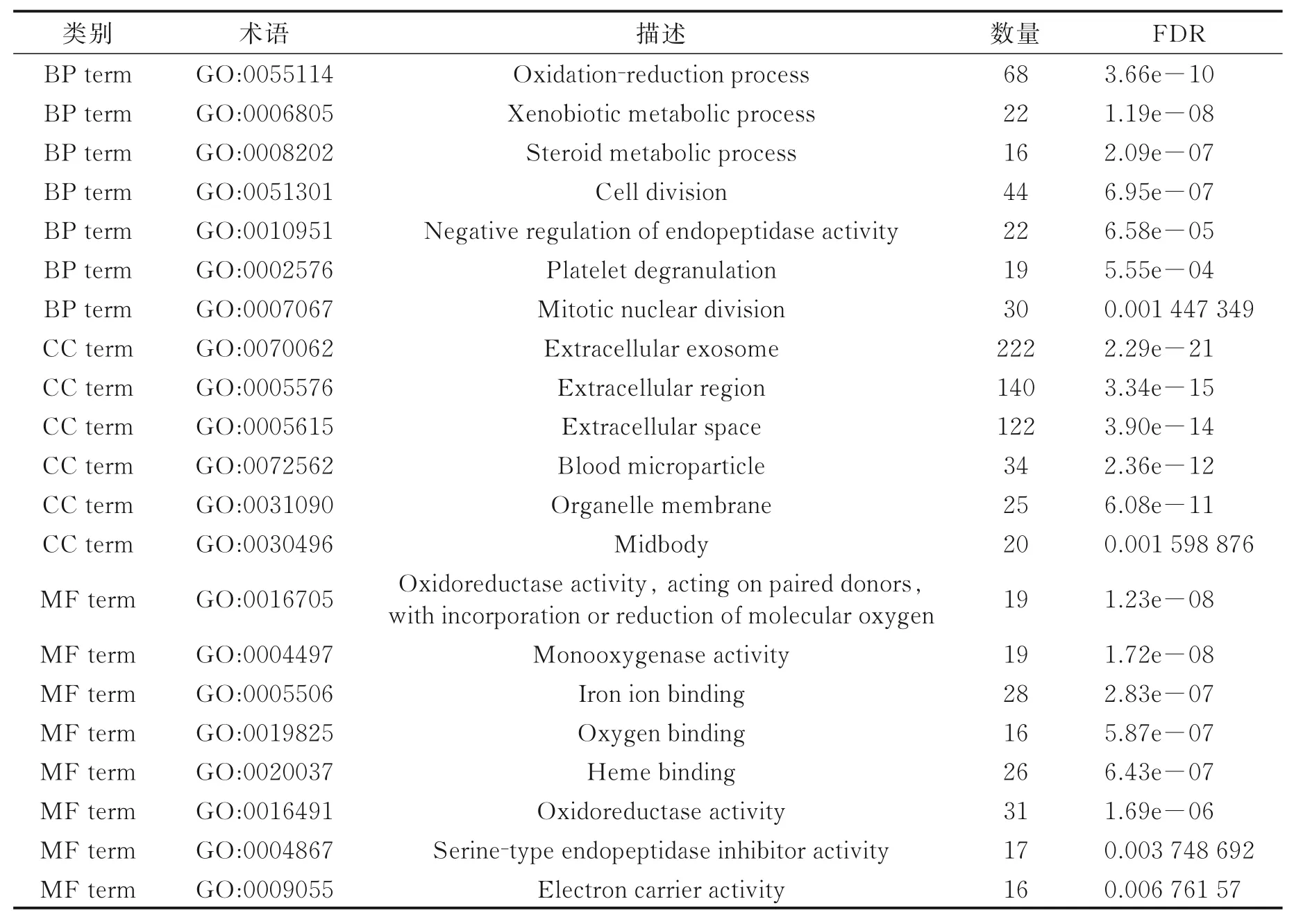
表1 肝癌相关差异基因的GO 功能分析结果Table 1 GO function analysis results of different genes related to liver cancer
对DEGs 进行KEGG 通路分析,以基因数量≥15 和FDR<0.01 为筛选条件,得到11 条相关通路,结果如表2 所示,用R 软件包对筛选结果可视化如图4 所示。由结果可知,差异表达基因主要在代谢途径、补体和凝血级联、视黄醇新陈代谢、化学致癌作用、色氨酸代谢、PPAR 信号通路、药物代谢-细胞色素P450、脂肪酸降解、抗生素的生物合成、细胞色素P450 代谢异种生物制剂、类固醇激素生物合成通路上显著富集。整理显著富集通路上的差异基因共有169 个。
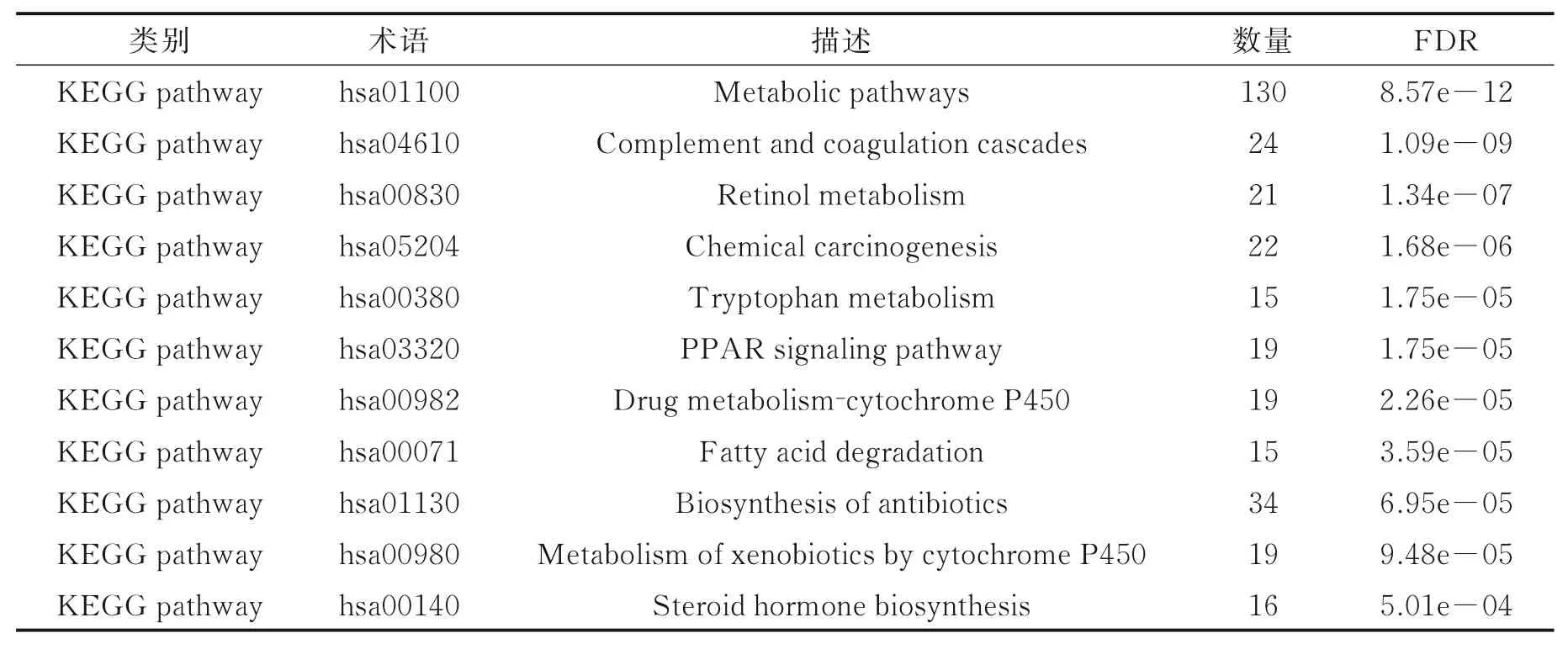
表2 肝癌相关差异基因的KEGG 通路分析结果Table 2 KEGG pathway analysis results of different genes related to liver cancer
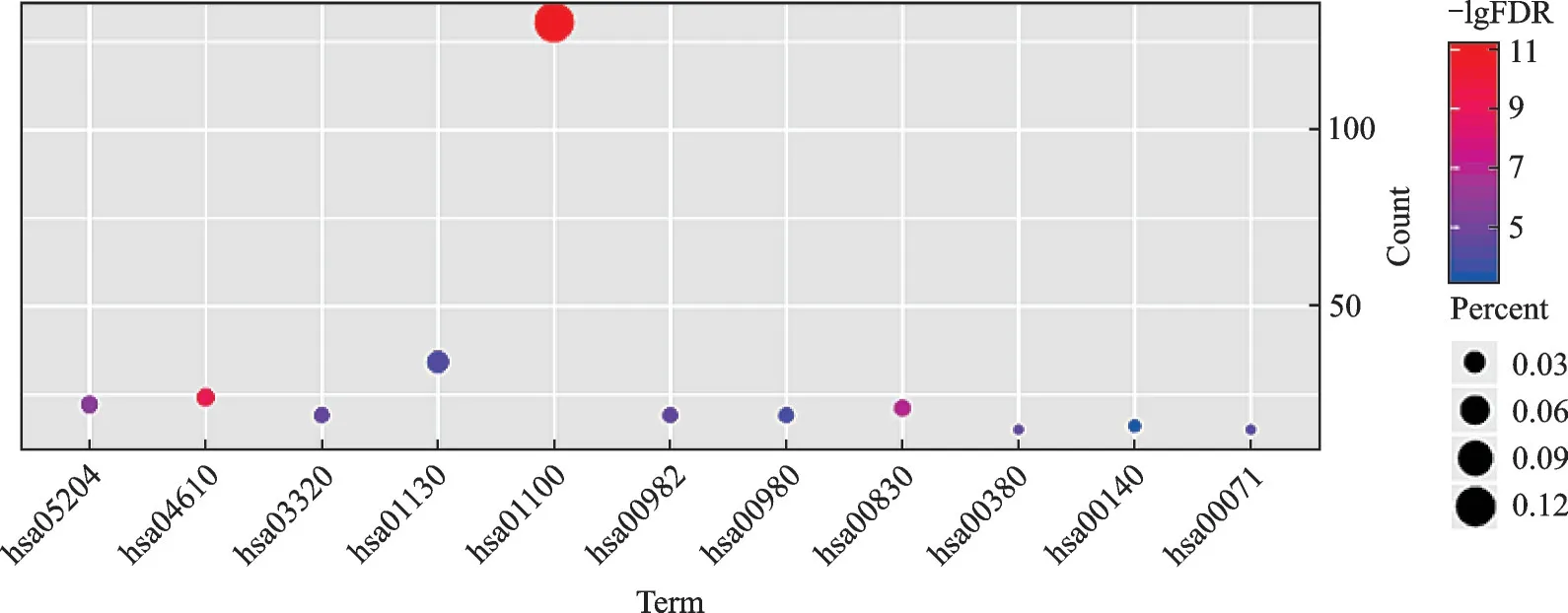
图4 肝癌相关差异基因的KEGG 通路分析结果可视化Fig.4 Visualization of KEGG pathway analysis results of different genes related to liver cancer
为了寻找与肝癌的发生和发展相关的关键基因,对同时在GO 功能和KEGG 通路上显著富集的128 个目标基因进行分析。目标基因的调控网络见图5,其中红色节点表示上调的差异基因,绿色节点表示下调的差异表达基因,共有126 个节点和678 条相互作用关系。使用CytoHubba 插件中的MCC 算法计算网络中每个节点的最大团中心性,筛选出最大团中心度排名前10 的关键基因:CYP3A4、CYP2C9、CYP2B6、CYP1A2、CYP3A5、CYP1A1、CYP2E1、HSD17B6、AOX1、CYP2C8。这10 个关键基因之间的相互作用网络如图6 所示,其中颜色表示节点度的变化。
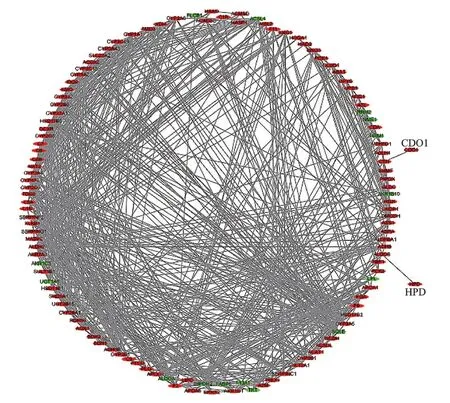
图5 肝癌相关差异基因的基因调控网络Fig.5 Gene regulatory network of different genes related to liver cancer
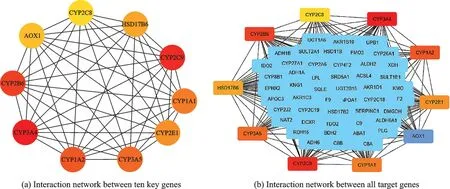
图6 基因调控网络Fig.6 Gene regulatory network
2.2 验 证
使用KM‑Plotter 数据库及Oncolnc 生存分析网站分别对10 个关键基因进行生存分析验证,研究这些关键基因与肝癌患者总生存期的预后价值,分析结果见表3。图7 和图8 分别展示了由On‑colnc 生存分析网站和KM‑Plotter 数据库进行生存分析得到的p值小于0.01 的基因生存曲线分析结果,可以看到两者都包含基因:CYP3A4、CYP3A5、CYP2C8 和CYP2C9。 根据两组不同肝癌临床数据进行生存分析的结果,有理由认为这4 个基因的高表达与肝癌的发生、发展和预后有着密切的关系。
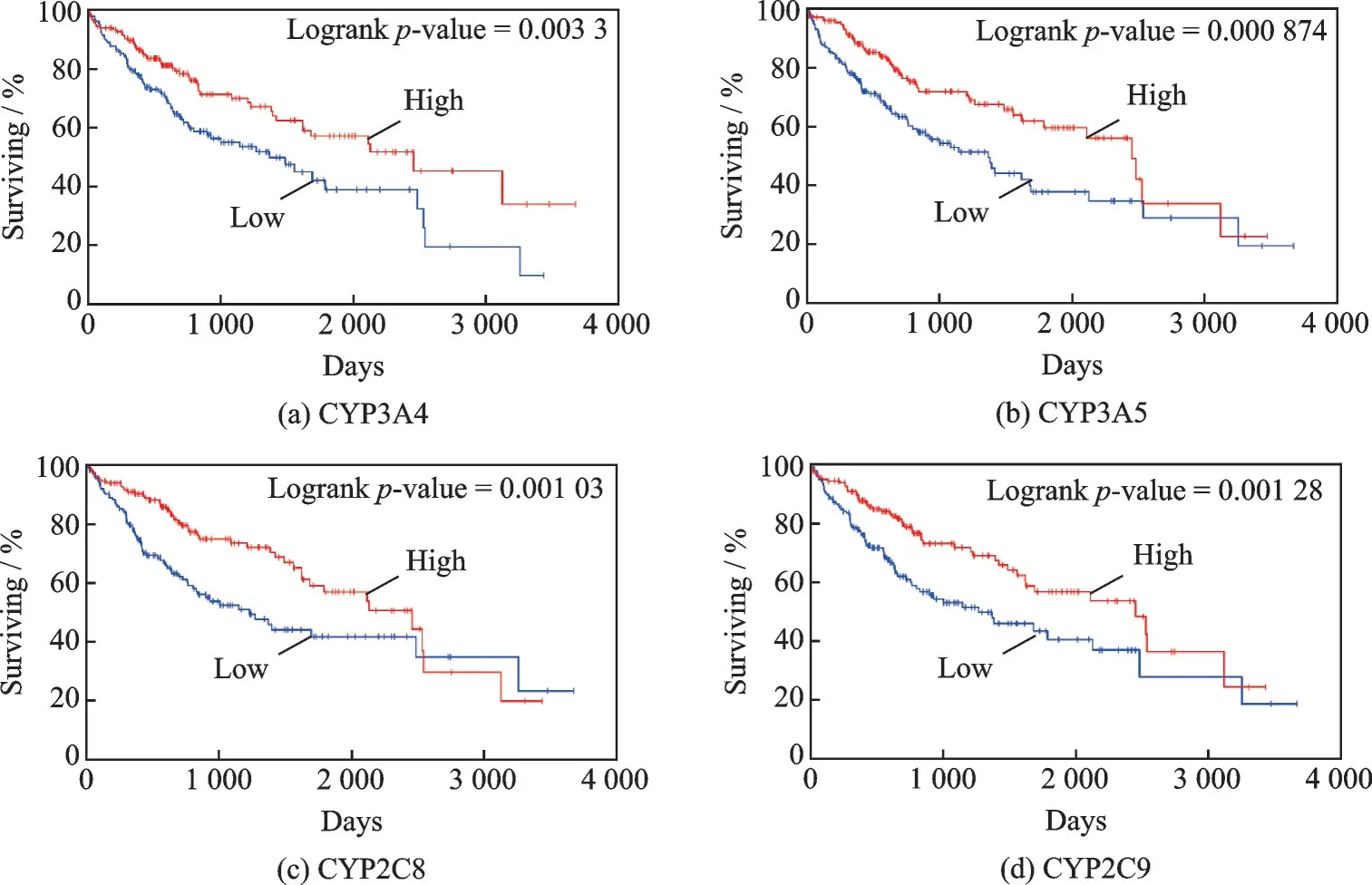
图7 CYP3A4,CYP3A5,CYP2C8,CYP2C9 基因的Oncolnc 在线生存分析结果Fig.7 Oncolnc online survival analysis results of CYP3A4, CYP3A5, CYP2C8, CYP2C9 genes
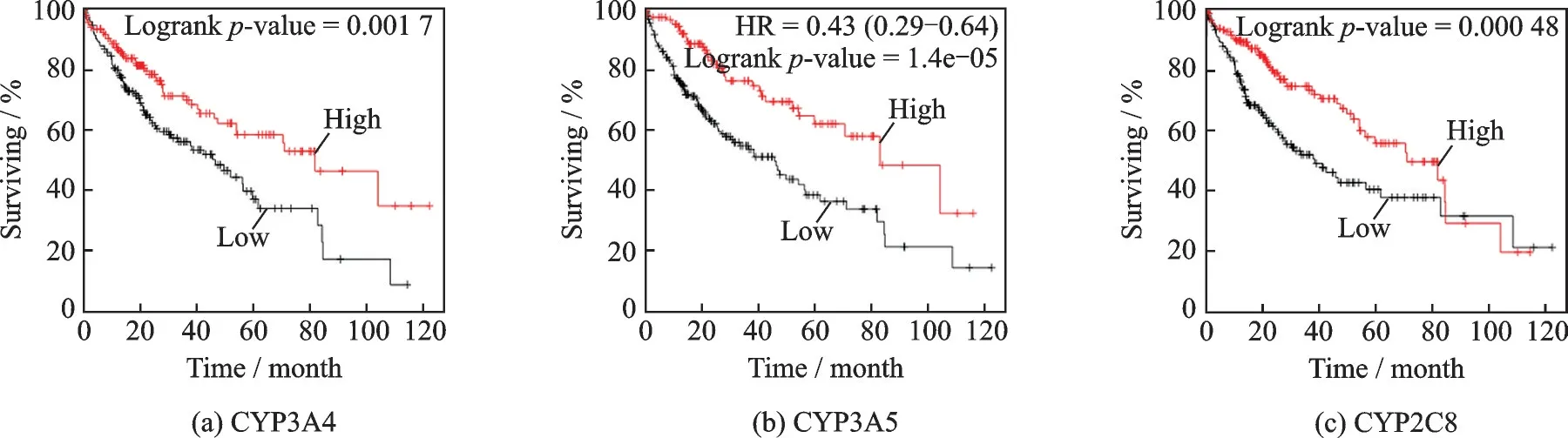
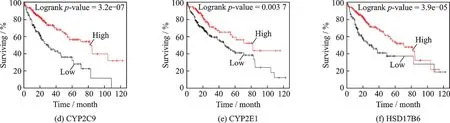
图8 CYP3A4,CYP3A5,CYP2C8,CYP2C9,CYP2E1,HSD17B6 基因的KM-Plotter 数据库生存分析结果Fig.8 KM-Plotter database survival analysis results of CYP3A4, CYP3A5, CYP2C8, CYP2C9, CYP2E1, HSD17B6 genes
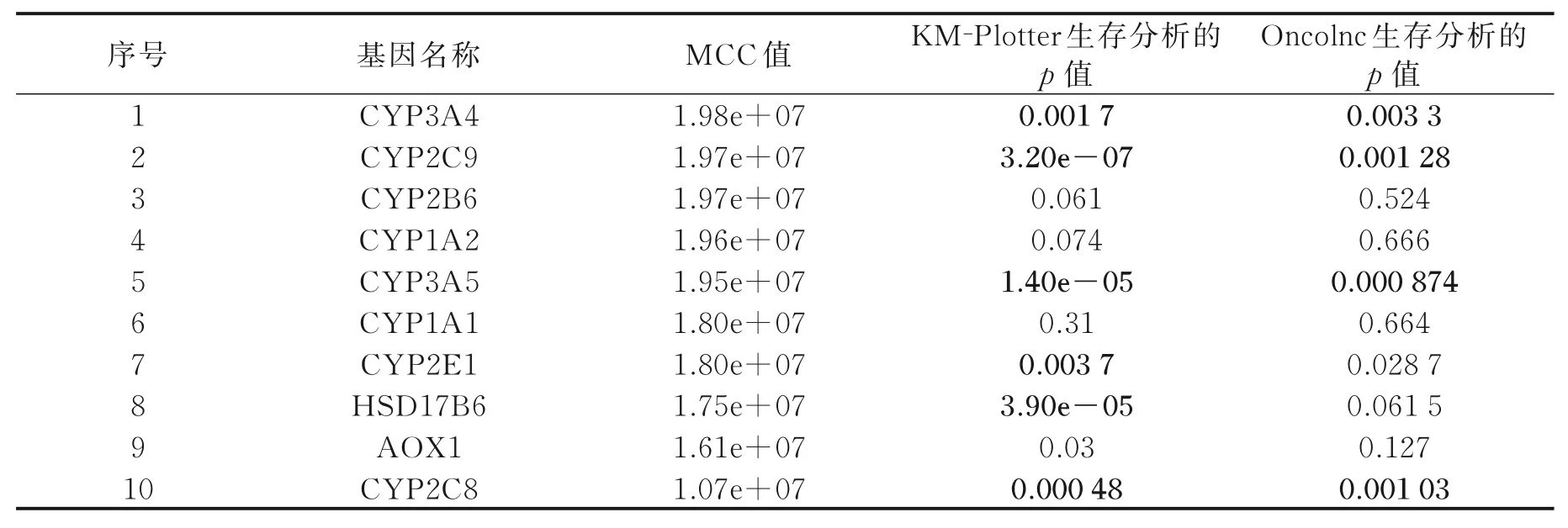
表3 10 个关键基因的两种生存分析结果Table 3 Two survival analysis results of ten key genes
3 讨 论
肝癌的靶向治疗研究进展缓慢,寻找新靶点、探索新药迫在眉睫。为深入了解肝癌发生、发展的分子机制,本文对GEO 数据库下载的肝癌组织和癌旁组织的基因表达谱数据进行系统分析,包括基因功能分析、通路分析、基因调控网络分析和生存分析,筛选可作为肝癌诊断的关键基因和药物治疗靶点。
使用R 语言软件对两组基因表达谱数据进行数据处理,取并集获得差异表达基因共807 个,其中上调基因496 个,下调基因311 个。通过GO 功能分析发现差异表达基因在氧化还原过程、细胞分裂、细胞外的外来体、细胞外区域、细胞外空间和氧化还原酶活性等方面显著富集并得到显著富集的基因433个;通过KEGG 通路分析发现差异表达基因在代谢途径、补体和凝血级联、视黄醇新陈代谢、化学致癌作用和抗生素的生物合成等方面显著富集并得到显著富集的基因169 个。对同时在GO 功能和KEGG通路上显著富集的基因取交集得到128 个差异表达基因作为目标基因(其中上调基因112 个,下调基因16 个),通过基因调控网络分析并结合MCC 算法计算网络中每个节点的最大团中心性筛选出排名前10的关键基因:CYP3A4、CYP2C9、CYP2B6、CYP1A2、CYP3A5、CYP1A1、CYP2E1、HSD17B6、AOX1、CYP2C8。使用KM‑Plotter 数据库及Oncolnc 生存分析网站分别对10 个关键基因进行生存曲线分析,发现CYP3A4、CYP3A5、CYP2C9 和CYP2C8 基因的高表达与肝癌的发生发展以及预后有着密切的关系,对于肝癌的研究具有重要意义。
CYP3A4 和CYP3A5 属于细胞色素P450 家族的成员。其中CYP3A4 主要分布于肝脏和小肠,是肝脏中最多的肝药酶(约占肝脏CYP450 酶总量的25%),临床中约有50% 的药物经由CYP3A4 酶代谢,还参与内源性化合物以及环境污染物的代谢[14]。CYP3A4 基因中已经发现了许多多态性,这些多态性影响CYP3A4 的表达和活性,且与致癌过程有关[15‑16]。同时,CYP3A4 又是一种激活剂,可代谢环境里的黄曲霉素B1(注:一种有效的肝癌原),以引起p53 基因的突变频率增加,并诱发肝癌发生[17‑18]。此外,慢性HBV 感染可降低肝脏CYP3A4 酶蛋白的表达,并导致酶活性下降,但是不影响酶的结构[19]。研究表明,CYP3A4 的过表达可诱导人肝癌细胞系Hep3B 的生长,在肿瘤进展中起到重要作用,且独立于致癌物的激活和抗癌药物的代谢[20]。Noreauh 等研究显示,亚砷酸盐通过降低孕烷X 受体的活性及核受体RXR 的表达,进而抑制人肝细胞CYP3A4 的转录,最终导致肝脏肿瘤和其他肝病的形成[17,21]。CYP3A5 是人体中最丰富、最重要药物代谢酶之一,参与到药物吸收、代谢和消除的整个过程。它可代谢黄曲毒素B1 成为致突变物——黄曲毒素B1 外8,9 环氧化物[22],后者是肝癌的主要危险因子,从而可影响个体患肝癌的风险[23]。同时,CYP3A5 作为一个保护性因子可抑制肝癌细胞的侵袭和转移[24‑25]。通过CYP3A5 基因型来检测口服双环醇的肝炎患者的治疗效果研究中,可以发现对CYP3A5*1 和CYP3A5*3 基因型患者均有显著疗效[26‑28]。
CYP2C9 和CYP2C8 都是CYP2C 亚家族中的成员。其中CYP2C9 是一种同功酶,主要分布在肝脏组织,且参与人体内多种药物的代谢[29],现有临床使用的药物大约16% 通过CYP2C9 代谢[30‑31]。具有功能意义的基因突变会导致CYP2C9 酶活性降低,可使CYP2C9 酶底物药物疗效下降或产生更多不良反应[29]。CYP2C8 是CYP2C 家族中最晚发现的一个成员,主要分布在肝脏,约占肝CYP 代谢酶总量的7%[32‑33]。CYP2C8 参与约5% 的临床药物的催化,并且存在着明显的基因多态性,对药物的代谢和清除有着显著影响[32,34],且伴随着肝癌疾病进程CYP2C8 活性也会显著降低[35]。 目前已经知道CYP2C8 的不同基因型对抗糖尿病药物[36]、抗肿瘤药物[37]和非甾体抗炎药[38]等药物的代谢有着重要影响。
4 结束语
本文对肝癌基因表达谱数据进行挖掘,从分子功能、生物学过程、网络相互作用的角度分析肝癌发生、发展过程中的关键基因,最后通过生存分析和文献挖掘验证,得出CYP3A4、CYP3A5、CYP2C9 和CYP2C8 基因适合作为肝癌标志物或有望成为靶向治疗靶点,为肝癌发生的机制研究、肿瘤标志物的筛选及药物靶点选择提供参考,为进一步开展相关功能研究提供了理论依据。
