基于分子对接技术筛选抗SARS-CoV-2海洋活性肽的研究
2021-09-17刘瑛楠朱文慧李学鹏仪淑敏徐永霞励建荣
步 营,刘瑛楠,何 玮,朱文慧,李学鹏,仪淑敏,徐永霞,励建荣
(渤海大学 食品科学与工程学院,辽宁锦州 121013)
0 引言
2019年12月,新型冠状病毒肺炎席卷全球。SARS-CoV-2作为一种新型病毒,突兀地登上了国际热点的舞台,威胁人民的生命安全,影响人民的正常生活。因此现阶段对于它的研究也在如火如荼的进行,例如对其的治疗、疫苗的研发、保健食品的生产等。
研究发现,SARS-CoV-2与SARS-CoV同属β型冠状病毒类,虽然现阶段SARS-CoV-2晶体结构不明,但其S蛋白RBD结构域的某些基因区域与SARS-CoV具有高度的同源性,它们的功能性受体皆为血管紧张素转化酶2(angiotensin converting enzyme 2,ACE2)[1],致病机理相同,皆为病毒的S蛋白与宿主细胞上的ACE2受体结合,从而致使病毒入侵宿主[2]。由此,可以选择与其同源性较高的SARS病毒的S蛋白和ACE2(即SARS-CoV-S/ACE2复合蛋白)作为代替的研究对象[3-5],进行新冠病毒抑制机理的研究。另一方面,由于2019-nCoV Mpro水解酶关系着SARSCoV-2前体蛋白的成熟,阻断其水解酶活性表达就可抑制病毒在宿主体内的复制过程。上海科技大学饶子和/杨海涛团队公开了2019-nCoV Mpro水解酶的高分辨率晶体结构,可以使其作为对SARS-CoV-2有抑制作用的另一研究对象,通过筛选抑制剂,有效遏制SARS-CoV-2在宿主体内的复制,阻断其增殖过程。
我国海洋资源丰富,蓝鳍金枪鱼属于大洋性中上层高度洄游鱼类,是金枪鱼类中经济价值最高的种类,而经酶解处理的蛋白质水解产物具有多种生物活性功能[6]。经数据库查询,蓝鳍金枪鱼的肌球蛋白序列已知。鉴于此,本文采用计算机酶解和分子对接的方法,对蓝鳍金枪鱼的肌球蛋白进行模拟酶解,进而筛选对新冠病毒有抑制作用的活性肽,为进一步应用于保健食品提供思路。
1 材料与方法
1.1 试验材料
蓝鳍金枪鱼的肌球蛋白可通过蛋白质数据库UniProt(https://www.uniprot.org/)查 询 得 到[7],UniProtKB ID号为G9M5T1的,其序列为FAST格式。
SARS-CoV病毒晶体结构的PDB代码为2AJF,选择仅保留晶体结构中的A链(ACE2)和E链(SARA-CoV-S)作为研究对象,结构如图1所示。2019-nCoV Mpro水解酶晶体结构的PDB代码为6LU7,如图2所示。

图1 SARS-CoV-S/ACE2复合蛋白的三维构型Fig.1 Three-dimensional configuration of SARS-CoV-S/ACE2 compound protein
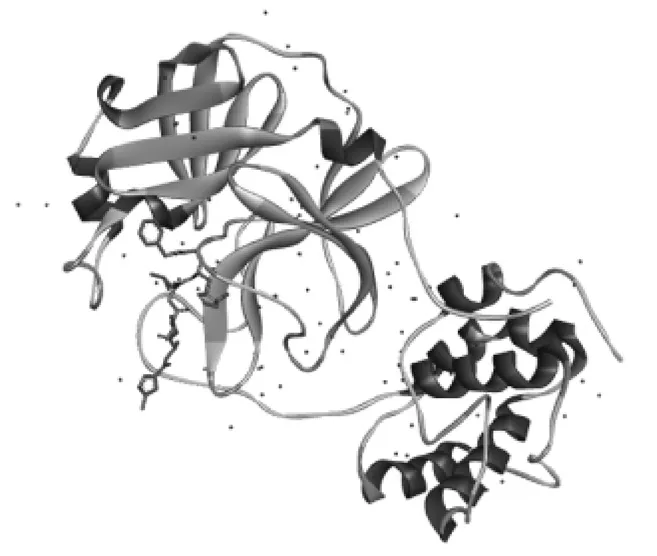
图2 2019-nCoV Mpro水解酶的三维构型Fig.2 Three-dimensional configuration of 2019-nCoV Mpro hydrolase
1.2 试验方法
1.2.1 计算机虚拟酶解
运用生物信息学的方法,以肌球蛋白G9M5T1序列作为原料,并通过酶切工具Peptide Cutter(https://web.expasy.org/peptide_cutter/)选择pepsin ph1.3,trypsin,chymotrypsin-low specificity(C-term to[FYW],not before P)3种酶对该蛋白进行模拟酶解[8]。
1.2.2 海洋活性肽的活性预测
使用活性预测工具 Peptide Ranker(http://distilldeep.ucd.ie/PeptideRanker/)对海洋多肽片段的活性进行预测,从中筛选高活性(>0.5)的肽片段。
1.2.3 海洋活性肽的毒性预测
对海洋活性肽使用毒性预测工具ToxinPred(http://crdd.osdd.net/raghava/toxinpred/)毒性预测,筛选无毒的海洋活性肽[9]。
1.2.4 筛选未知活性肽
使用生物活性肽数据库Peptides HGATSearch(http://www.peptides.be/?p=search)筛 选未知作用的海洋活性肽进行下一步的研究。
1.2.5 海洋活性肽三维结构的构建
使用 Discovery Studio 2017(DS,v17.2.016349)软件对酶解得到的高活性且无毒的海洋活性肽序列的三维结构进行构建。在DS软件中画出多肽结构,对其进行“Clean Geometry”处理,得到其三维结构。
1.2.6 分子对接
分子对接是一种用来研究分子间相互作用的一种方法[10]。去除SARS-CoV-S/ACE2复合蛋白和2019-nCoV Mpro水解酶的水分子和配体,并进行“Clean Protein”以及“Prepare Protein”的前处理操作。确定SARS-CoV-S/ACE2复合蛋白的关键氨基酸分别为ACE2片段上ASP38,GLN42,GLN325,GLU329,以 及 S蛋 白 上 的 TYR436,TYR491。确定2019-nCoV Mpro水解酶的关键氨基酸为 THR24,THR25,THR26,LEU27,ASN28以及ASN119[11]。对30条海洋活性肽片段进行“Full Minimization”和“Prepare Ligands”的前处理操作,与处理后的SARS-CoV-S/ACE2复合蛋白以及2019-nCoV Mpro水解酶分别进行Dock Ligands(LibDock)分子对接,选择“BEST”构象,其余参数均设置为默认状态[12]。与海洋活性肽配体对接结束后筛选结果中对接成功的化合物,选取分值(LibDockScore)最高的作为该化合物的对接结果。按照分值的大小将对接成功的配体排序,筛选与受体结合口袋中关键氨基酸结合作用力强的化合物,作为SARS-CoV-2与ACE2结合阻断剂以及2019-nCoV Mpro水解酶抑制剂的候选海洋活性肽[13]。
2 结果与分析
2.1 计算机模拟酶解结果
G9M5T1经trypsin模拟酶解后产生302条肽片段;经pepsin ph1.3酶解后产生369条肽片段;经chymotrypsin-low specificity (C-term to [FYW],not before P)酶解后产生392条肽片段。
2.2 活性预测结果
对所得到的肽片段使用Peptide Ranker工具分析,筛选活性肽,进行进一步的研究。筛选结果大于0.5的预测结果,经trypsin,pepsin ph1.3,chymotrypsin-low specificity(C-term to[FYW],not before P)酶解得到的肽片段中分别筛选出22条、17条和42条活性肽。
2.3 毒性预测结果
使用ToxinPred工具对活性肽进行毒性预测,筛选无毒活性肽,避免有毒物质进入人体从而对人体产生毒害作用。预测结果为所有活性肽均为无毒片段。
2.4 未知活性肽筛选结果
为避免与其他已知的活性肽进行重复性研究,使用Peptides HGAT-Search数据库对活性肽进行筛选,筛选经 trypsin,pepsin ph1.3,chymotrypsin-low specificity(C-term to[FYW],not before P)酶解后得到的未知无毒海洋活性肽,其结果如表1所示(分别用T*,P*,C*表示)。筛选结果即为对接过程最终所用的海洋活性肽配体序列,对其进行三维结构的构建。

表1 G9M5T1酶解后得到的未知海洋无毒活性肽Tab.1 Unknown marine non-toxic active peptides obtained after G9M5T1 enzymolysis
2.5 分子对接结果
2.5.1 SARS-CoV-2与ACE2结合抑制剂筛选结果
表1中的30条海洋活性肽中的19条与SARS-CoV-S/ACE2复合蛋白对接成功。氢键的分子作用力较大,结合作用较强,故筛选对接结果中与关键氨基酸以氢键作用相结合的10条海洋活性肽,作为潜在的对与ACE2的结合有阻断作用的活性肽片段,结果如表2所示。该10条多肽皆与SARS-CoV-S/ACE2蛋白对接口袋中关键氨基酸形成氢键化合物,其中,C4与TYR491形成3个氢键,分值为144.909;C8与TYR491形成2个氢键,T13与GLN325形成1个氢键,P5与GLN325形成2个氢键,P6与GLU329形成2个氢键外,它们还与其他关键氨基酸形成范德华力等其他作用力,分值分别为164.154、149.265、133.059、119.918,如图3所示。综上所述,活性肽C4,C8,T13,P5,P6 经分子对接后与对接受体结合作用较强,分析病毒入侵宿主的机理可知,以上活性肽对SARS-CoV与ACE2的结合有潜在的强阻止作用,能阻断S蛋白与ACE2受体结合,从而阻止SARS-CoV-2入侵宿主细胞。可进一步应用于对新型冠状病毒肺炎的预防等过程。

表2 对SARS-CoV-2结合ACE2阻断剂的筛选Tab.2 Screening of SARS-CoV-2 combined with ACE2 blocker
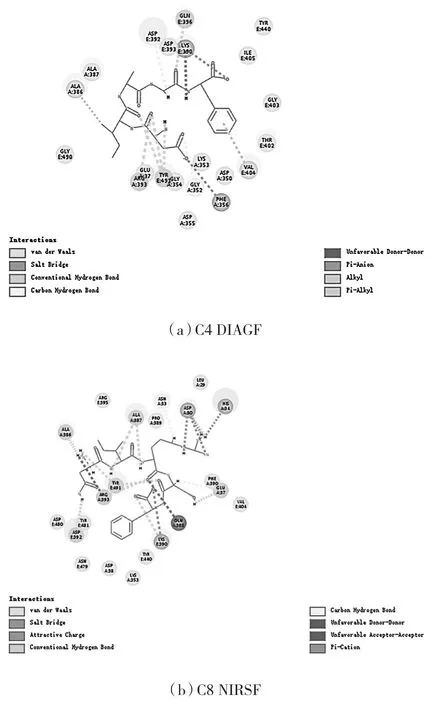
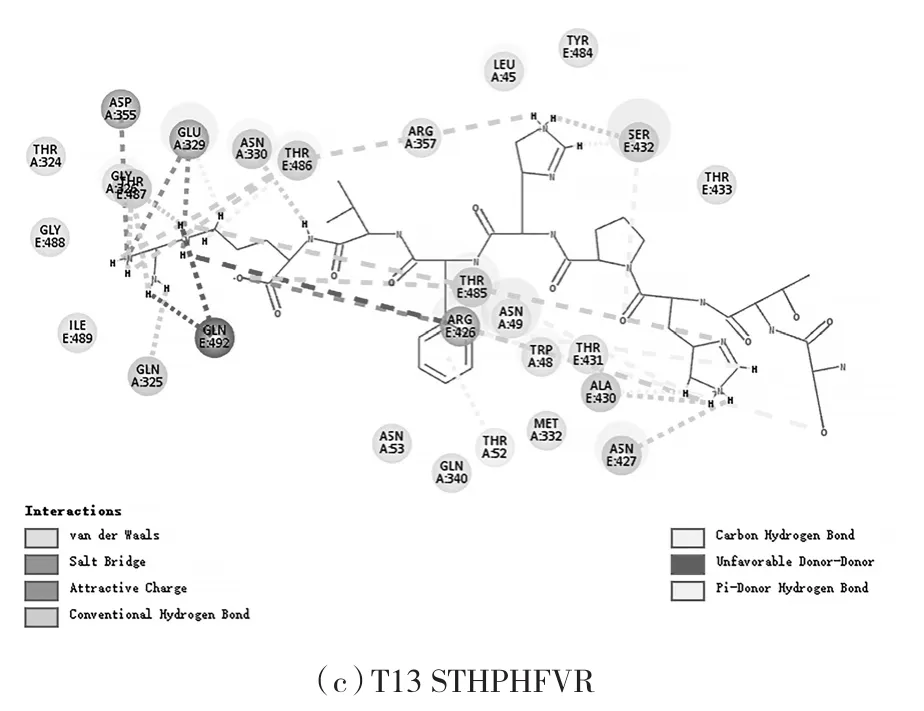
图3 活性肽段与SARS-CoV-S/ACE2复合蛋白对接平面图Fig.3 View of active peptides docking with SARS-CoV-S/ACE2 complex protein
2.5.2 2019-nCoV Mpro水解酶抑制剂筛选结果
海洋活性肽与2019-nCoV Mpro水解酶进行分子对接后,有25条活性肽成功与Mpro水解酶受体对接成功,其中18条肽片段与关键氨基酸以氢键的形式结合,结果如表3所示。其中C7,T13与 THR24,THR26,ASN119形成氢键,分值分别为 199.696 和 183.583;T9,P5,C8 与 THR26,ASN119形成氢键,分值分别为199.490、180.616和 175.280;P7、T11 与 THR24,THR26 形成氢键,分值 分别为 182.785和 162.997;T8与 THR24,THR25,THR26形成氢键,分值为177.758;P9与THR25,THR26形成氢键,分值为163.683。此外,这些海洋活性肽不仅与对接受体形成了多个氢键,还与关键氨基酸间形成范德华力等作用力。T3,C6与 THR26形成 2个氢键,且与 THR24,THR25,LEU27间形成范德华力,分值分别为163.379、156.337;C4与 THR24形成 2个氢键,与THR24,THR26,LEU27间有范德华力作用,分值为 150.652;P8,P6,T5,P3,T12,T10,除与关键氨基酸形成的氢键外,还与THR24,THR25,THR26,LEU27,ASN119等关键氨基酸间形成其他分子间作用力,分值分别为 191.320、188.958、179.405、177.136、143.197。对接结果见图4所示。综上所述,海洋活性肽片段 C7,T13,T9,P5,C8,P7,T11,T8,T5,P9,T3,C6,C4,P8,P6,P3,T12,T10 与受体对接后可形成较强的分子间作用力,可用作筛选2019-nCoV Mpro水解酶抑制剂[14],而抑制 2019-nCoV Mpro水解酶的活性表达可以影响SARSCoV-2前体蛋白的成熟过程,进一步影响病毒在宿主体内的复制过程[15-16],从而抑制SARSCoV-2的表达。
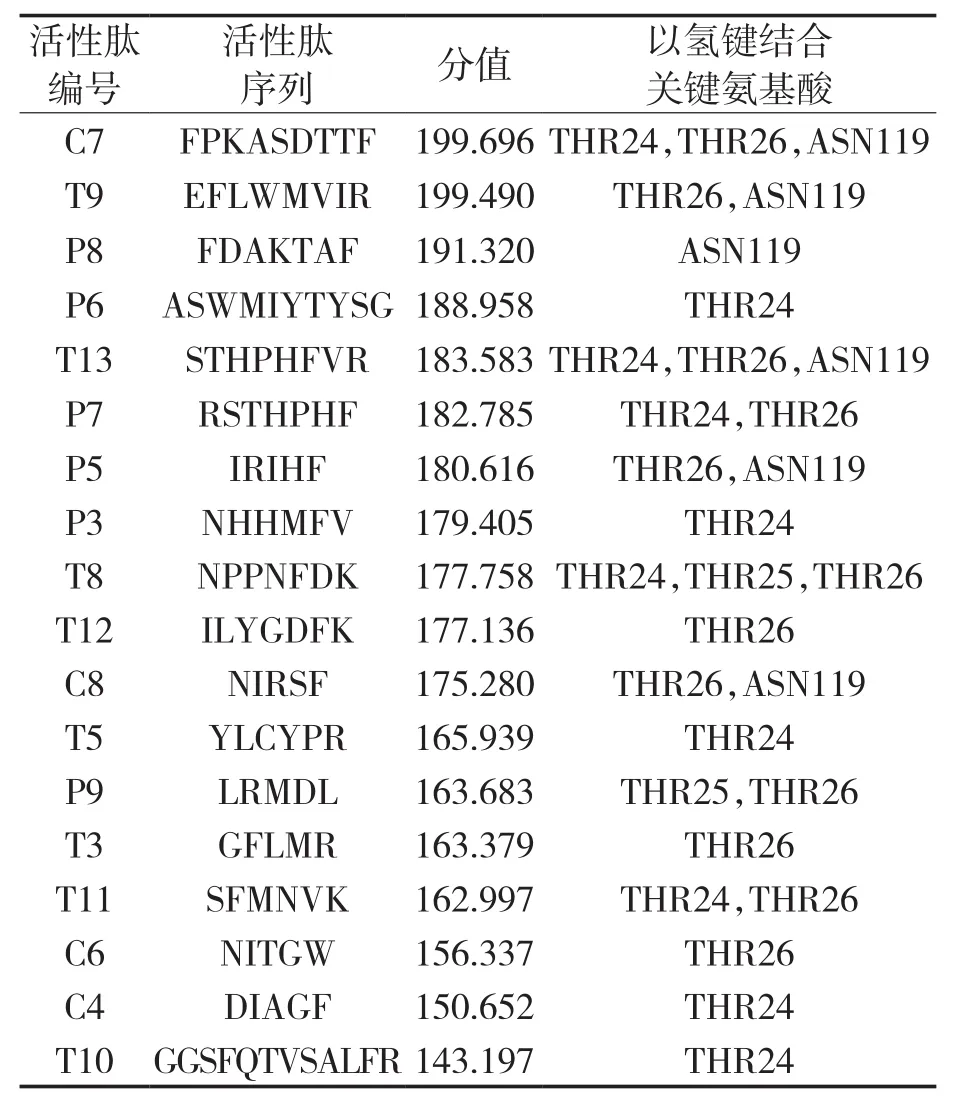
表3 对2019-nCoV Mpro水解酶阻断剂的筛选Tab.3 Screening of 2019-nCoV Mpro hydrolase blocker
3 结论
以SARS-CoV-S/ACE2复合蛋白以及2019-nCoV Mpro水解酶的晶体结构为受体,与30条未知的无毒海洋活性肽进行半柔性分子对接。结果可知,海洋活性肽片段 DIAGF,NIRSF,IRIHF,ASWMIYTYSG,STHPHFVR 与 SARS-CoV-S/ACE2复合蛋白受体结合紧密,是潜在的SARS-CoV-2与ACE2结合阻断剂;海洋活性肽片段FPKASDTTF,STHPHFVR,EFLWMVIR,IRIHF,NIRSF,RSTHPHF,SFMNVK,NPPNFDK,LRMDL,GFLMR,NITGW,DIAGF,FDAKTAF,ASWMIYTYSG,NHHMFV,ILYGDFK,GGSFQTVSALFR 与 Mpro 水解酶结合作用较强,是潜在的2019-nCoV Mpro水解酶抑制剂。
综上所述,活性肽段NIRSF,STHPHFVR,DIAGF,IRIHF和ASWMIYTYSG对两种对接受体的表达均有良好的抑制作用,是SARS-CoV-2的潜在抑制剂,可进一步应用于保健食品的开发制造。
