一株重组类NADC30猪繁殖与呼吸综合征病毒的分离鉴定及基因组分析
2021-09-16袁林茂张维达李自力
杨 康,袁林茂,张维达,李自力*
(1.农业微生物学国家重点实验室,华中农业大学动物医学院,湖北省预防兽医学重点实验室,武汉430070;2.上海新农饲料股份有限公司,上海201600)
猪繁殖与呼吸综合征俗称猪蓝耳病,是由猪繁殖与呼吸综合征病毒(Porcine Reproductive and Respiratory Syndrome Virus,PRRSV)引起猪繁殖障碍和呼吸系统损伤的高度接触性传染病,病猪和带毒猪是主要传染源[1]。该病最早于1987年在美国被报道,随后在许多国家出现。PRRSV分为两个基因型,即以LV为代表的欧洲型和以VR2332为代表的美洲型[2-3]。1996年,郭宝清等[4]在中国首次分离到PRRSV CH-1a株。2006年,田克恭等[5]首次报道中国暴发高热病,后确定是由高致病性PRRSV(Highly Pathogenic Porcine Reproductive and Respiratory Syndrome Virus,HP-PRRSV)所致,其Nsp2基因存在30(1+29)个氨基酸的不连续缺失(相对于CH-1a),这是判断HP-PRRSV的重要分子特征。2008年,在美国报道了一株 Nsp2 基因存在131个不连续氨基酸缺失的PRRSV,命名为NADC30[6]。2013年,周峰等[7]对河南省猪繁殖与呼吸综合征进行了流行病学调查,发现了与美国NADC30同源性较高的毒株,由于这类毒株与NADC30亲缘关系较近,国内现将该类病毒统称为类NADC30 PRRSV(NADC30-like PRRSV)。目前我国大部分省份均有类NADC30株检出,且阳性率逐年上升,类NADC30株现已成为多地的优势毒株。
PRRSV具有变异快的特点,特别是其非结构蛋白Nsp2、结构蛋白GP5变异最大,已有许多依据Nsp2及ORF5作为PRRSV遗传进化及基因分型的相关报道[8-9]。Shi等[10]对全球8624份PRRSV ORF5基因序列进行分析,将北美型PRRSV分为9个谱系(lineage1~9)。目前我国PRRSV分为4个谱系:lineage 1、lineage 3、lineage 5.1和lineage 8。也有国内学者根据Nsp2序列的差异,将我国PRRSV分为经典PRRSV、HP-PRRSV、类NADC30 PRRSV、疫苗毒株等。
目前商品化的疫苗主要针对经典PRRSV和HP-PRRSV。Bai等[11]建立了类NADC30 PRRSV的攻毒模型,评价了几种最主要的商品化疫苗的保护效力,证明现有疫苗不能对类 NADC30 PRRSV提供完全保护。因此,类NADC30株的分离工作意义重大。本研究旨在分离类NADC30株并分析其分子遗传进化特点,从而为上海地区的猪繁殖与呼吸综合征防控提供参考,为进一步研究其重组机制、致病特点及研制预防类NADC30 PRRSV疫苗等奠定基础。
1 材料与方法
1.1 主要实验材料 2份疑似猪繁殖与呼吸综合征肺脏病料采自上海某猪场发病猪;E.coliDH5α感受态细胞由华中农业大学湖北省预防兽医学重点实验室制备并保存;HP-PRRSV(XJ株)由华中农业大学湖北省预防兽医学重点实验室分离并保存;pMD18-T克隆载体购自大连宝生物有限公司;胎牛血清购自Gbico公司;DMEM培养基购自Hyclone公司;高保真DNA聚合酶(Phanta®Max Super-Fidelity DNA Polymerase,P505-d1)、反转录试剂盒(R223-01)购自南京诺唯赞生物科技有限公司;FITC标记羊抗鼠IgG购自ABclonal 公司;抗PRRSV GP5的单克隆抗体(MAb)由华中农业大学动科动医学院李建惠赠。
1.2 引物设计与合成 依照周峰[7]设计鉴别引物的原理,参考经典PRRSV CH-1a(AF132118)、HP-PRRSV JXA1(EF112445)、NADC30 PRRSV(MH500766)Nsp2基因序列,使用SnapGene设计Nsp2特异性鉴别引物(上游引物:5’-TGATTGGAATGTTGTGCTTCCTGGG-3,下游引物:5’-TGTCAAGCGTGAGAATCACACGCCC-3’),可扩增出经典PRRSV(1047 bp)、HP-PRRSV(957 bp)、类NADC30 PRRSV(654 bp);根据NADC30(MH500766)的基因组序列设计10对重叠基因片段引物(表1),引物由北京擎科生物科技有限公司合成。

表1 全基因扩增引物
1.3 猪肺泡巨噬细胞(PAMs)的获取 参照孟春花等[12]所采用的方法制备并保存猪肺泡巨噬细胞。
1.4 病料处理和RT-PCR检测 将1 g肺组织剪碎加入装有适量PBS的离心管中,加入钢珠后将离心管置于组织破碎仪中充分破碎,离心取上清后参照张维达等[13]报道的方法提取总RNA。按反转录试剂盒说明书将总RNA反转录为cDNA。PCR反应条件为95 ℃ 3 min;94 ℃ 15 s、60 ℃ 30 s、72 ℃ 1 min、35个循环;最后72 ℃ 10 min。取适量PCR产物经1%琼脂糖凝胶电泳检测。
1.5 病毒的分离、鉴定及纯化 将步骤1.4中经RT-PCR检测呈阳性的样品接种于铺满单层PAMs的6孔细胞培养板内。细胞培养板置于37 ℃ 5% CO2培养3~5 d。接种后每天观察细胞状态,盲传3代后收集细胞培养物经RT-PCR鉴定。经RT-PCR检测为阳性的细胞上清接种PAMs,培养48 h后,用4%甲醛室温固定30 min,0.1%Triton X-100室温穿透15 min,使用1∶200稀释抗PRRSV-GP5单克隆抗体于37 ℃孵育1 h,再用1∶500稀释FITC标记羊抗鼠 IgG 37 ℃孵育1 h,同时设正常细胞作为对照,经IFA鉴定。
按照王培培所采用的方法[14]进行病毒噬斑纯化。使用PAMs扩繁病毒,将收获的病毒液经超滤浓缩,蔗糖密度梯度离心后经透射电镜检测。
1.6 分离株的基因组扩增、克隆和测序 将分离株按照步骤1.4的操作提总RNA,经RT-PCR分段扩增,PCR产物纯化回收后连接到pMD18-T载体,转化,经PCR鉴定为阳性的重组质粒由上海生物工程股份有限公司测序。
1.7 基因序列同源性以及遗传进化分析 利用软件SeqMan(version 7.1.0)对测序结果进行重叠片段序列拼接,采用软件MegAlign(version 7.1.0)和MEGA(version 6.0)对分离株全基因组序列与GenBank登录的国内外毒株全基因组序列进行同源性和遗传进化分析,同时对分离株Nsp2与GP5氨基酸序列进行同源性及遗传进化分析,利用软件SimPlot(version 3.5.1) [200 bp window sliding along the genome alignment (20-bp step size)]对分离株全基因组序列和参考株基因序列进行重组分析。
1.8 分离株对Marc-145细胞的嗜性分析 将分离株接种Marc-145细胞,每天观察细胞状态,接种48~72 h后经RT-PCR检测PRRSV核酸,同时经IFA检测PRRSV GP5,将收获的细胞上清传代培养3次,评价分离株的细胞嗜性。
2 结果与分析
2.1 临床样品的检测 以患病猪肺脏RNA为模板,针对PRRSV Nsp2基因的鉴别引物进行RT-PCR,结果显示1号肺样为弱阳性,2号肺样为强阳性,且扩增条带为654 bp,阳性对照扩增出957 bp条带(图1)。将2号肺样的PCR产物送测序,经BLAST序列比对,发现样品中病毒和NADC30的部分Nsp2基因的同源性较高。
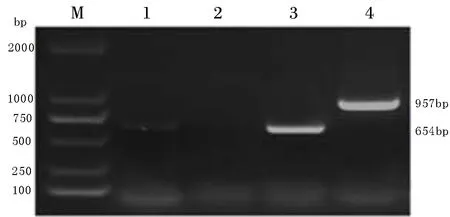
M:DL2000 Marker;1:1号肺样;2:阴性对照;3:2号肺样;4:阳性对照(XJ,HP-PRRSV)
2.2 病毒的分离鉴定 将检测为阳性的样品经0.22 μm滤器过滤后接种PAMs,在PAMs中连续传代3代后可出现稳定且典型的细胞病变(CPE),使用台盼蓝对接种72 h的细胞培养物染色,经普通光学显微镜观察,可见细胞脱落、破碎(图2B)。使用鼠抗PRRSV GP5单克隆抗体和FITC标记羊抗鼠IgG对感染48 h的PAMs进行间接免疫荧光鉴定,经荧光显微镜观察,可见接种阳性样品的PAMs呈现绿色荧光,为抗原阳性反应(图2D)。
将噬斑纯化后扩大培养并储存,将制备好的病毒样品经透射电镜观察,可见直径为50 nm左右的圆形颗粒(图2E)。以上实验数据表明成功分离出一株PRRSV,将其命名为SH2019。
2.3 PRRSV的全基因组扩增,同源性及遗传进化分析
2.3.1 PRRSV的全基因组扩增 将病毒约为15 kb的全基因组序列按参考株NADC30分为10个重叠基因片段,使用10对引物进行RT-PCR扩增,扩增结果如下所示(图3),序列拼接结果显示SH2019的全基因组大小为14677 bp(不含5'UTR和3'UTR)。
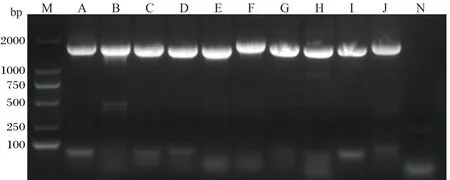
M:DL2000 Marker;A~J:单个基因片段;N:阴性对照
2.3.2 分离株全基因组的同源性分析及遗传进化分析 将拼接完的全基因组序列与GenBank登录的参考毒株序列进行同源性分析和遗传进化分析。同源性比对结果显示,分离株SH2019与欧洲型代表株LV的同源性为60.5%,与北美型PRRSV的同源性在82.5%~91.3%,与NADC30的同源性最高,为91.3%。遗传进化树分析结果显示,SH2019与中国本土类NADC30株HENAN-XINX、CHsx1401有很近的亲缘关系,这些分离株归属在流行于中国大陆的北美型PRRSV谱系1中(图4A和4B)。上述结果表明,分离株SH2019属于类NADC30 PRRSV。
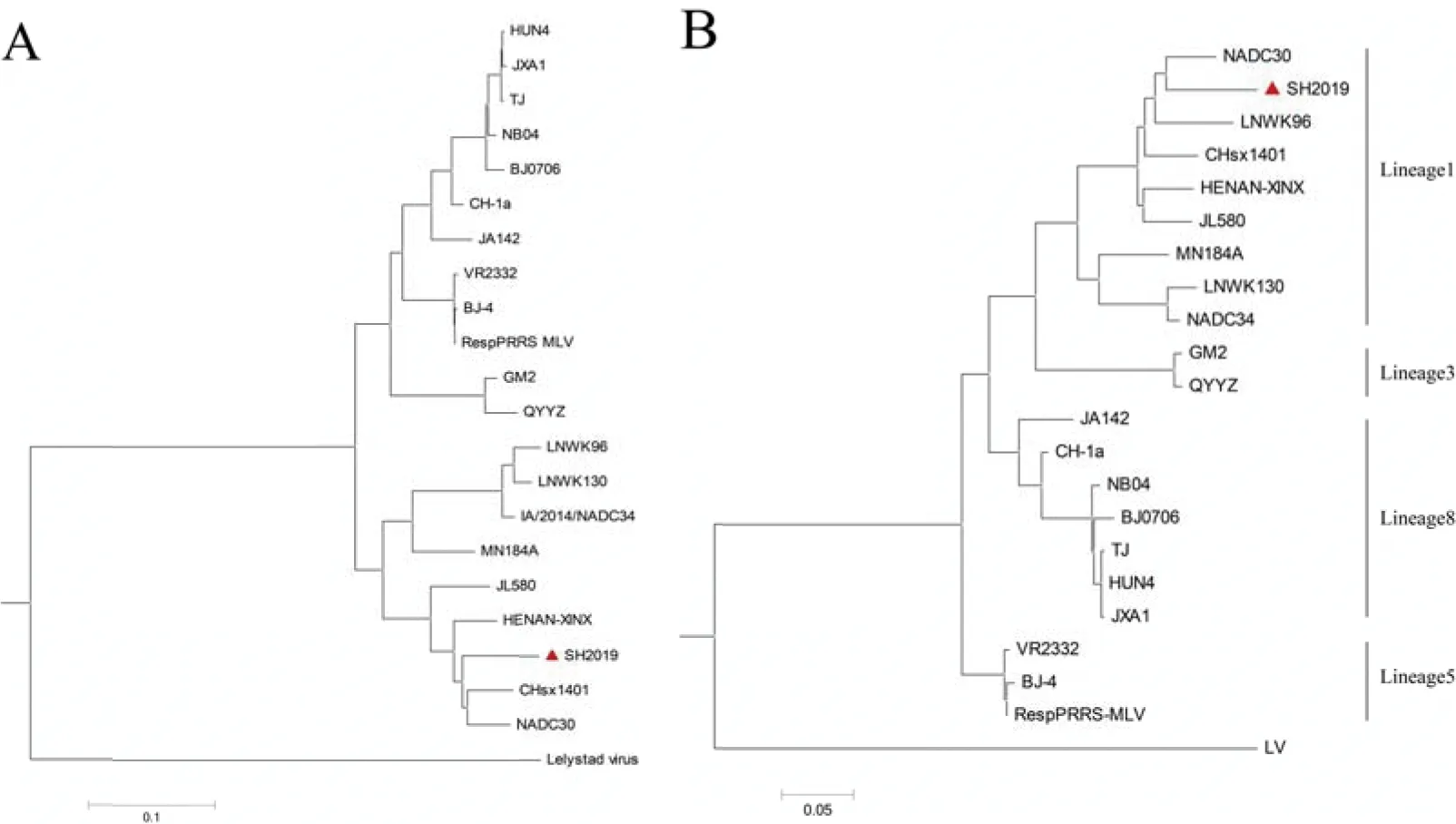
分离株用▲标注(the isolate was marked with ▲)
2.4 分离株Nsp2、ORF5基因编码的氨基酸序列分析
2.4.1 分离株Nsp2基因编码氨基酸序列的不连续缺失分析 氨基酸序列比对结果显示,分离株SH2019相对于经典毒株(CH-1a)在Nsp2中存在NADC30特征性的131(111+1+19)个氨基酸缺失(图5)。

分离株用_标注(the isolate was marked with_)
2.4.2 分离株Nsp2、ORF5基因编码氨基酸序列的同源性及遗传进化分析 根据Nsp2、GP5氨基酸序列同源性比对结果,分离株SH2019与欧洲代表株LV的Nsp2氨基酸序列同源性仅为49.8%,但与NADC30的同源性最高,为89.9%;分离株SH2019与欧洲型代表株LV的GP5氨基酸序列同源性最低,为54.6%,与NADC30的同源性最高,为89.4%。Nsp2、GP5氨基酸序列的遗传进化分析结果显示,分离株SH2019与中国本土类NADC30株HENAN-XINX、CHsx1401及北美株NADC30属于同一个谱系(图6)。上述结果进一步证实,分离株SH2019属于类NADC30 PRRSV。

分离株用▲标注(the isolate was marked with ▲)
2.5 分离株的基因组重组分析 以分离株SH2019全基因组序列为查询序列,根据软件Simplot分析结果,发现全基因组有3个重组断点(12601 nt、12761 nt、12901 nt),将分离株SH2019全基因组分为4个片段(图7A)。片段1~12600 nt和片段12902~14677 nt与NADC30的同源性最高,分别为91.0%和93.4%;片段12601~12761 nt与QYYZ的同源性最高,为91.3%;片段12762~12901 nt与BJ-4或JXA1的同源性最高,均为94.2%。进一步将4个基因片段分别制作遗传进化树,结果见图7B。由试验数据可知,分离株SH2019是以谱系1(代表株NADC30)的毒株为主要亲本,以谱系3(代表株QYYZ)、谱系5(代表株VR2332、BJ-4)或谱系8(代表株JXA1)的毒株为次要亲本,在12601~12901 nt(ORF2~ORF4)发生了基因重组。上述结果表明,分离株SH2019是发生基因重组的类NADC30 PRRSV。
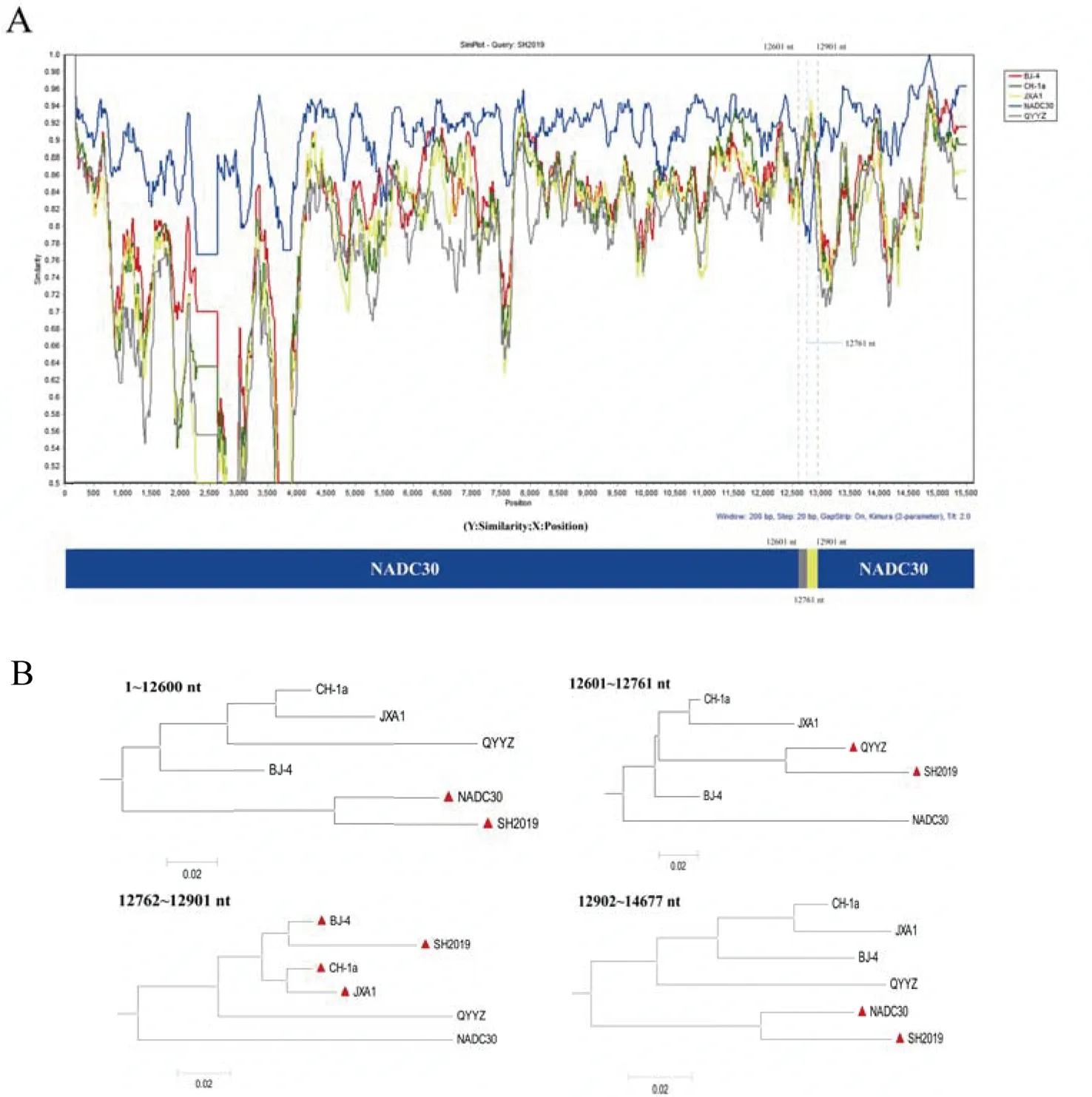
与分离株SH2019处于同一谱系的毒株均标注▲
2.6 分离株对Marc-145细胞的嗜性分析 将分离株SH2019接种Marc-145细胞48~72 h后,光学显微镜观察细胞是否出现CPE,同时经RT-PCR、IFA检测分离株能否感染Marc-145细胞。结果显示,并未观察到明显的CPE,RT-PCR、IFA检测也均为阴性,传3代每代检测都为阴性。第3代细胞培养物的IFA检测结果见图8。上述结果表明,分离株SH2019对Marc-145细胞无嗜性。
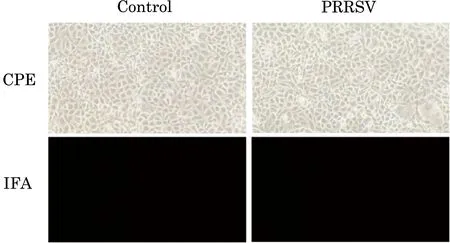
图8 分离株对Marc-145细胞的嗜性分析(100×)
3 结论与讨论
自2013年首次发现类NADC30 PRRSV以来[7],这类毒株便在我国迅速流行,对我国养猪业造成了较大的经济损失。由于现阶段商品化疫苗仅能对类NADC30 PRRSV提供部分保护力[11],因此分离类NADC30 PRRSV具有一定应用价值,可为以后研制相关疫苗奠定基础。
本研究最初采用Marc-45细胞分离PRRSV,将经RT-PCR检测为阳性的临床样品接种Marc-145细胞后,镜检未发现CPE,细胞培养物经RT-PCR检测也呈阴性,因此改用PAMs分离病毒。张洪亮等[15]报道的15株类NADC30 PRRSV中有13株病毒只能感染PAMs而不能感染Marc-145细胞;阮海宇等[16]用Marc-145细胞对类NADC30 PRRSV尝试进行分离,发现该毒株在Marc-145上适应性不好,不能直接用Marc-145分离该病毒。由于很多类NADC30株不能在Marc-145细胞中复制,而PAMs的获取成本较高,所以研制预防类NADC30株的疫苗具有较大难度。由此构建对类NADC30株高敏感性的细胞系可为疫苗的研制提供一条路径。
类NADC30株较经典PRRSV和HP-PRRSV具有更高的重组概率,目前很多类NADC30株以NADC30为主要亲本,以谱系3、5、8中的毒株为次要亲本,在不确定的基因组位置发生重组。例如Liu等[17]分离的类NADC30株(FJLIUY-2017)是以NADC30为主要亲本,以类VR2332株(12013 ~13282 nt)、类QYYZ株(13857 ~15140 nt)、类JXA1株(1~709 nt、1274~1829 nt、6890~9528 nt及11806~11524 nt)为次要亲本进行基因重组的毒株。目前关于类NADC30株发生重组的具体机制尚无准确认知,重组病毒的生物学特性可能会发生改变,越来越多重组毒株的出现给我国猪繁殖与呼吸综合征的防控和净化带来了巨大的挑战。
Zhang等[18]通过反向遗传学技术证明类NADC30株(MY-376,只能感染PAMs,而不能感染Marc-145细胞)的次要结构蛋白GP2a、GP3决定了其对Marc-145细胞的嗜性。目前一般认为PRRSV入侵细胞包含以下几步:首先PRRSV颗粒的异源二聚体(GP5/M)通过和胞膜上的硫酸乙酰肝素(pHS)互作而聚集在细胞表面,但是该作用较弱,接着唾液酸黏附素(pSn)与GP5/M相互结合强化第一步的吸附作用,并在网格蛋白介导的内吞作用下,病毒-受体复合物内化进入胞内体,并且这种配体-受体的互作十分依赖于pSn的唾液酸结合能力。随后病毒颗粒的次要结构蛋白复合物(GP2a/GP3/GP4)与必需受体pCD163互作并在胞内体酶的酸性条件下介导PRRSV脱衣壳和基因组释放,其中GP2a 184位天冬酰胺的N-糖基化对互作起了关键的作用[19],其他受体(vimentin、CD151、DC-SIGN)也在PRRSV侵入过程中发挥了一定作用。对分离株SH2019 GP2a糖基化位点预测,发现其同样具有GP2a 184位天冬酰胺的N-糖基化现象,由此推测不太可能是GP2a与CD163互作的改变而影响分离株的细胞嗜性。由于分离株在ORF2~ORF4的基因组位置上和类QYYZ、类VR2332或类JXA1株发生了重组,推测可能是基因重组导致了分离株SH2019不能感染Marc-145细胞,但是还需要进一步研究来解释该现象。也有实验室通过构建稳定表达PAMs CD163(pCD163)的Marc-145细胞系,发现与野生型细胞相比,Marc-145pCD163可成功感染类NADC30株(SD17-38),且经过多次重复验证,该毒株并不能在野生型Marc-145细胞中成功复制[20]。由于Marc-145细胞与PAMs均表达CD163蛋白,这提示不同物种间CD163蛋白的差异性也可能与类NADC30株的细胞嗜性有关。
本研究分离鉴定了一株重组类NADC30 PRRSV,丰富了类NADC30株的信息,可为上海地区猪繁殖与呼吸综合征的临床防控提供参考。
