胃癌VEGF通路相关基因的预后模型的构建
2021-09-16田莲莲章屹然王兆熹
田莲莲,朱 军,马 骞,章屹然,王兆熹,陈 睿
胃癌(gastric cancer,GC)是最常见消化道恶性肿瘤之一。据估计,中国平均每年约有200万人死于胃癌,占全球胃癌死亡人数的近1/2[1]。早期胃癌治疗效果较好,但检出率很低,大多数患者(>70%)检出时已发展为晚期胃癌。部分患者甚至失去进行手术切除的机会或者发生转移,因此晚期胃癌总体预后较差[2-4]。故对于胃癌的预后预测是临床工作中的重点内容且具有较大价值。除了分级分期系统,可以寻找一些新颖的手段进行预后预测。
研究表明肿瘤的生长和扩张与肿瘤内血管网络的发展密切相关[5]。血管生成受到某些关键分子的调节。其中,血管内皮生长因子(vascular endothelial growth factor,VEGF)是驱动肿瘤血管生成的最重要因素之一。VEGF家族由7个成员组成:VEGF-A,VEGF-B,VEGF-C,VEGF-D,VEGF-E,VEGF-F和胎盘生长因子。这些蛋白质主要通过内皮细胞上表达的特异性酪氨酸激酶受体起作用。这些分子中的大多数通过血小板运输,并在血液凝固过程中,由血小板脱颗粒后释放到血清中,只有小部分在血液中自由循环[6]。研究发现阻断血管生成是预防肿瘤生长的良好策略[5]。由于VEGF在大多数人类癌症中的表达增加以及与肿瘤微血管密度增加和预后不良相关,因此VEGF通路已成为抗血管生成治疗的主要靶标[7]。靶向VEGF治疗(如贝伐单抗、雷莫芦单抗)联合化疗已被证实具有抗肿瘤活性,具有较好的耐受性和安全性,可改善胃癌患者生存率[8-9]。因此筛选出胃癌VEGF通路相关基因,建立一个立足于胃癌VEGF通路相关基因预后模型,对于预后评估或为个体化治疗提供新视角,甚至作为靶向治疗的有效性指标,具有重大临床意义。
1 材料与方法
1.1 数据来源 从TCGA官网(https://portal.gdc.cancer.gov/)下载胃癌转录组数据及其相应的临床数据,其中包括癌组织375例和癌旁组织32例。将转录组数据与临床生存数据匹配,最终完整临床信息的胃癌患者共计371例。VEGF通路相关基因集在GSEA官网(https://www.gsea-msigdb.org/gsea/index.jsp)的MSigDB (Molecular Signatures Database)数据库下载。其中包括BIOCARTA VEGF PATHWAY和REACTOME SIGNALING BY VEGF。以这2个VEGF数据库为基础,进行GSEA富集分析。外部数据集来自GEO数据库,下载胃癌基因芯片数据GSE84437,用于验证VEGF通路基因预后模型的准确性。数据进行log2转换后提取模型基因,最后用生存曲线的方式验证模型准确性。
1.2 GSEA富集分析 GSEA富集分析是使用GSEA软件(版本4.0.1),并且使用1000次排列数。按照癌组织与癌旁组织分为2组,富集上述已经下载的2个VEGF通路基因。筛选标准为:基因数目大于15个,P值小于0.05,标准化富集分数绝对值(Standardized enrichment fraction,NES)大于1.2。
1.3 筛选差异表达的VEGF通路相关基因 在癌组织与癌旁组织的全转录组中提取VEGF通路相关基因219个。使用Wilcoxon秩和检验,对差异基因进行差异性分析。通过Benjamin-Hochberg方法对P值进行校正。差异基因筛选标准为:P<0.05。
1.4 构建预后模型及绘制ROC曲线 将上述VEGF通路差异表达基因与胃癌基本临床信息合并。使用生存曲线和单因素COX风险回归分析筛选出与预后相关基因。随后进行多因素COX风险回归分析,采取向前向后法,筛选出赤池信息量准则(Akaike information criterion,AIC)最小的模型,并且得到模型中各个基因的比例系数β。然后根据公式计算每位患者的风险值,以中位数为界限将患者人群分为高风险组(大于中位数)和低风险组(小于中位数),其中风险值的计算公式为:β1×expression(gene 1) +β2×expression(gene 2) + …βn×expression(gene n)。ROC曲线用来验证模型的预测能力。最后使用R语言中timeROC包分别绘制1、3年和5年的ROC曲线并且计算相应AUC。
1.5 构建VEGF通路基因相关列线图 使用R语言rms包绘制列线图和校正图进一步验证模型基因的临床价值。列线图被广泛用于癌症预后,主要是因为它们能够将统计预测模型简化为针对个别癌症患者情况而定的事件(例如死亡或复发)概率的单个数值估计[10]。用于生成这些估计值的用户友好型图形界面有助于使用列线图来告知患者临床决策[10]。
1.6 统计学处理 所有统计方式都是在R软件(版本:3.63)中完成。计量资料以±s表示,采用t检验或方差分析。非正态分布数据检验使用Wilcoxon秩和检验。生存分析结果采用Kaplan-Meier曲线,其检验方式为Log-Rank法。所有检验为双侧检验,以P<0.05为差异有统计学意义。单因素和多因素COX风险比例回归分析筛选并确定VEGF通路相关基因的胃癌预后模型。本研究中相应的R语言数据包有:survival包、edgeR包、rms包、timeROC包及一些R语言自带的基础绘图包。
2 结果
2.1 差异基因在VEGF通路显著富集 GSEA结果显示,与胃癌正常的癌旁组织(蓝色)比较,癌组织(红色)在2个VEGF通路上显著富集(P<0.05,NES>1.2)(图1)。这表明在胃癌的发生发展过程中,VEGF通路的异常激活起到了重要作用。
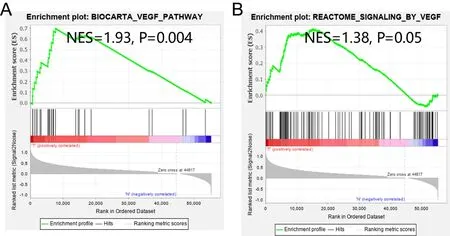
图1 癌组织与癌旁组织差异基因在VEGF通路显著富集
2.2 胃癌VEGF通路基因的差异表达 2个VEGF通路提取的基因共219个,使用差异分析的方式筛选出154个差异基因(P<0.05)。其中34个基因下降,120个基因上升。为了建立最优的模型,对logFC没有限定(|logFC|>0)。这也验证了大部分的VEGF基因在癌组织与癌旁组织异常表达。
2.3 预后模型的筛选与确定 首先对上述154个VEGF差异基因进行与生存数据的合并。使用单因素COX风险回归分析,确定了28个与预后相关的VEGF通路基因(P<0.05),用于后续模型的构建。通过多因素COX风险回归分析建立模型,前进后退法筛选最小AIC值,最终确定6个基因组成的预后模型,这6个基因分别为锚蛋白重复域1(ankyrin repeat domain 1,ANKRD1)、整联蛋白α-Ⅴ(integrin alpha-V,ITGAV)、内皮一氧化氮合酶(nitric oxide synthase,NOS3)、神经菌毛蛋白1(neuropilin-1,NRP1)、Ⅵ型胶原蛋白α3(collagen type Ⅵ alpha 3,COL6A3)、骨膜素(periosteal protein,POSTN)。可得,风险值的计算公式为:风险值=0.0269×ANKRD1+0.0181×ITGAV+0.0697×NOS3+0.0549×NRP1+ (-0.0078)×COL6A3 +0.005×POSTN。
2.4 预后模型的评估 根据计算公式得到每位患者的风险值,按照二分法(中位数)将患者分为高风险和低风险2组。Kaplan-Meier曲线显示:高风险组的预后要远远差于低风险组(P<0.05)(图2A)。高风险组的中位生存期为1.66年,低风险组的中位生存期为4.60年。绘制时间依赖的ROC曲线,结果显示,该模型的1、3年和5年AUC分别为65.4%、72.2%和73.0%(图2B)。这表明该模型可以较为准确的预测胃癌患者预后。
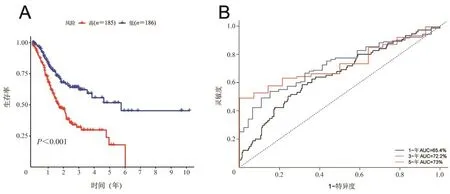
图2 预后模型的评估
2.5 预后模型独立于临床因素指导预后 首先通过单因素COX风险回归分析筛选胃癌预后相关临床因素,并绘制森林图。单因素森林图显示年龄(风险值=1.026,95%CI:1.008~1.044),TNM分期(风险值=1.534,95%CI:1.241~1.896)和本模型(风险值=1.235,95%CI:1.153~1.323)与胃癌预后息息相关(P<0.05)。随后将上述结果进一步纳入多因素COX风险回归分析,仍然显示年龄(风险值=1.033,95%CI:1.014~1.052),TNM分期(风险值=1.593,95%CI:1.279~1.985)和本模型(风险值=1.239,95%CI:1.146~1.339)与胃癌患者预后相关(P<0.05)(图3)。
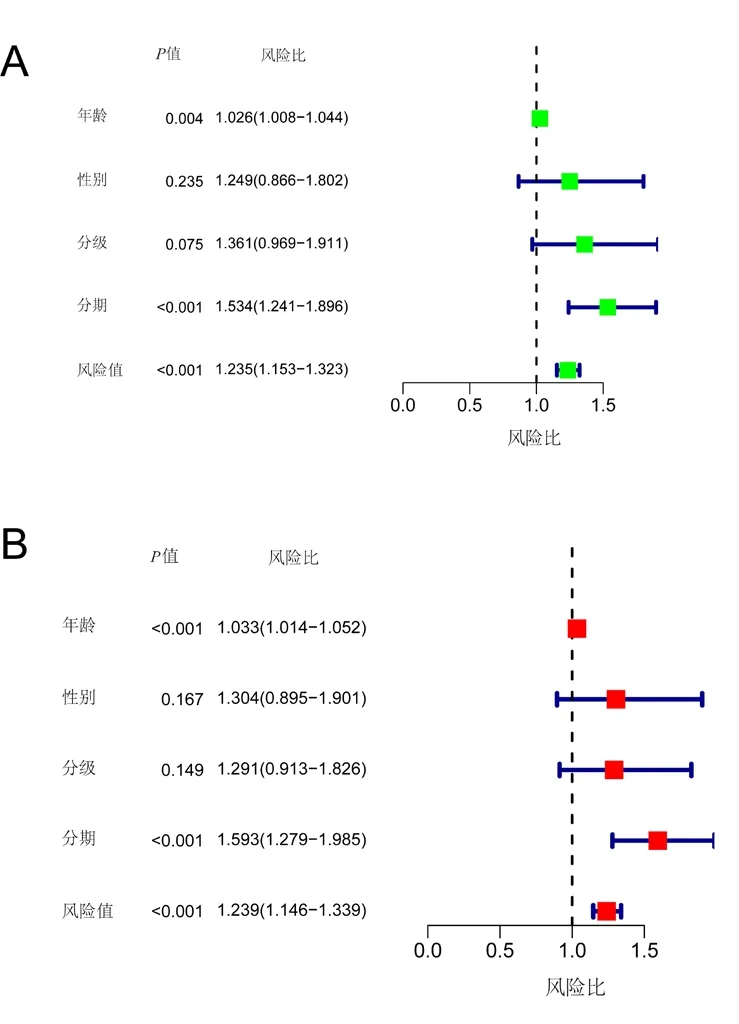
图3 预后模型独立于临床因素指导预后
2.6 GEO外部数据验证 为了进一步确认模型的预测能力,下载GEO数据库中胃癌数据集GSE84437,与临床资料合并后使用生存曲线的方式验证。生存曲线表明:高风险的患者(中位生存时间:3.5年)预后远远差于低风险的患者(中位生存时间:10.0年)(P<0.05)(图4)。提示在不同的环境下,该模型对胃癌的预后具有较好的预测能力。
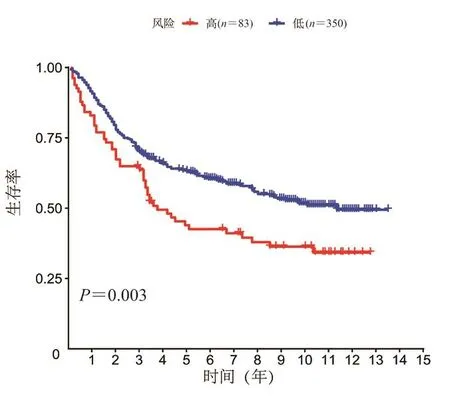
图4 GEO数据库验证预后模型:高风险组和低风险组的生存分析
2.7 临床运用 为了进一步将这6个VEGF通路相关基因在临床运用,绘制了列线图(图5A)。列线图显示,每个基因对应一个分数,最后得分是每个基因分数累加所得。根据每位患者的这6个基因的表达水平,可以通过查询列线图得到患者1、3年和5年生存率。同时为了验证列线图的预测准确性,绘制了校正图。校正图显示3年的预测与真实情况较为接近,说明VEGF通路相关基因预后模型在胃癌患者预后预测中具有较好的准确性(图5B)。
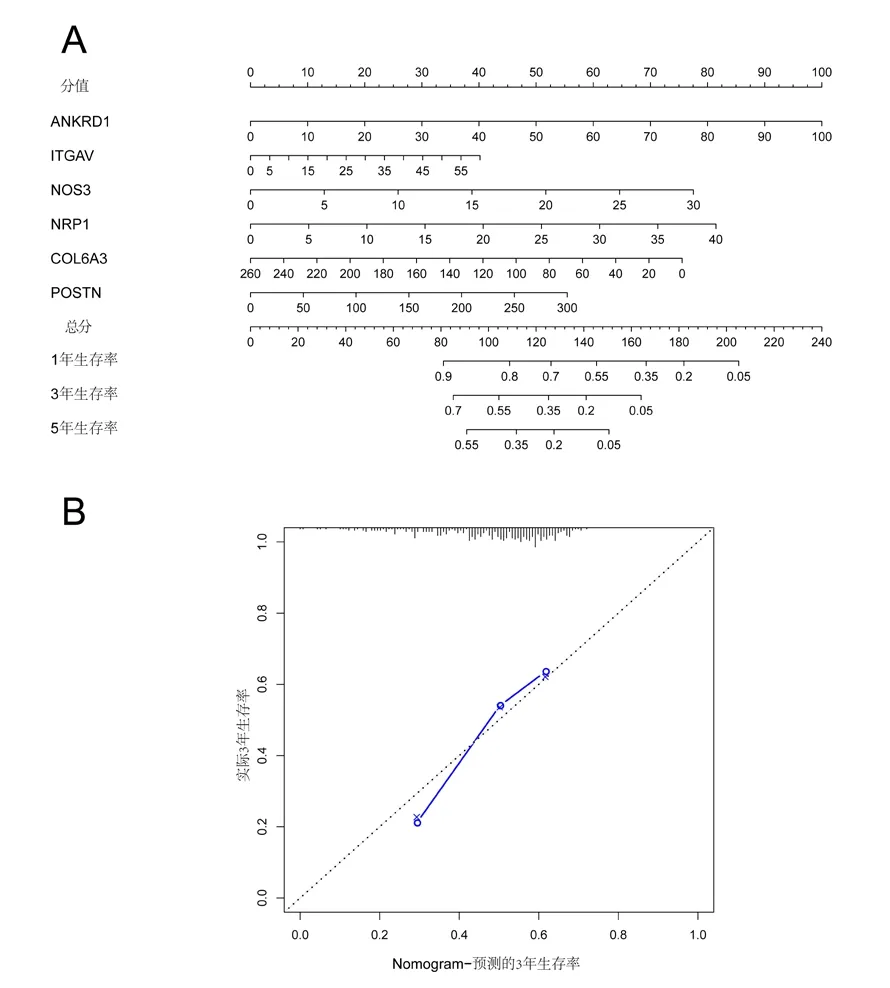
图5 列线图的构建和验证
3 讨论
胃癌是第五大最常见的恶性肿瘤,是全球癌症死亡的第三大主要原因[11]。胃癌患者表现出“三高三低”的特征:发病率、转移率和病死率高;早期诊断率、根治性切除率和5年生存率低[12-14]。早期胃癌患者接受根治性手术后再进行化疗,术后5年生存率为90%;晚期胃癌进行连续化疗治疗,但预后很差。多数患者诊断时已发展为晚期胃癌,因此对胃癌预后预测具有较大的临床运用价值。
肿瘤血管生成和淋巴管生成通过促进向肿瘤细胞输送氧气、营养物质和生长因子,在实体肿瘤生长、侵袭和转移扩散中起着至关重要的作用[6]。内皮抑素是内源性肿瘤血管生成抑制剂。它通过限制肿瘤的血液供应来抑制肿瘤血管生成和转移,从而剥夺肿瘤营养,被认为是治疗恶性肿瘤的潜在抗癌标志物[15]。VEGF参与肿瘤血管生成,研究表明靶向VEGF治疗具有抗肿瘤生长的作用[16]。在进展期胃癌中,分子靶向治疗已经成为联合治疗方案之一。贝伐单抗为重组人克隆抗体,通过抑制VEGF生物学活性对肿瘤血管生成起到抑制作用并可在一定程度上阻断肿瘤血供,诱导肿瘤细胞凋亡[16]。
本研究通过生物信息学确定了6个VEGF通路的胃癌预后基因。ANKRD1在卵巢癌中的高表达与生存不良有关,而在卵巢癌细胞系中与铂耐药相关[17]。ITGAV的过表达与经由神经周围浸润的结直肠癌的更高进展和扩散有关[18]。NOS3是钙依赖型酶,可调节血管功能,并且在激活的同时会产生一氧化氮[19]。NRP1是一种跨膜糖蛋白,可作为许多细胞外配体(包括Ⅲ/Ⅳ类信号蛋白,VEGF的某些同工型和转化生长因子β)的共受体[20]。NRP1对于介导VEGF对肿瘤干细胞的作用也至关重要,这主要是因为它们可以影响生长因子受体和整联蛋白功能。一些研究表明,靶向NRP1或NRP2可以抑制肿瘤发生并降低肿瘤对其他疗法的抵抗[21]。COL6A3是CRC的独立预后因子,主要在癌症相关成纤维细胞中表达[22]。而且独立于肿瘤淋巴结转移分期,COL6A3突变与总体生存期显著相关[23]。POSTN基因编码整联蛋白的配体,整联蛋白是促成细胞外基质与整联蛋白之间结构连接形成的关键粘着斑蛋白之一。POSTN基因高表达与许多人类恶性肿瘤相关[24]。总之,本研究模型中的VEGF通路相关基因都与肿瘤发生发展密切相关。结合本研究多因素COX风险回归分析建立的预后模型及生存分析和外部数据验证结果,提示本研究所构建的预后模型基本符合临床研究和基础研究,具有一定的临床预测能力。
本研究首次通过GSEA富集验证胃癌的发生发展与VEGF通路相关基因激活相关,并且建立了胃癌VEGF通路相关基因预后模型。使用生存分析和外部数据验证,进一步证实了该模型预测的准确性与稳定性。同时本研究通过绘制列线图的方式,将模型基因纳入列线图,使得该模型的临床运用更加简洁。挖掘的6个VEGF相关基因为胃癌的靶向治疗提供了新的启发和思路,并可能为胃癌的个体化治疗提供新视角。本研究的不足之处在于只是从生信角度出发,尚需临床试验及基础实验进一步验证。
