基因魔剪:改写生命密码新工具
2021-09-10王俏琦章元兵张子恒周爽刘冀珑
王俏琦 章元兵 张子恒 周爽 刘冀珑
2020年10月7日,2020年度诺贝尔化学奖授予德国马克斯·普朗克病原学研究室的生物化学家沙彭蒂耶(E. Charpentier)和美国加州大学伯克利分校的生物化学家杜德纳(J. A. Doudna),以表彰两人“开发了一种基因组编辑方法”。她们是第6位和第7位获得诺贝尔化学奖的女性。
沙彭蒂耶1968年出生于法国,1995年在巴黎巴斯德研究所获得博士学位,过去20年在5个不同国家9所不同大学工作过,已获得10项久负盛名的科学奖项,目前是德国马克斯·普朗克病原学研究所所长。杜德纳1964年出生于美国,1989年从哈佛医学院毕业获得博士学位,曾在2016年获得世界杰出女科学家成就奖,目前是加州大学伯克利分校教授、霍华德·休斯医学研究所研究员。
当细菌和病毒入侵人体,人体的免疫系统就会反击。同样,细菌在漫长的进化过程中,也形成了一套防御病毒入侵的“免疫系统”——CRISPR/Cas系统,CRISPR是成簇的规律间隔短回文重复序列(clustered regularly interspaced short palindromic repeats)的缩写,Cas是CRISPR相关(CRISPRassociated)基因的缩写。病毒侵入细菌后把自身的基因整合到细菌基因组中,利用细菌细胞复制自己的基因,而细菌的CRISPR/Cas系统可以识别病毒的基因,并将其从自己的基因组上切除。
CRISPR/Cas基因编辑技术就是由一段相当于GPS可精准定位基因组目的序列的向导RNA,将“上帝的手术刀”核酸内切酶Cas9指引到特定目的序列处,由核酸内切酶Cas9将基因组DNA切出缺口,然后利用机体自身的DNA双链断裂修复机制,产生插入(或)删除的突变来实现基因敲除,或者利用外源模板序列进行同源重组修复。
沙彭蒂耶发现了一种之前未知的分子——tracrRNA,而它则是细菌古老免疫系统CRISPR/Cas的一部分,它可通過裂解病毒DNA来对付病毒的入侵。沙彭蒂耶与杜德纳合作,又成功地再造了细菌的基因剪刀,简化了剪刀的分子组成,并对基因剪刀重新编程,使其可以被控制,可以在一个预定位置切断DNA分子。
利用CRISPR/Cas9技术可极其精确地改造动物、植物和微生物的DNA,它对生命科学产生了革命性的影响。比如,构建动物疾病模型,为癌症治疗提供新思路,使治愈遗传性疾病的梦想成为现实,甚至提供了创造新物种的可能性。
CRISPR研究的历程
一般认为CRISPR系统是在1987年被首次发现的。日本纳卡塔(A. Nakata)研究组在分析大肠杆菌基因组中与磷酸盐代谢相关基因的过程中,偶然发现了位于碱性磷酸酶同工酶(iap)基因的3’端有一段长度为29个碱基对的短序列会重复出现,当时他们对这个特征序列本身并没给予过多关注,甚至论文里也仅仅是提到这一序列,但并未将其命名[1]。
CRISPR系统被真正作为研究对象是在1993年。莫伊察(F. Mojica)在西班牙阿里坎特大学攻读博士学位期间发现:地中海富盐菌(Haloferax mediterranei),一种在西班牙圣波拉港口被发现的极度耐盐的古菌,其限制性内切酶会受到培养基中盐浓度的影响,而以不同方式切割自身的基因组。在研究这种现象时,莫伊察发现了一段与微生物中已知序列都不同的长约30个碱基的重复的近似回文的序列,这个固定序列之间由大约36个碱基间隔开。之后,莫伊察又陆续发现,在与地中海富盐菌亲缘关系近的沃氏富盐菌(Haloferax volcanii),以及与其亲缘关系较远的嗜盐古菌中,也有类似的保守序列。很快,莫伊察意识到这段序列与1987年纳卡塔研究组发表的文献中的细菌特征序列之间的联系,他认为在不同种微生物中,都存在这样一段序列,并且它们发挥着重要的作用,最终将其命名为短规律性间隔重复序列(short regularly spaced repeats)。到2000年,莫伊察已在20种微生物中发现这段重复序列。之后两年内,研究者们迅速将对这个基因座的研究拓宽到与这段序列有关的特定基因。2002年,詹森(R. Jansen)实验室发表论文,首次将这段序列命名为CRISPR,将与其相关的特定基因命名为Cas基因。
但是,这段特别序列的功能在很长一段时间里一直没有弄清楚。2003年莫伊察作为CRISPR领域的新领导者,开始将注意力放在将此重复序列隔开的间隔子(spacer)上。由于间隔子序列具有种内保守的特点,莫伊察将不同的间隔子序列进行大量比对,最终发现有一种间隔子的序列与感染大肠杆菌的P1噬菌体基因组某段序列相吻合。此后,他又通过一系列比对发现,CRISPR中的间隔子序列来自外源噬菌体或者质粒。值得注意的是,博洛廷(A. Bolotin)首次提出CRISPR基因座会通过反义RNA来抑制噬菌体基因的表达,虽然之后这个假说被证实是错误的,但它率先赋予了CRISPR在免疫学方面的意义。
CRISPR在细菌免疫系统中的作用是由霍瓦特(P. Horvath)揭示的。在克服困扰工业生产的噬菌体感染时,霍瓦特致力于开发基于DNA的菌株鉴定技术,并使用CRISPR对菌株进行基因分型。2004年,他同样注意到间隔子与细菌对噬菌体抗性之间的联系。2005年,霍瓦特及其同事试图证实CRISPR是一种细菌的适应性免疫系统。2007年,霍瓦特描述了一种嗜热链球菌(Streptococcus thermophilus)在被噬菌体入侵后,链球菌基因组内会整合进来自噬菌体基因组的间隔子序列,当该链球菌再次被同样的噬菌体入侵时,链球菌就可产生抗性,而如果改变这段间隔子序列,则会影响该链球菌的抗性。同时还发现,链球菌获得这种抗性需要Cas7蛋白的参与(但维持抗性不需要Cas7蛋白),而Cas9蛋白——其序列包含两种类型核酸酶结构域(HNH和RuvC)——及其产物,可能会切割核酸,它对抵抗噬菌体是必需的,所以,Cas9蛋白被认为是细菌免疫系统的活性成分。
科学家们很快开始进一步研究CRISPR/Cas的作用机理,第一个关键研究来自范德欧斯特(J. van der Oost)团队。他们通过将一种大肠杆菌的CRISPR系统插入另一种缺乏内源性CRISPR系统的大肠杆菌菌株中,从生化角度验证了该系统是由5种Cas蛋白组成的复合物,并将其命名为级联复合物(cascade)。然后通过依次敲除级联复合物每个组分,证明级联复合物对于切割从CRISPR基因座转录而来的前体CRISPR RNA(crRNA)是必要的,最终切割产物是具有61个碱基的crRNA,它由上一个重复序列最后8个碱基、完整间隔子序列和下一个重复序列的起始部分组成,可将Cas蛋白引导至目的DNA上。随后,范德欧斯特通过分别构建反义方向(与mRNA和编码链DNA的序列互补)和顺义方向(仅与另一条DNA互补)两种CRISPR序列,使得其可以靶向到λ噬菌体的必需基因上。他们的研究结果暗示了CRISPR不是作用于RNA,而是作用于DNA。
同年,玛拉菲尼(L. Marraffini)和松特海默尔(E. Sontheimer)证实了CRISPR的作用靶标是DNA。两年后,即2010年,穆瓦诺(S. Moineau)等人发现,CRISPR/Cas9在目的DNA的原间隔子邻近基序(protospacer adjacent motif, PAM)上游3个碱基对处进行切割,产生双链断裂缺口(double-strand break, DSB)。
而与此同时,沙彭蒂耶正着力于研究病原体化脓性链球菌是如何实现基因调控的。2011年,沙彭蒂耶等对链球菌内大量小RNA进行定位研究,发现其碱基序列非常接近已揭示的CRISPR基因座,一系列研究证实,一种未知的RNA分子对于CRISPR系统的功能实现具有重要作用,遂将其命名为反式激活的CRISPR RNA,简称tracrRNA(trans-activating crRNA),tracrRNA通过与crRNA形成双链体,将Cas9引导至靶标。
由于之前从未接触过CRISPR系统,沙彭蒂耶在一次參加在波多黎各召开的会议上,遇到来自美国加州大学伯克利分校的杜德纳博士,沙彭蒂耶邀请杜德纳进行合作研究,杜德纳欣然接受。杜德纳的同事们通过数字会议为合作制定了计划,此前他们猜测细菌中的Cas9最终执行切割DNA的作用,但在体外并没有得到验证。
2012年,她们将重组表达的Cas9与体外转录的crRNA和tracrRNA联合使用,成功构建出可体外切割纯化DNA的CRISPR/Cas系统:通过成熟的crRNA与tracrRNA形成双链RNA结构,引导Cas9蛋白靶向结合目的DNA片段,通过切割引起DNA双链断裂,从而使得CRISPR/Cas系统具有编辑基因的可能[2]。
在沙彭蒂耶和杜德纳的工作启发下,美国哈佛大学丘奇(G. Church)研究组与张锋研究组率先在哺乳动物中实现了基因编辑[3,4]。其后,在短短几年内这项技术风靡世界各地的实验室,成为生物学界最流行的基因编辑工具。
2013年,世界各地先后已经有多家实验室使用 CRISPR/Cas9,在包括人细胞系和多种模式生物中,比如斑马鱼、酵母、线虫、果蝇、小鼠、大鼠等,实现了对目的基因的编辑[5]。2015年CRISPR技术被美国《科学》周刊评为年度最佳科学突破。
2016年,张锋课题组率先将一种称为C2c2(也称Cas13a)的酶,用于CRISPR系统中,成功地对RNA进行了编辑。同年10月份,又有科学家首次利用CRISPR/Cas9编辑过的细胞开展人体临床试验,他们从患有转移性非小细胞肺癌患者血液中分离出的免疫细胞,通过CRISPR/Cas9技术特异性地敲除其中的PD-1基因,然后经体外扩增培养,再回输入患者体内,以期达到治愈癌症的目标。
2017年,美国迈阿密大学的课题组设计了CRISPR-RNA-靶向(CRISPR-RNA-targeting, CRISPR-RT)系统,对RNA编辑做出了很大贡献。此外,除了Cas13a,Cas13b和Cas13d的发现也推动了CRISPR系统的应用与发展。同年,刘如谦团队将tRNA腺嘌呤脱氨酶的蛋白定向进化后,与CRISPR/ Cas9系统融合,在不引起DNA链断裂的情况下实现了A-T到G-C的转换,且其在人体细胞中的编辑效率超过50%。为治疗多种单碱基突变遗传疾病提供了有效工具[6]。
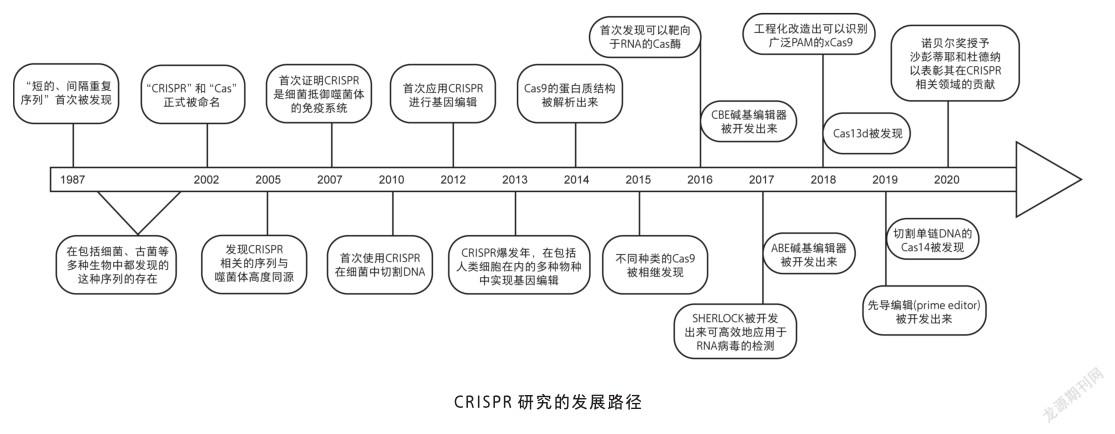
基于CRISPR/Cas9系统的基因编辑新工具层出不穷,2018年,刘如谦团队又进化出一种能够识别多种PAM序列的Cas9变体,提高了CRISPR/Cas9的剪辑效率。同年,杜德纳发布了CRISPR/Cas14系统。2019年,杜德纳成功地对Cas9蛋白的活性进行了改造,使之仅在特定组织中具有活性。张锋也开发出CRISPR/Cas12b系统,极大丰富了用于基因组编辑的CRISPR/Cas系统。
2020年初,新型冠状病毒肺炎爆发,科学家基于CRISPR/Cas13a技术开发了用于检测和定量SARSCoV-2病毒RNA的试剂盒,它无需对RNA进行反转录,而是直接对病毒RNA进行检测。正如沙彭蒂耶所说:“CRISPR/Cas9系统已经突破了界限,使基因工程技术更加地通用、有效和易操作。它的应用似乎真的没有什么限制[7]。”
CRISPR的工作原理
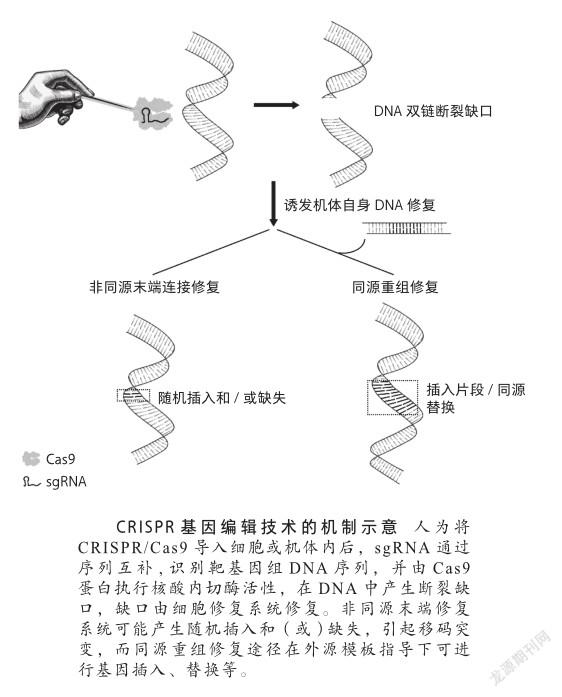
在噬菌体或质粒等外源DNA入侵宿主后,外来核酸被加工成短片段,并被作为新的间隔子整合到宿主染色体内的CRISPR重复间隔子序列中,这个过程相当于宿主的一次“免疫记忆”。在外源核酸上邻近间隔子序列下游有一段极其保守的基序(PAM),它对于间隔子序列的选择加工非常重要。当入侵者再次入侵时,CRISPR序列的转录产物被核酸内切酶加工为成熟crRNA。crRNA的5’端包含被整合进宿主基因组的间隔子序列,它可与外源序列互补配对,而3’端包含一段CRISPR的重复序列,与tracrRNA结合形成双链结构。带有Cas内切酶的crRNA-tracrRNA组装成核糖核蛋白复合体,通过间隔子序列与入侵核酸互补,进行特异性的破坏,从而实现宿主(细菌)的“二次免疫应答”[8]。
根据CRISPR基因座的不同,CRISPR系统可分为6种类型(Ⅰ-Ⅵ),它们有着不同的crRNA模式和特定的Cas蛋白[9]。与利用复杂的多Cas蛋白复合物进行crRNA结合和靶序列降解的Ⅰ型和Ⅲ型系统不同,Ⅱ型CRISPR系统采用单一核酸内切酶Cas9来识别双链DNA的靶位点,核酸内切酶Cas9的两个结构域(HNH和RuvC)分别切割两条DNA链。这两个不同结构域在PAM上游3个碱基对处剪接双链DNA,HNH结构域负责切割与crRNA互补的DNA链,RuvC结构域负责切割另一条DNA链。其间,tracrRNA与crRNA中的重复序列碱基互补配对,形成独特的双链RNA结构。这个双链RNA分子中约20个碱基靶向与其序列互补的DNA序列,并由Cas9进行DNA位点特异性切割,产生平末端的双链断裂缺口。然后,通过机体自身的DNA修复机制进行修复。一种是极易出错的非同源末端连接修复(nonhomologous end joining, NHEJ),在切割位点产生短的随机插入和(或)缺失;一种是通过高保真的同源重组修复(homology directed repair, HDR),在同源修复模板指导下,对DSB位点进行精确的修复。后来,通过人为地将crRNA和tracrRNA进行组合,形成单条RNA进行转录表达,得到嵌合的单链向导RNA(singleguide RNA, sgRNA),就极大地简化了CRISPR基因编辑方法。
CRISPR的优势
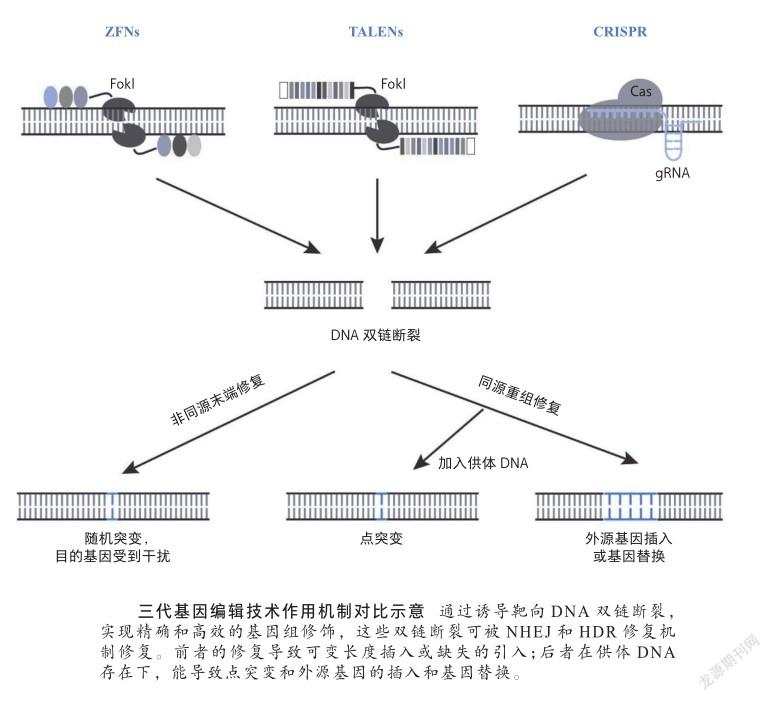
基因编辑技术从本质上来讲,是对DNA双链断裂损伤和修复机制的应用。外在因素如辐射和内在因素如细胞代谢产物,都会使DNA产生不同程度的损伤,DNA双链断裂是真核细胞中常见的一种DNA损伤类型,DNA双链断裂后,机体会通过两种修复机制进行及时修复:非同源末端修复和同源重组修复[10]。截至目前,基因编辑技术已经过三代更迭,从锌指核酸酶(zinc finger nucleases, ZFNs)技术,转录激活子样效应子核酸酶(transcription activator like effector nucleases, TALENs)技术,再到CRISPR/Cas技术[11]。
ZFNs是融合蛋白,由锌指蛋白和细菌的FokⅠ限制酶组成。锌指蛋白包括串联的锌指结构域,它们属于DNA结合结构域。每个锌指结构域识别3个碱基,串联的锌指结构域使得ZFNs可潜在地结合到长的核苷酸序列上。使用时需成对设计,一个切割位点在侧翼的正义链,另一个切割位点在侧翼的反义链。当切割位点两侧的ZFNs结合后,FokⅠ限制酶在该位点发生二聚化并进行切割,造成具5’突出端的双链断裂。ZFNs技术使得DSB不必依赖自然发生,从而为基因工程发展奠定了基础。
TALENs的构造与ZFNs类似,由串联的转录激活因子样效应子(TALE)的DNA结合结构域和FokI限制酶组成。與ZFNs不同的是,每个TALE蛋白识别单个特异性核苷酸,因此其结构更复杂,但设计更简单。然而,ZFNs和TALENs技术操作难度大、构建组装时间长,容易在细胞中积累,产生细胞毒性,动物实验已证明它们会引发机体的免疫反应。
CRISPR/Cas系统由向导RNA和Cas核酸酶组成,向导RNA结合到目的基因上,引导Cas蛋白切割目的序列产生双链断裂,根据不同实验目的选择不同Cas蛋白。通过修饰向导RNA的序列,就可以切割所选的任何靶序列。类似于ZFNs和TALENs系统,CRISPR/ Cas系统可用于通过非同源末端连接修复方式,在DNA切割位点处引入随机突变,也可通过共同注射与DNA同源的工程化DNA载体,通过同源重组修复方式引入特定突变或插入。CRISPR/Cas系统使基因定点修饰变得更加高效。
与ZFNs和TALENs相比,CRISPR/Cas系统有很多优势:①设计简单。CRISPR-Cas系统不像ZFNs和TALENs是依赖于蛋白与靶DNA之间的识别,而是由向导RNA和靶DNA之间形成核糖核苷酸复合物,利用核糖核苷酸识别靶向基因。②效率高,成本低。如创建突变小鼠时,仅需要将编码Cas蛋白和向导RNA的RNA直接注射到小鼠胚胎中,相较转染和选择小鼠胚胎干细胞注射小鼠、再进行同源重组的方法,可节省几个月甚至几年时间。③可同时进行多基因突变操作。Cas9蛋白单体可与任意数量不同序列的特异性向导RNA组合,轻松实现用CRISPR/Cas9文库进行多重基因组编辑。
CRISPR的应用与展望
在短短不到10年时间里,CRISPR得到了前所未有的高速发展,基于CRISPR的各类技术已在科研、医疗、农业等诸多方面得到广泛应用。研究人员可以精确编辑引发疾病的突变基因,以使从根源上治愈此前束手无策的疾病。比如,通过特殊的病毒将CRISPR系统递送到特定细胞中,针对性地修复致病的突变基因,目前这已在镰刀型贫血症、杜氏肌萎缩症等疾病中成功验证,并寻求临床实验[12]。在农业基因育种上,CRISPR技术可以对农作物基因组进行更便捷而又精确的修饰,以获得更好的遗传性状,比如增强农作物抗性、改善农产品品质、提高产量。
CRISPR尽管其在基因编辑上展示出了巨大的潜力,但也存在不可忽视的风险。自CRISPR技术问世以来,关于其造成脱靶(即可能切割非目的序列的风险)的讨论就从未停止过,尽管也在不断地进行优化,降低脱靶率,但至今尚未从根本上解决问题,错误地修饰其他基因可能会造成一系列无法想象的严重后果,这是CRISPR应用所面临的一大障碍。
基因编辑导致的伦理问题,除了在医疗方面有待进一步讨论和解决外,还有对于基因改造的伦理问题,比如用于强化人的某些生理机能,甚至造出一个“定制人”,都是需要我们认真思考和对待的。现今在人类胚胎上的应用应当受到严格管控,在关注其带来的增益效果时,其带来的风险一样应是每个人必须深思熟虑。
管控风险、避免违反伦理道德,并将其合理应用到生活中,是人类共同的责任。我们要在风险和伦理问题上时刻保持警惕和谨慎,也期待CRISPR在将来为人类带来更多的意想不到的惊喜。
[1]Ishino Y, Shinagawa H, Makino K, et al. Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J Bacteriol, 1987, 169(12): 5429-5433.
[2]Jinek M, Chylinski K, Fonfara I, et al. A programmable dual-RNA–guided DNA endonuclease in adaptive bacterial immunity. Science, 2012, 337(6096): 816-821.
[3]Mali P, Yang L, Esvelt K M, Et al. RNA-guided human genome engineering via Cas9. Science, 2013, 339(6121): 823-826.
[4]Cong L, Ran F A, Cox D, et al. Multiplex genome engineering using CRISPR/Cas systems. Science, 2013, 339(6121): 819-823.
[5]Hsu P D, Lander E S, Zhang F. Development and applications of CRISPR-Cas9 for genome engineering. Cell, 2014, 157(6): 1262-1278.
[6]Lander Eric S. The heroes of CRISPR. Cell, 2016, 164(1): 18-28.
[7]Doudna J, Charpentier E. The new frontier of genome engineering with CRISPR-Cas9. Science (New York, NY), 2014, 346, 1258096.
[8]Barrangou R, Marraffini L A. CRISPR-Cas systems: prokaryotes upgrade to adaptive immunity. Mol Cell, 2014, 54(2): 234-44.
[9]Makarova K S, Wolf Y I, Alkhnbashi O S, et al. An updated evolutionary classification of CRISPR-Cas systems. Nat Rev Microbiol, 2015, 13(11): 722-736.
[10]Ensminger M, L?brich M. One end to rule them all: nonhomologous end-joining and homologous recombination at DNA double-strand breaks. Br J Radiol, 2020, 93(1115): 20191054.
[11]Gaj T, Gersbach C A, Barbas C F. ZFN, TALEN, and CRISPR/ Cas-based methods for genome engineering. Trends Biotechnol, 2013, 31(7): 397-405.
[12]Xu Y Y, Li Z J. CRISPR-Cas systems: overview, innovations and applications in human disease research and gene therapy. Comput Struct Biotechnol J, 2020, 18: 2401-2415.
關键词:诺贝尔化学奖 基因编辑 CRISPR 修复 ■
