基于级联3D U-Net的CT和MR视交叉自动分割方法
2021-08-31沈镇炯彭昭孟祥银汪志徐榭裴曦
沈镇炯,彭昭,孟祥银,汪志,2,徐榭,3,裴曦,4
1.中国科学技术大学核医学物理研究所,安徽合肥230025;2.安徽医科大学第一附属医院肿瘤放疗科,安徽合肥230022;3.中国科学技术大学附属第一医院放疗科,安徽合肥230001;4.安徽慧软科技有限公司,安徽合肥230088
前言
放疗要求确保肿瘤靶区接受足够的处方剂量照射的同时,避免靶区周边危及器官正常组织受到过多的损伤[1-2]。因此,精准可靠的危及器官分割是制定放疗计划的一个必备条件。传统的手工分割非常耗时,而且准确性受医生个体经验所限[3-4]。因此,近年来基于多图谱和深度学习的自动分割方法逐渐受到了更多的关注,其中基于深度学习的方法在图像分割领域取得了更好的成果[5-7]。在医学图像分割领域,绝大多数的深度学习分割算法都基于卷积神经网络(CNN)[8]和U-Net[9]。Zhou 等[10]为解决3D 图像内存制约计算性能的问题,提出先以一个点提取感兴趣区域(ROI)进行分割后,再进行更精确的ROI提取,反复迭代至收敛。Han等[11]则使用粗分割结果提取肝脏的ROI。Isensee 等[12]提出级联U-Net 用于心脏、肝脏等器官的分割。Sun 等[13]将级联模型应用于眼睛及周边器官的分割,提出先定位后分割的方法可以提高小体积危及器官的分割性能。但是,他们都没有将级联模型应用于视交叉的分割。在垂体腺瘤和视神经胶质瘤的放射治疗中,视交叉的定位对于评估视交叉受压情况和预后的视觉恢复非常重要[14-15]。Bekes 等[16]提出的依据CT 图像的半自动分割算法在眼球和晶体上表现很好,但是分割视交叉的效果不佳,准确度只有眼球的65%。MICCAI2015头颈自动分割挑战赛中视交叉分割的最优DSC 结果仅为0.57[17]。对于视交叉这类软组织危及器官,磁共振图像(MRI)比CT 具备更好的图像对比度,因此针对病人个体同时采集CT 和MRI,有可能进一步提高分割的精准度[18]。因此,采用多模态图像逐渐广泛应用于医学图像分割领域[18-20]。Noble等[18]基于多图谱和模型的方法分割CT 和配准的MRI中的视交叉,但是需要手动调整参数。基于此,本文拟通过改进的级联3D U-Net网络研究利用多模态数据提高自动分割视交叉的精度。
1 资料与方法
1.1 实验数据及预处理
实验使用的数据来自2019年8月至2020年8月于安徽医科大学第一附属医院已接受放疗的60例头颈癌患者,同一患者的CT 及MRI采集的时间间隔均不超过3 个月,其中MRI包含T1、T2两种成像模态。患者均采取热塑面膜固定,CT 图像的分辨率从(2.500×0.488×0.488)mm3至(5.000×0.976×0.976)mm3不等,MRI的分辨率从(4.000×0.687×0.687)mm3至(7.000×0.532×0.532)mm3不等。主管医生在Eclipse计划系统(Varian, USA)中仅依据CT 图像手工分割患者视交叉,并交由上级资深医生审核修改。此外,该主管医生使用MIM Maestro 软件(Ver 6.8.7)将MRI形变配准到CT 图像上,然后结合配准后的MRI在CT上重新手工分割视交叉,如图1所示,并交由上级资深医生审核修改。将CT 图像及配准后的MRI重新采样到统一分辨率(1.250×0.800×0.800)mm3,预裁剪为256×256×80 大小的3D 图像。这些预处理可减少计算量,减少由于成像扫描参数不同引起的差异,加速级联3D U-Net网络的收敛。
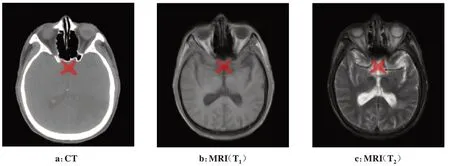
图1 弹性配准后的CT与MRI(T1、T2)图像及视交叉手工分割结果Fig.1 CT and MRI(T1,T2)images after elastic registration and manual segmentation of the optic chiasm
1.2 自动勾画模型
本文提出一种级联3D U-Net模型,具体如图2所示。该级联3D U-Net 由两部分组成,第一部分为基础的3D U-Net 网络模型,包括一个捕获上下文信息的收缩路径和一个支持精确本地化的对称扩展路径,每一个路径均包含5 个卷积块,每个卷积块包含两个3×3 卷积层和一个Leaky ReLU 激活函数,下采样过程使用一个步长为2 的3×3 卷积层和一个Leaky ReLU 激活函数来代替原始的最大池化层,来提升网络性能[21]。第二部分为改进的3D D-S U-Net,在基础的3D U-Net 模型上添加深度监督机制。Lee 等[22]提出的深度监督网络(DSN),在基础的CNN 的隐藏层中引入了分类器(SVM 或Softmax)来提高分类网络的性能。Zeng 等[23]将深度监督机制应用于3D UNet,提出深度监督3D U-net-like 网络,并用于3D 磁共振图像中股骨头的分割,提高网络训练效率。Zeng 等[23]将深度监督3D U-net-like 网络在扩展路径中引入了低、中、高3 个不同级别的深度监督,每一层深度监督中均包含一个解卷积块和分支分类器。本网络在此基础上改进了3D D-S U-Net,在低级别(24×24×4)深度监督中,经过卷积和上采样操作,得到与中级别(48×48×8)隐藏层相同分辨率的图像并与其叠加,再一次经过卷积和上采样操作,得到与高级别(96×96×16)隐藏层相同分辨率的图像并与其叠加,通过分类器(Softmax)输出分割结果。

图2 级联3D U-Net模型结构Fig.2 Cascaded 3D U-Net model structure
1.3 训练方法
模型以256×256×80 大小的预处理3D 图像作为原始输入,实现视交叉ROI的提取,并重新自动裁剪为96×96×16大小的3D 图像,作为第二部分网络的输入,最终输出结果至大小为512×512×Sz 的原始临床图像中,其中Sz为原始临床图像的层数。
模型训练过程中,将CT 图像像素值约束为-160~240 HU,T1图像像素值约束为200~800,T2图像像素值约束为200~1 700。模型采用Pytorch 框架,以DSC Loss 作为损失函数,训练迭代次数采用早停法(Early Stopping),当损失函数连续30 个迭代期不减少时停止训练。模型采用Adam 优化器,学习率为0.000 2,Batch 的大小为1。在单个NVIDIA GTX 1070TI(8 GB)上,训练消耗显存约为7 GB,训练耗时约20 h。
1.4 评价标准
本研究以医生手工分割的视交叉轮廓为“金标准”,使用形状相似性系数(Dice Similarity Coefficient,DSC)评价网络自动分割的视交叉轮廓的准确性,该值为0~1,数值越大表示自动分割与手工分割越相似,表明预测精度越高,DSC 计算公式如下所示,其中A为医生手工分割的视交叉区域,B为自动分割的视交叉区域,公式如下所示:

2 结果
2.1 自动分割精度
级联3D U-Net的一阶段分割结果得到视交叉的三维中心,据此获得像素为96×96×16 大小的外扩轮廓,如图3a~图3c 所示。依据外扩轮廓,对原始图像进行定位裁剪,减少背景类的信息[24]。将自动定位裁剪后的图像输入二阶段的分割网络后,得到自动分割的最终结果,如图3d~图3f所示。以随机选取的15 例患者作为测试集,一阶段分割的五折交叉验证DSC 系数及标准差为0.564±0.159,最终分割的五折交叉验证结果为0.645±0.085,经过定位裁剪后,DSC系数得到了约0.08 的提升,说明一定程度上减少背景类的占比可以提升网络模型的自动分割精度。本实验还尝试将定位裁剪像素大小调整为64×64×16,发现结果并没有提升,这可能是因为ROI太小,虽然背景类占比更少,但同时也减少了有用的图像特征信息。
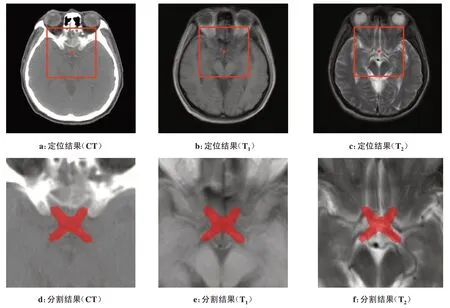
图3 级联3D U-Net的定位和分割结果Fig.3 Positioning and segmentation of cascaded 3D U-Net
图4中展示了测试集中某一例病例的视交叉分割情况,图4a 和图4b 分别是正反两个视角下的分割对比情况,其中手工分割结果用蓝色表示,自动分割结果用粉色表示,二者交叉重合的部分用红色表示。通过观察图4中手工分割和自动分割的情况,我们发现自动分割结果能够较为准确地包含完整的视交叉区域,对于视交叉核心区域分割非常准确,只有在视交叉延伸至视神经及视束的边界区域稍有误差。

图4 自动分割与手工分割结果对比Fig.4 Comparison between automatic segmentation and manual segmentation
由于目前尚无文献同时利用CT 和配准的MRI数据用于视交叉的自动分割研究,我们对比了本研究中的级联3D U-Net 方法与其他采用CT 数据分割方法的DSC 值。Ren 等[25]采用交错式的3D-CNN,其视交叉的平均DSC 结果为0.580±0.170,田娟秀等[26]采用基于3D 深度残差FCN 的SV-Net,其视交叉的平均DSC 结果为0.583±0.286,这两个方法均采用了MICCAI2015 头颈自动分割挑战赛的数据[17],共包含48例CT,其中15例患者作为测试。本研究提出的级联3D U-Net 自动分割模型,其视交叉的平均DSC结果为0.645±0.085,通过对比发现,本研究提出的模型在视交叉自动分割领域取得了更好的结果。
同时,本研究进行不同模态数据的实验对比,其中采用多模态数据(包含CT、MRI的T1和T2模态)的自动分割结果为0.645±0.085,采用单模态数据(仅CT 图像)的结果为0.552±0.096。通过对比发现,在视交叉的自动分割中,采用多模态数据相比单模态CT数据能得到更好的分割结果。
本实验采用多种数据增强方法来扩充数据,将图像进行添加高斯噪声、随机翻转、弹性形变(Elastic deformation)[27],最终分割DSC 结果得到0.03 的提升。
2.2 自动分割效率
测试集中15 例患者的视交叉自动分割耗时约42 s,平均每例患者的视交叉自动分割仅需2.8 s,可以大大提升临床手工分割的效率。
3 讨论及总结
本研究不需要借助其他器官的分割算法用于辅助定位,直接针对视交叉进行二级分割,所需手工分割标签更少,临床应用更加简单。相比CT图像,MRI具备更高的空间分辨率和软组织对比度,临床上MRI已经被用于检查视交叉及相关病变情况,利用MRI的信息进行自动分割视交叉逐渐成为一种有效趋势,Sun等[15]在研究中进行基于MRI的视交叉分割实验,取得较好的结果,但是他们借助了垂体瘤患者的垂体位置信息进行辅助,而且最终分割未应用于CT。在基于深度学习的视交叉自动分割的相关工作中,本研究首次结合CT 和MRI进行自动分割,发现相比仅采用CT 图像进行自动分割,采用多模态数据的策略更好,得到更高的分割精度。
在本研究中发现,常规头颈放疗中使用的MRI层厚通常为6~7 mm,视交叉的可见层数少,边界难以界定,是目前结合MRI进行视交叉自动分割的一个难点,未来结合深度学习技术,针对层厚较大的MRI进行重采样,重建成层厚较小,信息更丰富的图像,是一种可考虑的数据增强方式。
综上所述,本研究提出一种新的基于级联3D U-Net 的多模态视交叉自动分割方法,首次结合CT 和MRI应用于视交叉的自动分割,比通常仅采用CT 图像进行自动分割的方法,在平均DSC 评估上高出约0.093,取得更好的精度,该方法可以作为临床中医生手工分割视交叉的辅助手段。未来将本方法拓展至其他视神经、晶体及海马等脑部小体积危及器官的通用性研究中,并集成至中国科学技术大学核医学物理研究所团队研发的一款危及器官自动勾画软件——DeepViewer 系统中[28],从而提高放疗计划制定的工作效率。
