甘薯NAC转录因子家族的全基因组鉴定与分析
2021-08-26黄小芳毕楚韵王和寿陈其俊胡韵卓陈选阳林世强
黄小芳 毕楚韵 王和寿 陈其俊 胡韵卓 陈选阳 林世强


摘 要:利用生物信息學方法对甘薯(Ipomoea batatas)基因组中NAC转录因子进行鉴定、保守结构域分析、motif查找、染色体定位、系统进化树分析以及逆境胁迫下基因表达分析。结果表明:甘薯全基因组序列中含有91个NAC转录因子基因,非均匀分布于甘薯15条染色体上;系统进化树及motif分析结果显示,91个甘薯NAC家族成员可分为16个亚组,共包含20个motif,其中大部分家族成员中均含有motif 2、motif 4、motif 1、motif 8和motif 3,这5个motif分别对应NAC结构域中的5个子域A、B、C、D和E。在与拟南芥(Arabidopsis thaliana)NAC转录因子共同构建的进化树中,有64个甘薯NAC家族成员被归入拟南芥NAC基因家族的14个亚组,其中,NAC2亚组包含的成员数最多,有9个,TIP和AtNAC3亚组均仅有1个。转录组数据分析结果显示,在蔓割病菌(Fusarium oxysporum f. sp. batatas, Fob)胁迫下甘薯NAC基因家族中有10个基因的表达量发生变化;而在低温环境下有25个NAC基因差异表达。本研究结合利用基因组与转录组数据分析,该结果为进一步研究甘薯NAC基因家族的功能提供参考。
关键词:甘薯;NAC;转录因子;生物信息学
中图分类号:S531 文献标识码:A
Genome-wide Identification and Analysis of NAC Transcription Factor Family of Ipomoea batatas
HUANG Xiaofang1,2, BI Chuyun1,2, WANG Heshou3, CHEN Qijun4, HU Yunzhuo1, CHEN Xuan-yang1,2,5*, LIN Shiqiang1,6*
1. Key Laboratory of Crop Biotechnology, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 2. College of Agriculture, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 3. Ningde Agricultural and Rural Bureau, Ningde, Fujian 352100, China; 4. Seed Centre of Fujian, Fuzhou, Fujian 350002, China; 5. Key Lab of Genetics, Breeding and Multiple Application of Crops, Ministry of Education, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China; 6. College of Life Science, Fujian Agriculture and Forestry University, Fuzhou, Fujian 350002, China
Abstract: The bioinformatic methods were used to conduct the identification, conservative domain analysis, motif search, chromosomal localization, phylogenetic analysis and gene expression analysis under stress with regard to the NAC transcription factors of the Ipomoea batatas genome. The results showed that there existed 91 genes of NAC transcription factors within the I. batatas genome, which were distributed unevenly in the 15 chromosomes. Phylogenetic and motif analysis indicated that the 91 members of NAC transcription factors could be classified into 16 subgroups and there were 20 motifs in total. Most members of the NAC family contained motif 2, motif 4, motif 1, motif 8 and motif 3, which corresponded to the subdomains A, B, C, D and E of the NAC domain. Within the phylogenetic tree constructed with the transcription factors of I. batatas and Arabidopsis thaliana, there were 64 I. batatas NAC transcription factors categorized into the 14 subgroups of A. thaliana NAC transcription factors, of which the NAC2 subgroup was the largest in number and the TIP subgroup and AtNAC3 subgroup both had only one member. Analysis of the transcriptomics data demonstrated that 10 genes of the I. batatas NAC transcription factors were differentially expressed under Fusarium oxysporum f. sp. batatas stress; whereas under the low temperature condition, there were 25 NAC genes differentially expressed. This study combined the analysis of genomic and transcriptomics data of I. batatas and the results provided reference for the functional inquiry into the I. batatas NAC transcription factors.
Keywords: Ipomoea batatas; NAC; transcription factor; bioinformatics
DOI: 10.3969/j.issn.1000-2561.2021.07.004
近几年来,对植物转录因子的研究越来越广泛。转录因子根据其DNA结合结构域的不同可分为WRKY、MYB、bHLH、bZIP和NAC等多个转录因子家族[1]。NAC转录因子是植物中特有的一类转录因子超家族,最早在矮牵牛和拟南芥中发现,Aida等[2]研究发现矮牵牛NAM基因和拟南芥ATAF1、ATAF2以及CUC2的3个基因所编码的蛋白序列N端均有一段高度保守的氨基酸序列,因此将这一段高度保守的区域命名为NAC结构域。典型的NAC转录因子蛋白序列的N端包含一个大约由150个氨基酸组成的NAC结构域,NAC结构域由A、B、C、D和E共5个亚结构域组成,其中,A、C和D亚结构域高度保守,B和E保守性较弱;序列C端为转录激活区,在氨基酸组成和功能方面均呈现高度多样化[3]。
目前,越来越多的研究表明NAC转录因子在植物生长发育、激素调节以及生物与非生物胁迫中发挥着重要作用。Ooka等[4]以水稻和拟南芥为研究对象,将这2种植物的NAC转录因子分为Ⅰ组和Ⅱ组,2个大组又进一步分为18个亚类,其中I组中含有TERN、ONAC022、SENU5、NAP、AtNAC3、ATAF、OsNAC3、NAC2、ANAC011、TIP、OsNAC8、OsNAC7、NAC1和NAM等14个亚类,Ⅱ组中含有ANAC001、ONAC003、ONAC001和ANAC063等4个亚类。分类后的水稻和拟南芥NAC转录因子根据结构域相似性对其功能进行分析,拥有motif x和motif xi的NAM及NAC1亚类与植物形态发生有关;含有motif v的ATAF亚类则在植物响应压力刺激的反应中起着重要作用。
甘薯(Ipomoea batatas)中关于NAC转录因子的研究还比较少,张欢等[5]利用cDNA末端快速扩增技术从甘薯品种栗子香中鉴定得到一个NAC转录因子IbNAC72,在烟草中过表达IbNAC72基因发现转基因烟草植株的抗旱性显著提高。甘薯属于六倍体植物,基因组高度多态且非常难以组装,Yang等[6]通过使用新型单倍型基因组装配方法,以甘薯品种‘泰中6号为研究对象进行测序,获得了甘薯栽培种的全基因组测序结果。本研究基于甘薯‘泰中6号测序得到的全基因组序列,应用隐马尔可夫方法,从中挖掘出甘薯NAC转录因子家族序列,分析NAC转录因子的结构域保守性、基因之间的进化关系以及结构特征等,为研究NAC转录因子在甘薯生长发育和环境胁迫等过程中的调控作用提供参考。
1 材料与方法
1.1 材料
从NCBI数据库(https://www.ncbi.nlm.nih. gov/genome/?term= Ipomoea+batatas)下载甘薯‘泰中6号[6]15条染色体的基因组序列。利用snap程序分别对甘薯15条染色体的基因组序列进行CDS区检索,检索所选用的HMM模型为拟南芥(At.hmm)、线虫(Ce.hmm)和水稻(Os.hmm),最终得到178 458个甘薯全基因组CDS序列,翻译后得到蛋白组数据[7]。
1.2 方法
1.2.1 甘薯NAC转录因子挖掘与筛选 从Pfam数据库网站(http://pfam.xfam.org/)下载NAC结构域的隐马尔可夫模型NAC.hmm(PF01849)和NAM.hmm(PF02365)。下載的2种HMM模型利用hmmsearch程序分别检索甘薯全基因组蛋白序列,获得甘薯NAC基因家族候选蛋白,从中选取特异的序列构建甘薯特异的NAC结构域HMM模型,再次对甘薯全基因组蛋白序列进行检索,将2次检索得到的候选蛋白合并去除重复序列及氨基酸序列长度小于150的序列(已知NAC结构域大约由150个氨基酸组成[8]),剩余序列提交到NCBI保守结构域数据库(conserved domain database,CDD)进行NAC结构域验证。经CDD验证后的候选蛋白通过Interproscan(https://www.ebi.ac.uk/interpro/search/sequence/)、Augustus[9]以及结合转录组数据[10]利用IGV软件进行比对确认,最终得到甘薯完整的NAC转录因子蛋白序列。
1.2.2 甘薯NAC转录因子保守结构域分析 研究表明,NAC结构域的5个亚结构域中以A、C和D亚结构域较为保守[11]。使用Clustal Omega程序[12]对筛选到的甘薯NAC转录因子蛋白序列进行多重序列比对,截取较保守区域并对其氨基酸组成特征进行分析。
1.2.3 甘薯NAC转录因子分类及motif分析 应用MEME程序[13]预测甘薯NAC蛋白序列的保守基序,查找的保守基序最大数目为20,其他参数设为默认值。甘薯NAC蛋白序列经ClustalO比对后,利用MEGA X[14]构建甘薯NAC转录因子系统进化树,采用邻接法(Neighbor- joining),p-distance模型,空位选项为pairwise deletion,重复次数设为1000。
1.2.4 NAC转录因子染色体定位及重复基因筛选 从甘薯基因的ZFF文件中,提取NAC基因在染色体上的位置信息,通过circos程序[15]将其标注在染色体对应的位置上。进一步利用Blastn计算甘薯NAC家族中各基因之间的序列相似度,根据2个基因之间的相似度是否大于75%及比对区域覆盖度是否超过较长序列的75%,筛选出具有潜在复制关系的基因对,在圈图中用线条进行连接[16]。
1.2.5 甘薯及拟南芥NAC转录因子系统进化树构建 从拟南芥数据库网站(https://www. rabidopsis.org/)下载得到99条拟南芥NAC转录因子蛋白序列。将甘薯和拟南芥的NAC蛋白序列合并,经多重序列比对后,通过MEGA X软件[14]中的Neighbor-joining方法构建系统进化树(Bootstrap=1000)。构建成功的甘薯及拟南芥NAC转录因子系统进化树,参考拟南芥NAC基因家族已知的分类信息[4],对甘薯NAC基因家族成员进行整理和归类。
1.2.6 甘薯NAC转录因子基因表达分析 从EBI(https://www.ebi.ac.uk/)数据库下载得到甘薯蔓割病菌(Fusarium oxysporum f. sp. batatas, Fob)胁迫和低温胁迫下的转录组测序数据。利用Hisat2[17]将下载的转录组数据与甘薯NAC转录因子DNA序列进行比对,手动统计每个NAC转录因子DNA序列所比对上的read数目,并通过DESeq2[18]对其进行标准化。筛选出其中log2FoldChange大于1或小于?1的值,p adjust<0.05,利用pheatmap(https://cran.r-project. rg/web/packages/pheatmap/index.html)构建甘薯差异基因的表达模式聚类图。
2 结果与分析
2.1 甘薯基因组中NAC转录因子鉴定
利用NAM.hmm和NAC.hmm检索甘薯全基因组蛋白序列,分别得到161个和8个甘薯NAC家族成员。从NAM.hmm模型检索得到的161个家族成员中选取47个(Evalue<1e-30)作为高特异的序列构建甘薯特异的NAM.hmm模型(Ib-nam.hmm),再次检索甘薯全基因组蛋白序列,得到197条NAC蛋白序列,新增36条;以NAC.hmm检索得到的8个家族成员为基础构建的甘薯特异的NAC.hmm模型(Ib-nac.hmm),再次检索后得到10条NAC蛋白序列,新增2条,合并全部结果后得到207条甘薯NAC转录因子候选蛋白。因此对筛选到的207条甘薯NAC蛋白序列进行排序,去除60条长度小于150个氨基酸的序列,余下的147条去除其中不含有NAC结构域或结构域缺失严重的序列后,最终得到91条含有完整NAC结构域的甘薯NAC转录因子序列。
2.2 甘薯NAC保守结构域分析
利用ClustalO软件将甘薯91条NAC蛋白进行多重序列比对,参考拟南芥和水稻中NAC转录因子蛋白序列的比对结果,用不同的颜色显示甘薯NAC蛋白中NAC结构域的氨基酸组成特征,同时截取A、B、C、D和E亚结构域的序列范围(图1),其中,C亚结构域最长,A、D亚结构域次之,B和E亚结构域相对较短(包括gap区域);A、C和D亚结构域中含有较明显的空位(gap)区域,主要是因为比对过程中个别序列长度过长或其中的NAC结构域过短所造成的,例如A亚结构域中,有2个明显的gap区域,而在这2个区域中分别只有CM008342.1-snap.9597和CM008344. 1-snap.11179 2个转录因子的氨基酸序列。
对甘薯NAC转录因子NAC结构域保守性进行分析,在甘薯NAC蛋白的NAC结构域中发现68个保守性大于50%的位点,其中A亚结构域中有17个,B亚结构域中有6个,C亚结构域中有21个,D和E亚结构域中分别有19个和5个。A、C和D亚结构域保守位点明显多于B、E亚结构域。
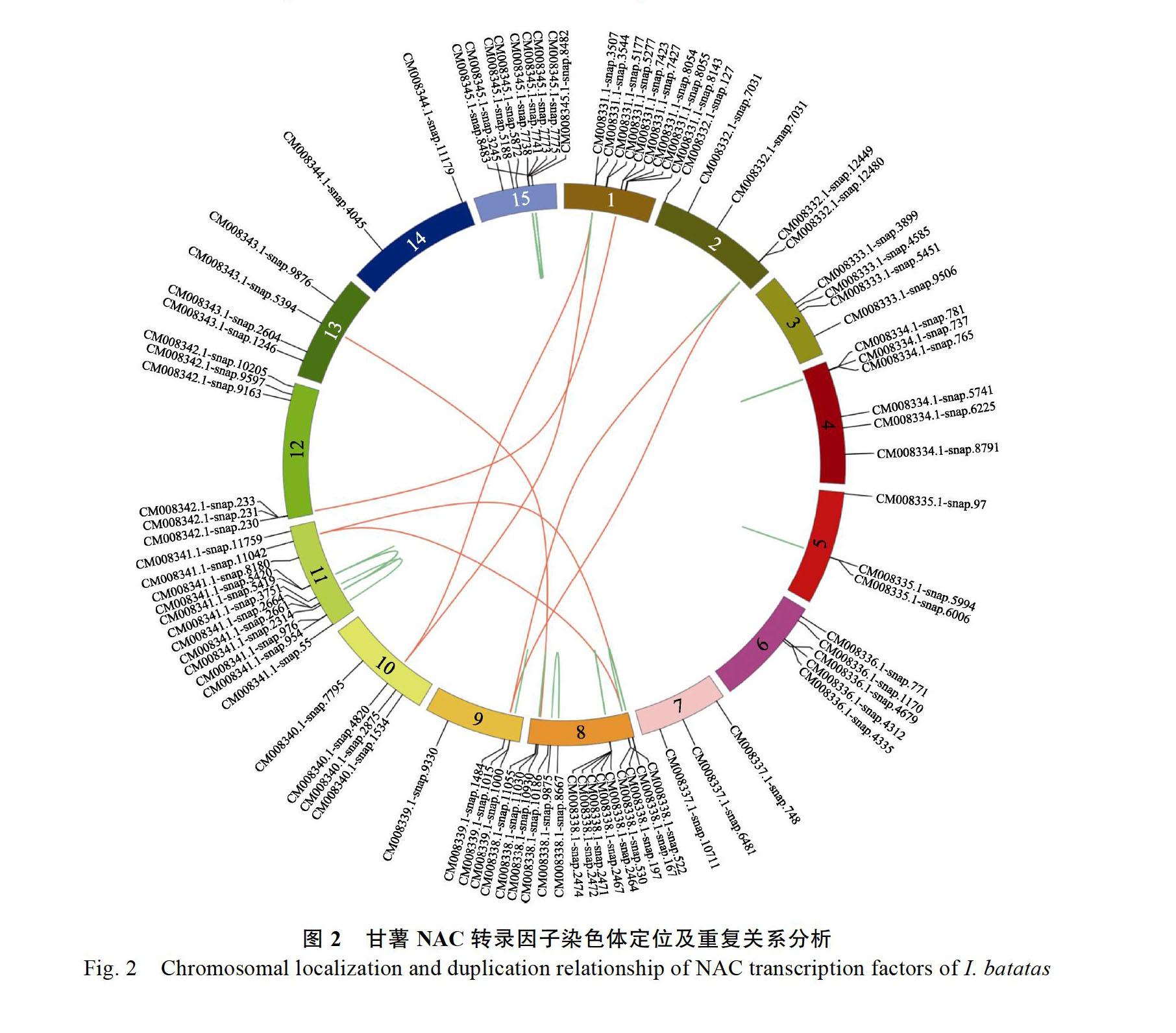
2.3 甘薯NAC基因家族在染色体上的分布
利用circos分析甘薯NAC基因在染色体上的分布(图2),91个NAC转录因子在甘薯15条染色体上均有分布,但并不均匀。8号染色体上分布数量最多,为15个,1、11、15号染色体上分别含有9、12、9个;而在3、5、7、9、10、13以及14号染色体上所分布的NAC家族成员數则明显偏少,仅为2~4个。为了研究甘薯NAC各家族成员之间的联系,通过比对获得各个基因序列之间的序列相似性,从中找到了46对含有潜在复制关系的基因并用线条在图中连接。其中属于染色体间的基因对有8对(红色连线),分别为CM008339.1-snap.1484和CM008332.1-snap. 2449、CM008339.1-snap.1484和CM008332.1-snap. 2480、CM008340.1-snap.4820和CM008331.1-snap. 507、CM008340.1-snap.4820和CM008331.1-snap. 544、CM008341.1-snap.11042和CM008338.1-snap. 97、CM008341.1-snap.11042和CM008338.1-snap. 30、CM008342.1-snap.230和CM008331.1-snap. 423、CM008343.1-snap.5394和CM008338.1-snap. 0930。染色体内的基因对有38对(图2中绿色连线),且部分染色体内的基因对在染色体上的排列位置相近,形成基因簇,例如第15号染色体上的CM008345.1-snap.7738、CM008345.1-snap.7741、
CM008345.1-snap.7773、CM008345.1-snap.7775、CM008345.1-snap.8482和CM008345.1-snap.8483每2个基因之间均存在潜在重复关系,形成15个基因对,从图2可看出,这6个基因在染色体上的分布位置相近。
2.4 甘薯NAC基因家族分类及基序分析
对甘薯91个NAC转录因子构建系统进化树,根据序列之间Bootstrap值的大小分为16个亚组,以I~ⅩⅥ命名。利用MEME软件对甘薯NAC蛋白进行保守基序分析,得到20个motif信息,将甘薯NAC转录因子motif的组成结果根据基因名字对应到进化树的相应位置上。从图3中可发现,甘薯NAC转录因子motif的组成存在一定的规律,例如motif 1、motif 2、motif 3、motif 4和motif 8存在于50%以上的甘薯NAC家族成员中,含有motif 2、motif 3和motif 8的家族成员更是高达90%。另外,这5个高频率出现的基序在甘薯NAC转录因子蛋白序列中呈motif 2-motif 4- motif 1-motif 8-motif 3的排布顺序。
进一步对这5个保守基序的氨基酸组成进行分析(表1),发现motif 2、motif 4、motif 1、motif 8、motif 3分别对应NAC结构域中的A、B、C、D、E这5个亚结构域,与图1中甘薯NAC蛋白序列的比对结果相一致。结合进化树的分组结果,发现同一亚组中的成员具有极为相似的motif组成特征,个别亚组成员拥有其他亚组成员所不含有的motif类型,如VII组中的7个NAC转录因子均含有其他亚组所没有的motif 9;motif 17和motif 19分别仅存在于XIV亚组以及XVI亚组中;XV亚组和XVI亚组motif组成高度相似,且均含有其余亚组所没有的motif 6,在进化树分支上的位置也极为相近,推测这2个亚组中的NAC蛋白具有相似的功能。
2.5 甘薯NAC转录因子系统进化树分析
本研究将甘薯91条NAC转录因子蛋白序列和拟南芥99条NAC转录因子蛋白序列合并后共
同构建系统进化树,根据拟南芥NAC基因家族中
已知的家族成员分类信息对甘薯NAC转录因子进行归类。结果表明(图4),甘薯NAC转录因子中有64个家族成员被归入拟南芥NAC基因家族的14个亚组中,NAC2亚组中包含的甘薯NAC家族成员数最多,有9个;而TIP和AtNAC3亚组中均只有1个甘薯NAC家族成员。II组的4个亚组中,除了ANAC001亚组并未在图中显示,其余的亚组ANAC063和ANAC001中不含有甘薯NAC家族成员,仅ONAC003亚组中含有7个甘薯NAC家族成员。
2.6 甘薯NAC转录因子在生物胁迫和非生物胁迫条件下的差异表达
利用转录组学分析方法获得了甘薯NAC转录因子在尖孢镰刀菌胁迫下表达量的变化(图5A)。
从图5中可发现,甘薯响应尖孢镰刀菌胁迫的NAC转录因子数目较少,91个NAC家族成员中仅10个基因的表达量明显变化,其中JS57_F07组中未发现NAC基因表达量的变化;JS57_F04和XZH_F07中分别有4个(CM008?331.1-snap.8055、CM008336.1-snap.1170、CM008337.1-snap.10711、CM008342.1-snap.9597)和6个(CM008331.1- snap.5177、CM008332.1-snap.3271、CM008332.1- snap.7031、CM008333.1-snap.3899、CM008338.1- snap.10930、CM008343.1-snap.5394)NAC转录因子的表达量发生上调或下调。
本研究利用Xushu15-1和Xushu15-4在低温胁迫下的转录组测序数据,绘制甘薯NAC转录因子在低温胁迫下的基因表达模式聚类图(图5B)。甘薯91个NAC家族成员中有25个成员响应低温胁迫,其中有11个家族成员在4个分组中表达量均有明显变化。从图5B中可发现,大部分响应低温胁迫的NAC转录因子表达量呈现下调趋势,如CM008333.1-snap.3899、CM008336.1-snap.1170、CM008338.1-snap.197及CM008343.1-snap.1246等均出现明显的表达量下调;其中CM00?8333.1- snap.3899在Xushu15_1和Xushu15_4中表达量虽均有下调,但相比于Xushu15_4 (log2Fo?ld?Change值2w和6w分别为?1.78和?1.52),CM008333.1- snap.3899在Xushu15_1中表达量下调更加显著,在Xushu15_1中log2FoldChange值2w和6w分别为?5.13和?4.38。另外,部分甘薯NAC转录因子在低温胁迫下表达量出现明显上调,如CM008331.1- snap.5177、CM008332.1-snap.7031、CM008335.1- snap.5994、CM008341.1-snap.976和CM008345.1- snap.5188,这些在低温环境下表达量显著提高的基因可能在植物响应低温胁迫时发挥着重要作用。
3 讨论
NAC转录因子是植物中特有的一类重要的转录因子,在调控植物生长发育、响应生物与非生物胁迫以及调节植物体内激素信号传导等方面发挥重要的作用。本研究利用生物信息学方法从甘薯全基因组蛋白序列中鉴定得到91个含有完整NAC结构域的NAC转录因子,并对其进行染色体定位、保守基序查找与分析、系统进化树构建以及逆境胁迫下基因表达量的变化等进行了研究,以期为研究甘薯NAC转录因子的功能提供数据参考。
NAC转录因子广泛存在于多种植物中。目前,对NAC转录因子的研究越来越多,除了在模式植物拟南芥(105)、烟草(152)以及水稻(151)[11]中鉴定出了一定数量的NAC转录因子,在玉米[19]、大豆[20]以及毛竹[21]的基因组数据中分别鉴定出了128、152和125个NAC转录因子。相比于其他植物中鉴定得到的NAC蛋白家族成员数量,甘薯基因组中NAC转录因子数量(91)偏少。进一步对甘薯的NAC转录因子构建系统进化树,将其分为16个亚组,对每个亚组氨基酸序列保守基序的组成进行分析,发现大多数甘薯NAC转录因子拥有相同的若干保守基序,但在不同的亚组之间保守基序的组成存在不同程度的变化。NAC2亚组中包含的9个甘薯NAC家族成员,推测它们可能与拟南芥NAC2亚组功能相似,与植物氧化应激反应相关[22];ONAC003亚组中含有7个甘薯NAC家族成员,推测其与拟南芥中ONAC003亚组的NAC有相同的功能,参与调节开花时间[23]。
在对拟南芥NAC基因家族功能的研究过程中,Jensen等[24]研究表明ANAC019蛋白的NAC结构域可通过识别并结合DNA的核心序列调控拟南芥体内与低温胁迫相关基因的表达。低温会导致植物体内生理代谢活动的紊乱,转录因子的转录调控是植物响应低温胁迫的重要部分[25]。Ji等[26]将具有不同低温贮藏能力的甘薯品种‘Xushu15-1(高)和‘Xushu15-4(低)的块根在4 ℃条件下贮藏0周、2周和6周后进行转录组测序,比较分析2个品种在低温胁迫下的基因差异表达。本研究将甘薯与拟南芥NAC转录因子序列合并共同构建系统进化树,发现拟南芥中参与调节低温胁迫的ANAC019基因所在的AtNAC3亚组中包含有甘薯CM008333.1-snap. 9506基因,该基因在对甘薯低温胁迫的转录组测序数据分析的结果中表达量下调;另外,在对甘薯NAC基因家族进行分类的过程中发现,CM008333.1-snap.9506基因所在的I组成员还包括CM008332.1-snap.7031、CM008338.1-snap. 10186、CM008335.1-snap.5994、CM008335.1-snap. 6006、CM008341.1-snap.976、CM008333.1-snap. 3899以及CM008344.1-snap.4045,7个基因与CM008333.1-snap.9506在进化树上位置相近,motif的分布模式相似,说明这7个NAC基因家族成员与CM008333.1-snap.9506基因可能具有類似的功能;比较低温胁迫的转录组测序结果,发现这7个基因在转录组测序的分析结果中均出现明显的上调/下调表达。
為研究甘薯在病菌胁迫下其基因组中基因表达量的变化,Lin等[9]将高感品种‘新种花(XZH)和高抗品种‘金山57(JS57)植株接种蔓割病菌生理小种F04和F07,取样测定了转录组。为了更全面地分析甘薯NAC转录因子在生物与非生物胁迫下的基因表达模式,本研究分别以蔓割病菌胁迫和低温胁迫下的2个转录组测序数据进行分析。转录组分析结果中有多个NAC转录因子表达量出现变化,响应低温胁迫的NAC基因数量(25)明显多于蔓割病菌胁迫下的数量(10)。这些表达量出现明显上调或下调变化的NAC转录因子,可为后续的功能研究包括基因克隆、基因过表达、干扰、敲除和编辑等提供数据参考。
参考文献
[1] 荣玉萍, 张文香, 邓 娇, 等. 苦荞NAC基因家族的生物信息学分析[J]. 湖南农业大学学报(自然科学版), 2019, 45(3): 273-280.
[2] Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation in Arabidopsis: an analysis of the cup-shaped Cotyledon mutant[J]. Plant Cell, 1997, 9(6): 841-857.
[3] Olsen A N, Emst H A, Leggio L L, et al. NAC transcription factors: structurally distinct, functionally diverse[J]. Trends in Plant Science, 2005, 10(2): 79-87.
[4] Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes in Oryza sativa and Arabidopsis thaliana[J]. DNA Research, 2003, 10(6): 239-247.
[5] 张 欢, 杨乃科, 商丽丽, 等. 甘薯抗旱相关基因IbNAC72的克隆与功能分析[J]. 作物学报, 2020, 46(11): 1649-1658.
[6] Yang J, Moeinzadeh M H, Kuhl H, et al. Haplo-type-resolved sweet potato genome traces back its hexaploidization history[J]. Nature Plants, 2017, 3(9): 696-703.
[7] 黄小芳, 毕楚韵, 石媛媛, 等. 甘薯基因组NBS-LRR类抗病家族基因挖掘与分析[J]. 作物学报, 2020, 46(8): 1195-1207.
[8] 张 丹, 马玉花. NAC转录因子在植物响应非生物胁迫中的作用[J]. 生物技术通报, 2019, 35(12): 144-151.
[9] Hoff K J, Stanke M. Predicting genes in single genomes with AUGUSTUS[J]. Current Protocols in Bioinformatics, 2018, 65(4): e57.
[10] Lin Y L, Zou W K, Lin S Q, et al. Transcriptome profiling and digital gene expression analysis of sweet potato for the identification of putative genes involved in the defense response against Fusarium oxysporum f. sp. batatas[J]. PLoS One, 2017, 12(11): e0187838.
[11] 王 芳, 孙立娇, 赵晓宇, 等. 植物NAC转录因子的研究进展[J]. 生物技术通报, 2019, 35(4): 88-93.
[12] Sievers F, Higgins D G. Clustal Omega for making accurate alignments of many protein sequences[J]. Protein Science, 2018, 27(1): 135-145.
[13] Bailey T L, Elkan C. Fitting a mixture model by expectation maximization to discover motifs in biopolymers[J]. Proceedings International Conference on Intelligent Systems for Molecular Biology, 1994, 2: 28-36.
[14] Kumar S, Stecher G, Li M, et al. MEGA X: molecular evolutionary genetics analysis across computing platforms[J]. Molecular Biology and Evolution, 2018, 35(6): 1547-1549.
[15] Krzywinski M, Schein J, Birol I, et al. Circos: an infor-mation aesthetic for comparative genomics[J]. Genome Research, 2009, 19(9): 1639-1645.
[16] Vatansever R, Koc I, Ozyigit I I, et al. Genome-wide identification and expression analysis of sulfate transporter (SULTR) genes in potato (Solanum tuberosum L.)[J]. Planta, 2016, 244(6): 1167-1183.
[17] Kim D, Langmead B, Salzberg S L. HISAT: a fast spliced aligner with low memory requirements[J]. Nature Methods, 2015, 12(4): 357-360.
[18] Love M I, Huber W, Anders S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2[J]. Genome Biology, 2014, 15(12): 550.
[19] 葛姍姗, 唐桂英, 毕玉平, 等. 玉米全基因组中NAC基因家族的鉴定与分析[J]. 山东农业科学, 2015, 47(2): 1-6.
[20] 王 洋, 柏 锡. 大豆NAC基因家族生物信息学分析[J]. 大豆科学, 2014, 33(3): 325-333.
[21] 黎帮勇, 胡尚连, 曹 颖, 等. 毛竹NAC转录因子家族生物信息学分析[J]. 基因组学与应用生物学, 2015, 34(8): 1769-1777.
[22] De Clercq I, Vermeirssen V, Van Aken O, et al. The membrane-bound NAC transcription factor ANAC013 functions in mitochondrial retrograde regulation of the oxidative stress response in Arabidopsis[J]. Plant Cell, 2013, 25(9): 3472-3490.
[23] Fujiwara S, Mitsuda N. ANAC075, a putative regulator of VASCULAR-RELATED NAC-DOMAIN7, is a repressor of flowering[J]. Plant Biotechnology, 2016, 33(4): 255-265.
[24] Jensen M K, Kjaersgaard T, Nielsen M M, et al. The Arabidopsis thaliana NAC transcription factor family: structure-function relationships and determinants of ANAC019 stress signalling[J]. Biochemical Journal, 2010, 426(2): 183-196.
[25] 肖玉洁, 李泽明, 易鹏飞, 等. 转录因子参与植物低温胁迫响应调控机理的研究进展[J]. 生物技术通报, 2018, 34(12): 1-9.
[26] Ji C Y, Kim H S, Lee C J, et al. Comparative transcriptome profiling of tuberous roots of two sweetpotato lines with contrasting low temperature tolerance during storage[J]. Gene, 2020, 727: 144244.
责任编辑:黄东杰
