应用Illumina Miseq测序技术分析传统酸马奶细菌多样性
2021-08-26宋凯
宋凯
(徐州生物工程职业技术学院药品食品学院,江苏 徐州 221006)
乳制品是全世界共认的高营养食品,千百年来成为人民喜爱的食品,同时也为不同的外源微生物提供了理想的繁殖生境,形成了复杂多样的微生物群落,促进了发酵乳制品的形成[1-2]。酸马奶是其中一种非常著名的发酵乳制品,其以马奶为原材料与发酵剂混合,在开放的环境中自然发酵2 d~3 d即可,且具有略带酸味、沁人心脾、清爽适口等特性[3]。此外,由于酸马奶含有丰富的微生物资源及营养,使其对结核病为主的传染性疾病、消化系统疾病、心血管系统疾病等均有明显的辅助治疗作用[4-5]。但是,酸马奶的这些治疗作用与其化学成分和菌群结构密切相关。酸马奶的品质不仅取决于原料的差异,也与其微生物组成有密切关系。菌群结构容易受到地理和气候条件变化的影响,同时也受温度和发酵时间变化的影响。这些因素的差异导致了不同地区酸马奶的化学成分、菌群结构和风味的差异[6]。因此,了解传统酸马奶的微生物多样性至关重要。
以往对酸马奶微生物多样性的研究多采用传统分离鉴定方法,发现酸马奶中的主要微生物是乳酸菌和酵母菌,其对风味、酸度和口感特别重要[5,7]。鉴于一些微生物具有不可培养的特性,传统分离鉴定方法会严重低估微生物多样性。近年来,16S rRNA高通量测序技术被广泛应用于传统酿造食品中复杂微生物群落的研究,高通量测序不仅具有高通量、高覆盖、高准确率等特点,还可以全面地揭示样本微生物群落的组成及多样性等信息[8-9]。为了解传统酸马奶中细菌群落多样性,本研究利用Illumina Miseq高通量测序技术解析传统酸马奶的细菌群落结构及组成,为工业化生产提供理论依据及筛选潜在价值的益生菌提供数据参考。
1 材料与方法
1.1 材料与试剂
酸马奶:取自新疆塔城和丰县5个不同农场生产的样品(命名为 C1、C2、C3、C4、C5)和周边 5 户牧民手工酿造的样品(命名为 M1、M2、M3、M4、M5),取样后立即装入采样管中封口,用液氮冷冻后带回实验室置于-80℃储存,以备后续试验。
FastDNA®Spin Kit for Soil试剂盒:美国 MP Biomedicals公司;Truseq®DNA PCR-Free Sample Preparation Kit试剂盒、Illumina Miseq PE250型测序仪:美国Illumina公司;Agencourt AMPure XP核酸纯化磁珠:美国Beckman Coulter公司;细菌16S rRNA V3-V4区 PCR 扩增引物(338F:5′-ACTCCTACGGGAGGCAGCA-3′和 806R:5′-GGACTACHVGGGTWTCTAAT-3′):北京诺禾致源公司。
1.2 仪器与设备
Illumina Miseq PE250型测序仪:美国Illumina公司;S1000型聚合酶链式反应(polymerase chain reaction,PCR) 仪、CFX96 型荧光定量 PCR 仪、Gel DocTMXR+型凝胶成像系统、低温冷冻离心机:美国Bio Rad公司;DYY-8C型水平电泳槽:北京六一仪器厂;VORTEX-3型旋涡混匀器:德国IKA公司;NaneDrop 2000型超微量分光光度计:美国Thermo公司。
1.3 试验方法
1.3.1 酸马奶DNA提取及PCR建库
利用FastDNA®Spin Kit for Soil试剂盒提取酸马奶样本中微生物的基因组DNA。使用NaneDrop 2000型超微量分光光度计测定其溶度和纯度。将合格的DNA用引物338F和806R进行PCR扩增,PCR扩增体系(20 μL):模板 DNA 10 ng,正向及反向引物各10 pmol,2×高保真 DNA聚合酶混合物10 μL。扩增条件为94℃预变性2 min;94℃变性20 s,55℃退火30 s,72℃延伸1 min,共进行25个循环;最后72℃延伸10 min[10]。扩增产物使用2%的琼脂凝胶进行电泳检测。加入等体积的Agencourt AMpure XP核酸纯化磁珠对产物进行纯化。利用Truseq®DNA PCR-Free建库试剂盒制备测序文库并添加接头序列,经Nane-Drop 2000确定文库质量,使用Illumina Miseq PE250测序平台进行高通量测序。
1.3.2 生物信息学分析
采用FLASH软件[11]对测序的原始数据进行拼接,利用QIIME软件[12]对低质量的拼接序列进行过滤,使用UCHIME软件[13]去除嵌合体,得到优质序列。根据UCLUST软件[14]在相似性97%的水平上对优质序列进行聚类,得到各可操作分类单元(operational taxonomic unit,OTU),以各OTU中丰度最高的序列作为代表序列。在80%置信水平下,通过Silva 132数据库[15]对OTU进行分类学注释,将蓝藻门和不能注释到门水平的OTU删除。基于OTU得到分析结果,采用随机抽平的方法,利用QIIME软件[12]对样品进行多样性分析。基于OTU的主坐标分析(principal co-ordinates analysis,PCoA)、样本聚类分析、箱线图绘制、热图绘制、火山图绘制均通过R软件实现。
2 结果与分析
2.1 样本测序数据处理结果
传统酸马奶样本测序数据处理结果统计见表1。

表1 传统酸马奶样本测序数据处理结果统计Table 1 Statistics of sequencing data processing results in traditional koumiss
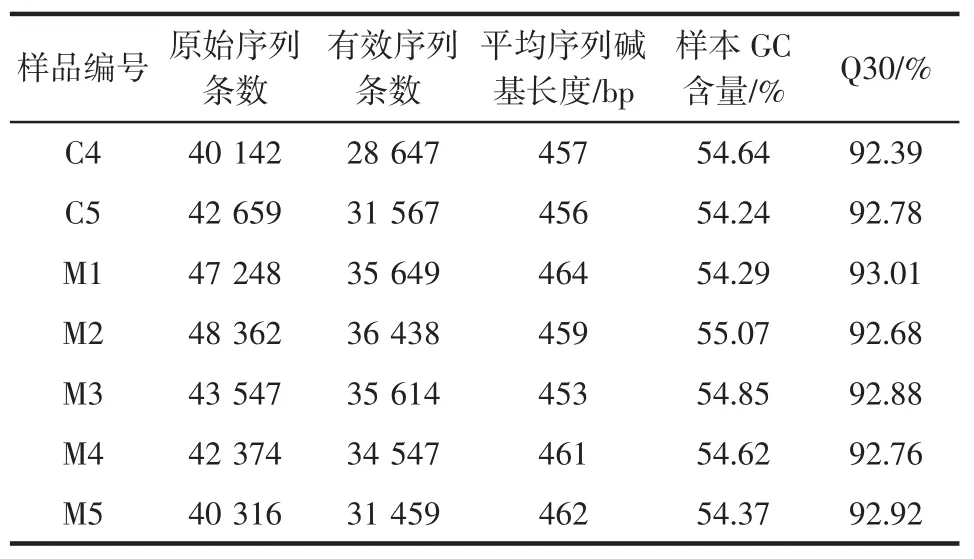
续表1 传统酸马奶样本测序数据处理结果统计Continue table 1 Statistics of sequencing data processing results in traditional koumiss
根据表1可知,10份酸马奶样品共检出429 155条原始序列,通过相关软件过滤后最终得到331 327条有效序列。去除条形码(barcode)和引物后,10份酸马奶样品的平均碱基长度均在450 bp以上,且GC含量为54.49%。此外,酸马奶样品的Q30值均高于90%,说明测序数据可靠且质量较好。
稀疏性曲线是判定样本的取样大小是否合理及确定测序深度是否可以覆盖整个微生物体系。酸马奶样品细菌群落的稀疏性曲线见图1。

图1 酸马奶样品细菌群落的稀疏性曲线Fig.1 Rarefaction curves of bacterial communities in traditional koumiss
由图1可知,随着测序深度的增加,被检测发现的OTU呈先迅速增加,随后平稳增加。当测序深度>25000时,酸马奶样品中能检测到新OTU数量越来越少。这些结果表明虽然通过增加测序深度可以识别一些新的微生物,但在分析中已捕获了样品的大多数微生物,进一步说明Illumina Miseq高通量测序技术可有助于全面了解酸马奶的细菌多样性。
2.2 细菌群落的多样性分析
在97%的相似度阈值下,对331 327条有效序列进行聚类分析,根据QIIME软件进行OTU划分,并根据得到OTU进行α-多样性分析。酸马奶样品细菌Alpha多样性结果统计见表2。

表2 酸马奶样品细菌Alpha多样性结果统计Table 2 Statistics of α-diversity results of bacteria in traditional koumiss
由表2可知,通过OTU聚类后可得到1 083个OTU,OTU平均为 175个/样本~265个/样本,其中农场生产样品的平均OTU数量为217个/样本,而牧民手工酿造样品的平均OTU数量为225个/样本,两者之间没有显著差异。Chao1指数是反映物种丰富度的指标,它通过观测到的结果推算出一个理论的丰富度,而这个丰富度更接近真实的丰富度,通常能观测到的物种丰富度肯定会比实际少。根据表2可知,10份酸马奶样品所检测到OTU数与Chao1指数的差异均不超过20,从而进一步说明当前测序深度是可以基本覆盖整个酸马奶样品的微生物体系。Shannon指数和Simpson指数是评价群落多样性和均匀度的常用指数。农场生产的5份酸马奶样品的Shannon指数和Simpson指数分别在1.4~1.89和0.49~0.55之间,其平均值分别为1.57和0.51;而牧民手工酿造的5份酸马奶样品的Shannon指数和Simpson指数分别在1.34~1.99和0.5~0.6之间,其平均值分别为1.78和0.57。牧民手工酿造的样品中Shannon指数和Simpson指数要显著高于农场生产的样品(P<0.05),表明酸马奶的细菌多样性会受到地理和发酵器具的影响[16]。
为了更好地了解不同地域生产酸马奶的微生物结构的变化,采用主坐标分析法和非加权配对算术平均(unweighted pair group method with arithmetic mean,UPGMA)聚类法进行微生物群落多样性研究。酸马奶样品细菌群落结构的主坐标分析和非加权配对算术平均聚类分析见图2。

图2 酸马奶样品细菌群落结构的主坐标分析和非加权配对算术平均聚类分析Fig.2 PCoA and UPGMA clustering analysis of bacterial communities in traditional koumiss
如图2A所示,PCoA的第一主坐标和第二主坐标分别占总变异的93.01%和4.39%。农场生产的样品可明显聚集在一起,位于PCoA图的左上方,而牧民手工酿造的样品也可聚集在一起,位于PCoA图的右侧,表明两者的细菌群落结构存在明显差异。此结果与UPGMA聚类分析结果(图2B)相一致,表明不同来源的酸马奶的微生物组成易受到当地环境因素的影响。
2.3 细菌群落的结构分析
在划分OTU后,挑选不同OTU中丰度最高的序列作为该OTU的代表序列,随后与微生物参考数据库进行97%相似性比对分析,从而对酸马奶样品测序得到的每个OTU进行分类学鉴定。在分类学门水平揭示酸马奶样品细菌群落的结构差异。酸马奶样品细菌群落结构在门水平的分布见图3。
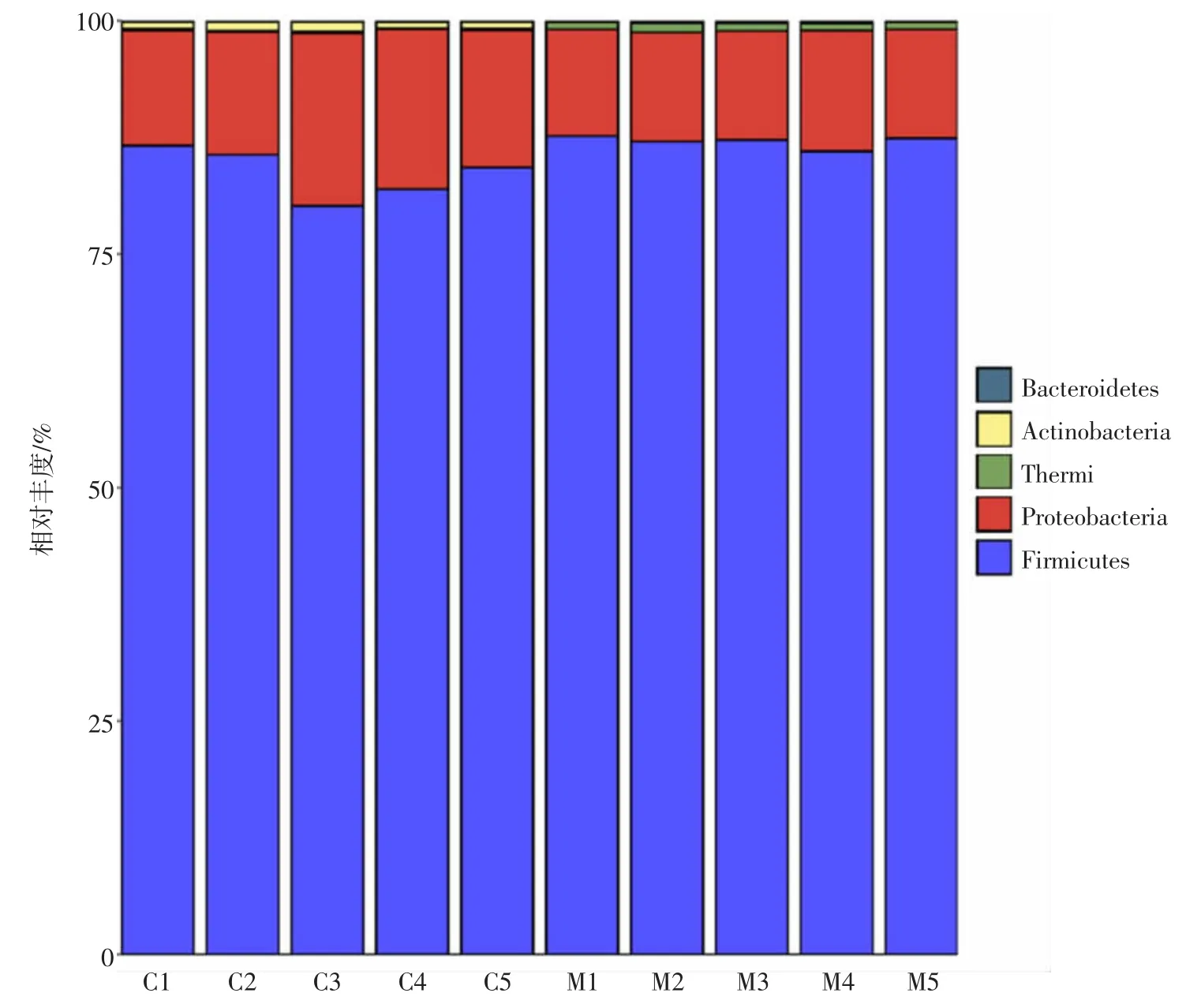
图3 酸马奶样品细菌群落结构在门水平的分布Fig.3 Distribution of bacterial communities at phylum level in traditional koumiss
如图3所示,酸马奶样品的细菌群落分别来自厚壁菌门(Firmicutes)、变形菌门(Proteobacteria)、放线菌门(Actinobacteria)、异常球菌—栖热菌门(Deinococcus-Thermus)和拟杆菌门(Bacteroidetes)。其中厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)占整个测序序列的99%左右。这与BAO等[17]的研究结构相一致,说明这些门水平的微生物决定酸马奶的成熟。此外,厚壁菌门在酸马奶中相对丰度可达80.19%~97.65%,说明这些来自厚壁菌门的微生物具有厌氧、耐酸和分解蛋白等特性。
为了进一步准确揭示酸马奶细菌群落的结构,故从分类学的属水平探究其组成,并揭晓不同来源样品的差异。酸马奶样品细菌群落结构在属水平的分布见图4。
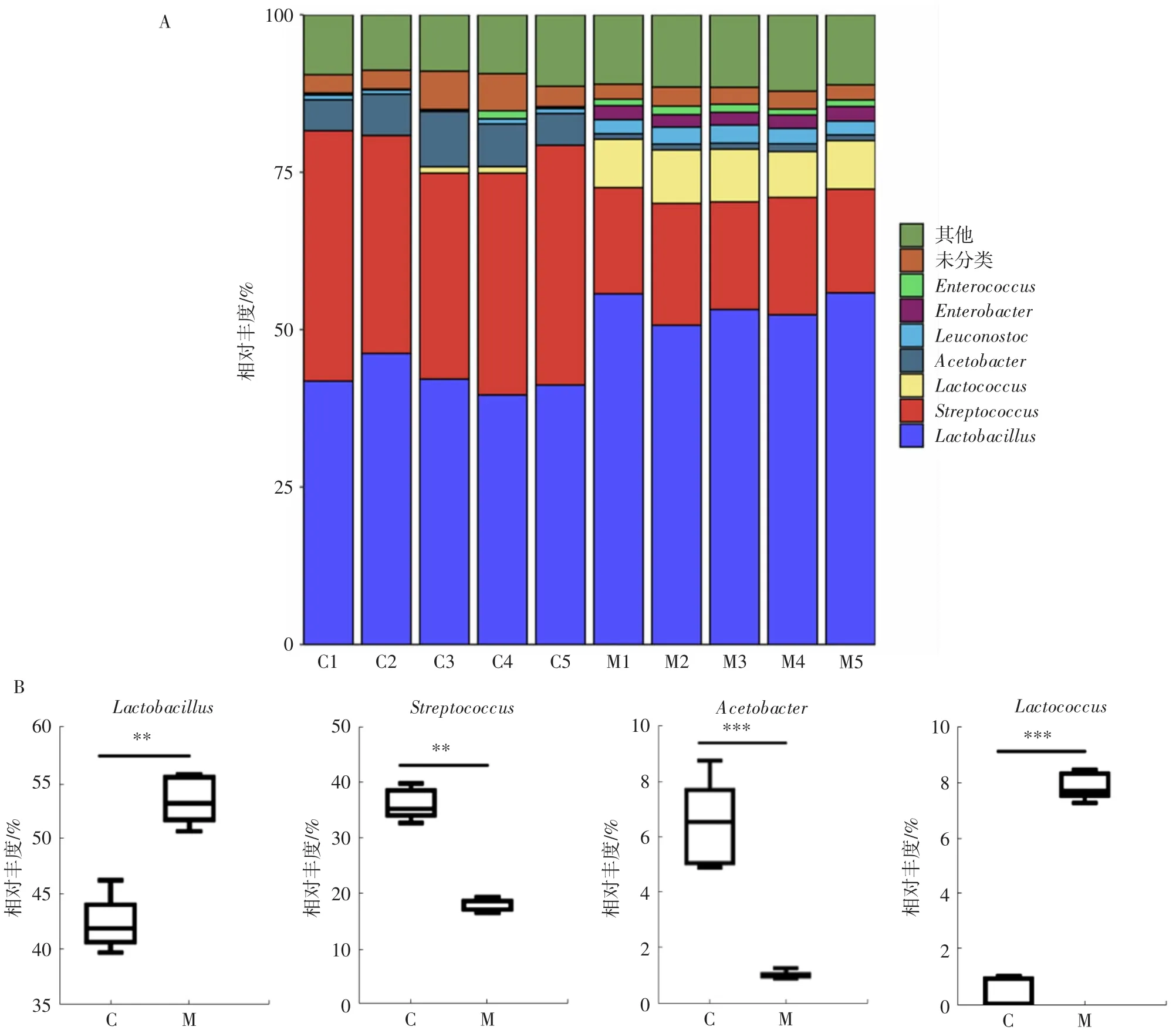
图4 酸马奶样品细菌群落结构在属水平的组成和主要乳酸菌及醋酸菌的差异Fig.4 Composition of bacterial communities at genus level in traditional koumiss and the difference of main lactic acid bacteria and acetic acid bacteria
如图4A所示,共鉴定出78个属,其中所有样品中平均相对丰度>1%的属有6个,分别为乳杆菌属(Lactobacillus)(47.87%)、链球菌属(Streptococcus)(26.88%)、乳球菌属(Lactococcus)(4.16%)、醋杆菌属(Acetobacter)(3.7%)、明串珠菌属(Leuconostoc)(1.56%)和肠杆菌属(Enterobacter)(1.09%)。这些乳酸菌的丰富度促使厚壁菌门在酸马奶中具有显著优势。乳酸菌可能来源于发酵原料和自然环境,其可将原料中的小分子糖类物质转化为酸类化合物,从而赋予酸马奶柔和的酸感;部分糖类物质被转化为醇类化合物,这些醇类物质和酸类代谢物反应形成酯类物质,进而赋予产品具有酯香和醇香等特征[18]。此外,乳酸菌可通过蛋白水解系统分解发酵基质中的可溶性蛋白形成短肽和氨基酸等小分子代谢产物,赋予酸马奶独特的鲜味特征[18]。众所周知,乳杆菌属在发酵乳制品中起着重要作用[19]。根据图4A可知,乳杆菌属是绝对优势属,所有酸马奶样品均以乳杆菌属为主,其中占每个样品细菌总数的41.20%~55.82%。不同来源的酸马奶样品中乳杆菌属的相对丰度存在明显差异,其中农场生产酸马奶样品中乳杆菌属的平均相对丰度为42.2%,显著低于(P<0.01)牧民手工酿造酸马奶样品中乳杆菌属的平均相对丰度(53.55%)(图4B)。上述结果与TANG等[20]的研究结果相一致,证实了乳杆菌属广泛存在酸马奶中,且在不同地域来源酸马奶样品之间存在差异。此外,链球菌属在酸马奶样品中相对丰度仅次于乳杆菌属,其占每个样品细菌总数的16.49%~39.76%,不同样品之间差异明显。农场生产酸马奶样品中链球菌属的平均相对丰度为36.07%,显著高于(P<0.01)牧民手工酿造酸马奶样品中乳杆菌属的平均相对丰度(17.69%)(图4B),表明链球菌属和乳杆菌属在酸马奶发酵体系中可能存在互利共生的关系。这个与丁秀云[21]的研究结果相一致,此研究还发现嗜热链球菌与保加利亚乳杆菌组合发酵可增加胞外多糖的含量。如图4B所示,醋杆菌属和乳球菌属的相对丰度在不同地域来源酸马奶样品中差异较大,其在农场生产酸马奶样品的平均相对丰度分别为6.42%和0.42%,而在牧民手工酿造酸马奶样品的平均相对丰度分别0.98%和7.9%。相比牧民手工酿造酸马奶样品,农场生产酸马奶样品中醋杆菌属的平均相对丰度提高了5.5倍(P<0.001),但乳球菌属的平均相对丰度却降低了95%(P<0.01)。醋杆菌属是食醋和果醋中的一种重要微生物,由于其可产生乙醇脱氢酶,能够将酵母产生的酒精转化为乙酸[22],也能在酸马奶中被检测到[17]。YIN等[23]发现添加谷氨酸可以增加醋杆菌属细胞生长及可培养细胞数。正是由于乳酸菌将原料中蛋白分解为短肽和氨基酸,从而促进醋杆菌的生长繁殖。乳球菌也被认为是一种重要的乳制品微生物,其可将多余的糖转化为甘露醇和葡聚糖,而这两种物质非常适合乳酸菌的发酵。上述结果表明不同地域采集的不同样品中细菌属及相对丰度含量存在显著差异,与GESUDU等[24]的研究结果相一致。根据传统酸马奶发酵工艺的总体情况,分析得出:(1)自制发酵剂、马奶原料、发酵环境和器具中的微生物是酸马奶微生物体系的来源;(2)酸马奶的品质和生产的工艺参数(如温度、发酵时间、搅拌强度等)可能会影响不同地区的酸马奶中细菌群落的变化。
2.4 差异微生物的分析
差异表达分析是比较两组不同样品基于表达异同的基本方法,可获得一组样品相对于另一组样品表达显著上调和下调,从而进一步研究这些差异表达基因的功能,多用芯片和转录组测序,但也可用于16S rRNA高通量测序技术探究不同类型样品之间微生物的差异,从而揭示其差异的原因。两组不同样品的差异微生物结果见图5。
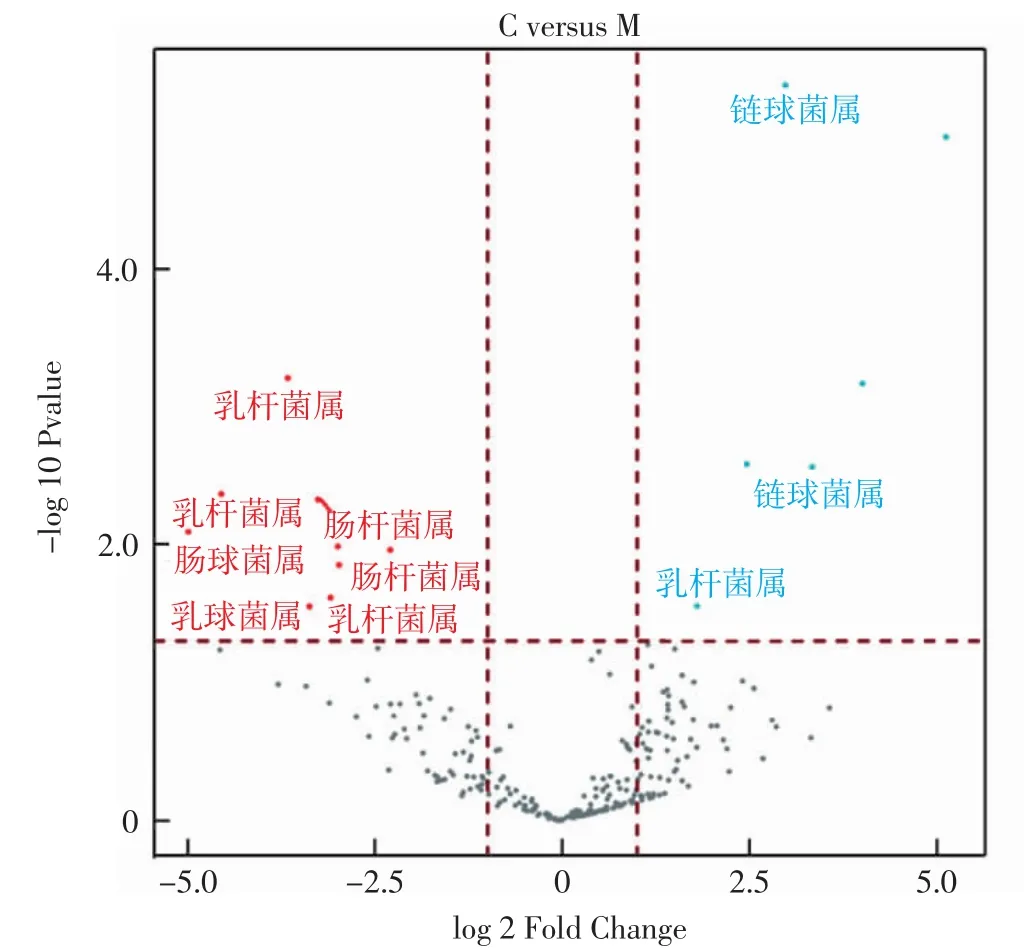
图5 火山图显示了两个不同环境来源酸马奶样品出现不同程度的OTU富集Fig.5 Volcano plot showing differentially abundant OTUs enriched in traditional koumiss from two different places
由图5可知,共发现16个差异显著的OTU,其中6个OTU可显著在农场生产酸马奶样品中富集 [2个来自链球菌属、2个来自醋杆菌属、1个来自乳杆菌属、1个来自罗斯氏菌属(Rothia)],而剩下10个差异显著的OTU则显著在牧民手工酿造酸马奶样品中富集[1个来自肠杆菌属、1个来自肠球菌属(Enterococcus)、1个来自微小杆菌属(Exiguobacterium)、2个来自乳杆菌属、2个来自乳球菌属、1个来自明串珠菌属(Leuconostoc)、1个来自假单胞菌属、1个来自栖热菌属(Thermus)]。上述结果说明牧民手工酿造的酸马奶具有较高的细菌多样性,这也与表2的结果相一致。
3 结论
本研究利用Illumina Miseq高通量测序技术探究了两个不同来源酸马奶样品细菌群落的多样性及其组成。研究发现牧民手工酿造的样品中Shannon指数和Simpson指数要显著高于农场生产的样品(P<0.05),且两者的细菌群落结构存在明显差异。厚壁菌门(Firmicutes)和变形菌门(Proteobacteria)是主要优势菌门,两者相对丰度之和可达98%。在属水平上,乳杆菌属、乳球菌属、醋杆菌属、明串珠菌属和肠杆菌属是酸马奶样品的优势菌属,尤其是乳杆菌属的相对丰度可达41.20%~55.82%。农场生产酸马奶样品中链球菌属和醋杆菌属的相对丰度含量要显著高于(P<0.01)牧民手工酿造酸马奶样品。此外,通过差异分析发现两个不同来源酸马奶样品中有16个OTU存在显著差异,这些OTU中绝大部分都来自乳酸菌。这些结果有助于了解环境异质性对传统酸马奶细菌群落的影响,但本研究是在同一个城市采集的样品,未来可进一步探究酸马奶微生物多样性的生物地理分布模式。综上所述,本研究全面了解传统酸马奶的细菌种群分布,为工业化生产提供理论依据及筛选潜在价值的益生菌提供数据参考。
