Illumina MiSeq高通量测序技术研究原料乳冷藏过程中微生物群落演替规律
2021-08-11张彤彤
苟 萌,胡 婕,张彤彤,剧 柠
(宁夏大学农学院 银川 750021)
原料乳丰富的营养物质及其偏中性的pH值,是微生物生长的绝佳环境[1]。原料乳中的微生物可能来自牛棚、饲料、乳头表面和顶端或乳制品设备的再污染[2]。这些微生物包括对乳及乳制品的感官与风味有直接影响的乳球菌属、乳酸杆菌属和链球菌属;对人体健康造成影响的李斯特菌属、志贺氏菌属和产生霉菌毒素的真菌等病原菌;以及对原料乳的腐败造成影响的有害微生物,特别是假单胞杆菌属及黄杆菌属等嗜冷菌[3]。原料乳中的微生物群落结构受地理环境、乳种类、泌乳期及贮存条件的影响[3-7]。
原料乳挤出后因无法立刻投入生产链,故运至工厂冷藏。全世界不同地区的生产商对原料乳加工前的储藏时间(2~5 d)也不同[2]。冷藏虽能抑制大多数微生物的生长,但嗜冷微生物如黄杆菌属、假单胞杆菌属仍持续生长,它们产生的蛋白酶和脂肪酶作用于乳中蛋白质和脂肪[8-10],不仅导致原料乳的腐败,而且在工业热加工处理后仍保持活性,对加工后的乳及乳制品的营养价值、风味及感官品质产生影响,例如奶酪中产生苦味或UHT乳中产生凝胶与沉淀[4,5,11-12]。由于利用现有技术手段很难使这些微生物酶失活,因此降低它们在原料乳中被污染的风险具有非常重要的意义。
近年来,有研究者针对某特定贮藏时间的原料乳中微生物的菌群多样性进行研究。然而,原料乳中的微生物随着冷藏时间发生改变,嗜冷微生物分解乳成分的同时,乳环境也影响它的存在。了解生乳加工前其中微生物菌群随冷藏时间的改变至关重要。
本研究利用高通量技术研究原料乳在4 ℃冷藏条件下微生物群落的演替规律,为原料乳冷藏期间品质控制提供理论支持。
1 材料与方法
1.1 材料与试剂
原料乳,取自宁夏回族自治区银川市某奶牛场乳罐。
1.2 主要仪器与试剂
DYY-6C 电泳仪,北京六一生物科技有限公司;C200 凝胶成像分析系统,北京百晶生物技术有限公司;核酸纯度测定仪,德国TITERTEK BERTHOLD 公司。
食品DNA 提取试剂盒(DNeasyRPowerFood Microbial Kit),德国QIAGEN 公司。
1.3 方法
1.3.1 样品采集及处理 无菌采集新鲜牛乳。原料乳的体细胞数≤2.0×105细胞/mL,细菌总数<1.0×105CFU/mL。严格无菌采集乳罐原料乳样品,装入灭菌(121 ℃,15 min)的采样瓶中,置放有冰袋的隔热背包,2 h 内运回实验室。
将采集的样品分装后4 ℃保存。根据冷藏时间编号。0 h 开始,乳罐中乳样每24 h 取出1 次,直至细菌总数达到2×106CFU/mL。根据取出时间标记为A1~A7。所有编号样品取出后-20 ℃保存,以备细菌总DNA 的提取。每个时间点样品取3 个平行样本。
1.3.2 DNA 提取检测 对食品DNA 提取试剂盒提取DNA 的方法进行改良。具体操作是:原料乳混匀后取10 mL,5 000×g、4 ℃离心10 min,去除上清液和脂肪。加入3 mL 无菌水,用涡旋混匀器混匀,5 000×g、4 ℃离心5 min,去除上清液和脂肪。加入2 mL PBS 缓冲液,混匀后吸取2 mL 菌液,按照试剂盒说明书操作,在第(4)步(转移重悬的细胞到PowerBead Tube 上)后,55 ℃水浴10 min,在第(19)步(在白色滤膜中加入100 μL EB 溶液,13 000×g 离心1 min)中加入80 μL EB 溶液。
将提取的DNA 用1.0%琼脂糖凝胶电泳和核酸纯度测定仪进行浓度和纯度检测。所检测DNA质量浓度>20 ng/L,OD260nm/OD280nm检测范围在1.8~2.0 之间的样品为合格样品。所有提取的DNA 样品置-20 ℃冰箱冻存,送样,备用。
1.3.3 PCR 扩增及Illumina MiSeq 测序 选择细菌的16S rRNA 基因的V4-V5 区作为目的扩增段,将样品统一稀释到20.0 ng/L,使用引物515 F(GTGCCAGCMGCCGCGGTAA)和引物907 R(CCGTCAATTCMTTTRAGTTT)进行扩增[13],添加Barcode 序列进行引物区分。扩增体系(25.0 L):5×反应缓冲液5.0 L,5×GC 缓冲液5.0 L,dNTP(2.5 mmol/L)2.0 L,正向引物(10 μmol/L)1.0 L,反向引物(10 μmol/L)1.0 L,DNA 模板2.0 L,dd H2O 8.7 L,Q5 高保真DNA 聚合酶0.3 L。扩增程序:98 ℃预变性2 min,25 次循环(98 ℃变形15 s,55 ℃退火30 s,72 ℃延伸30 s),72 ℃延伸5 min。使用2.0 %琼脂糖凝胶电泳检测PCR 扩增产物,采用AXYGEN 公司的凝胶回收试剂盒对目标片段进行切胶回收,将扩增回收产物进行荧光定量。制备测序文库后,使用MiSeq 平台测序仪进行2×300 bp 双端测序。PCR 扩增、测序工作由上海派森诺公司完成。
1.3.4 数据处理及分析
1.3.4.1 测序数据预处理及质控 对于原始数据,利用Trimmomatic 软件采用滑动窗口法对FASTQ 格式的双端序列逐一筛查过滤;根据重叠碱基,利用FLASH 软件对通过质量初筛的双端序列配对连接,调整序列方向,最终获得每个样本的有效序列。
1.3.4.2 数据处理 对细菌的16S rRNA V4-V5区测序,利用软件Uparse 7.0 进行OTU 聚类分析。按照97%相似性对非重复序列(不含单序列)进行OTU 聚类,在聚类过程中去除嵌合体,得到OTU 的代表序列。对有效序列进行去杂,并归类至OTU 代表序列,选出与代表序列相似性在97%以上的序列,生成OTU 表格。使用QIIME 软件,参考Silva 数据库,采用RDP classifier 贝叶斯算法对OTU 代表序列进行分类学分析,得到物种分类信息。利用R 语言制作丰度等级曲线图、稀释曲线、Venn 图、群落柱形图。
通过SAS 8.1 软件分析数据,使用最低显著差异(LSD)方法以0.05 水平进行多重比较。
2 结果与分析
2.1 提取DNA 的质量
21 个样本提取均采用试剂盒改良法,所提样本DNA 浓度范围在30.3~71.9 ng/L,OD260nm/OD280nm为1.7~2.3,均满足后续试验要求。
2.2 OTU 基本情况
21 个原料乳样本中共产生665 619 个原始reads,平均每个样本22 011 个reads。通过对原序列的质量控制,共获得462 231 个平均长度411 bp 的高质量序列,用于后续分析。按97%的序列相似度进行OTU 划分,共聚成673 个OTU。丰度曲线反映样品中所含物种丰富度和均匀程度,由图1可知,样本间重复效果较好。A1 的曲线平坦且下降缓慢,说明原料乳的初始微生物物种组成较丰富且均匀,A2~A7 曲线跨度较小,下降陡峭,说明OTU 之间丰度差异大,均匀度低,物种组成比较单一,且优势物种明显。
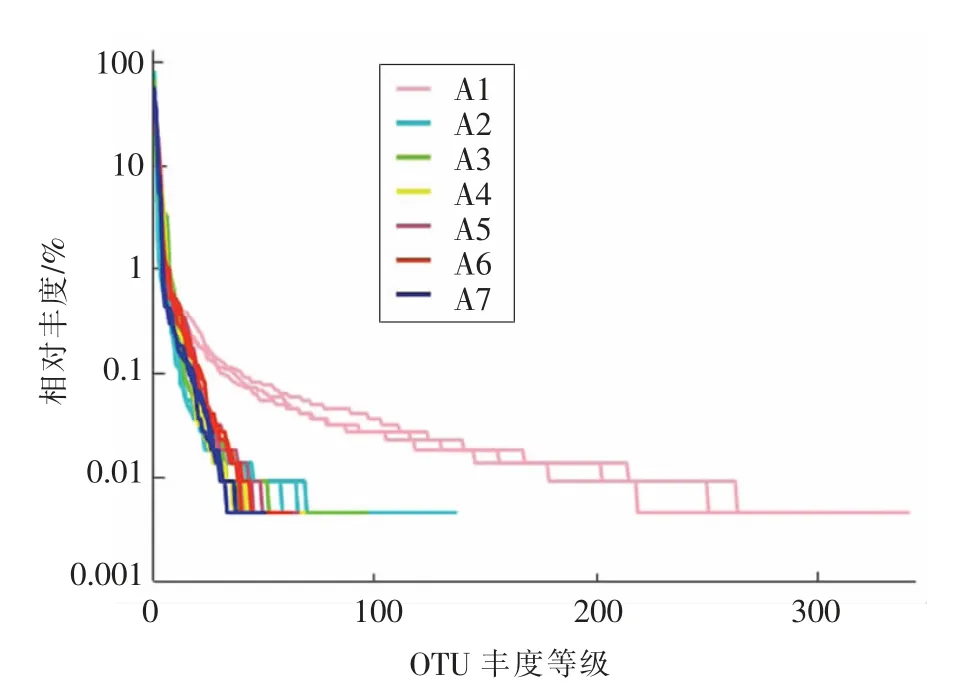
图1 相对丰度曲线Fig.1 The curve of relative abundance
2.3 Alpha 多样性分析
稀释曲线(Rarefaction curve)是利用各样本在不同测序深度时的微生物Alpha 多样性指数构建,以此反映各样本在不同测序数据量时的微生物多样性,也可以用来说明样本的测序数据量是否合理[14]。由图2可知,随着序列数的增加,稀释曲线趋于平缓,说明试验的测序深度反映样品中细菌的群落信息。
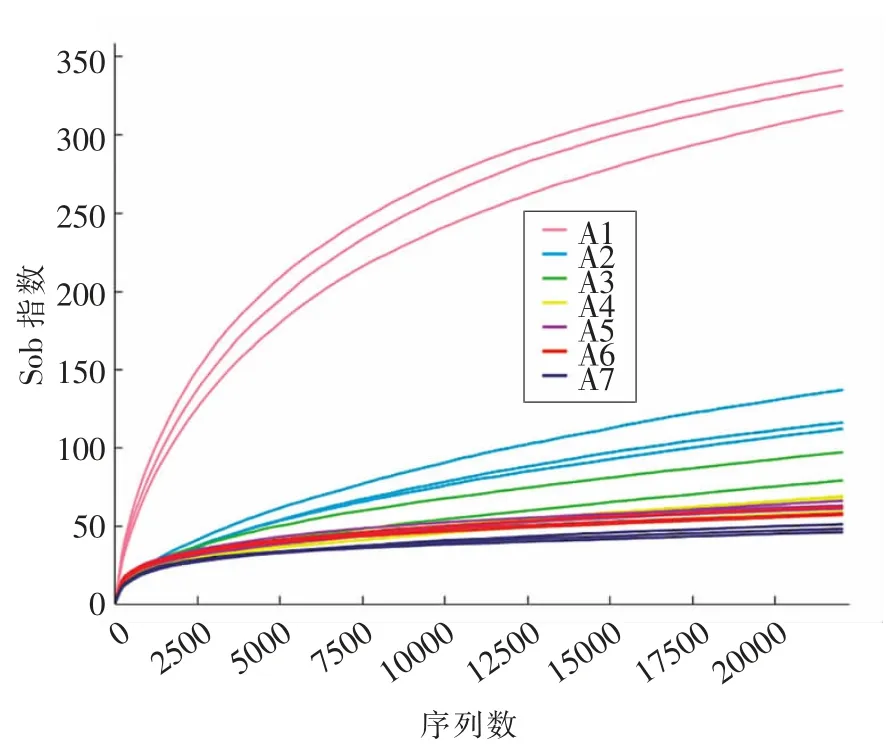
图2 稀释曲线Fig.2 Rarefaction curve
从Alpha 多样性指数(表1)可知,每个样品的覆盖率(Coverage)都在99%以上,说明测序深度基本覆盖所有细菌,测序结果能够反映每个样品中微生物的真实情况。在4 ℃冷藏期内,原料乳的Shannon 指数、Ace 指数和Chao 1 指数总体下降,与OTU 结果相似,说明罐内原料乳微生物种群多样性随冷藏时间的延长而下降。
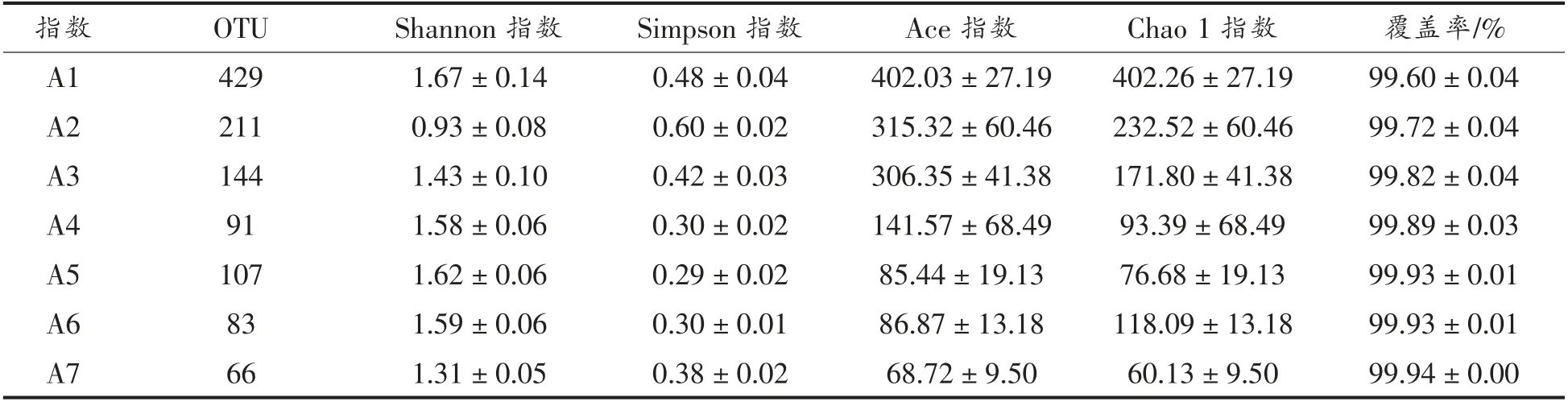
表1 Alpha 多样性指数Table 1 Alpha diversity indices
2.4 原料乳样品微生物群落组成及变化规律
2.4.1 Venn 图分析 Venn 图可用于统计多组或多个样本中所共有和独有的物种数目,可比较直观地表现环境样本的物种组成相似性及重叠情况[15]。选用相似水平为97%的OTU,统计各组样本中共有和独有OTU 数目。图3可知,冷藏过程中各样本共有OTU 数为29。冷藏初期原料乳中微生物特有OTU 个数最多,为157。冷藏过程中随时间的变化乳中微生物OUT 数目也发生变化,特有OTU 数均小于10,表明在4 ℃下,随着冷藏时间的延长,原料乳中未有新细菌出现。
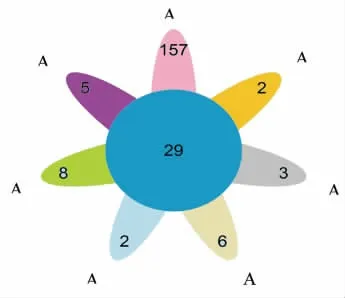
图3 基于OTU 水平的Venn 图Fig.3 Venn diagram based on OTU
2.4.2 菌群门水平多样性及演替规律分析 利用ILLumina MiSeq 测序平台研究原料乳的微生物及其4 ℃冷藏6 d 的菌群变化。整个冷藏过程中,在所有样本中共检出14 个菌门,261 个菌属。在门水平上,原料乳中存在的主要细菌菌门有变形菌门(Proteobacteria)、厚壁菌门(Firmicutes)、拟杆菌门(Bacteroidetes)和放线菌门(Actinobacteria),其变化规律及相对丰度占比见图4和表2。其中,放线菌门占比最高,相对丰度高于50%。这与张敏等[16]、Li 等[2]、Kim 等[3]、Weber 等[17]的研究结果一致,说明原料乳中的微生物受奶牛品种、地理环境及冷藏时间的影响,变形菌门是原料乳中主要的细菌菌门。
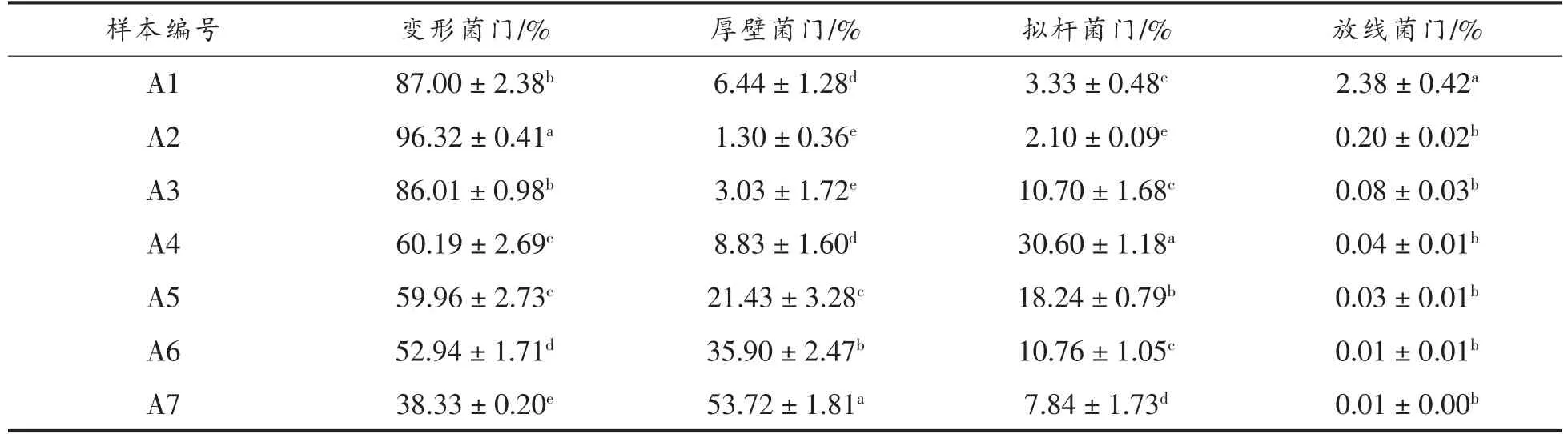
表2 原料乳冷藏过程中门水平上的菌群组成及丰度Table 2 Species composition and abundance map at phylum level in raw milk
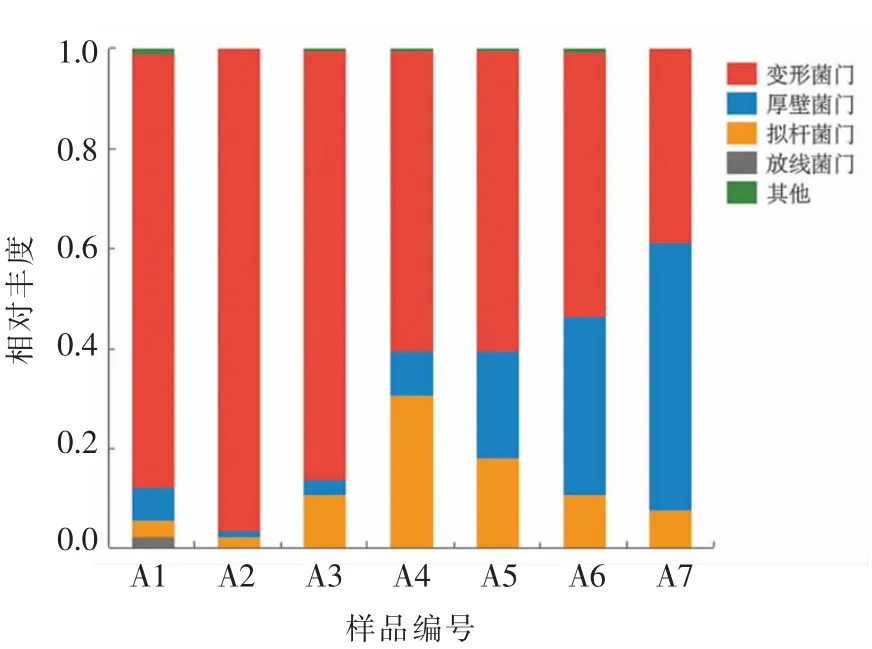
图4 原料乳冷藏过程中门水平上的菌群分布Fig.4 The flora distribution in raw milk at phylum level
在检出的细菌门中,变形菌门的占比在冷藏1 d 后显著增加(P<0.05),之后持续降低,直至冷藏结束,相对丰度从起初的87.00%(A1)降至38.33%(A7),变化显著(P<0.05)。冷藏2 d,厚壁菌门占比显著减少(P<0.05),从起初的6.44%减至1.3%,随后逐渐增加,冷藏6 d 后成为优势菌门,相对丰度达到53.72%(A7)。拟杆菌门在冷藏过程中相对丰度呈先增后降的趋势。冷藏3 d 时,拟杆菌门从起初的3.33%增到30.60%(A3);随后逐渐降低为7.84%(A7)。此时,原料乳中最主要的优势细菌由变形菌门转变为厚壁菌门。值得注意的是,Li 等[2]的研究表明,原料乳在1~4 ℃冷藏24 h 后,其优势菌门由变形菌门转变为厚壁菌门,72 h 后,变形菌门成为优势菌门。而本试验显示:3 d 内,原料乳中厚壁菌门占比虽有所上升,但变形菌门仍是主要优势菌门,直至冷藏6 d,优势菌门才转变为厚壁菌门。出现这种差异的原因可能是由于Li等[2]用原料乳为水牛乳,其中营养成分与本试验用的牛乳有很大不同,从而造成初始微生物状态存在差异。此外,冷藏过程中微生物变化又受乳环境影响,导致最终微生物群落演替产生差异。
2.4.3 菌群属水平多样性及演替规律分析 在属水平上,从原料乳中检出的主要菌属为不动杆菌属(Acinetobacter)、乳球菌属(Lactococcus)、黄杆菌属(Flavobacterium)和假单胞菌属(Pseudomonas),且各菌属相对丰度随冷藏时间发生变化(见图5和表3)。此结果与于国萍等[18]、Neubeck等[19]的研究报道一致。此外,Kim 等[3]、Doyle 等[6]研究发现原料乳中存在弯曲杆菌属及粪肠球菌属,而在本试验中未检出。
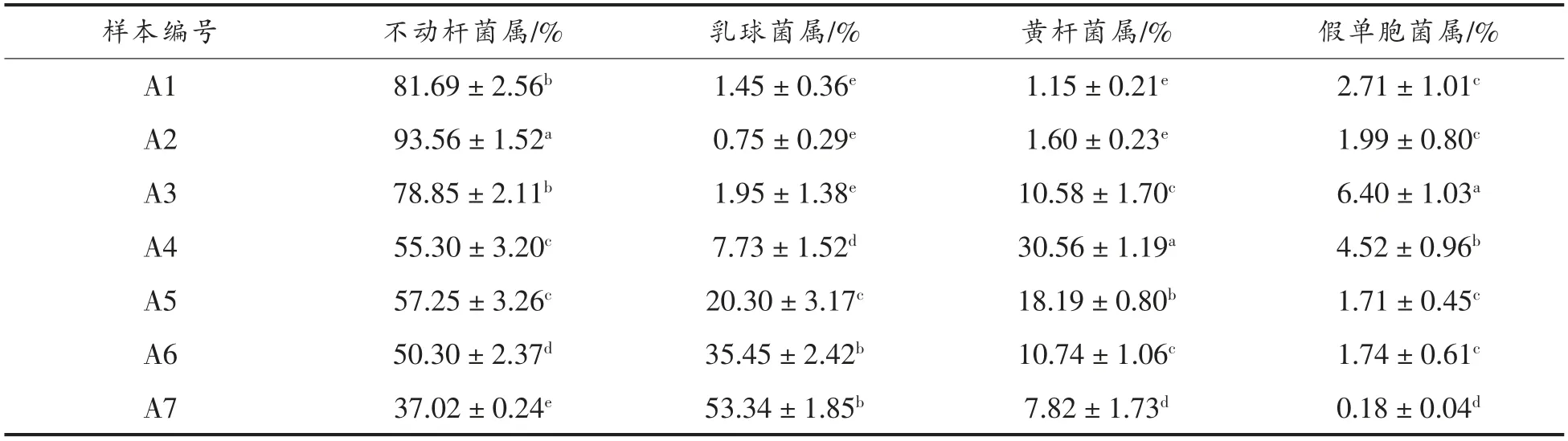
表3 样本冷藏过程中属水平上的菌群组成及丰度Table 3 Species composition and abundance map at genus level in raw milk
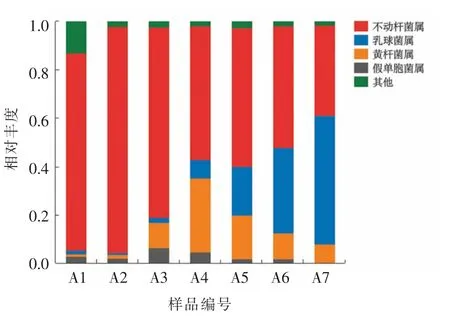
图5 原料乳冷藏过程中属水平上的菌群分布Fig.5 The flora distribution at genus level in raw milk during cold storage
冷藏5 d 内,原料乳中的菌属以不动杆菌属为主,占细菌总数的50%以上。最初,不动杆菌属占比81.69%(A1),然后显著增至93.56%(A2,P<0.05),又逐渐降到55.30%(A4),第4 天时升至57.25%(A5),之后突然降至37.02%(A7),变化显著(P<0.05)。乳球菌属初期含量仅为1.45%(A1),稍下降后从第2 天开始逐步上升,至冷藏末期升至53.34%(A7)。黄杆菌属的相对丰度从冷藏初期的1.15%增至第3 天的30.56%后下降,直至冷藏末期其丰度降为7.82%,期间变化显著(P<0.05)。而假单胞菌属的变化呈现先下降后上升再下降的趋势。相对丰度最高时在第2 天,为6.40%(A3),冷藏末期仅占0.18%(A7,P<0.05)。可见,整个冷藏过程中,优势菌属从不动杆菌属逐渐转化为乳球菌属。群落组成分析表明,冷藏过程中,原料乳微生物群落结构发生明显而复杂的变化。Li 等[2]报道,原料乳中初始优势菌属为链球菌属,72 h 后转变为假单胞菌属,同时不动杆菌属的相对丰度也有所升高。而本试验中,乳球菌属、黄杆菌属和假单胞菌属3 种优势菌属在3 d 内的占比有所增加,而不动杆菌属始终是这个阶段的优势菌属。
先前的研究显示原料乳是一个复杂的体系,其中微生物的多样性取决于泌乳期、储存时间、环境、温度、季节以及初始微生物群落。一些特有菌属的出现与这些因素密不可分。假单胞菌属、黄杆菌属及部分不动杆菌属所产生的蛋白酶和脂肪酶导致原料乳的腐败。而导致其腐败的关键菌属不仅与嗜冷微生物(如假单胞菌属)有关,也与这些特有菌属有关。虽然各研究中均能检测到一些常见嗜冷菌的存在与变化,但是不同原料乳的微生物组成与变化趋势在多种因素的影响下并不完全一致。本试验仅研究特定原料乳冷藏过程中的微生物,而对牛乳体细胞数、原料乳初始微生物含量及原料乳营养成分等因素与冷藏过程中微生物变化间的关系还有待更深入的研究。此外,本试验中还存在部分未知菌,它们占比虽不大,但与原料乳的品质变化也可能相关。这些未知菌需采用其它方法确定名称。
3 结论
本试验对4 ℃冷藏6 d 期间原料乳中的细菌群落变化进行研究。通过高通量测序,对不同阶段原料乳中存在的微生物进行门水平和属水平表征。原料乳冷藏初始阶段其菌群多样性最为丰富,随冷藏时间的延长群菌丰富度减少。同时优势菌属——不动杆菌属、乳球菌属、黄杆菌属和假单胞菌属随时间的变化而改变。了解原料乳中微生物群落演替有助于揭示原料乳品质变化的原因。本研究结果为原料乳的冷藏提供技术依据。
