桃PpMADS1的克隆及对类胡萝卜素合成基因的调控研究
2021-08-11柴吉钏方筱琴曹士锋施丽愉杨震峰
余 凡 秦 娟 柴吉钏 方筱琴 曹士锋 陈 伟 施丽愉 杨震峰,*
(1 浙江万里学院生物与环境学院,浙江 宁波 315100;2 上海海洋大学食品学院,上海 201306)
类胡萝卜素是黄色、橙色和红色果实中重要的呈色物质,是一类天然色素的总称。类胡萝卜素作为有效清除单线态氧和自由基的抗氧化剂[1],不仅可以提高健康成年人大脑认知能力[2],还能够降低白内障等眼部疾病[3]及癌症[4]的发病率。近年来,随着人们对营养健康关注度的提高,人们对类胡萝卜素等功能性物质需求也越来越大,因此,深入揭示果实中类胡萝卜素的形成与调控研究正日益受到人们的广泛关注。
高等植物类胡萝卜素的生物合成在质体中进行,遵循类异戊二烯途径,涉及的酶包括八氢番茄红素合成酶(phytoene synthase,PSY)、八氢番茄红素脱氢酶(phytoene desaturase,PDS)、ζ-胡萝卜素脱氢酶(ζ-carotene desaturase,ZDS)、番茄红素ε环化酶(lycopene ε-cyclase,LCYE)、番茄红素β环化酶(lycopene β-cyclase,LCYB)、β-羟基酶(β-hydroxylase,CHYB)和玉米黄质环氧酶(zeaxanthin epoxidase,ZEP)。目前编码这些酶的结构基因已经在柑橘[5]、越桔[6]、榴莲[7]、番木瓜[8]以及芒果[9]等许多果实中得到克隆和鉴定,且发现不同物种果实中参与类胡萝卜素合成代谢的关键基因存在差异。
MADS-box转录因子是植物中研究最广泛的转录因子家族之一[10]。MADS-box转录因子在植物所有器官的形成发生及植物生长发育的全过程都扮演了重要角色[11]。近年来,有关MADS-box转录因子参与果实成熟及品质形成调控的研究已经在番茄(Solanumlycopersicum)及多年生果树上陆续报道。但有关MADS-box转录因子参与调控果实类胡萝卜素合成与积累的研究还主要集中在模式植物番茄中。研究发现,番茄果实成熟过程中类胡萝卜素的积累由多个MADS-box转录因子共同调控,且不同MADS-box成员在果实成熟期间的表达模式存在差异,对类胡萝卜素合成的调控机制也不同。TAGL1、SlMADS-RIN、SlMBP15和SlCMB1都是番茄中正调控果实类胡萝卜素合成的转录因子,它们的沉默表达均减少了果实中类胡萝卜素的积累,降低了PSY1和PDS表达水平[12-15]。但这4个MADS-box基因中只有TAGL1和SlMADS-RIN的表达量与果实成熟期间类胡萝卜素的积累相一致;SlMBP15表达量在果实成熟期间呈先上升后下降趋势,并在果实转色期达最高值;而SlCMB1表达量在整个成熟过程中逐渐下降。SlMADS1、SlFYFL和SlMBP8是负调控番茄果实类胡萝卜素积累的转录抑制因子,但它们在果实成熟期间表达模式不同,SlMADS1和SlFYFL表达量逐渐下降,而SlMBP8表达量反而逐渐上升[16-18]。SlMADS1通过抑制PSY1基因表达降低类胡萝卜素的含量,SlMBP8对PSY1、PDS和ZDS基因均有转录抑制作用。
类胡萝卜素物质的合成与积累是黄肉桃果实呈色的重要原因[19]。目前,有关桃果实类胡萝卜素生物合成的分子机制已有一些研究。Cao等[20]报道黄肉桃果实采后贮藏期间类胡萝卜素积累与PpPSY和PpCHYB的高表达密切相关。PpCHYB基因的差异表达也是导致套袋和不套袋桃果肉中类胡萝卜素含量差异的重要原因[21]。转录调控层面进行的桃果实类胡萝卜素合成的有关研究鲜见报道。为此,本研究以黄肉桃金丽和白肉桃湖景为试验材料,克隆得到1个MADS-box基因PpMADS1,并对该基因进行生物信息学分析,通过实时荧光定量PCR(quantitative real-time PCR,qRT-PCR)检测PpMADS1基因在桃不同组织以及不同发育阶段的表达情况,并利用双荧光素酶试验分析PpMADS1对类胡萝卜素合成基因PpPSY和PpCHYB启动子的转录调控作用,旨在为揭示MADS-box转录因子对桃果实类胡萝卜素合成的转录调控机制提供理论依据。
1 材料与方法
1.1 试验材料
供试材料为浙江省宁波市德馨园生态农业科技有限公司种植的金丽及湖景水蜜桃品种。采集试样芽、幼叶、花、软核果实、硬核果实等不同组织样,S1(盛花后35~45 d)、S2(盛花后55~65 d)、S3(盛花后95~105 d)、S4(盛花后105~120 d)和S5(盛花后120~150 d)期不同成熟度果实。采集完的样品于1 h内运送回实验室,将各组织及成熟度的样品于液氮中研磨成粉末状,储存于-80℃超低温冰箱中备用。
1.2 试验方法
1.2.1 RNA的提取及cDNA的合成 采用改进的植物RNA提取试剂盒(Omega Bio-Tek公司,美国),根据使用说明提取不同组织和不同成熟度果实的总RNA。采用无RNase活性的DNaseI(Omega Bio-Tek公司,美国)去除总RNA中微量DNA污染,以备后续cDNA合成。采用NanoDro-p2000核酸蛋白分析仪(Thermo Fisher Scientific,美国)测定RNA样品的浓度和纯度。通过Gel DOC XR+凝胶成像仪(美国BIO-RAD)观察提取的RNA条带的完整性、大小和清晰度。按照SuperRT cDNA第一链合成试剂盒(江苏康为世纪生物科技有限公司)说明书进行cDNA合成。
1.2.2 桃果实PpMADS1基因的克隆 利用桃基因库(http://www.plantgdb.org/PeGDB/)以及桃果实发育时期转录组数据库筛选获得1个MAD-box基因家族候选基因ppa010595m,对其进行BLAST(https://blast.ncbi.nlm.nih.gov/Blast.cgi)比对,发现ppa010595m与预测的桃AGx3(GeneBank登录号为XM_020561783.1)序列一致,因此将ppa010595m命名为PpMADS1基因。利用Primer 5.0软件根据基因序列设计PCR扩增特异性引物(表1),委托上海生物工程技术有限公司进行引物合成。以合成的cDNA为模板,使用PCR技术扩增PpMADS1基因的开放阅读框(oper reading frame, ORF)。反应体系:5.8 μL ddH2O,0.2 μL dNTP Mix,2 μL SF Buffer,1 μL cDNA模板(浓度约为250 ng·μL-1),上下游引物各0.4 μL,Phanta Super-Fidelity DNA pdynerase 0.2 μL。PCR反应程序:94℃预变性3 min;95℃变性8 s,58℃退火30 s,72℃延伸1 min,30个循环;72℃终延伸10 min。PCR扩增产物经电泳检测后,回收纯化连接到pMD18-T 载体上,转化大肠杆菌DH5α,过夜培养,经过菌落PCR挑取筛菌板上阳性克隆子,通过摇瓶培养提质粒送华大基因测序。
1.2.3PpMADS1的生物信息学分析 利用ExPASy Protparam在线软件(https://web.expasy.org/protparam/)对PpMADS1转录因子的氨基酸序列、蛋白质分子量、氨基酸组成、理论等电点、脂溶指数、亲水指数等一级理化性质进行分析。利用SOPMA在线网站对PpMADS1蛋白的二级结构进行预测。利用在线工具Protcomp9.0进行亚细胞定位分析。利用swissmodel在线软件(http://swissmodel.expasy.org)对蛋白的三级结构进行预测。利用NCBI网站的BLAST工具对PpMADS1进行同源性比对分析,利用NCBI、ClustalX2.0、GeneDoc软件对PpMADS1与同源基因进行氨基酸序列比对分析。利用进化树分析软件MEG4.1(http://www.megasoft-ware.net)中的邻接(neighbor-joining,NJ)法[执行参数:Poission correction correction、pairwise deletion和boot-strap(1 000次重复)]对PpMADS1转录因子进行进化树分析。
1.2.4 实时荧光定量PCR分析 采用CFX96 Touch Real-Time PCR系统(Bio-Rad,美国),利用荧光定量引物进行3步法qRT-PCR扩增。qRT-PCR 程序设置为95℃初始变性7 min,95℃保持15 s,45℃退火30 s,75℃保持15 s,进行39个循环。以PpTEF2基因(引物见表1,退火温度为59℃)作为内参基因,设置阴性对照。采用2-ΔΔCT方法进行qRT-PCR的数据分析,设置3次生物学重复。
1.2.5 双荧光素酶试验 利用高保真酶Phanta® Super-Fidelity DNA Polymerase(南京诺唯赞生物科技有限公司)和ClonExpress Ⅱ One Step Cloning Kit试剂盒(南京诺唯赞生物科技有限公司)将PpPSY和PpCHYB启动子序列插入pGreenII 0800-LUC载体的荧火虫荧光素酶(firetty luciferase, LUC)上游作为报告基因,将PpMADS1的ORF序列重组到pGreenII 62-SK载体上作为效应基因。阴性对照为空载体pGreenII 62-SK。构建载体所需引物见表1。

表1 引物序列
农杆菌侵染烟草及转录激活活性测定试验参考Ba等[22]的方法,效应基因和报告基因培养液的混合比例为8∶1,用医用针筒吸取混合液侵染叶片背面,置于恒温培养箱3 d,利用双荧光素酶报告分析试剂盒(Promega,美国)和Luminoskan Ascent化学发光分析仪(Thermo,美国)测定叶片侵染区中萤火虫光素酶(firefly luciferase,LUC)与海肾荧光素酶(renilla luciferase,REN)催化产生的荧光信号的比值。PpMADS1对PpPSY和PpCHYB启动子的转录调控活性通过LUC/REN比值来计算,归一化为阴性对照,每个处理重复4次。
2 结果与分析
2.1 PpMADS1基因的克隆及序列分析
以桃果实cDNA为模板克隆得到长度为732 bp的PpMADS1的ORF序列见图1。PpMADS1基因编码的氨基酸一级结构预测值见表2,PpMADS1蛋白酸碱性氨基酸组成比例和理论等电点显示其呈碱性。不稳定指数和脂溶指数表明PpMADS1不稳定,具有较好脂溶性。亲水指数表明PpMADS1表现出亲水性。

Note: M: Marker.

表2 PpMADS1蛋白基本理化性质
2.2 PpMADS1蛋白的结构分析
如图2所示,PpMADS1蛋白的二级结构预测结果中α-螺旋占55.97%,β-转角占4.12%,延伸链约占8.64%,无规则卷曲占31.28%,说明PpMADS1蛋白的二级结构以α-螺旋为主要构成元件,这与PpMADS1蛋白的三级结构预测结果相一致(图3)。对PpMADS1蛋白进行亚细胞定位分析发现,PpMADS1定位在细胞核中的可能性最大,符合转录因子的功能定位特征。
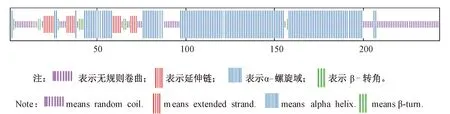
图2 PpMADS1蛋白的二级结构预测
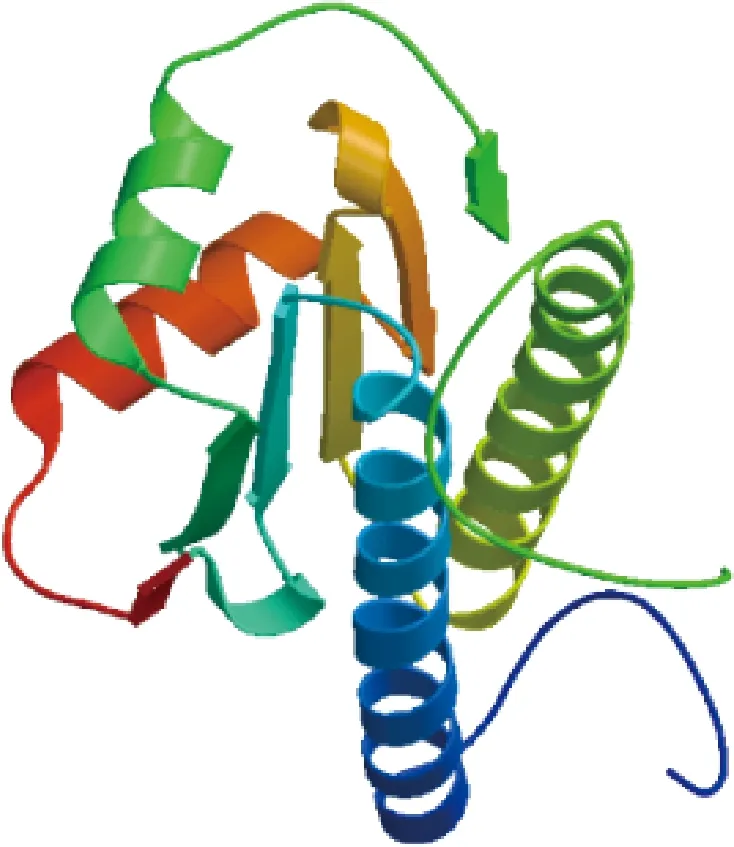
图3 PpMADS1蛋白的三级结构预测
2.3 PpMADS1氨基酸序列分析
从NCBI数据库中下载桃PpMADS1基因的同源序列,物种包括乌梅(Prunusmume),中国樱桃(Prunuspseudocerasus),野黑樱桃(Prunusserotina),粳稻(Kerriajaponica),苹果(Malusdomestica),沙梨(Pyruspyrifolia),白梨(Pyrusxbretschneideri),番茄等。同源性分析发现PpMADS1与乌梅(XP_008225496.1)的氨基酸相似率最高,达到100%,与中国樱桃(AIU94283.1)氨基酸序列相似率达到99.59%,仅有少量氨基酸存在差异。将PpMADS1与番茄中已被证明对类胡萝卜素合成代谢有调控作用的基因SlMADS1[16](NP_001234380.1)、SlCMB1[15](XP_004237061.1)、SlMADS-RIN[13](NP_001234670.1)、SlFUL1[23](NP_001234173.2)、SlFYFL[17](AHG98096.1)、TAGL1[24](NP_001300859.1)、SlMBP15[14](NP_001352611.1)进行同源分析,他们的同源性分别为43.95%、46.12%、44.50%、47.54%、46.55%、65.82%、38.64%。利用ClustalX及Genedoc软件对PpMADS1氨基酸序列与其他6个物种的MADS蛋白氨基酸序列进行比对(图4),结果发现PpMADS1蛋白的N端包含MADS转录因子家族共有的特征MADS结构域,同时包含了Ⅰ(intervening)region、k(keratin-like)-box以及相似的C端结构[25-26],PpMADS1蛋白C端有含有植物AG-motif特征序列,这说明桃PpMADS1属于MADS-box转录因子AG亚族。

注:PpS:Prunus pseudocerasus. Ps: Prunus serotina. Kj: Kerria japonica. Ppy: Pyrus pyrifolia; Pxb: Pyrus x brets chn eideri. Md: Malus domestica.
2.4 PpMADS1系统进化树分析蛋白
为进一步研究桃PpMADS1蛋白的系统进化关系,用NJ法分析了与其同源性较高的扁桃、中国樱桃、乌梅、苹果、欧洲樱桃、野黑樱桃、白梨、东京樱花、粳稻、沙梨相关MADS家族基因的进化关系。由图5可知,与PpMADS1蛋白亲缘关系较近的是扁桃、东京樱花和中国樱桃。同为李属的扁桃、乌梅、欧洲樱桃、野黑樱桃、中国樱桃、东京樱花聚为一类,粳稻、苹果、沙梨和白梨聚于一类,表明不同植物间的系统进化关系具有明显的种属特征。
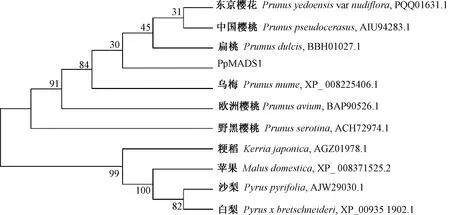
图5 PpMADS1基因系统发育树
2.5 PpMADS1时空表达分析
PpMADS1基因在金丽和湖景不同组织中的表达模式一致,即PpMADS1在芽、叶中几乎不表达,在花和果实中大量表达,湖景花中PpMADS1表达量显著高于金丽,金丽软核和硬核果实中PpMADS1表达量都显著高于湖景(图6)。在2个品种桃果实成熟过程中PpMADS1表达量整体呈下降趋势;成熟度S1时期,金丽果实中PpMADS1表达量显著高于湖景;成熟度S2时期,湖景果实中PpMADS1表达量显著高于金丽;成熟后期(S4~S5),湖景果实中PpMADS1表达量都显著低于金丽果实(图7)。

注:不同小写字母表示差异显著(P<0.05)。下同。
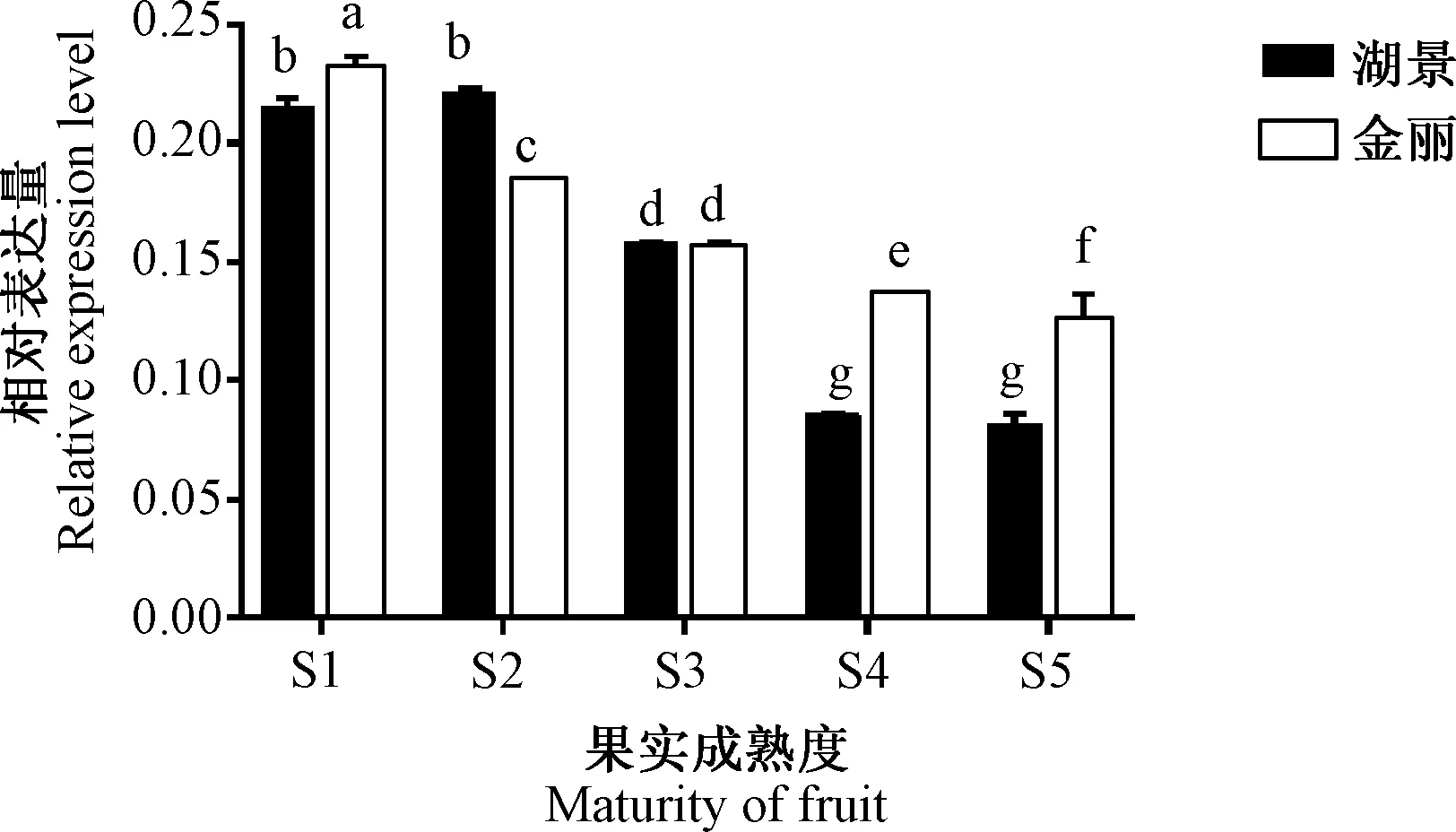
图7 不同成熟度果实中PpMADS1基因的表达
2.6 PpMADS1转录因子对PpPSY和PpCHYB基因启动子的转录调控特性
利用同源重组方法将PpPSY和PpCHYB基因启动子融合LUC报告基因,将PpMADS1融合62-sk效应基因(图8-A)。双荧光素酶试验结果如图8-B所示,与空载体相比,PpMADS1转录因子显著提高了PpPSY和PpCHYB启动子的转录活性(P<0.05),说明PpMADS1转录因子对PpPSY和PpCHYB启动子具有转录激活作用。

注:A: 载体构建原理图;B: PpMADS1对PpPSY、PpCHYB启动子的调控作用。
3 讨论
MADS-box转录因子家族是目前在植物领域研究最广泛的转录因子家族之一,调控着植物的营养生长和生殖生长过程,在果实成熟与品质调控方面具有显著作用[27]。本研究从桃果实中克隆得到一个桃MADS-box基因,将其命名为PpMADS1。MADS-box家族不同成员基因具有相对保守的序列,蛋白结构存在相似性。生物信息学分析表明,PpMADS1编码蛋白呈碱性,二级结构中α-螺旋、β-转角和无规则卷曲及三级结构中的蛋白质折叠的空间构象具有较高的保守性,具有较好的脂溶性,蛋白质结构不稳定,且为亲水蛋白,PpMADS1蛋白特征与其他植物MADS基因家族的研究结果基本一致[28]。亚细胞定位预测显示PpMADS1定位于细胞核,具有转录因子特征。同源性分析表明,不同物种来源的MADS-box蛋白具有较高同源性,其中桃PpMADS1与扁桃、乌梅、中国樱桃、东京樱花等李属MADS-box蛋白相似性较高。
在模式植物番茄中研究发现果实成熟过程中类胡萝卜素的积累是由多个MADS-box转录因子共同调控,包括正调控转录因子TAGL1、SlMADS-RIN、SlMBP15、SlCMB1以及负调控转录因子SlMADS1、SlFYFL和SlMBP8[12-18]。在这些番茄MADS-box转录因子中,桃PpMADS1与TAGL1序列相似度最高,达65.82%;PpMADS1与TAGL1氨基酸序列上都含有AG亚族特有的典型基序,因此都属于MADS-box转录因子AG亚族[29];在不同组织中,PpMADS1与TAGL1基因在芽、叶中几乎不表达[12],在花和果实中大量表达;且成熟后期黄肉桃金丽果实比白肉桃湖景有更高的PpMADS1表达水平,这与Cao等[20]报道黄肉桃金丽果实含有更高类胡萝卜素含量相一致,上述结果暗示PpMADS1可能具有与TAGL1相似的基因功能,即可能也对果实类胡萝卜素的积累起正调控作用。八氢番茄红素合成酶PSY被认为是类胡萝卜素合成的限速酶[30]。番茄果实中PSY基因超量表达可有效促进果实类胡萝卜素的积累[31]。TAGL1转录因子正是通过上调PSY1和PDS表达水平,促进番茄果实类胡萝卜素的积累[12]。董新甜等[21]研究发现套袋和不套袋桃果肉间PpCHYB基因的差异表达可能是导致两种果实类胡萝卜素含量差异的重要原因。Cao等[20]报道黄肉桃金丽果实采后贮藏期间类胡萝卜素积累与PpPSY和PpCHYB基因的高表达密切相关。这些结果表明PpPSY和PpCHYB是黄肉桃果实类胡萝卜素合成的关键基因。在此基础上,本研究通过双荧光素酶试验发现,PpMADS1转录因子对PpPSY和PpCHYB基因启动子具有转录激活作用,进一步验证了PpMADS1转录因子可能通过调控PpPSY和PpCHYB基因表达促进桃果实类胡萝卜素合成。但PpMADS1转录因子调控桃果实类胡萝卜素合成与积累的具体分子机制有待进一步深入研究。
4 结论
本研究从金丽和湖景2个品种桃果实中克隆得到PpMADS1的ORF序列,长度为732 bp,编码243个氨基酸。生物信息学分析表明PpMADS1蛋白可能为碱性,具有较好的脂溶性,不稳定,表现出亲水性,定位于细胞核。PpMADS1与乌梅的氨基酸序列相似率最高,达到100%,氨基酸序列具有典型的MADS家族特征结构域及AG亚族特种基序,属于MADS-box转录因子家族AG亚族基因。qRT-PCR和双荧光素酶试验结果表明,PpMADS1转录因子可能通过调控PpPSY和PpCHYB基因表达促进桃果实类胡萝卜素合成。本研究为进一步揭示PpMADS1转录因子在果实成熟过程中调控类胡萝卜素合成代谢的分子机制提供了理论依据。
