动植物环状RNA研究进展与展望
2021-07-29孙铭阳李永光徐世强李静宇王继华
孙铭阳 李永光 徐世强 梅 瑜 顾 艳 周 芳 李静宇 王继华*
(1.广东省农业科学院 作物研究所/广东省农作物遗传改良重点实验室,广州 510640;2.东北农业大学 大豆生物学教育部重点实验室/农业农村部东北大豆生物学与遗传育种重点实验室,哈尔滨 150030)
环状RNA(Circular RNAs)是由前体mRNA经反向剪切(Back-splicing, BS)产生的具有组织表达特异性的单链共价闭环RNA分子。其参与基因转录或转录后水平的调控,普遍涉及生长发育、病理反应、激素诱导及胁迫应激等进程[1]。早在二十世纪七十年代末,学者便已发现环状RNA,但当时其被描述为类病毒的遗传物质[2]。直至1991年,Nigro等[3]报道,由抑癌候选基因“Deleted in colorectal cancer(DCC)”转录产生的前体mRNA在进行可变剪切(Alternative splicing, AS)时,可产生由外显子组成且定位于细胞质的环形转录本。这是证明环状RNA可由DNA转录的首篇报道。然而,因其无Poly(A)尾,环状RNA无法被借助Oligo dT完成反转录的传统高通量测序技术识别并与mRNA区别开来,导致其后续研究受限。RNase R是一类3’-5’特异性识别并降解线状RNA的外切酶。总RNA样品经RNase R处理后,可去除其中线性RNA的干扰,为环状RNA的测序提供可靠模板。最近,针对环状RNA的高通量测序技术及BS位点特异性识别工具(如circMarker)已成功研发并得到广泛应用[4]。环状RNA特异性注释软件(如CIRIquant)已发现真核生物体内大量存在的环状RNA[5-6]。
部分环状RNA可通过碱基互补配对结合miRNA并抑制其功能,称为miRNA海绵。因部分miRNA参与人类癌细胞的扩散和转移进程,所以miRNA海绵已成为临床研究热点[7]。此外,临床研究还发现一些可翻译多肽或充当蛋白支架的环状RNA,现已阐明它们在癌症发展过程中的作用[8-9]。除在转录后水平发挥功能外,环状RNA还可通过结合来源基因的DNA序列调控转录效率[10]。虽然植物领域有关环状RNA作用机理的研究还很少,但有学者在植物体内过量表达特定环状RNA后,发现转基因株系可抗逆境胁迫[11]。可见,动物和植物环状RNA均体现出它们的应用潜能。
生物标志物是科技应用的基础。环状RNA的结构稳定且表达于各类组织及细胞,具有生物标志物的特征。临床研究发现,其可作为诊疗癌症的候选生物标志物[12]。植物生物标志物是作物遗传改良的重要工具。Conn等[13]表明,拟南芥(Arabidopsisthaliana)的特定环状RNA可成为目标基因AS事件的候选生物标志物。但若要将某环状RNA确定为真正的生物标志物并应用于生产生活中,仍需广泛且准确的验证数据作为支撑。目前,围绕动植物环状RNA的分子机理研究已得到深入开展,为解析它们在生命进程中发挥的作用提供了大量实验线索和判定依据。但截至如今,相对于动物环状RNA系统性的研究,有关植物环状RNA的功能验证很少,且多数还停留在表型水平。若植物环状RNA的研究能够在借鉴动物研究经验的同时,根据自身特有或共有的性质制定合理的试验思路,将提高重大突破出现的可能性。鉴于此,本研究旨在对动植物环状RNA的生成机制、剪切识别位点、发挥功能、研究手段和应用潜力等方面进行异同点比较,以期为植物环状RNA研究提供参考。
1 环状RNA的分类
环状RNA主要来自基因区(少数位于基因间区),无5’帽和3’尾的共价结构使其比线性RNA更加稳定,半衰期更长[14]。通常将覆盖基因区的环状RNA划分为三大类,即仅含外显子的环状RNA、仅含内含子的环状RNA及同时包含外显子和内含子的环状RNA。无论在动物还是植物体内,根据BS位点形成的位置,环状RNA共细化为10种类型(图1):(1)外显子型(仅含外显子);(2)外显子-内含子复合型;(3)内含子型(仅含内含子);(4)内含子-外显子复合型;(5)非编码区型;(6)非编码区-外显子复合型;(7)非编码区-内含子复合型;(8)基因间区型;(9)基因间区-基因区复合型;及(10)跨基因型。

1.外显子型;2.外显子-内含子复合型;3.内含子型;4.内含子-外显子复合型;5.非编码区型;6.非编码区-外显子复合型;7.非编码区-内含子复合型;8.基因间区型;9.基因间区-基因区复合型;10.跨基因型1.Exon type; 2.Exon-intron type; 3.Intron type; 4.Intron-exon type; 5.Non-coding region type; 6.Non-coding region-Exon type; 7.Non-coding region-intron type; 8.Intergenic region type; 9.Intergenic-gene type; 10.Cross-genotype图1 环状RNA类型示意图(修改自参考文献[15])Fig.1 Types of circRNAs (Modified from reference [15])
2 环状RNA生成机制的同源性和特异性
为形成仅包含外显子的成熟体mRNA,真核细胞会识别前体mRNA内含子两侧的特定碱基,对其进行AS。真核生物AS事件是保守的,过程主要依赖RNA聚合酶II(Pol II)和U2蛋白,可使一种前体mRNA产生多种成熟体mRNA亚型[16]。除产生类型丰富的线性RNA外,当外显子的3′端与5′端反向相遇时,RNA连接酶可将其反向连接成环,形成仅包含或部分包含外显子的环状RNA。被切掉的内含子呈套索结构,经进一步修饰形成内含子型环状RNA[17](图2)。

图2 环状RNA生物合成示意图(修改自参考文献[18])Fig.2 Biosynthesis of circRNAs(Modified from reference [18])
2.1 环状RNA生成相关蛋白的同源性
部分动物RNA结合蛋白可充当环状RNA生成过程的激活物或抑制物。如quaking(QKI)蛋白可结合在外显子两侧的内含子上,在外显子的3′端和5′端反向接近时促进成环[19]。有研究者在植物中鉴定出QKI的同源蛋白(QKI-like),但其是否在植物环状RNA生物发生过程中发挥功能仍有待验证。已有研究表明,拟南芥QKI-like蛋白除参与前体mRNA的加工[20],还响应开花调节[21]、应激反应[22]及激素信号转导过程[23]。经氨基酸序列比对发现,拟南芥QKI-like蛋白与动物QKI蛋白高度同源,可能参与植物环状RNA的生成[19]。
2.2 外显子型环状RNA侧翼序列的特异性
动物外显子型环状RNA的生成受两侧内含子上的长重复序列、长反向互补序列及RNA结合蛋白等元件的调控[24-25]。植物外显子型环状RNA的侧翼内含子在接近时并不依赖长重复或长反向互补序列[26]。超过33%的拟南芥环状RNA在距离BS位点4~11 nts的位置会出现至少两段短反向互补序列,表明促进植物环状RNA生成的侧翼元件可由多段短互补序列弥补序列长度上的不足[27]。此外,玉米(ZeamaysL.)转座子及其反向互补产物可富集于BS位点的侧翼并促进生成环状RNA[28]。根据侧翼序列及其上结合分子的特性可得出结论,植物外显子型环状RNA的形成过程更倾向于部分相似或完全区别于动物外显子型环状RNA形成方式的其他调控机制。
3 环状RNA剪切信号的多样性及影响因素
3.1 动物环状RNA的经典型剪切信号
高等真核生物体内积累最多的核蛋白复合体为催化剪切前体mRNA的U2依赖型剪切体,其可识别剪切位点5′端和3′端的经典型剪切信号(GU-AG序列),即动物环状RNA的主要剪切信号。在特定序列被切掉的同时,RNA连接酶可催化两端连接为成熟mRNA或环状RNA。此外,部分动物环状RNA可由非典型剪切位点(NN-NN)生成(由U2蛋白或小剪接体介导)[29]。但在早期的环状RNA高通量测序研究中,由非典型剪切信号产生的环状RNA候选片段常被归类为假阳性并被多数测算工具过滤掉,为相关分析带来阻碍。
目前,KNIFE是最可靠的环状RNA识别工具,其算法以统计学为基础,仅针对环状RNA的BS位点[30]。KNIFE已在动物体内发现少数具有非典型剪切信号的环状RNA,但大多数动物环状RNA均由经典型GU-AG信号剪切而来。
3.2 植物环状RNA的差异剪切信号
多数植物环状RNA侧翼内含子上均存在短互补或短重复序列,目前无法确定这些短序列是否与它们剪切信号的类型有关[29]。现已发现,不同的双子叶植物之间或单/双子叶植物之间环状RNA的非典型剪切信号占比有所不同。如在双子叶植物拟南芥中,99%的环状RNA均形成于经典型GU-AG识别信号[31]。而针对同为双子叶植物黄瓜(CucumissativusL.)的一项研究显示,只有少数(约11.7%)环状RNA含有经典型GU-AG剪切信号[32]。同时,在单子叶植物水稻(OryzasativaL.)中,超过90%的环状RNA来自不同的非典型剪切信号,这与circseq_cup工具对水稻环状RNA的鉴定结果一致[33]。由此可知,与动物相比,不同植物物种环状RNA的剪切识别信号差异较大。
经典或非经典型剪切信号出现的频率也受到不同预测工具、过滤标准和转录本测序程序的影响。虽然这些标准可单独用于评估环状RNA的数量和质量,但未来仍需利用多种识别工具和过滤标准来分析同一份环状RNA的测序结果,以此来减少或消除不同工具间的识别误差。
4 动植物环状RNA的功能研究进展
通常情况下,外显子型环状RNA在生成后定位于细胞质,内含子型和复合型环状RNA定位于细胞核。环状RNA的主要功能为:(1)MiRNA海绵;(2)滚环式翻译新型多肽;(3)形成环状RNA-蛋白复合体调控细胞进程;(4)调控来源基因的转录水平。
4.1 MiRNA海绵
MiRNA是内源短序列(21~24 nts)非编码RNA(non-coding RNAs,ncRNAs),其抑制靶蛋白积累水平的功能可根据不同的碱基互补率分为:(1)在动物体内结合靶mRNA和AGO蛋白来抑制肽链的合成(与靶mRNA之间的碱基互补率较低);(2)在植物体内与靶mRNA结合并借助AGO蛋白触发RNA链的裂解(与靶mRNA之间的碱基互补率较高)[34]。随研究深入,学者发现越来越多的环状RNA具有miRNA海绵的功能[35]。环状RNA与miRNA碱基互补配对后,通过抑制miRNA的活性,间接上调miRNA靶基因的蛋白积累[36]。
类似环状RNA这类可吸附miRNA的转录本在动物界被称作竞争性内源RNA,在植物界被称为模拟靶标[37]。因不易被多数RNA外切酶裂解,环状RNA的半衰期大于48 h[38],而线性RNA半衰期不足20 h[39]。所以,环状RNA作为miRNA海绵的优点是它们在细胞内高度稳定。
4.1.1动物miRNA海绵的特征及研究进展
2013年,有研究证实环状RNA-CDR1as(或ciRS-7)上存在70多个同一miRNA(miR-7)的吸附位点。但这些结合位点并未显示出较高的碱基互补率,互补区仅位于5’端种子区[40]。随后,研究者在基因编辑小鼠体内发现了一个环状RNA与2种miRNA之间的互作关系,表明动物环状RNA吸附的miRNA类型具多样化[41]。医学领域有关miRNA海绵的研究最为深入,2018—2020年最新研究结果见表1(影响因子>15.0)。

表1 miRNA海绵功能在临床上的验证研究进展Table 1 Research of miRNA-sponge function verification in clinic
4.1.2植物miRNA海绵预测及验证
与动物领域的研究类似,植物环状RNA的功能研究也集中在miRNA海绵或调控miRNA通路的潜在作用上。PlantCircNet(http:∥bis.zju.edu.cn/plantcircnet/)是一类预测植物环状RNA-miRNA-靶mRNA网络的数据库,可对多来源数据进行生物信息学整合,形成可视化环状RNA-miRNA-靶mRNA网络图,有助于识别环状RNA的代谢效应和潜在机制[63]。Liu等[64]假设mRNA和环状RNA都是同一miRNA的靶标,在叶片衰老过程中差异表达的环状RNA、miRNA和靶mRNA中,发现其中任意两种RNA之间的表达模式存在相反的情况,以此作为环状RNA参与miRNA和靶基因调控网络的间接证据。有研究曾尝试从植物中鉴定出吸附位点丰富的miRNA海绵。但截至目前,植物中并未出现类似CDR1as那样拥有高密度miRNA结合位点的环状RNA。与动物环状RNA相比,预测为miRNA海绵的植物环状RNA数量很少(约5%),且它们的miRNA结合位点也相对较少[65]。虽然拟南芥研究发现了不同生长阶段潜在的环状RNA-miRNA-靶mRNA调控网络,但由于缺乏实验验证,这些调控网络的可信度不高[64]。2018—2020年预测到植物环状RNA-miRNA-靶mRNA网络的最新研究见表2。

表2 预测环状RNA-miRNA-mRNA网络的植物研究Table 2 Plant research on prediction of circRNA-miRNA-mRNA networks
植物miRNA与环状RNA之间的碱基互补率比动物更高,且主要功能为诱导靶RNA的断裂[70]。虽然高互补率更有利于识别植物miRNA的靶基因,但裂解RNA的功能可能导致环状RNA的断裂。因此,植物miRNA海绵的验证工作会比动物研究更加艰难,相关互作实验进展缓慢。可喜的是,CLIP-seq技术已在植物中得到实现,可识别植物miRNA海绵[71]。通过针对不同RNA结合蛋白(如AGO蛋白)的CLIP-seq实验,不难想到其中会活跃着环状RNA。
4.2 翻译新型多肽
经典的cap依赖型蛋白翻译机制需先识别mRNA的5’帽或3’尾,随后启动多肽合成。故缺乏5’帽和3’尾的闭合环状RNA在早期被归类为无法翻译蛋白的ncRNA。但近期研究发现,动物环状RNA可通过cap非依赖型蛋白翻译机制的识别序列——内部核糖体进入位点(Internal ribosome entry sites, IRESs)来激活翻译[72],且翻译过程可由N6-甲基腺苷RNA(m6A)修饰来推进[73]。CircInteractome网站(https:∥circinteractome.irp.nia.nih.gov/)可检测环状RNA序列中潜在的IRESs序列,预测其可能生成的功能蛋白[74]。同时,某些环状RNA可能含有无限开放阅读框,允许肽链进行滚动式无限延伸。Abe等[72]人工合成了一个具有无限开放阅读框的环状RNA分子,用以测试其在真核细胞中的翻译能力;该环状RNA上不含IRESs和m6A修饰,取而代之的是一段包含起始密码子的Kozak consensus序列;结果显示该合成环状RNA可以进行滚动式无限翻译。由此可知,IRESs序列和m6A修饰并非环状RNA启动翻译的必要因素。目前,环状RNA的翻译功能较为罕见。
由表3可知,部分人类环状RNA翻译产物的功能已在癌症研究中得到验证。如circ-FBXW7翻译的多肽(FBXW-185aa)可抑制脑癌细胞的增殖[8]。FBXW-185aa可与去泛素化酶USP28相互作用,避免USP28与肿瘤发生关键调节因子FBXW7α结合,破坏USP28诱导的c-Myc稳态,阻止FBXWα诱导的降解反应,在脑肿瘤治疗过程中发挥积极作用。
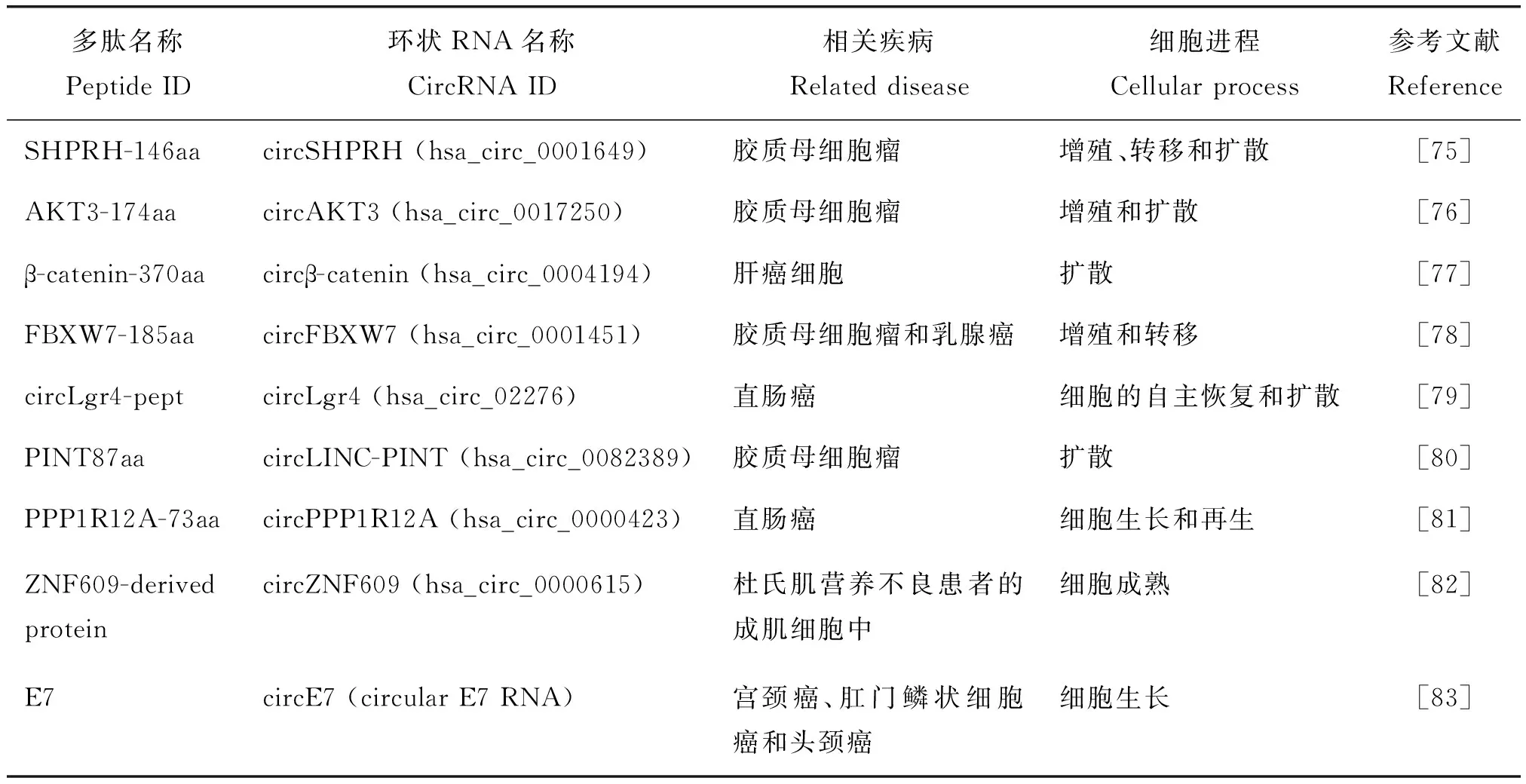
表3 人类癌症相关环状RNA的翻译蛋白和功能Table 3 Functions of proteins encoded by circRNAs in human cancers
虽已有研究指出拟南芥的特定环状RNA存在特异性m6A修饰,但尚未有植物环状RNA翻译多肽的公开报道[85]。随后续研究的深入,植物环状RNA的翻译功能也将得到注释。
4.3 与蛋白结合并调控细胞进程
环状RNA的功能不止于吸附miRNA和编码多肽,闭环结构使其能够稳定地与功能蛋白结合。虽然目前未出现植物环状RNA是否可与蛋白结合的报道;但临床研究已证实,部分人类环状RNA可作为分子支架与功能蛋白结合,协同调控疾病的发展。小鼠活体试验表明,circ-Amotl1可作为PDK1蛋白和AKT1蛋白的支架,帮助它们在细胞核中移动和定位,减缓细胞凋亡,提高AKT心肌细胞的存活率,最终帮助治愈心肌炎[9]。同时,circ-Amotl1还可从细胞质中募集信号转导因子和转录因子,保证它们与Dnmt3a启动子稳定结合,提高细胞的黏附率、迁移率和增殖率,促进伤口愈合[86]。
对于疾病的治疗,同一环状RNA在不同组织内发挥的功能未必都是积极的。如circ-Foxo3可作为p53蛋白和MDM2蛋白的结合支架来增强MDM2依赖型的p53泛素化,促进癌细胞凋亡,抑制癌症扩散[87]。但circ-Foxo3的蛋白结合功能却在治疗心肌细胞衰老时发挥负调控作用。心肌组织中的circ-Foxo3通过扣留抗衰老相关蛋白ID1和E2F1来加速心肌细胞衰老。敲除circ-Foxo3成为了保护心肌细胞的潜在治疗方法[88]。因此,未来应谨慎考虑将环状RNA应用于临床治疗,避免为患者带来更大副作用。
其他人类环状RNA的结合功能也不容忽视。Ⅲ类先天淋巴细胞(Group 3 innate lymphoid cells,ILC3)是肠道免疫系统中的负调控因子。位于细胞核的circ-Kcnt2可通过自身的过量表达来抑制ILC3的活性,维持肠道的免疫稳态。内在分子机理为:circ-Kcnt2可与核小体重构脱乙酰酶(Nucleosome remodeling deacetylase,NuRD)复合物共同结合在促结肠癌发展的T细胞功能调控基因Batf的启动子上;抑制Batf蛋白的表达;进而减少白细胞间介素-17(Interleukin-17,IL-17)的积累;最终阻止ILC3的激活;帮助治疗先天性结肠炎[89]。该研究在circ-Kcnt2缺失型突变小鼠的肠道中检测到过量积累的ILC3及结肠炎加重的表型,反向证明了circ-Kcnt2的免疫保护作用。另一项研究指出,INK4基因座可编码多个肿瘤抑制相关蛋白,而circANRIL及其亚型可通过募集polycomb蛋白抑制INK4蛋白的积累。适当地调整circANRIL及其亚型与polycomb蛋白的比例后,可使前者由INK4蛋白的抑制物转化为激活物,帮助遏制癌细胞的扩散[90]。
4.4 调控来源基因的转录水平
上述环状RNA的功能均处在转录后水平,关于环状RNA在转录过程中发挥作用的研究成果相对较少。
4.4.1动物环状RNA调控来源基因转录的最新进展
研究发现,特定的同时包含外显子和内含子的复合型环状RNA可通过结合U1蛋白来调控来源基因的转录效率[10]。另外,由SMARCA5基因(参与DNA的损伤修复)产生的环状RNA(circSMARCA5)可与来源基因SMARCA5的DNA序列结合并形成R-loop结构,最终致SMARCA5基因的15号外显子暂停转录[91]。该研究显示,无论将circSMARCA5在体外肿瘤组织中诱导还是在患病小鼠活体内过量表达,均能检测到乳腺癌细胞对药物的显著敏感性及癌细胞中SMARCA5转录本的下调。该结果为乳腺癌患者提供了全新的治疗靶点。
4.4.2植物环状RNA调控来源基因转录的主要成果
Conn等[13]更早地在植物体内发现环状RNA的R-loop依赖型转录抑制功能。拟南芥SEPALLATA3(SEP3)基因的第6个外显子形成的环状RNA可通过形成RNA:DNA杂合R-loop结构与SEP3基因的DNA序列紧密结合,最终暂停转录。该研究是环状RNA参与转录水平调控的第一项成就。随后,Cheng等[92]发现拟南芥基因AT5G37720的第1个内含子形成的套索型环状RNA也可竞争性调控来源基因的表达,同时影响拟南芥的生长发育。综合动植物环状RNA的研究进展可知,三大类环状RNA均可调控来源基因的转录效率。
最近,Philips等[93]为确定控制植物环状RNA转录的主效基因,选用了转录调控关键基因CBP80、C2H2和FLK的T-DNA插入式拟南芥突变体进行环状RNA的积累水平分析。结果发现,以上三种突变体体内积累的环状RNA均显著高于野生型;表明植物CBP80、C2H2及FLK基因是成熟mRNA外显子能够保持正确连接顺序所不可缺少的管控基因;缺失其中任意一个均可导致常规转录本的剪切失调并促使环状RNA的生成。由此可得,植物RNA的成环机制控制着线性转录本的积累。
5 环状RNA功能验证技术的研究述评
5.1 系统化的动物环状RNA功能验证方法
5.1.1反义寡核苷酸沉默技术
传统的RNA干扰载体无法产生环形转录本,为环状RNA特异性沉默增添了难度。反义寡核苷酸(Antisense oligonucleotide,ASO)是脱氧核苷酸短序列(15~20 nts)的类似物,可通过碱基互补配对结合目标RNA,最终依赖RNase H降低其表达水平[94]。Song等[95]将特定ASOs转染入果蝇体内,提前终止了目标基因的转录。并且,该研究在转染针对BS位点设计的环状RNA特异性ASOs时,实现了在沉默目标环状RNA的同时,不影响对应线性mRNA积累水平。因此,BS位点特异性ASOs在未来可应用于产生环状RNA基因座的功能研究。
5.1.2短发夹RNA敲除技术
短发夹RNA(Short hairpin RNA,ShRNA)可通过瞬时转染降低环状RNA的表达水平。有研究利用慢病毒转导法向小鼠眼窝皮层导入针对环状RNA的shRNA后,成功靶向敲除目标环状RNA(表达水平降低40%)[96]。但该方法效率较低,有脱靶风险。2020年,Pamudurti等[97]将shRNA技术进一步优化,在不改变来源基因mRNA积累水平的前提下,高效且特异性地沉默了果蝇的5个高表达环状RNA。
其他的环状RNA沉默技术也得以实现。如Xia等[98]通过敲除特定环状RNA的侧翼内含子序列,沉默了该环状RNA的转录。但该方法操作困难、效率低、不具广泛性、不可用于组织特异性分析且无法同时分析顺反式环状RNA的功能,仅适用于单个环状RNA的功能分析。随干扰技术的不断改进,有关环状RNA的研究成果将更加完善。
5.1.3MiRNA海绵的验证方法
动物领域已建立成熟的miRNA海绵验证体系,相关技术有:(1)利用环状RNA特异性探针对样品进行pull-down实验,分析沉淀成分;(2)对生物素化的野生型和miRNA突变体进行miRNA的pull-down实验,对比野生型和miRNA突变体内捕获环状RNA的情况;(3)将环状RNA与miRNA之间的互补区序列和突变碱基的互补区序列分别构建到荧光素报告酶载体中,与目标miRNA过表达载体分别共转染动物细胞,观察两组荧光信号强弱以确定互补区域的沉默效率;(4)利用荧光定量PCR技术分析环状RNA敲除前后的miRNA表达水平[42]。
5.1.4环状RNA翻译产物的验证方法
质谱分析、蛋白印迹技术和免疫荧光染色分析已应用于环状RNA蛋白产物的验证。质谱分析结合免疫共沉淀技术可确定环状RNA蛋白产物与其他蛋白之间的互作关系[66-69]。同时,低表达环状RNA的积累量几乎无法被检测到,验证它们的功能十分困难。Mo等[99]构建出一个内含子介导的增强系统,成功将环状RNA的表达水平提升5倍。该系统已识别部分先前未检测到的环状RNA,且发现它们具有翻译功能。此外,该作者还构建了一种多功能环状RNA表达载体——pCircRNA-DMo,未来将成为环状RNA生物发生和翻译产物研究的重要工具。
5.2 有待开发的植物环状RNA功能验证手段
5.2.1培育环状RNA过表达株系
植物环状RNA功能研究仍停留于表型水平。了解环状RNA的形成机制后,研究人员可根据特异性的侧翼序列实现人为构建促进成环的植物环状RNA过表达载体[25]。如已有研究成功在水稻的不同组织中过表达特定的环状RNA,结果发现该环状RNA的来源基因mRNA积累水平显著下降[26]。Zhang等[11]在拟南芥中过表达circGORK(来源基因功能为保卫细胞外流钾离子通道蛋白)后,转基因植株表现出ABA敏感性及耐旱性。Gao等[100]在拟南芥中过表达Vv-circATS1(来源基因功能为甘油-3-P酰基转移酶)后,转基因植株体现出耐寒性,然而将与Vv-circATS1序列相同的线状RNA转入拟南芥后,转基因株系并不耐寒。这表明植物环状RNA有区别于同源线性RNA的特异性功能。
5.2.2培育环状RNA敲除株系
培育植物环状RNA缺陷型株系也是其功能验证的重要手段。因仅需一段20 nts的特异性片段即可实现基因敲除,CRISPR/Cas9技术可用于植物环状RNA的功能研究。动物研究领域已利用CRISPR/Cas9技术成功将一个可吸附miRNA的环状RNA进行特异性敲除,培育环状RNA缺陷型突变体[101]。虽然CRISPR/Cas9已广泛应用于植物分子生物学研究,但其在植物环状RNA领域的应用还未见报道,相关细节仍需探索。
6 环状RNA作为生物标志物的应用前景
6.1 临床诊断生物标志物的设想与现实之间的差距
目前,临床仍缺乏有效检测癌细胞的生物标志物。环状RNA稳定且广泛存在于各类组织和细胞中,具备生物标志物的特性[102]。部分可作为食道癌和直肠癌候选生物标记物的环状RNA见表4(影响因子>4.0),结果初步体现了环状的临床应用价值。
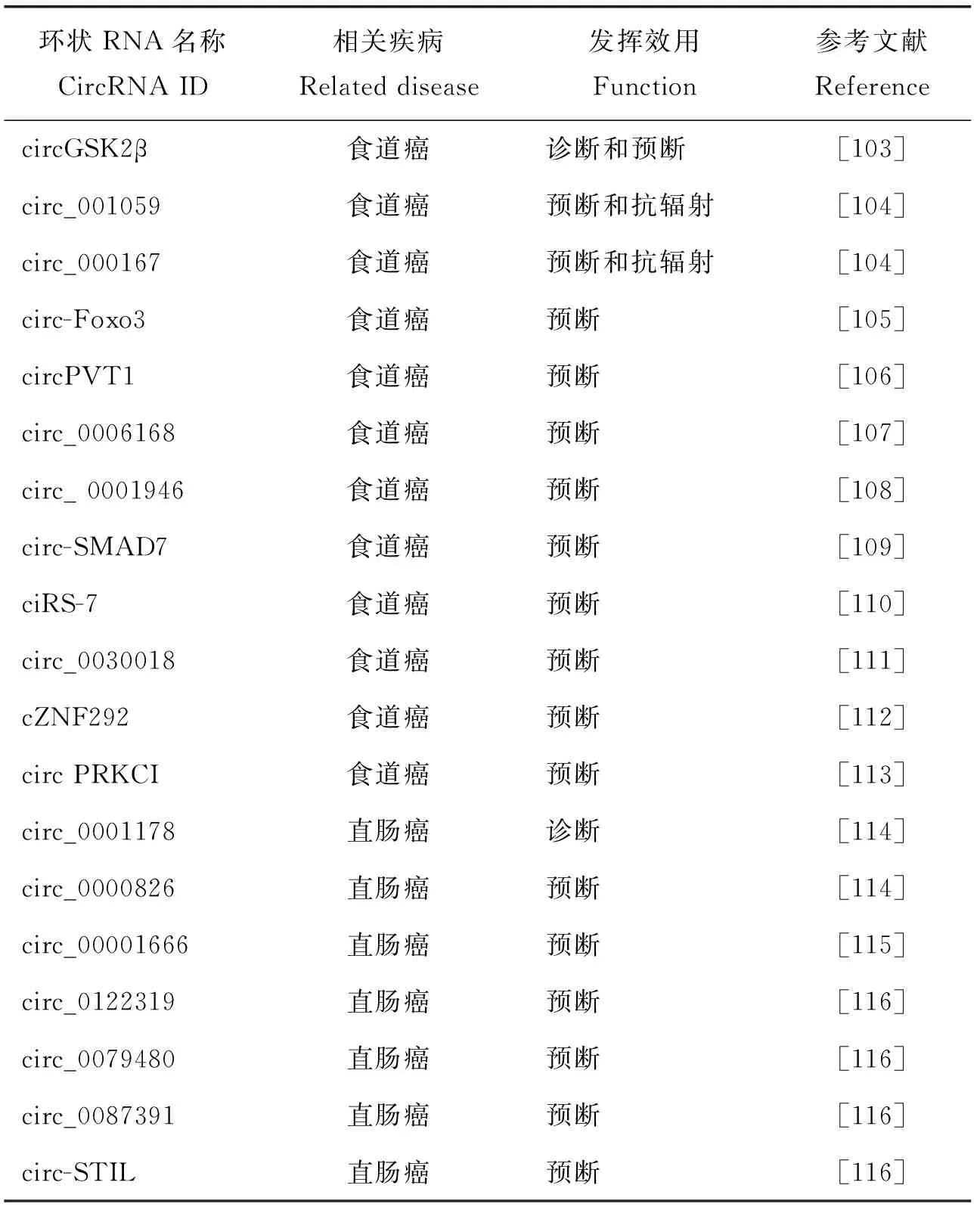
表4 诊疗食道癌和直肠癌的环状RNA候选生物标志物Table 4 Candidate circRNA biomarkers for diagnosis ofesophageal and rectal cancer
虽然,环状RNA作为临床生物标志物的优势明显。但Rochow等[118]提出,部分环状RNA在临床标本中并不稳定,也会经历类似mRNA降解的分解过程,诊断时不可单纯利用荧光定量PCR结果来判断患者组织内环状RNA的积累水平。因此,未来需要对环状RNA的降解机制进行深入探究,用以得到更可靠的环状RNA表达数据。
6.2 作物遗传育种生物标志物的新方向
生物标志物是作物遗传育种和实践应用的基础,其可预测作物产品的质量,评估植物对不同施肥条件的反应,是监测和优化肥料供给的潜在农艺工具[119]。虽然,植物环状RNA作为生物标记物仅为一个新的概念,但其具有半衰期长、不易降解、检测手段简便和序列特异性等优点。现已发现,由拟南芥SEP3基因产生的环状RNA可作为MADS-box相关基因AS事件的候选标志物[13]。今后植物环状RNA功能的研究重点应追随动物环状RNA的脚步,筛选可帮助推进作物遗传育种进程的生物标志物。
7 环状RNA研究的问题与展望
环状RNA普遍存在于真核生物体内,参与生命进程的各个阶段[120]。学者在描述环状RNA发挥功能时,相当于身处另一个“平行世界”,概括其调控能力既具广泛性又与线性RNA的作用机制完全不同[31]。虽然,有关环状RNA的信息仍存在大量空白,但其无疑已成为分子生物学研究领域划时代的标志。
癌症是威胁人类生命健康的障碍之首,而miRNA几乎在所有人类癌症细胞中差异表达。因此,环状RNA作为miRNA海绵的功能得到了临床研究的普遍关注。部分环状RNA可通过吸附miRNA调节肿瘤细胞的耐药性[121]。目前,将环状RNA列为候选的疾病诊断标志物所面临的一个重要问题为:一些功能性环状RNA具有丰富的空间表达特异性,在细胞的不同位置可发挥完全相反的作用。如位于癌细胞内的hsa_circ_0000338可抑制癌细胞扩散,但当其被分泌到细胞外时,hsa_circ_0000338可致癌[122]。因此,筛选环状RNA作为生物标记物的工作任重道远,更全面且稳定的检测数据是必不可少的。
与系统化的动物环状RNA研究相比,植物环状RNA的研究十分匮乏,多数还停留在测序和生物信息学预测阶段。因植物miRNA特殊的裂解机制,miRNA海绵功能的验证工作进展缓慢。另外,有关植物环状RNA的翻译及蛋白支架功能的研究也尚未发表。笔者建议,与动物环状RNA差异较大的生物发生机制及miRNA海绵功能均可成为植物环状RNA研究的切入点;通过借鉴动物环状RNA的研究手段;制定符合实际情况的实验路线;根据它们在不同发育阶段、逆境胁迫和激素刺激等条件的差异表达模式;由宏观筛选细化到单个环状RNA的功能验证;最终确定它们参与上述条件中发挥的具体作用。本文仅从内在分子发生机理到外在功能验证等方面综述了动植物环状RNA最新研究成果之间的特异性和同源性,旨为植物环状RNA的后续深入研究提供参考。
