全基因组蛋白质的标签细胞和小鼠资源库建设
2021-07-28蒋婧赵安麒谢甜陈淑藯李劲松
蒋婧,赵安麒,谢甜,陈淑藯,李劲松
资源与平台
全基因组蛋白质的标签细胞和小鼠资源库建设
蒋婧,赵安麒,谢甜,陈淑藯,李劲松
中国科学院分子细胞科学卓越创新中心(生物化学与细胞生物学研究所), 基因组标签计划研发中心,上海 200031
小鼠是最广泛使用的模式生物,构建基因改造的小鼠模型是研究基因功能和疾病发生机制的重要手段。从20世纪80年代发展至今,小鼠基因改造技术更新迭代,主要包括胚胎干细胞介导的同源重组策略、配子介导的转基因策略、以及最新基于CRISPR/Cas9技术的遗传改造策略等。由我国科学家自主研发的全新小鼠基因改造技术——“人造精子细胞”(或“类精子干细胞”)介导的半克隆技术与以往策略相比,具有小鼠构建周期短、效率高、成本低、应用场景兼容性强等特点。为了摆脱我国小鼠实验动物“卡脖子”风险,填补标签蛋白质小鼠资源库的国际空白,中国科学院分子细胞科学卓越创新中心进行了战略资源布局,于2017年启动了基因组标签计划(genome tagging project, GTP),并成立了GTP研发中心致力于建立全新的标签细胞和小鼠战略资源库。GTP研发中心通过对“人造精子细胞”基因组上编码蛋白质的基因原位系统性地添加标签序列,建立了可用液氮保存的标签细胞资源库,并作为生产标签小鼠的种子贮备。当需要标签小鼠时,利用半克隆技术将标签细胞注射到小鼠卵子中,一步法获得标签小鼠。标签小鼠可通过标准化的标签抗体监测特定蛋白质从而实现蛋白质的在体、实时、动态研究。目前,GTP研发中心已构建1532个标签细胞和277个标签小鼠,初步形成了具有特色标签产品战略资源库;已有242个标签小鼠品系被分发到3个囯家15个地区的32个研究机构,供66个研究团队使用。同时,GTP研发中心已建立完备的标签产品信息,定期在GTP官网上发布更新,供科研人员查询、订购和定制。后续标签小鼠的配繁规律、标签蛋白质组织图谱描绘、发表文献等也会陆续更新到GTP官网。GTP研发中心将对标行业尖端,打造全球独特性战略资源,为蛋白质功能研究提供标准化平台,推动生命科学的发展和临床转化。
基因组标签计划;“人造精子细胞”;半克隆技术;标签小鼠;战略资源
在生命科学研究领域,为揭示生命现象的普遍规律,科学家们往往需要借助模式生物来进行研究验证。其中,小鼠()是最佳且使用范围最广泛的模式动物。作为哺乳动物的代表,小鼠体型小、易饲养、世代周期短、繁殖力强、性成熟早、遗传操作简便、实验方便快捷,并且小鼠基因组计划已测序完成,基因组序列清晰,99%的人类蛋白质编码基因都有小鼠同源基因[1]。小鼠生理生化情况和发育进程也与人类相似,是研究人类基因功能、模拟人类疾病发病过程及对药物反应的优秀动物模型[2]。目前小鼠已在生命科学、医学研究和药物研发中被大量使用。利用PubMed数据库统计发现,在2000~2020年间,全球利用小鼠模型发表相关研究论文共484,076篇,呈现逐年递增趋势(图1)。
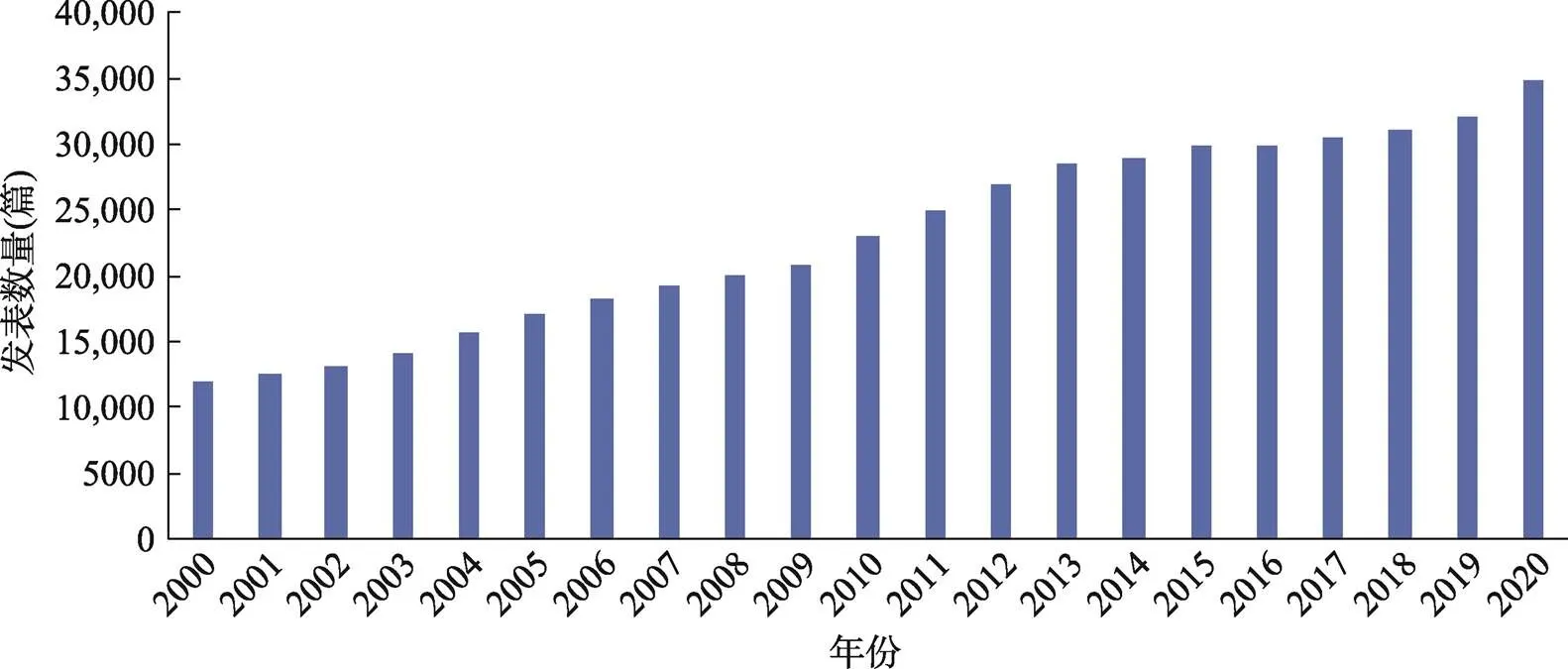
图1 统计2000~2020年以小鼠模型为研究材料发表的科研论文
小鼠成为最佳模式动物还受益于小鼠基因改造技术的成熟。20世纪七八十年代,利用显微操作技术进行的基因改造技术诞生[3~5]。1982年,第一个人工设计的表达大鼠生长激素基因的MGH (metallothionein-growth hormone)小鼠诞生,标志着转基因动物的开端[6]。随后,转基因技术不断地发展和成熟,形成了多种小鼠基因改造策略,主要包括胚胎干细胞介导的同源重组策略、配子介导的转基因策略等。由于早期改造策略构建的转基因小鼠转入的外源基因在基因组中随机整合,导致整合位点和插入拷贝数不确定等问题,需要不断对后代进行交配筛选才能获得蛋白表达稳定的小鼠。美国杰克逊实验室(The Jackson Laboratory,简称JAX)通过收录全美各家实验室蛋白表达稳定的转基因小鼠而发展成为全球最大的基因修饰小鼠品系供应商,我国科研工作者不得不花费大量经费从国外引进别人已报道的工具小鼠品系。近年来随着基因编辑技术的发展,尤其是CRISPR/Cas9基因编辑技术的发明,加速了整个基因改造领域的发展,小鼠资源库的全球格局也发生变化,呈现出小鼠模型标准化、精准化,供应规模化的特点。
在这关键发展时期,一种由我国科学家独立自主研发的全新小鼠基因改造技术——“人造精子细胞”介导的半克隆技术孕育而生,并入选为2012年“中国科学十大进展”[7,8]。为了摆脱我国小鼠实验动物“卡脖子”风险,中国科学院分子细胞科学卓越创新中心(Center for Excellence in Molecular Cell Science, CEMCS,原生物化学与细胞生物学研究所)进行了战略资源布局,于2017年启动了基因组标签计划(genome tagging project, GTP),成立了GTP研发中心来建立全新的标签细胞和小鼠战略资源库[9]。本文将对GTP研发中心标签产品资源库的技术优势、建设理念、建设情况以及对外服务等工作内容进行简要介绍,以增强研究者和公众的了解。
1 小鼠基因改造主流技术
早期受技术限制,基因敲入(knockin, KI)大多采用制备转基因技术小鼠来实现。但是由于转入的基因是随机整合的,导致产生整合位点和插入拷贝数不确定等问题,因此需要不断对后代进行交配筛选才能获得蛋白表达稳定的小鼠。另外随着交配传代,由于插入拷贝数稀释以及插入位点容易导致周围基因沉默等原因,首建鼠的表型容易在传代过程中丢失,给后期建系和重复实验带来诸多困难。并且,同一基因片段在不同位点插入的转基因小鼠品系可能会出现表型不一致的情况[2]。综上所述,筛选到具有稳定表型的转基因小鼠耗费时间长、效率低,严重影响获得易用工具小鼠的效率,这也是造成早期国内科研工作者需要花费大量经费从国外引进已报道的工具小鼠品系的主要原因。
CRISPR/Cas9基因编辑技术建立于2012年,通过sgRNA引导Cas9核酸酶对基因组进行剪切和编辑,从而能够在细胞和受精卵中直接进行高效的基因修饰,大幅提高了基因编辑效率,降低了基因编辑成本[10~12]。其简便性使得科研工作者可以根据自身科学研究需求来定制各种基因改造小鼠模型。CRISPR/Cas9基因编辑技术在基因组上的精准定位和高效改造,促使小鼠基因改造领域升级,呈现出小鼠模型标准化和精准化的特点。目前小鼠基因改造行业普遍使用的主流技术都借力于CRISPR/Cas9基因编辑技术,根据产生小鼠的方式不同,分为受精卵注射法和胚胎干细胞(embryonic stem cell, ES细胞)介导法。其中受精卵注射法由于其简便性,市场占比更高。
受精卵注射法是将sgRNA、Cas9核酸酶和同源重组片段利用显微注射技术直接注入小鼠受精卵中,从而获得阳性基因改造F0代小鼠[13]。由于CRISPR/ Cas9在小鼠受精卵发育过程中都可能进行基因编辑,经常会产生基因型嵌合的个体。因此该方法获得的F0代雄鼠需要与野生型雌鼠进行交配,从而筛选鉴定到稳定遗传的阳性F1小鼠。该方法主要用于基因敲除(knockout, KO)和点突变等基因修饰,基因改造效率高,生产周期短。该方法在获得F0小鼠后才能鉴定小鼠的基因型,并且由于CRISPR/Cas9切割随机修复后的序列会有所不同,因此筛选出序列确定的纯合子KO小鼠还需要花费较多工作量。另外由于注射大片段DNA的胚胎毒性高、同源重组效率低等特点,因此在插入片段大、同源重组效率低的KI基因改造上基本不采用该方法,而主要采用ES细胞介导法来进行。
ES细胞介导法建立于20世纪80年代[14~18]。现行的技术路径是先将sgRNA、Cas9核酸酶和同源重组质粒导入到小鼠ES细胞中,在细胞水平上进行同源重组,体外筛选阳性基因改造ES细胞。再将阳性ES细胞通过显微注射技术注入小鼠囊胚中,利用ES细胞的多能性获得嵌合体小鼠,最后通过交配繁殖传递给下一代,获得阳性基因改造小鼠。其中,仅有当ES细胞嵌合入生殖细胞,才能获得阳性小鼠。因此嵌合体的形成和生殖细胞传递是该方法的限速步骤。通常选用雄性ES细胞获得较高嵌合率(70%以上)的嵌合体雄鼠,与野生型雌鼠进行交配,从而在大量子代中鉴定筛选到阳性小鼠。该方法顺利的话也需要花费8个月到1年左右时间才能构建一个基因改造小鼠。该方法除了需要熟练掌握显微操作技术外,对ES细胞培养和打靶的要求也很高,所以目前只在插入片段大、同源重组效率低的KI基因修饰改造才使用。
2 GTP研发中心的技术优势
GTP研发中心采用的技术是我国科学家自主知识产权的“人造精子细胞”介导的半克隆技术。“人造精子细胞”,又称为孤雄单倍体胚胎干细胞(androgenetic haploid ESCs, AG-haESCs)或“类精子干细胞”,是从孤雄单倍体囊胚中建立的携带精子遗传物质的单倍体胚胎干细胞。将其注入到成熟的卵母细胞中, 能像精子一样支持重构胚胎的发育, 并产生健康的小鼠。孤雄单倍体胚胎干细胞注射入卵子的技术(intracytoplasmic AG-haESC injection, ICAHCI)被称为半克隆技术(semi-cloning technology)[7,8]。进而将半克隆技术结合CRISPR/Cas9的基因编辑能力,能高效获得基因改造小鼠[8]。
与ES细胞介导法相比,“人造精子细胞”同样是一种ES细胞,可在体外进行基因改造;并且其单倍体属性使得打靶效率比二倍体的ES细胞更高,能更好实现多个位点在同一套染色体上的改造。其次,“人造精子细胞”又具有精子的属性,可通过ICAHCI注射一步法获得基因改造小鼠,F0小鼠基因型全是阳性小鼠。由于“人造精子细胞”仅携带X染色体,无法携带Y染色体,所以该技术获得的F0全是阳性杂合雌鼠,并且无法对Y染色体进行基因编辑。但是因为半克隆技术小鼠出生率很高(~20%),一次ICAHCI注射即能获得较多F0雌鼠,所以这个缺陷能够通过超排4周龄F0雌鼠,并与野生型雄鼠进行IVF体外受精来弥补,从而比常规8周龄雄鼠交配能更快速获得杂合F1雄鼠和雌鼠。因此“人造精子细胞”介导的半克隆技术既拥有ES细胞介导法的体外编辑优势,又弥补了嵌合体小鼠的缺陷,能快速获得基因型确定的F1雄鼠和雌鼠。“人造精子细胞”同ES细胞相似培养要求高,并且还需要维持单倍体,技术瓶颈高,不易掌握和推广,需要通过建立专业化的技术服务平台来发挥其优势(表1)。
与受精卵注射法相比,首先“人造精子细胞”介导半克隆技术的阳性筛选步骤是在细胞水平,而不是小鼠水平。该步骤阳性筛选的成本就降低了许多。其次“人造精子细胞”作为中间媒介形成的资源库能像ES细胞库一样,体外进行大规模制备,而不受限于显微注射仪、小鼠笼位等资源限制,并且储存成本也低。再次,受精卵注射法构建的F0小鼠为基因型嵌合小鼠,需要交配才能获得基因型确定的F1小鼠。并且基因敲除的F0小鼠由于DNA的随机修复,每只小鼠的基因型序列不一致,也为后续鉴定和交配带来了许多麻烦。相比之下,半克隆技术获得的F0小鼠基因型确定,阳性率为100%;并利用快速扩繁的方式获得阳性F1小鼠,阳性率为50%,时间至少缩短1个月。最后,受精卵注射法在插入片段大、同源重组效率低的KI基因修饰改造上基本不采用,应用场景受限。而“人造精子细胞”介导的半克隆技术则无此限制,反而由于单倍体属性,高难度的多个位点改造更具优势(表1)。

表1 3种基因改造小鼠方法的比较
综上所述,“人造精子细胞”介导的半克隆技术相比主流的两种技术具有构建基因改造小鼠周期短、效率高、成本低、应用场景兼容性强等特点。并且“人造精子细胞”更利于全基因组大规模的改造和筛选,形成中间媒介的“人造精子细胞”资源库,作为种子战略资源生产和贮存的成本也低。当需要活体小鼠时再进行ICAHCI注射获得,可高效利用有限资源,节约科研经费。但该技术的技术瓶颈高,需要通过建立专业化的技术服务平台发挥竞争优势。
3 国内外主要小鼠资源库
JAX成立于1929年,是全球最大的基因修饰大小鼠品系供应商,主要收集全美各实验室小鼠品系,早期各种各样基因组随机插入的转基因小鼠多收录其中,因此小鼠品系繁多。截止2021年4月,JAX 在国际小鼠品系资源库(International Mouse Strain Resource,IMSR,http://www.findmice.org/repository)数据显示有11,465个小鼠品系(排第3,图2),其中有超600种Cre重组酶表达品系和近300种报告小鼠,并自主研发了22种高度免疫缺陷的人源化小鼠模型。JAX最大的亮点是严格质控保证小鼠质量,不计成本的细化所有品系的鉴定和繁殖工作,数据全球共享。因此经过90多年的扎实积累,使得JAX小鼠成为实验动物模型的行业标准,定量标识数据表征最完整,并被广泛使用。JAX不仅向全球提供小鼠资源,还致力于维护两大权威数据库:小鼠基因组信息学(Mouse Genome Informatics)和小鼠表型组数据库(Mouse Phenome Database),二者都被科研工作者广泛使用。

图2 各机构在IMSR上传小鼠资源的数目和占比
IMSR上拥有的小鼠品系最多的是美国突变小鼠区域资源中心(Mutant Mouse Regional Resource Centers, MMRRC),共有17,392个,其ES细胞数排第二,为42,614个。ES细胞数排第一的是美国德州农工大学基因组医学研究所,共142,543个。这表明这两家机构主要依靠传统方法在ES细胞上进行随机或目的突变来获得突变小鼠,因此有较高的ES细胞资源储量。另外,德国的欧洲突变小鼠存储库(European Mouse Mutant Archive, EMMA)有7119个小鼠品系(排第4,图2)。日本理化所生物资源中心(RIKEN BioResource Research Center, RBRC)有5505个小鼠品系(排第5,图2)。美国Taconic公司主要为欧洲和北美的制药和生物医学公司提供啮齿类实验动物模型及相关产品和服务,有2725个小鼠品系(排第7,图2)。美国Charles River Laboratories是全球知名的CRO公司和动物模型服务提供商,有56个小鼠品系(排第20,图2)。
国内主要有江苏集萃药康生物科技股份有限公司,成立于2017年,脱胎于国家遗传工程小鼠资源库,在IMSR数据显示有14,259个小鼠品系(排第2,图2)。该公司主打小鼠全基因组的条件性敲除CKO/KO模型制作,即“斑点鼠”计划,后还推出“免疫检查点人源化计划”及“人源肿瘤免疫资源库”,目标是发展成为全球最大的模式大小鼠资源供应商。利用CRISPR/Cas9受精卵注射法的简便性,集萃药康公司快速扩增小鼠全基因组的CKO/KO资源库,使其小鼠资源品系已赶超JAX。另外国内规模较大的还有上海南方模式生物科技股份有限公司,以用户定制服务为主,有5481个小鼠品系(排第6,图2)。
4 GTP研发中心的发展历程
在2017年5月的CEMCS头脑风暴会议上,基因组标签计划(GTP)被第一次提出,引起了与会专家的共鸣。蛋白质研究作为继人类基因组计划后广受关注的重要科学问题,功能研究进展缓慢,接近50%的蛋白质从未被研究,同时大量已知蛋白质缺乏揭示其功能的研究手段。如果能将全基因组蛋白质在模式动物体内带上容易识别的标签,就能使得追踪和研究蛋白质功能如同使用手机GPS在茫茫人海中进行对象识别和追踪一般简便、精准、高效[9]。GTP设想提出后,CEMCS就一直大力推动GTP的实施和发展,通过召开多场学术研讨会来探讨其可行性与重要性,并通过中国科学院战略性先导科技专项(B类)“细胞命运可塑性的分子基础与调控”(项目编号:XDB19010204)首席调控费拨付200万元用于支持开展50个蛋白质编码基因标签的尝试。上海市科学技术委员会对GTP高度重视,在听取相关介绍后,于2017年给予了第一期的资助。在此背景下,CEMCS于2017年11月专门成立GTP研发中心来实施GTP项目和生产GTP标签产品。由于GTP项目的迅速推进,于2019年再次获得上海市科委的资助。上海市I期项目《基于“人造精子”技术的重要基因标签研究》(项目编号:17411954900;执行期:2017.12~2019.6,2000万)的顺利完成证明大规模开展蛋白质标签的可行性;II期项目《基于基因组标签计划的蛋白质图谱研究》(项目编号:19411951800;执行期:2019.12~2020.11,1000万)的顺利完成表明GTP中试调试成功,标志着GTP在产业化方面又向前迈进了一大步。2019年5月23日,CEMCS在上海举办以“基因组标签计划(GTP)”为专题的香山科学会议(第S47次学术研讨会),共31家单位的50多位专家学者出席,会议围绕“GTP与基础生物学”和“GTP与精准医学”两个中心议题,进行了广泛而深入的讨论,就GTP实施的可行性、重要性以及潜在的应用前景达成了共识。
GTP研发中心利用“人造精子细胞”介导的半克隆技术来系统性在基因组上给编码蛋白质加上标签,建立液氮保存的标签细胞资源库。用户需要标签小鼠时,再复苏标签细胞,ICAHCI注射到卵子中一步法获得标签小鼠,从而建立标签小鼠资源库[9]。2018年5月GTP研发中心正式运营,建立了165 m2的独立实验室,用于标签细胞的生产和质量控制检测,已配备了齐全的仪器设备,如流式细胞分选仪、生物安全柜、荧光显微镜、PCR仪、DNA/蛋白电泳仪、DNA/蛋白成像仪等;建立了实验动物基地,用于标签小鼠的生产,共有3000个小鼠笼位,11间独立饲育室、1间胚胎移植室、1间显微注射室和4套显微操作注射仪。GTP研发中心已培养一批核心技术骨干15人,建立起成熟的技术方案、完善的标准化生产操作流程和信息化管理体系,确保GTP生产流程和产品质量可控。在3年逐步发展的过程中,这支短小精悍的队伍建立起1532个标签细胞资源库和277个标签小鼠资源库。
5 GTP标签产品战略资源库的建设
为了资源信息透明化,资源共享国际化,GTP研发中心建立了标签产品共享平台,为全球科研工作者提供有力的信息支撑,方便用户在线查询和订购。网址为http://www.sibcb.ac.cn/gtp/。官网上发布GTP研发中心已构建完毕的标签细胞和标签小鼠的资源库信息,详尽记录了每个标签细胞的标签插入位置、序列和标签蛋白质的表达情况,以及每个标签小鼠的基因型鉴定、发表文献等信息,方便研究者更好地了解和选择。
5.1 GTP标签细胞资源库
在GTP官网的标签细胞资源库模块下,点击单个标签细胞可以看到如下信息:(1)目的基因:统一为打靶构建时的NCBI基因名,随着后续NCBI数据库的更新,也会更换为最新基因名;(2) GTP编号:每种标签细胞生成唯一的GTP编号;(3)标签插入位点:通常为C端或N端,部分N端插入在信号肽后面,具体信息详见打靶质粒序列;(4)插入标签的类别:主要为HA标签,其他还有EGFP、mCherry、Flag等;(5)NCBI基因序列:统一为打靶构建时参考的NCBI mRNA序列,用以区分同一蛋白质的不同isoform形式;随着NCBI数据库的更新isoform信息可能会变化,但这一项信息仍然保持不变,用以定位该标签细胞的具体标签插入位点;(6)标签序列:可供下载插入的标签DNA序列;(7)同源重组质粒序列:可供下载标签细胞打靶所用同源重组质粒的序列,指示该标签细胞携带标签的位置和具体序列;(8) sgRNA序列:标签细胞打靶所选用的sgRNA序列;(9)标签细胞的Western blot (WB)结果:利用标签对应的抗体进行WB,显示在“人造精子细胞”中被标记蛋白质的表达情况,可直观分辨蛋白质分子量大小和表达强弱;值得注意的是如果被标记蛋白质本身不在“人造精子细胞”中表达,则无法被检测到;(10)标签细胞中被标记蛋白质的表达强弱:由于所有标签细胞都用少数几个对应的标签抗体来检测,所以可根据WB结果的曝光时长并对比抗体杂带的强弱程度来半定量蛋白质表达强度,共分为4种类别,定义为+++、++、+和–。
目前GTP标签细胞资源库的产品主要分为两大类:一类是根据客户需求提交订单来定制;二类是优先聚焦转录因子和重要信号通路因子等基因来进行标记,用于前沿科学研究。标签细胞由细胞生产技术员打靶完成后,会经过多轮质控确认无误后再发布到GTP官网。所有标签细胞经过跨同源臂PCR测序,排除转基因并核对DNA序列正确;标签细胞经过支原体检测为阴性;细胞水平蛋白质表达WB质控检测,评估蛋白质表达强度;细胞生产技术员将所有信息上传至GTP内网,并进行质粒和细胞入库;细胞主管审核所有细胞数据;最后由信息管理部统一进行信息审查,审查无误后再对外发布到GTP官网。
综上所述,GTP标签细胞资源库为自主研发建立,数据信息全,专业度高,数据更新快,产品质量高,并具有独特性、技术瓶颈高、不易被复制的特点,致力于发展成为我国标签细胞种子战略资源。
5.2 GTP标签小鼠资源库
为了充分利用“人造精子细胞”介导半克隆技术的优势,不浪费科研经费,仅有实际研究需求提交的订单才会安排标签小鼠的生产。标签小鼠生产与官网信息发布的流程为:复苏库存里液氮保存的标签细胞,进行ICAHCI注射,获得F0标签雌鼠;优化小鼠基因型鉴定引物,再次复核F0雌鼠基因型并测序验证;超排4周龄F0雌鼠与野生型雄鼠进行IVF体外受精,快速获得阳性杂合F1标签雄鼠和雌鼠;安排标签小鼠出库,并发送客户该标签小鼠名片;小鼠主管审核所有小鼠数据,确认无误后将信息上传至GTP内网;最后由信息管理部再次进行信息审查,审查无误后再发布到GTP官网。在GTP官网的标签小鼠资源库模块下,点击单个标签小鼠可以看到如下信息:标签细胞信息(1)~(10);(11)标签小鼠基因型鉴定信息和胶图:通常用两对PCR引物来区分纯合子、杂合子和野生型小鼠;(12)参考文献:记录使用该标签小鼠进行相关研究发表的研究论文,供用户参考。
除了以上严格生产流程外,GTP研发中心对标签小鼠资源库做了详尽的配套服务。首先,对所有标签小鼠都制作了名片,实现小鼠基因型鉴定的PCR条件全优化和PCR试剂全国产化。每张标签小鼠名片包含有:标签小鼠名称,GTP编号,基因型鉴定引物序列,测序引物序列,PCR反应体系和程序,PCR结果胶图与说明,并特殊标记X染色体上的基因。需要特别注意的是,当打靶基因位于X染色体时,其雄鼠鉴定结果仅能是纯合子或野生型。其次,GTP研发中心在标签小鼠资源库建立的过程中,不是一味地求多求快,而是每步稳扎稳打,对每个标签小鼠品系进行质量控制,掌握每个品系标签小鼠的繁殖规律。尽管杂合子标签小鼠已能满足实验需求,可通过检测标签蛋白质来反映内源目的蛋白质的时空表达、蛋白质相互作用网络等。但基于GTP的科学性,需要进一步明确标签蛋白质的插入是否会对小鼠机体造成影响,因此GTP研发中心将每个品系标签小鼠的繁殖规律一直记录到是否能由纯合子繁殖笼产生全纯合后代才终止。由于杂合小鼠繁殖笼交配获得纯合子的概率为25%,因此每个品系的纯合配繁都需要花费几个月的时间。经过细化和统计繁殖情况,在277个标签小鼠品系中,已成功配繁至全纯合终点的有134个品系,表明这134个内源蛋白质携带标签蛋白质后对其本身功能并不会产生影响,更加坚定了GTP的科学性。此项工作会持续进行,后续每个标签小鼠品系的繁殖规律也会细化更新到GTP官网上。
综上所述,GTP标签小鼠资源库同样为自主研发建立,数据信息全,专业度高,数据更新快,产品质量高,并具有独特性、技术瓶颈高、不易被复制的特点,致力于发展成为我国标签小鼠种子战略资源。
5.3 GTP标签蛋白质组织图谱数据库
基因组标签计划(GTP),不仅希望利用“人造精子细胞”介导半克隆技术为全基因组蛋白质打上相同标签,获得首创的标签细胞和标签小鼠战略资源库。还希望利用该战略资源做出0到1的科学研究突破,仅用一个标签抗体即可识别所有的蛋白质,开展基于标签抗体的蛋白质在体、实时、动态研究,促进生命活动规律及疾病发生发展机制的揭示,实现蛋白质研究的标准化。要实现这一过程,不仅依托于广大科研工作者使用GTP标签小鼠做出前沿科学研究,还要求GTP研发中心在这两大战略资源库上做出特色蛋白质数据库,而不只是个生产基地。随着标签战略资源库的逐步发展扩大,GTP研发中心一步步深入挖掘蛋白质信息。GTP标签细胞的蛋白质表达WB质控检测已经给我们第一层蛋白质信息,标签小鼠的组织图谱描绘能给我们第二层信息。
GTP研发中心利用标签抗体来系统性平行性描绘GTP标签蛋白质组织图谱。从标签小鼠上分离脑、肝、心脏、肾脏、肌肉、脾脏、肺、肠、胸腺、白色脂肪和睾丸11个器官组织,利用HA或其他标签抗体进行WB分析,共检测了227个标签小鼠品系,其中组织WB阳性为160个品系,阳性率70.48%。组织分布率见图3,睾丸和胸腺阳性比例最高,分别为82.5%和78.1%。由于目前GTP生产的标签小鼠主要为各个科学家研究迫切所需,具有偏好性,不具有广谱代表性,因此该数据仅供参考。组织WB检测阴性的标签小鼠,也不代表标记失败。部分由于蛋白质自身表达的时空特异性,不在我们检测的这11个成体器官组织中表达,又或者表达细胞类别少、表达丰度低等原因,需要根据每个蛋白质的属性在不同时期的器官组织中进一步分析。因此现阶段的组织WB阳性率仅是一个基本参考指标。标签小鼠的蛋白质组织表达WB质控检测阳性结果,表明该蛋白质在对应组织中表达丰度高。为了确保每个标签小鼠的标记正确性,GTP研发中心后续会与科学家保持密切沟通,或自行开发下游技术平台来进行验证,确保每个标签小鼠都能用于研究。此项工作首次大规模绘制小鼠蛋白质组织图谱,填补国内相关数据的空白,并会持续进行和升级,细化每个标签小鼠品系的蛋白质组织图谱更新到GTP官网上,为科学研究提供强有力的自主数据支持。

图3 标签小鼠在11个组织中的WB检测阳性分布图
从8周龄以上的雄性标签小鼠身上分离脑、肝、心脏、肾脏、肌肉、脾脏、肺、肠、胸腺、白色脂肪和睾丸11个器官组织,利用HA或其他对应标签抗体进行WB分析,其中组织WB结果出现阳性信号的共有160个品系。图中显示在这160个品系的标签小鼠中,携带标签的蛋白质分别在11个组织中内源性表达的分布情况及其比例。
6 技术开发与服务
经过3年的努力,GTP研发中心已建立了初具规模的特色标签产品战略资源库,定期在GTP官网上发布更新标签细胞和标签小鼠的资源库信息。科研人员可登陆官网,查询并订购感兴趣的标签产品。对于官网上无法搜索到的基因,GTP研发中心提供专属订制服务,为广大用户提供专业的个性化标签产品。
GTP研发中心的标签小鼠资源库,已有242个标签小鼠品系被分发到3个囯家15个地区的32个研究机构,供66个研究团队使用,为推动科学研究和临床应用转化作出了重大贡献。其中包含22个标签小鼠品系发货到国外3个研究机构的3个研究团队,开展国际合作,逐步扩大国际影响力。
基于“人造精子细胞”介导半克隆技术的强大拓展性,在其他基因改造小鼠的业务中具有很强优势,因此GTP研发中心也将加强市场宣传推广,接受各种基因改造小鼠业务订单。在服务中体现半克隆技术省时高效的优势,打好口碑,逐步拓展市场,丰富资源库类别,扩大自主资源库体量。实现以专业技术取胜,充分发挥本技术优势,聚焦高端用户的高难度模型小鼠构建,解决用户现有科研难题,推动基因改造小鼠行业发展,实现科学研究理论创新和技术突破。
7 结语与展望
GTP研发中心利用自主知识产权的“人造精子细胞”介导半克隆技术来大规模生产基因改造细胞和小鼠,具有技术首创性。并且建立了全新种子战略资源,主打标签细胞和标签小鼠战略资源,促进蛋白质功能研究,具有资源全球独特性。在3年的发展历程中,GTP研发中心已证明GTP技术的可行性和优越性,也在一步步以专业性打造全新蛋白质研究工具的战略资源库。JAX目前是全球最大的基因改造小鼠品系供应商,是全球实验小鼠“黄金标准”,拥有优秀遗传纯度和遗传稳定性,以丰富的基因型与表型数据享誉世界。在后续发展中,GTP研发中心将对标行业尖端,秉承“实事求是、独立自主、科技为本”的原则,积极努力走向产业化。在市场化运营过程中持续优化生产体系,降低成本,提高核心技术人员业务水平。同时吸收市场化企业的经验,提升创新积极性,增强产品市场竞争力,着实提高自身的行业竞争力,努力打造中国JAX。全力开发独有标签产品战略资源,优先支撑我国科学家进行创造性蛋白质组功能研究,将全基因组的蛋白研究提升到新高度,并作为关键基础提出重大科学问题,培育一系列前沿热点,加快国内科研队伍的建设,推动国际生命科学领域的发展,做出中国科学家的贡献,引领国际生命科学研究模式,带动一系列产业化平台建设和临床应用,最终改善人类健康。
[1] Mouse Genome Sequencing C, Waterston RH, Lindblad- Toh K, Birney E, Rogers J, Abril JF, Agarwal P, Agarwala R, Ainscough R, Alexandersson M, An P, Antonarakis SE, Attwood J, Baertsch R, Bailey J, Barlow K, Beck S, Berry E, Birren B, Bloom T, Bork P, Botcherby M, Bray N, Brent MR, Brown DG, Brown SD, Bult C, Burton J, Butler J, Campbell RD, Carninci P, Cawley S, Chiaromonte F, Chinwalla AT, Church DM, Clamp M, Clee C, Collins FS, Cook LL, Copley RR, Coulson A, Couronne O, Cuff J, Curwen V, Cutts T, Daly M, David R, Davies J, Delehaunty KD, Deri J, Dermitzakis ET, Dewey C, Dickens NJ, Diekhans M, Dodge S, Dubchak I, Dunn DM, Eddy SR, Elnitski L, Emes RD, Eswara P, Eyras E, Felsenfeld A, Fewell GA, Flicek P, Foley K, Frankel WN, Fulton LA, Fulton RS, Furey TS, Gage D, Gibbs RA, Glusman G, Gnerre S, Goldman N, Goodstadt L, Grafham D, Graves TA, Green ED, Gregory S, Guigó R, Guyer M, Hardison RC, Haussler D, Hayashizaki Y, Hillier LW, Hinrichs A, Hlavina W, Holzer T, Hsu F, Hua A, Hubbard T, Hunt A, Jackson I, Jaffe DB, Johnson LS, Jones M, Jones TA, Joy A, Kamal M, Karlsson EK, Karolchik D, Kasprzyk A, Kawai J, Keibler E, Kells C, Kent WJ, Kirby A, Kolbe DL, Korf I, Kucherlapati RS, Kulbokas EJ, Kulp D, Landers T, Leger JP, Leonard S, Letunic I, Levine R, Li J, Li M, Lloyd C, Lucas S, Ma B, Maglott DR, Mardis ER, Matthews L, Mauceli E, Mayer JH, McCarthy M, McCombie WR, McLaren S, McLay K, McPherson JD, Meldrim J, Meredith B, Mesirov JP, Miller W, Miner TL, Mongin E, Montgomery KT, Morgan M, Mott R, Mullikin JC, Muzny DM, Nash WE, Nelson JO, Nhan MN, Nicol R, Ning Z, Nusbaum C, O'Connor MJ, Okazaki Y, Oliver K, Overton-Larty E, Pachter L, Parra G, Pepin KH, Peterson J, Pevzner P, Plumb R, Pohl CS, Poliakov A, Ponce TC, Ponting CP, Potter S, Quail M, Reymond A, Roe BA, Roskin KM, Rubin EM, Rust AG, Santos R, Sapojnikov V, Schultz B, Schultz J, Schwartz MS, Schwartz S, Scott C, Seaman S, Searle S, Sharpe T, Sheridan A, Shownkeen R, Sims S, Singer JB, Slater G, Smit A, Smith DR, Spencer B, Stabenau A, Stange-Thomann N, Sugnet C, Suyama M, Tesler G, Thompson J, Torrents D, Trevaskis E, Tromp J, Ucla C, Ureta-Vidal A, Vinson JP, Von Niederhausern AC, Wade CM, Wall M, Weber RJ, Weiss RB, Wendl MC, West AP, Wetterstrand K, Wheeler R, Whelan S, Wierzbowski J, Willey D, Williams S, Wilson RK, Winter E, Worley KC, Wyman D, Yang S, Yang SP, Zdobnov EM, Zody MC, Lander ES. Initial sequencing and comparative analysis of the mouse genome.2002, 420(6915): 520–562.
[2] García-García MJ. A History of Mouse Genetics: From Fancy Mice to Mutations in Every Gene.2020, 1236: 1–38.
[3] Jaenisch R. Germ line integration and Mendelian transmission of the exogenous Moloney leukemia virus.1976, 73(4): 1260–1264.
[4] Gordon JW, Scangos GA, Plotkin DJ, Barbosa JA, Ruddle FH. Genetic transformation of mouse embryos by microinjection of purified DNA.1980, 77(12): 7380–7384.
[5] Gordon JW, Ruddle FH. Integration and Stable Germ Line Transmission of Genes Injected into Mouse Pronuclei.1981, 214(4526): 1244–1246.
[6] Palmiter RD, Brinster RL, Hammer RE, Trumbauer ME, Rosenfeld MG, Birnberg NC, Evans RM. Dramatic Growth of Mice That Develop from Eggs Micro-Injected with Metallothioneine-Growth Hormone Fusion Genes.1982, 300(5893): 611–615.
[7] Yang H, Shi LY, Wang BA, Liang D, Zhong CQ, Liu W, Nie YZ, Liu J, Zhao J, Gao X, Li DS, Xu GL, Li JS. Generation of genetically modified mice by oocyte injection of androgenetic haploid embryonic stem cells.2012, 149(3): 605–617.
[8] Zhong CQ, Yin Q, Xie ZF, Bai MZ, Dong R, Tang W, Xing YH, Zhang HL, Yang SM, Chen LL, Bartolomei MS, Ferguson-Smith A, Li DS, Yang L, Wu YX, Li JS. CRISPR-Cas9-Mediated Genetic Screening in Mice with Haploid Embryonic Stem Cells Carrying a Guide RNA Library.2015, 17(2): 221–232.
[9] Jiang J, Yan M, Li DS, Li JS. Genome tagging project: tag every protein in mice through 'artificial spermatids'.2019, 6(3): 394–396.
[10] Wiedenheft B, Sternberg SH, Doudna JA. RNA-guided genetic silencing systems in bacteria and archaea.2012, 482(7385): 331–338.
[11] Jinek M, Chylinski K, Fonfara I, Hauer M, Doudna JA, Charpentier E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity.2012, 337(6096): 816–821.
[12] Cong L, Ran FA, Cox D, Lin SL, Barretto R, Habib N, Hsu PD, Wu XB, Jiang WY, Marraffini LA, Zhang F.Multiplex Genome Engineering Using CRISPR/Cas Systems.2013, 339(6121): 819–823.
[13] Wu YX, Liang D, Wang YH, Bai MZ, Tang W, Bao SM, Yan ZQ, Li DS, Li JS. Correction of a Genetic Disease in Mouse via Use of CRISPR-Cas9.2013, 13(6): 659–662.
[14] Evans MJ, Kaufman MH. Establishment in culture of pluripotential cells from mouse embryos.1981, 292(5819): 154–156.
[15] Martin GR. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells.1981, 78(12): 7634–7638.
[16] Bradley A, Evans M, Kaufman MH, Robertson E. Formation of germ-line chimaeras from embryo-derived teratocarcinoma cell lines.1984, 309(5965): 255–256.
[17] Robertson E, Bradley A, Kuehn M, Evans M. Germ-Line Transmission of Genes Introduced into Cultured Pluripotential Cells by Retroviral Vector.1986, 323(6087): 445–448.
[18] Gossler A, Doetschman T, Korn R, Serfling E, Kemler R: Transgenesis by Means of Blastocyst-Derived Embryonic Stem-Cell Lines.1986, 83(23): 9065–9069.
Construction of genome-wide protein tagging cell and mouse libraries
Jing Jiang, Anqi Zhao, Tian Xie, Shuwei Chen, Jinsong Li
Mice are the most widely used model organism for the study of gene functions and disease mechanisms through the generation of gene-modified mice. Since the 1980s, different genetic manipulation technologies have been developed to reveal gene functions, including homologous recombination strategies mediated by embryonic stem cells, transgenic strategies mediated by gametes, and the latest genetic modification strategies based on CRISPR/Cas9 technology. Semi-cloning technology mediated by “artificial spermatids” (androgenetic haploid embryonic stem cells, also termed sperm-like stem cells) is developed by Chinese scientists in 2012. In combination with CRISPR/Cas9, semi-cloning technology enables one-step generation of gene-modified mice through injection of “artificial spermatids” with specific gene modifications into oocytes. It has the characteristics of short construction cycle, high efficiency, low cost, and high application compatibility. In 2017, the Center for Excellence in Molecular Cell Science (CEMCS) of CAS has launched the genome tagging project (GTP) based on “artificial spermatid”-mediated semi-cloning technology. The ambitious goal of GTP is to tag every protein in mice and construct a unique mouse library that maintains the genome-wide protein-tagging mouse models. Subsequently, the GTP center was established at CEMCS to pursue the project. GTP center developed strategies to generate protein-tagging cells and mice. Briefly, a tag sequence is precisely inserted in a specific protein- coding gene endogenously in cultured “artificial spermatids”to build a cell library, in which, each cell line carrying a specific protein tag. The tagged cells could be further used as a sperm replacement to produce tagged mice in one step upon injection into oocytes. The tagged mouse library enables global analysis of protein expression, localization, and complexes using standard tag-based assays. By April 2021, the GTP center has generated 1532 tagged cell lines, 277 of which have been successfully used to produce tagged mice through oocyte injection. A total of 242 tagged mouse strains have been distributed to 66 research teams in 32 research institutions of 15 districts in 3 countries. The database of tagging product resources has been established and released regularly on the GTP website for scientists to inquire and order. Later, more information about GTP products, such as mouse breeding, protein tissue expression map, published literature, etc., will also be successively published on the GTP website. The GTP center will provide a standardized platform for protein function research, which may dramatically promote the development of life science and clinical transformation.
genome tagging project; artificial spermatid; semi-cloning technology; protein tagging mouse; strategic resource
2021-04-29;
2021-06-07
上海市科学技术委员会科技创新行动计划项目(编号:17411954900和19411951800),中国科学院战略性先导科技专项(编号:XDB19010204)和国家自然科学基金项目(编号:31801057)资助[Supported by Shanghai Municipal Commission for Science and Technology Grants (Nos. 17411954900, 19411951800), the Strategic Priority Research Program of Chinese Academy of Sciences (No. XDB19010204), and the National Natural Science Foundation of China (No. 31801057)]
蒋婧,博士,副研究员,研究方向:基因组标签计划。E-mail: jiangjing@sibcb.ac.cn
10.16288/j.yczz.21-163
2021/6/25 13:30:57
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210625.1238.003.html
(责任编委: 徐璎)
