作物氮肥利用效率遗传改良研究进展
2021-07-28李姗黄允智刘学英傅向东
李姗,黄允智,刘学英,傅向东,3
优博专栏
作物氮肥利用效率遗传改良研究进展
李姗1,黄允智1,刘学英2,傅向东2,3
1. 南京农业大学,作物遗传与种质创新国家重点实验室,南京 210095 2. 中国科学院遗传与发育生物学研究所,植物细胞与染色体工程国家重点实验室,北京 100101 3. 中国科学院大学,北京 100049
氮素是植物生长发育所需的大量元素之一,施用氮肥是农业生产中提高农作物产量的重要手段。自20世纪60年代以来,“绿色革命”半矮秆农作物品种的育成和大面积推广有效地解决了“高产与倒伏”之间的矛盾,提高了农作物的收获指数和产量。然而半矮秆水稻和小麦品种也表现出生长发育对氮肥响应减弱、根系对铵态氮和硝态氮的吸收能力下降以及氮肥利用效率(nitrogen use efficiency, NUE)低的弊病,其产量增加依赖于氮肥的大量投入,这不仅提高了种植成本还导致了严重的环境污染问题。因此,提高农作物氮肥利用效率对于保障国家粮食安全和农业可持续发展具有重要战略意义。本文概述了“绿色革命”与赤霉素的作用机理,系统总结了植物氮素吸收、同化和代谢调控方面的研究进展,并介绍了提高作物氮肥利用效率的最新研究发现,以期为作物氮肥高效利用的遗传改良提供参考。
水稻;绿色革命;氮肥利用效率;可持续农业
自20世纪60年代以来,以半矮秆小麦(L.)和水稻(L.)等农作物品种的培育和推广为标志的“绿色革命”带来了全球粮食产量的飞跃,解决了由于人口快速增长而引发的粮食危机。水稻半矮秆基因()和小麦半矮秆基因()的应用解决了因密植和大量施肥而导致的倒伏问题,提高了收获指数,使作物单产大幅度提升。然而,半矮秆品种也伴随着生长发育对氮肥响应减弱、根系对氮素吸收能力下降以及氮肥利用效率(nitrogen use efficiency, NUE)降低等问题[1]。联合国粮食及农业组织(Food and Agriculture Organization of the United Nations, FAO)的数据显示,在过去10年中,世界范围的氮肥消耗量显著增加,但粮食产量增速缓慢,氮肥的过量投入不但没有大幅度地提高产量,反而导致了经济效益和生态效益的下滑。目前农田氮肥利用效率仅为40.2%,大部分氮肥在土壤中累积,并随着水土而流失进江河湖海或经过微生物反硝化作用排入大气,这不仅浪费了资源和能源,还引起了土壤酸化、大气和水体污染等一系列生态环境问题。因此,在保证产量不减的基础上如何提高氮肥的利用效率,减少氮肥的使用量是我国农业可持续发展亟待解决的重要问题。
1 “绿色革命”与赤霉素的作用机理
“绿色革命”的分子本质归结于赤霉素(gibberellic acid, GA)的生物学效应。小麦“绿色革命”基因编码赤霉素信号途径的负调控因子DELLA蛋白,育种中应用比较广泛的和均编码N端截断突变形式的DELLA蛋白[2],突变形式的DELLA蛋白本身不受GA诱导降解而积累,使小麦表现出半显性的、GA不敏感的半矮化表型[3]。水稻“绿色革命”基因位于1号染色体的长臂上,编码GA合成途径的GA20氧化酶2 (GA20ox2)。在籼稻生产上广泛应用的是该基因缺失了383 bp的突变类型,突变导致蛋白翻译提前终止,阻断了GA53至GA20的合成过程,降低了水稻内源活性GA的含量,使水稻DELLA蛋白SLR1 (SLENDER RICE 1)高水平积累,进而导致水稻株高降低[4,5]。
DELLA蛋白为植物特有的GRAS家族(由最初发现的3个家族成员GAI、RGA和SCR的特征字母命名)的一个亚家族,是GA信号途径的关键元件,在胞内起到阻遏植物生长发育的作用[6~8]。GA信号首先被赤霉素受体蛋白GID1 (GIBBERELLIN- INSENSITIVE DWARF1)感知并与之结合[9]。GID1中存在一个可以结合GA的口袋和一个可延展的N端区域[10],具有生物活性的GA与GID1结合后,GID1蛋白的N端区域就会盖住口袋,然后与DELLA蛋白的TVHYNP结构域互作形成GA-GID1-DELLA复合体[10,11],进一步使DELLA蛋白的GRAS结构域发生变化,增强DELLA蛋白与F-BOX蛋白(SCFSLY1/GID2)的互作强度[12],加速了DELLA蛋白的泛素化和26S蛋白酶体降解进程。植物生长阻遏蛋白DELLA被降解后,释放与其互作的转录因子,游离的转录因子启动GA信号途径相关基因的表达,进而促进植物的生长发育。DELLA蛋白N端的两个保守结构域DELLA和TVHYNP在拟南芥()、水稻、小麦、玉米(Linn.)和大麦(L.)等不同物种中高度保守。拟南芥基因组中的DELLA蛋白有5个成员,分别是GAI (GA-INSENSITIVE)、RGA (REPRESSOR OF)、RGL1 (RGA-LIKE1)、RGL2 (RGA-LIKE2)和RGL3 (RGA-LIKE3)。小麦绿色革命基因、玉米矮秆基因()、水稻和大麦()均为的直系同源基因[6,7,13,14]。
水稻生长调节因子OsGRF4 (growth-regulating factor 4)能与GIF1 (GRF-interacting factor 1)蛋白形成转录激活复合体,该复合体可以直接结合多个氮代谢相关基因的启动子,并促进基因表达,从而促进氮的吸收同化和转运。而DELLA蛋白能与OsGRF4互作,并抑制OsGRF4-GIF1复合体的形成,进而抑制了氮代谢相关基因的表达[15]。()是一个控制水稻分蘖响应土壤供氮水平变化的关键基因,其编码一个AP2 (APETALA2)结构域的转录因子。NGR5能够招募PRC2复合体(polycomb repressive complex)并通过介导组蛋白H3K27m3甲基化修饰来调控水稻分蘖相关基因的表达。此外,基因表达受氮素调控,并且NGR5蛋白还是GA-GID1促进的蛋白酶体降解的新靶标。DELLA能与NGR5蛋白互作并与之竞争性结合GID1蛋白来抑制NGR5降解,从而促进水稻分蘖增加[16]。
因此,“绿色革命”半矮秆的水稻和小麦因DELLA蛋白高水平积累而获得了3个方面的特性:第一,DELLA蛋白的积累因抑制植物的生长而降低了株高,提高了抗倒伏能力;第二,高水平的DELLA蛋白抑制了OsGRF4对下游氮响应因子的激活作用,降低了作物的氮肥利用效率;第三,高丰度的DELLA提高了NGR5蛋白的稳定性,促进了植物分蘖,提高了产量。因此,半矮秆的水稻、小麦品种在高水肥条件下能保持其半矮化、抗倒伏和多分蘖等高产特性,但同时也降低了作物的氮素响应和氮肥利用效率(图1)。
2 植物的氮素吸收、转运与同化
氮素是植物生长发育所必需的大量元素之一,是体内氨基酸和蛋白质的重要构成组分,其平均含量约占植物干重的1.5%。为了获取充足的氮源以满足生长发育的需要,植物根系需要从土壤中吸收不同形式的氮素,包括硝态氮(NO3−)、铵态氮(NH4+)、可溶性多肽和复杂的不溶性含氮化合物等,其中以吸收NO3−和NH4+为主。
2.1 NO3−的吸收和转运
在通气良好的旱田土壤中,植物主要以NO3−为氮源,植物根系吸收NO3−主要依靠硝酸盐转运蛋白(nitrate transporters, NRTs)来完成。从植物中分离鉴定出来的NO3−转运体大致分为4类:NRT1/PTR (nitrate transporter 1/peptide transport family)、NRT2 (nitrate transporter 2)、CLC (chloride channel)和SLAC1/SLAH (slow anion channel/slow anion channel homolog)。其中,大多数NRT1蛋白主要发挥低亲和力NO3−转运蛋白功能,但拟南芥AtNPF6.3/ AtNRT1.1、水稻OsNPF2.42和蒺藜苜蓿() MtNRT1.3例外;NRT2主要负责低浓度NO3−的转运;CLC蛋白部分成员是液泡膜定位的NO3−/H+的逆向转运蛋白,在液泡的NO3−积累方面发挥作用[17];SLAC1/SLAH家族蛋白定位在保卫细胞和根的中柱细胞,可能参与气孔的关闭和NO3−从根到茎的运输[17,18]。
在模式植物拟南芥中,有关NO3−吸收的分子机制研究比较深入。NRT1和PTR蛋白家族也被称为NPF (NRT1 PTR family)[19]。除AtNPF6.3/AtNRT1.1/ AtCHL1外,拟南芥NPF家族成员均属于低亲和力转运体。AtNPF6.3/AtNRT1.1/AtCHL1属于双亲和力转运体,在高浓度和低浓度的NO3−条件下均参与根系对NO3−的吸收,这主要取决于AtNRT1蛋白的第101位苏氨酸是否发生磷酸化。当T101位被磷酸化时,AtNPF6.3/AtNRT1.1作为高亲和力的NO3−转运体在低浓度NO3−的条件下发挥作用;当T101位没有被磷酸化时,AtNPF6.3/AtNRT1.1作为低亲和力的NO3−转运体在高浓度NO3−的条件下发挥作用[20],在此过程中蛋白激酶CIPK23 (calcineurin B-like interacting protein kinase 23)感应NO3−信号,能够对AtNRT1.1进行磷酸化修饰,进而调控AtNPF6.3/ AtNRT1.1对NO3−的亲和活性和吸收[21]。拟南芥NRT2家族至少有7个成员,其中6个成员发挥吸收NO3−的功能需要伴侣蛋白NAR2 (nitrate assimilation related protein 2)的存在,NRT2和NAR2家族成员均是NO3−高亲和力运输系统的组成部分[22,23]。
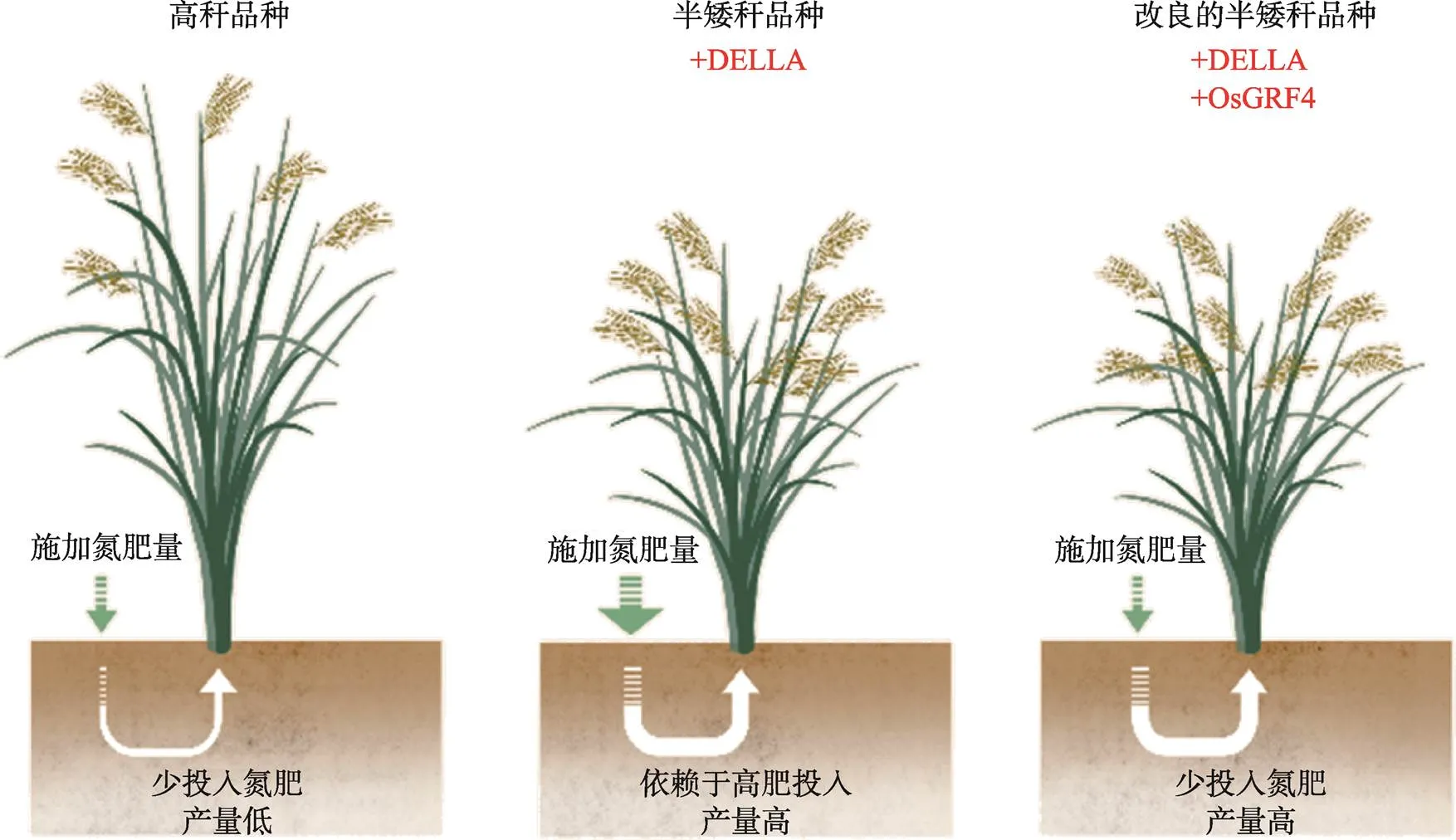
图1 新“绿色革命”品种协同提升氮肥利用效率与产量
在厌氧条件下土壤中的氮素多以NH4+形式存在,但由于根系周围发生的硝化作用,仍有大量的氮以NO3−的形式被作物吸收,因此NO3−对于水稻等喜铵作物也十分重要[24]。水稻中NO3−转运蛋白大致可以分为低亲和力转运体OsNRT1和高亲和力转运体OsNRT2两个家族,而OsNPF6.5 (OsNRT1.1B)和OsNPF6.1例外。编码双亲和的NO3−转运蛋白,受硝酸盐诱导表达,在高、低NO3−浓度下都会发挥吸收NO3−的功能,高表达后可以显著促进根系对NO3−的吸收,增加株高、产量和籽粒的氮积累[25]。此外,能影响根系富集的、具有氮转化能力的微生物组,从而改变根际微环境,进而影响水稻的氮肥利用效率[26]。OsNPF6.1也是一个双亲和硝酸根转运蛋白,定位于质膜,在根部高表达,在硝酸盐的吸收和再分配中发挥作用,并且受氮响应的转录因子OsNAC42的调控[27]。编码一个定位于液泡膜上的低亲和力NO3−转运蛋白,其表达不受硝酸盐的诱导,而受铵盐的诱导,主要参与细胞内硝酸盐、铵盐利用的调节[28]。在高浓度NO3−条件下低亲和力转运体OsNPF8.9 (OsNRT1.1) 和OsNPF2.4 (OsNRT1.6)发挥吸收NO3−的功能[29,30]。在低浓度NO3−条件下,NRT2家族成员的高亲和力转运蛋白OsNRT2.1、OsNRT2.2及其伴侣蛋白OsNAR2.1 起主导作用[31,32]。OsNRT2家族成员OsNRT2.3具有OsNRT2.3a和OsNRT2.3b两种不同的剪接形式。编码一个由516个氨基酸残基组成的膜定位蛋白,负责NO3−在韧皮部的长距离运输;编码一个由486个氨基酸残基组成的膜定位蛋白,在茎韧皮部高表达,在根部低表达,是一个pH敏感的NO3−转运蛋白,其发挥功能不需要OsNAR2.1的协助[33,34]。
2.2 NH4+的吸收和转运
在淹水环境中生长的植物主要以NH4+为氮源。NH4+的摄入主要由NH4+转运体家族(ammonium transporter/methylammonium permease/rhesus, AMT/ MEP/Rh)来完成。在植物中,NH4+转运体主要有AMT1和AMT2/MEP两个蛋白亚家族。目前研究表明,AMT家族成员在拟南芥中有6个、衣藻()中有8个、水稻中有10个、杨树(L.)中有14个[35,36]。
拟南芥的根系吸收NH4+主要由、、和四个基因负责,它们均编码定位于细胞质膜的转运蛋白,并在根表皮细胞高表达,在氮源缺乏或供应蔗糖的情况下,这4个基因的表达均上调[37,38]。这4个成员的NH4+吸收能力排序为:AtAMT1.1 = AtAMT1.3 > AtAMT1.2 > AtAMT1.5,其中AtAMT1.1、AtAMT1.2 和AtAMT1.3三个成员承担了拟南芥根系对NH4+吸收量的90%[39,40]。
水稻主要以吸收NH4+为主。目前发现水稻中至少有10个OsAMT成员,分为OsAMT1、OsAMT2、OsAMT3和OsAMT4四个家族[41]。其中OsAMT1、OsAMT2、OsAMT3各包含3个成员,OsAMT4只有1个成员。OsAMT1家族成员属于高亲和力NH4+转运体(high-affinity transport system, HATS)。在NH4+浓度较低时,高亲和力的铵转运体负责植物根系对NH4+的吸收,会表现饱和动力学特征;而当NH4+浓度较高(1~40 mmol/L)时,低亲和力NH4+转运体起主导作用,不会表现出饱和动力学特征[42]。在水稻根部和地上部组成型表达,其表达水平受底物积累的反馈调节;在根部特异性表达,其表达水平受NH4+的诱导;同样在根部特异性表达,其表达水平受高浓度铵的抑制。根部游离的谷氨酸盐抑制的表达,但可以促进和的表达[36]。
2.3 植物的氮素同化途径
植物根系吸收的不同形式的氮源需要经过一系列的同化作用才能被植物体利用。植物吸收的NO3−大部分被运送至地上部分进行同化与利用。首先在细胞质中,NO3−在硝酸还原酶(nitrate reductase, NR)的作用下被还原成NO2−,随后NO2−被运送至细胞质体中,在亚硝酸还原酶(nitrite reductase, NiR)的作用下转变成NH4+,最后NH4+在谷氨酰胺合成酶/谷氨酸合酶(glutamine synthetase/ferredoxin-glutamate synthase, GS/Fd-GOGAT)的作用下进入谷氨酸循环。首先GS催化NH4+与谷氨酸结合生成谷氨酰胺,随后谷氨酰胺与三羧酸循环的中间产物2-酮戊二酸(2-OG)在GOGAT催化作用下生成两个谷氨酸[43]。参与GS/GOGAT循环的酶主要是定位于叶绿体的GS2和Fd-GOGAT以及定位在细胞质中的GS1和NADH-GOGAT (nicotinamide adenine dinucleotide- glutamate synthase)。
水稻GS1家族有3个成员。对水稻的生长和灌浆非常重要,几乎在水稻的所有组织中都能检测到的表达,在叶片中表达最高。在根部特异性表达,不仅参与NH4+在根部的同化过程,还参与水稻对高温的响应;的突变体其分蘖数、穗粒数和千粒重均显著减少,总氨基酸、谷氨酸和天冬酰胺含量显著下降,表明其在根部的NH4+同化过程中起着重要作用[44]。主要在小穗中表达,并且和均不能补偿的功能[45]。主要在叶片的叶绿体中表达,在光呼吸作用释放出来的NH4+的再同化过程中起主导作用[46]。
由于NH4+对植物具有毒害作用,因此植物根系从土壤吸收的NH4+大部分在根部进行同化,然后主要以谷氨酸和谷氨酰胺的形式向地上部运输,在水稻中OsGS1.2和OsNADH-GOGAT1在这个过程中起主要作用。当外界NH4+浓度升高时,和在根部表皮细胞和外皮层细胞中的表达量会显著上升,从而将NH4+快速同化,生成的谷氨酰胺和谷氨酸被运输到地上部[47]。
3 植物的氮素代谢调控
氮代谢是植物生命活动中最基本的物质代谢过程之一。植物在长期的进化过程中,形成了一套复杂的、精细的氮素代谢调控网络来感知、响应氮素水平的动态变化以快速获取生长所需的氮营养。目前研究发现,植物的氮素代谢也受到多种转录因子、miRNA、植物激素等的共同调控。
3.1 转录因子调控植物氮代谢
转录因子是一类能识别基因上游启动子特异性基序,调控基因表达的蛋白质分子。研究表明,NAC (NAM, ATAF, CUC)、NLP (NIN-LIKE protein)、BTB (Bric-a-Brac/Tramtrack/Broad)、Dof (DNA-binding with one finger)、bZIP (basic leucine zipper)和MYB (v-Myb avian myeloblastosis viral oncogene homolog)等转录因子均参与植物氮代谢的调控过程。NAC是植物特有的一类转录因子。水稻基因表达受低氮诱导,OsNAC42蛋白可与启动子结合进而激活的表达[27]。NLP是一类调控氮诱导相关基因表达的转录因子[48],能够调控、()、和()等NO3−响应基因的表达。此外,Ca2+依赖蛋白激酶III亚族(subgroup III Ca2+- sensor protein kinases, CPKs) 中的成员CPK10/30/32能转导NO3−信号诱导产生的Ca2+浓度变化,通过磷酸化激活NLP蛋白的活性,组成NO3−-CPK-NLP信号传导网络,进而促进根系和叶片发育[49]。拟南芥/编码BTB家族蛋白,其表达受转录因子NLP调控。在低氮条件下,BT1/BT2通过负调控基因的表达来影响氮吸收[50]。Dof是植物特有的一类转录因子。水稻中OsDOF18能调控下游多个基因和氮同化基因的表达,从而调节NH4+的运输和氮分配[51]。bZIP转录因子在植物氮信号传导、氮代谢和碳–氮平衡过程中均发挥重要作用,如小麦TabZIP60负调控氮素利用[52]。水稻中的生长调控因子OsGRF4蛋白能够直接激活氮吸收和同化相关基因的表达,同时也能激活光合作用相关基因的表达,实现对植物碳–氮代谢平衡的调控[15]。MYB是一类包含Myb保守结构域、具有调控植物生长发育功能的转录因子。水稻MYB61是一种调节纤维素合成的转录因子,其基因表达受低氮诱导并受OsGRF4的调控,因此OsGRF4-MYB61信号通路将纤维素合成与氮响应代谢联系在一起[53]。Varala等[54]将机器学习引入到基于时间序列的氮反应转录组数据分析中,明确了氮信号相关转录因子的时序性调控关系,并发现146个能够响应氮信号的新转录因子。Gaudinier等[55]利用大规模酵母单杂交(yeast one- hybrid, Y1H)筛选调节氮代谢的转录因子,并结合大数据分析得到了一个氮代谢相关的酵母单杂交网络(yeast one-hybrid network for nitrogen-associated metabolism, YNM),并通过计算预测了在氮代谢中起关键作用的转录因子。这些研究为深入研究植物氮吸收、代谢调控网络提供了很大的帮助。
3.2 MicroRNA调控植物氮代谢
MicroRNA (miRNA)是在真核生物中发现的一类内源性的具有调控功能的非编码RNA,长度约为20~25 bp。miRNA对靶基因mRNA稳定性及其翻译过程的作用,是植物在转录后水平调控基因功能的重要方式之一[56]。
第一个将miRNA与氮素利用联系起来的基因是miRNA167靶标基因()。研究发现,在根的中柱鞘中,硝酸盐和谷氨酰胺/谷氨酸抑制的表达,从而增强的转录水平,最终导致侧生根数目增多[57]。()的表达受硝酸盐诱导,高表达能抑制主根生长而促进侧根生长,而氮的代谢物促进的表达,随后会抑制的表达[58]。和以及它们的下游基因形成了一个改变植物根系结构来响应氮信号的反馈机制。
在氮饥饿状态下,(,)家族成员的表达量上升,这是由于的表达下降引起的。在低氮条件下,在韧皮部的表达量下降,暗示着能够在植物体内的氮含量信号从茎至根的长距离传输过程中发挥作用[59]。过表达会抑制氮诱发的侧根生长,但能够通过增强磷转运基因的表达来提高作物的含磷量,这说明能够联系氮、磷信号通路[60]。磷饥饿能诱导的表达,进而降低靶基因()的表达,而NLA参与植物低氮逆境胁迫响应。在低氮条件下,与野生型相比,突变体中能积累更多的磷[61]。因此,miRNA不仅通过改变植物根系构型和氮信号的长距离传输,还通过与磷信号通路互作来调节植物根系对氮的响应。
3.2 植物激素信号与氮信号的互作
环境中的氮源还是植物生长发育和响应环境胁迫的重要信号物质。许多氮代谢与激素信号转导相关的最新发现都将氮信号与植物激素信号联系在一起。
生长素(Indole-3-acetic acid, IAA)的极性运输是由顶部运送至基部,促进侧根的起始和发育。因此,IAA一直被认为是介导氮信号从地上部传递至地下部的媒介[62]。NRT1.1可以通过调节硝酸盐依赖的生长素运输和硝酸盐信号途径来调控侧根的生长。模拟磷酸化的AtNPF6.3/AtNRT1.1T101D的突变表现出较强生长素运输调节能力,促进生长素快速横向流动,而非磷酸化的AtNPF6.3/AtNRT1.1T101A的突变表现出较弱的生长素运输调节能力,导致生长素在根尖积累,表明低NO3−诱导的T101磷酸化影响了生长素运输从而调节生长素介导的侧根生长[63]。NLP7能诱导生长素生物合成基因()和生长素外排基因()的表达[49]。在低氮条件下,突变体在侧根原基中的生长素积累减少,侧根的形成受到抑制[64]。Gaudinier等[55]构建的酵母单杂交网络表明生长素响应因子ARF9能调控PNR (primary nitrate responses)基因(例如和())的表达;ARF18能调控下游氮代谢基因和的表达。在水稻中,()编码一个负调控生长素合成的酶,其表达水平受外界氮供应的调控从而改变植物体内的生长素含量,进而影响生长素响应因子OsARF6和OsARF17对下游氮代谢相关基因的调控作用,最终影响植物的氮代谢[65]。
脱落酸(abscisic acid, ABA)被认为是一种参与植物响应胁迫的应激激素。外界高浓度的氮素能够抑制拟南芥侧根发育,ABA信号途径可能参与其中[66]。ABA不敏感突变体(、、)和ABA合成缺失突变体(、、和)的侧根生长对高浓度NO3−的抑制作用不敏感。在ABA信号转导关键调节因子ABI2的突变体中,NO3−诱导表达和侧根生长的效应减弱,说明ABI2在NO3−信号传递途径中发挥作用[21]。赤霉素信号途径的DELLA蛋白调控植物生长发育对氮肥的响应。一方面,DELLA蛋白可以与OsGRF4互作,抑制OsGRF4-GIF1转录激活复合体的形成,从而抑制OsGRF4对下游氮代谢基因的激活作用,降低作物的氮吸收能力[15];另一方面,DELLA蛋白结合GID1蛋白抑制氮响应因子NGR5降解,而NGR5能促进植物分蘖,调控植株在高氮水平下的分蘖响应。氮响应负调控因子LBD可以直接结合在启动子上调控的表达,而OsTCP19作为转录因子可以抑制油菜素内酯(brassinosteroids, BRs)信号途径中的关键组分()的表达,从而调控水稻分蘖。LBD-OsTCP19-DLT通路证明了BR信号途径直接参与氮调控植物生长发育的过程[67]。茉莉酸甲酯(methyl jasmonate, MeJA)处理使水稻根系中和的转录水平和蛋白质水平均降低,并且根系中的NO3−和NH4+转运相关基因的表达水平显著下降,抑制了根中氮素的吸收和同化,并且MeJA 还会导致质体分解和谷氨酸脱氢酶上调,促进叶片中氮的再利用[68]。
4 提高作物氮肥利用效率
提高作物品种的氮肥利用效率,减少氮肥投入和环境污染已成为农业可持续发展中一个艰巨的挑战。影响作物氮利用效率的主要因素包括两个部分:氮吸收效率(nitrogen uptake efficiency, NUpE)和氮利用效率(nitrogen utilization efficiency, NUtE)。氮吸收效率是指成熟农作物地上部分的含氮量与土壤中含氮量的比值;氮利用效率是指单株粒重与成熟农作物地上部分的含氮量之比[69]。一般来说,在高氮条件下,氮吸收效率与氮效率的关联较大,而在低氮条件下,氮利用效率与氮效率的关联性较大[70]。
提高氮的吸收、同化效率是提高作物氮效率最有效的途径。例如,高表达能够增加作物产量并提高NUE,因此,将该基因表达水平较高的籼稻等位基因导入到表达水平较低的粳稻品种中,可以显著改良作物的NUE[25]。籼稻类型基因编码具有更强活性的硝酸还原酶NR2,将籼型等位基因导入到粳稻品种中能有效地提高分蘖数、产量和NUE[71]。野生稻来源的优异等位基因OsNPF6.1能提高氮吸收和NUE,并在低氮条件下提高水稻的产量[27]。过表达和均可以提高水稻的产量和NUE[28,33];使用的启动子驱动基因在根和茎中特异性高表达可以显著提高水稻的NUE和谷物产量[72]。过表达后能够促进侧根的形成,提高水稻NUE和产量[73]。和过表达后能增加水稻的分蘖、提高NUE和产量[74,75]。通过增加亚硝酸还原酶基因启动子中的硝酸盐反应顺式元件(nitrate-responsive cis-element, NRE)可以显著地增强其氮同化的功能[76]。使用拟南芥的启动子驱动一个由和嵌合产生的硝酸盐转运蛋白基因的表达可以激活植物体内硝酸盐的再利用,从而提高NUE和产量[77]。在水稻中过量表达能够增强水稻根系对NH4+的吸收,在低浓度的NH4+环境下能促进水稻生长,提高NUE和产量;但在高浓度的NH4+环境下植株会表现出铵中毒[78]。过量表达可以在高氮条件下协同提高水稻的NUE和产量[79],但也有研究表明过量表达GS后会破坏碳–氮代谢平衡,从而影响水稻的生长和产量[44]。
NUE是一个复杂性状,受植物生命周期中不同的代谢、发育和环境信号网络的协同控制。因此,仅仅通过提高植物的氮素吸收和同化来提高NUE存在着局限性,而通过优化植物氮素响应、代谢信号网络,系统性协调植物的生长发育和代谢过程来提高作物NUE是一个更加理想的策略。()基因编码的植物G蛋白的γ亚基,其通过与水稻Gα亚基(RGA1)和Gβ亚基(RGB1)互作共同调控植物的氮响应。显性等位基因可使植物在营养生长期的地上部生长发育对氮不敏感,但能增强植物根系的氮吸收和同化能力,提高氮肥利用效率,进而提高收获指数和产量[80]。MYB61是联系碳–氮代谢的关键节点,受到OsGRF4的调控,其启动子区域存在一个helitron转座子插入的籼粳分化,将不含转座子插入的、籼稻来源的导入到粳稻品种中可以同时提高作物的纤维素合成水平和NUE,并在低氮条件下促进增产[53]。编码一个叶绿体蛋白,其功能缺失突变可延迟植物衰老,在低氮条件下提高NUE,携带启动子小片段插入的低表达的突变类型可在低氮条件下提高NUE和产量[81]。能够响应外界的氮信号,通过改变生长素的水平来调控氮代谢,启动子存在520 bp的片段插入/缺失的籼粳差异,将片段缺失型低表达的籼稻等位基因导入到粳稻中可以显著提高其NUE和产量[65]。启动子在不同品种中存在29 bp的插入/缺失,缺失型的启动子可以使氮响应负调控因子LBD蛋白高效地结合在该位点并抑制的表达,从而促进水稻的分蘖发育。而在我国现代水稻品种中这一氮高效的变异类型几乎全部丢失,将该等位基因导入现代水稻品种中可以在少施氮肥的条件下显著提高水稻的NUE[67]。DELLA蛋白是“绿色革命”的关键蛋白,其在作物中的高积累量赋予了“绿色革命”品种半矮化、抗倒伏、高产、低NUE等特性,其效应与关键作用因子OsGRF4和NGR5密切相关。的优异等位基因GRF4能显著增加自身的转录水平和蛋白水平,使得DELLA-OsGRF4平衡向OsGRF4丰度增加倾斜,可在不增加作物株高的情况下协同提高作物的光合作用和氮肥利用效率[15];能响应土壤的氮素水平变化,正向调控水稻的生长发育,提高的表达能够保持其半矮秆、耐倒伏、多分蘖等高产特性的同时,提升水稻的NUE;在“绿色革命”品种中聚合应用和优异等位基因可以进一步提高水稻产量和NUE[16]。
5 结语与展望
氮肥是促进作物生长发育和产量提高的重要因素。通过遗传改良培育氮肥高效利用的农作物新品种对农业可持续发展至关重要。利用各种种质资源材料和遗传材料,深入挖掘氮肥利用和吸收效率相关的关键基因、解析其遗传调控网络,并将优异等位基因用于育种,是培育氮高效品种的重要途径。近年来,随着功能基因组学的不断发展,植物的氮吸收、代谢及其信号转导和调控网络得到了不断的完善,越来越多具有改良作物NUE和提升产量潜力的基因被发现,但如何结合植物生长和代谢平衡的调控网络,将这些优良的基因应用到育种实践中,培育出“少投入、多产出、环境友好”的资源高效型作物品种还需要很大努力。
总之,随着氮素吸收、代谢和响应的信号传导分子机制的不断解析,必将极大地促进农作物NUE和产量的协同改良、推动新一轮的“绿色革命”。
[1] Pingali PL. Green Revolution: Impacts, limits, and the path ahead.,2012, 109(31): 12302–12308.
[2] Van De Velde K, Thomas SG, Heyse F, Kaspar R, Van Der Straeten D, Rohde A. N-terminal truncated RHT-1 proteins generated by translational reinitiation cause semi-dwarfing of wheat Green Revolution alleles., 2021, 14(4): 679–687.
[3] Peng J, Richards DE, Hartley NM, Murphy GP, Devos KM, Flintham JE, Beales J, Fish LJ, Worland AJ, Pelica F, Sudhakar D, Christou P, Snape JW, Gale MD, Harberd NP. 'Green revolution' genes encode mutant gibberellin response modulators., 1999, 400(6741): 256–261.
[4] Sasaki A, Ashikari M, Ueguchi-Tanaka M, Itoh H, Nishimura A, Swapan D, Ishiyama K, Saito T, Kobayashi M, Khush GS, Kitano H, Matsuoka M. Green revolution: a mutant gibberellin-synthesis gene in rice., 2002, 416(6882): 701–702.
[5] Zhang QF. Strategies for developing green super rice., 2007, 104(42): 16402–16409.
[6] Chandler PM, Marion-Poll A, Ellis M, Gubler F. Mutants at thelocus of barley cv Himalaya. Molecular and physiological characterization., 2002, 129(1): 181–190.
[7] Ikeda A, Ueguchi-Tanaka M, Sonoda Y, Kitano H, Koshioka M, Futsuhara Y, Matsuoka M, Yamaguchi J., a constitutive gibberellin response mutant, is caused by a null mutation of thegene, an ortholog of the height-regulating gene., 2001, 13(5): 999–1010.
[8] Ogawa M, Kusano T, Katsumi M, Sano H. Rice gibberellin-insensitive gene homolog,, encodes a nuclear-localized protein capable of gene activation at transcriptional level., 2000, 245(1): 21–29.
[9] Ueguchi-Tanaka M, Ashikari M, Nakajima M, Itoh H, Katoh E, Kobayashi M, Chow TY, Hsing YIC, Kitano H, Yamaguchi I, Matsuoka M.encodes a soluble receptor for gibberellin., 2005, 437(7059): 693–698.
[10] Shimada A, Ueguchi-Tanaka M, Nakatsu T, Nakajima M, Naoe Y, Ohmiya H, Kato H, Matsuoka M. Structural basis for gibberellin recognition by its receptor GID1., 2008, 456(7221): 520–523.
[11] Ueguchi-Tanaka M, Nakajima M, Katoh E, Ohmiya H, Asano K, Saji S, Hongyu X, Ashikari M, Kitano H, Yamaguchi I, Matsuoka M. Molecular interactions of a soluble gibberellin receptor, GID1, with a rice DELLA protein, SLR1, and gibberellin.,2007, 19(7): 2140–2155.
[12] Hirano K, Asano K, Tsuji H, Kawamura M, Mori H, Kitano H, Ueguchi-Tanaka M, Matsuoka M. Characterization of the molecular mechanism underlying gibberellin perception complex formation in rice., 2010, 22(8): 2680–2696.
[13] Fu X, Richards DE, Ait-Ali T, Hynes LW, Ougham H, Peng J, Harberd NP. Gibberellin-mediated proteasome- dependent degradation of the barley DELLA protein SLN1 repressor., 2002, 14(12): 3191–3200.
[14] Peng J, Carol P, Richards DE, King KE, Cowling RJ, Murphy GP, Harberd NP. Thegene defines a signaling pathway that negatively regulates gibberellin responses., 1997, 11(23): 3194– 3205.
[15] Li S, Tian YH, Wu K, Ye YF, Yu JP, Zhang JQ, Liu Q, Hu MY, Li H, Tong YP, Harberd NP, Fu XD. Modulating plant growth–metabolism coordination for sustainable agriculture., 2018, 560(7720): 595–600.
[16] Wu K, Wang SS, Song WZ, Zhang JQ, Wang Y, Liu Q, Yu JP, Ye YF, Li S, Chen JF, Zhao Y, Wang J, Wu XK, Wang MY, Zhang YJ, Liu BM, Wu YJ, Harberd NP, Fu XD. Enhanced sustainable green revolution yield via nitrogen-responsive chromatin modulation in rice., 2020, 367(6478): eaaz2046.
[17] Bergsdorf EY, Zdebik AA, Jentsch TJ. Residues important for nitrate/proton coupling in plant and mammalian CLC transporters.,2009, 284(17): 11184–11193.
[18] MacMillan J. Occurrence of gibberellins in vascular plants, fungi, and bacteria.2001, 20(4): 387–442.
[19] Léran S, Varala K, Boyer JC, Chiurazzi M, Crawford N, Daniel-Vedele F, David L, Dickstein R, Fernandez E, Forde B, Gassmann W, Geiger D, Gojon A, Gong JM, Halkier BA, Harris JM, Hedrich R, Limami AM, Rentsch D, Seo M, Tsay YF, Zhang MY, Coruzzi G, Lacombe B. A unified nomenclature of NITRATE TRANSPORTER 1/PEPTIDE TRANSPORTER family members in plants., 2014, 19(1): 5–9.
[20] Liu KH, Tsay YF. Switching between the two action modes of the dual-affinity nitrate transporter CHL1 by phosphorylation.. 2003, 22(5): 1005–1013.
[21] Léran S, Edel KH, Pervent M, Hashimoto K, Corratgé- Faillie C, Offenborn JN, Tillard P, Gojon A, Kudla J, Lacombe B. Nitrate sensing and uptake inare enhanced by ABI2, a phosphatase inactivated by the stress hormone abscisic acid., 2015, 8(375): ra43.
[22] Kiba T, Feria-Bourrellier AB, Lafouge F, Lezhneva L, Boutet-Mercey S, Orsel M, Bréhaut V, Miller A, Daniel-Vedele F, Sakakibara H, Krapp A. Thenitrate transporter NRT2.4 plays a double role in roots and shoots of nitrogen-starved plants., 2012, 24(1): 245–258.
[23] Kotur Z, Mackenzie N, Ramesh S, Tyerman SD, Kaiser BN, Glass ADM. Nitrate transport capacity of theNRT2 family members and their interactions with AtNAR2.1., 2012, 194(3): 724–731.
[24] Li YL, Fan XR, Shen QR. The relationship between rhizosphere nitrification and nitrogen-use efficiency in rice plants., 2008, 31(1): 73–85.
[25] Hu B, Wang W, Ou SJ, Tang JY, Li H, Che RH, Zhang ZH, Chai XY, Wang HR, Wang YQ, Liang CZ, Liu LC, Piao ZZ, Deng QY, Deng K, Xu C, Liang Y, Zhang LH, Li LG, Chu CC. Variation incontributes to nitrate-use divergence between rice subspecies., 2015, 47(7): 834–838.
[26] Zhang JY, Liu YX, Zhang N, Hu B, Jin T, Xu HR, Qin Y, Yan PX, Zhang XN, Guo XN, Hui J, Cao SY, Wang X, Wang C, Wang H, Qu BY, Fan GY, Yuan LX, Garrido-Oter R, Chu CC, Bai Y.is associated with root microbiota composition and nitrogen use in field-grown rice., 2019, 37(6): 676–684.
[27] Tang WJ, Ye J, Yao XM, Zhao PZ, Xuan W, Tian YL, Zhang YY, Xu S, An HZ, Chen GM, Yu J, Wu W, Ge YW, Liu XL, Li J, Zhang HZ, Zhao YQ, Yang B, Jiang XZ, Peng C, Zhou C, Terzaghi W, Wang CM, Wan JM. Genome-wide associated study identifies NAC42-activated nitrate transporter conferring high nitrogen use efficiency in rice., 2019, 10(1): 5279.
[28] Wang W, Hu B, Yuan DY, Liu YQ, Che RH, Hu YC, Ou SJ,Liu Y, Zhang ZH, Wang HR, Li H, Jiang ZM, Zhang ZL, Gao XK, Qiu YH, Meng XB, Liu YX, Bai Y, Liang Y, Wang YQ, Zhang LH, Li LG, Sodmergen, Jing HC, Li JY, Chu CC. Expression of the nitrate transporter geneconfers high yield and early maturation in rice., 2018,30(3): 638–651.
[29] Li YG, Ouyang J, Wang YY, Hu R, Xia KF, Duan J, Wang YQ, Tsay YF, Zhang MY. Disruption of the rice nitrate transporter OsNPF2.2 hinders root-to-shoot nitrate transport and vascular development., 2015, 5(1): 9635.
[30] Lin CM, Koh S, Stacey G, Yu SM, Lin TY, Tsay YF. Cloning and functional characterization of a constitutively expressed nitrate transporter gene,, from rice., 2000, 122(2): 379–388.
[31] Feng HM, Yan M, Fan XR, Li BZ, Shen QR, Miller AJ, Xu GH. Spatial expression and regulation of rice high-affinity nitrate transporters by nitrogen and carbon status., 2011, 62(7): 2319–2332.
[32] Liu XQ, Huang DM, Tao JY, Miller AJ, Fan XR, Xu GH. Identification and functional assay of the interaction motifs in the partner protein OsNAR2.1 of the two-component system for high-affinity nitrate transport., 2014, 204(1): 74–80.
[33] Fan XR, Tang Z, Tan YW, Zhang Y, Luo BB, Yang M, Lian XM, Shen QR, Miller AJ, Xu GH. Overexpression of a pH-sensitive nitrate transporter in rice increases crop yields., 2016, 113(26): 7118– 7123.
[34] Feng HM, Li B, Zhi Y, Chen JG, Li R, Xia XD, Xu GH, Fan XR. Overexpression of the nitrate transporter, OsNRT2.3b, improves rice phosphorus uptake and translocation., 2017, 36(8): 1287–1296.
[35] González-Ballester D, Camargo A, Fernández E. Ammonium transporter genes in Chlamydomonas: the nitrate-specific regulatory geneis involved inexpression., 2004, 56(6): 863–878.
[36] Sonoda Y, Ikeda A, Saiki S, von Wirén N, Yamaya T, Yamaguchi J. Distinct expression and function of three ammonium transporter genes () in rice., 2003, 44(7):726–734.
[37] Lejay L, Gansel X, Cerezo M, Tillard P, Müller C, Krapp A, von Wirén N, Daniel-Vedele F, Gojon A. Regulation of root ion transporters by photosynthesis: functional importance and relation with hexokinase., 2003,15(9): 2218–2232.
[38] Gazzarrini S, Lejay L, Gojon A, Ninnemann O, Frommer WB, von Wirén N. Three functional transporters for constitutive, diurnally regulated, and starvation-induced uptake of ammonium intoroots., 1999, 11(5): 937–948.
[39] Sohlenkamp C, Shelden M, Howitt S, Udvardi M. Characterization ofAtAMT2, a novel ammonium transporter in plants., 2000, 467(2–3): 273–278.
[40] Yuan LX, Loqué D, Kojima S, Rauch S,Ishiyama K, Inoue E, Takahashi H, von Wirén N. The organization of high-affinity ammonium uptake inroots depends on the spatial arrangement and biochemical properties of AMT1-type transporters., 2007, 19(8): 2636–2652.
[41] Li C, Tang Z, Wei J, Qu HY, Xie YJ, Xu GH. The OsAMT1.1 gene functions in ammonium uptake and ammonium-potassium homeostasis over low and high ammonium concentration ranges., 2016, 43(11): 639–649.
[42] Gaur VS, Singh US, Gupta AK, Kumar A. Understanding the differential nitrogen sensing mechanism in rice genotypes through expression analysis of high and low affinity ammonium transporter genes., 2012, 39(3): 2233–2241.
[43] Masclaux-Daubresse C, Daniel-Vedele F, Dechorgnat J, Chardon F, Gaufichon L, Suzuki A. Nitrogen uptake, assimilation and remobilization in plants: challenges for sustainable and productive agriculture., 2010, 105(7): 1141–1157.
[44] Bao A, Zhao ZQ, Ding GD, Shi L, Xu FS, Cai HM. Accumulated expression level of cytosolic glutamine synthetase 1 gene (or) alter plant development and the carbon-nitrogen metabolic status in rice., 2014, 9(4): e95581.
[45] Tabuchi M, Sugiyama K, Ishiyama K, Inoue E, Sato T, Takahashi H, Yamaya T. Severe reduction in growth rate and grain filling of rice mutants lacking OsGS1;1, a cytosolic glutamine synthetase1;1., 2005, 42(5): 641–651.
[46] Wallsgrove RM, Turner JC, Hall NP, Kendall AC, Bright SW. Barley mutants lacking chloroplast glutamine synthetase-biochemical and genetic analysis., 1987, 83(1): 155–158.
[47] Tamura W, Kojima S, Toyokawa A, Watanabe H, Tabuchi- Kobayashi M, Hayakawa T, Yamaya T. Disruption of a novel NADH-Glutamate synthase2 gene caused marked reduction in spikelet number of rice.,2011, 2: 57.
[48] Konishi M, Yanagisawa S.NIN-like transcription factors have a central role in nitrate signalling., 2013, 4:1617.
[49] Liu KH, Niu YJ, Konishi M, Wu Y, Du H, Sun Chung H, Li L, Boudsocq M, McCormack M, Maekawa S, Ishida T, Zhang C, Shokat K, Yanagisawa S, Sheen J. Discovery of nitrate-CPK-NLP signalling in central nutrient-growth networks., 2017, 545(7654): 311–316.
[50] Araus V, Vidal EA, Puelma T, Alamos S, Mieulet D, Guiderdoni E, Gutierrez RA. Members of BTB gene family of scaffold proteins suppress nitrate uptake and nitrogen use efficiency., 2016, 171(2): 1523–1532.
[51] Wu YF, Yang WZ, Wei JH, Yoon H, An G. Transcription factor OsDOF18 controls ammonium uptake by inducing ammonium transporters in rice roots., 2017, 40(3): 178–185.
[52] Yang J, Wang M, Li W, He X, Teng W, Ma W, Zhao X, Hu M, Li H, Zhang Y, Tong Y. Reducing expression of a nitrate-responsive bZIP transcription factor increases grain yield and N use in wheat., 2019, 17(9): 1823–1833.
[53] Gao YH, Xu ZP, Zhang LJ, Li SC, Wang SG, Yang HL, Liu XL, Zeng DL, Liu QQ, Qian Q, Zhang BC, Zhou YH. MYB61 is regulated by GRF4 and promotes nitrogen utilization and biomass production in rice., 2020, 11(1): 5219.
[54] Varala K, Marshallcolón A, Cirrone J, Brooks MD, Pasquino AV, Léran S, Mittal S, Rock TM, Edwards MB, Kim GJ, Ruffel S, Mccombie WR, Shasha D, Coruzzi GM. Temporal transcriptional logic of dynamic regulatory networks underlying nitrogen signaling and use in plants., 2018, 115(25): 6494–6499.
[55] Gaudinier A, Rodriguez-Medina J, Zhang L, Olson A, Liseron-Monfils C, Bågman AM, Foret J, Abbitt S, Tang M, Li B, Runcie DE, Kliebenstein DJ, Shen B, Frank MJ, Ware D, Brady SM. Transcriptional regulation of nitrogen-associated metabolism and growth., 2018, 563(7730): 259–264.
[56] Ha M, Kim VN. Regulation of microRNA biogenesis., 2014, 15(8): 509–524.
[57] Gifford ML, Dean A, Gutierrez RA, Coruzzi GM, Birnbaum KD. Cell-specific nitrogen responses mediate developmental plasticity., 2008, 105(2): 803–808.
[58] Vidal EA, Araus V, Lu C, Parry G, Green PJ, Coruzzi GM, Gutiérrez RA. Nitrate-responsive miR393/regulatory module controls root system architecture in.,2010, 107(9): 4477– 4482.
[59] Pant BD, Musialak-Lange M, Nuc P, May P, Buhtz A, Kehr J, Walther D, Scheible WR. Identification of nutrient-responsiveand rapeseed microRNAs by comprehensive real-time polymerase chain reaction profiling and small RNA sequencing., 2009, 150(3): 1541–1555.
[60] Yan YS, Wang HC, Hamera S, Chen XY, Fang RX.miR444a has multiple functions in the rice nitrate- signaling pathway., 2014, 78(1): 44–55.
[61] Lin WY, Lin YY, Chiang SF, Syu C, Hsieh LC, Chiou TJ.Evolution of microRNA827 targeting in the plant kingdom., 2018, 217(4): 1712–1725.
[62] Fukaki H, Tasaka M. Hormone interactions during lateral root formation., 2009, 69(4): 437–449.
[63] Zhang X, Cui YN, Yu M, Su BD, Gong W, Baluška F, Komis G, Šamaj J, Shan XY, Lin JX. Phosphorylation- mediated dynamics of nitrate transceptor NRT1.1 regulate auxin flux and nitrate signaling in lateral root growth., 2019, 181(2): 480–498.
[64] Ma WY, Li JJ, Qu BY, He X, Zhao XQ, Li B, Fu XD, Tong YP. Auxin biosynthetic geneis involved in low nitrogen-mediated reprogramming of root architecture in., 2014, 78(1): 70–79.
[65] Zhang SY, Zhu LM, Shen CB, Ji Z, Zhang HP, Zhang T, Li Y, Yu JP, Yang N, He YB, Tian YN, Wu K, Wu JY, Harberd NP, Zhao YD, Fu XD, Wang SK, Li S. Natural allelic variation in a modulator of auxin homeostasis improves grain yield and nitrogen use efficiency in rice., 2021, 33(3): 566–580.
[66] Signora L, De Smet I, Foyer CH, Zhang H. ABA plays a central role in mediating the regulatory effects of nitrate on root branching in., 2001, 28(6): 655–662.
[67] Liu YQ, Wang HR, Jiang ZM, Wang W, Xu RN, Wang QH, Zhang ZH, Li AF, Liang Y, Ou SJ, Liu XJ, Cao SY, Tong HN, Wang YH, Zhou F, Liao H, Hu B, Chu CC. Genomic basis of geographical adaptation to soil nitrogen in rice.. 2021, 590(7847): 600–605.
[68] Wu XY, Ding CH, Baerson SR, Lian FZ, Lin XH, Zhang L Q, Wu CF, Hwang SY, Zeng R, Song YY. The roles of jasmonate (JA) signaling in nitrogen uptake and allocation in rice (L.)., 2019, 42(2): 659–672.
[69] Moll RH, Kamprath EJ, Jackson WA. Analysis and interpretation of factors which contribute to efficiency of nitrogen utilization., 1982, 74(3): 562–564.
[70] Sasakawa H, Yamamoto Y. Comparison of the uptake of nitrate and ammonium by rice seedlings influences of light, temperature, oxygen concentration, exogenous sucrose, and metabolic inhibitors., 1978, 62(4): 665–669.
[71] Gao ZY, Wang YF, Chen G, Zhang AP, Yang SL, Shang LG, Wang DY, Ruan BP, Liu CL, Jiang HZ, Dong GJ, Zhu L, Hu J, Zhang GH, Zeng DL, Guo LB, Xu GH, Teng S, Harberd NP, Qian Q.The indica nitrate reductase geneallele enhances rice yield potential and nitrogen use efficiency., 2019, 10(1): 5207.
[72] Chen JG, Zhang Y, Tan YW, Zhang M, Zhu LL, Xu GH, Fan XR. Agronomic nitrogen-use efficiency of rice can be increased by drivingexpression with the OsNAR2.1 promoter., 2016, 14(8): 1705–1715.
[73] Fang ZM, Xia KF, Yang X, Grotemeyer MS, Meier S, Rentsch D, Xu XL, Zhang MY. Altered expression of the PTR/NRT1 homologue OsPTR9 affects nitrogen utilization efficiency, growth and grain yield in rice., 2013, 11(4): 446–458.
[74] Fang ZM, Bai GX, Huang WT, Wang ZX, Wang XL, Zhang MY. The rice peptide transporter OsNPF7.3 is induced by organic nitrogen, and contributes to nitrogen allocation and grain yield., 2017, 8:1338.
[75] Huang WT, Bai GX, Wang J, Zhu W, Zeng QS, Lu K, Sun SY, Fang ZM. Two Splicing Variants of OsNPF7.7 Regulate shoot branching and nitrogen utilization efficiency in rice., 2018, 9:300.
[76] Yu J, Xuan W, Tian YL, Fan L, Sun J, Tang WJ, Chen GM, Wang BX, Liu Y, Wu W, Liu XL, Jiang XZ, Zhou C, Dai ZY, Xu DY, Wang CM, Wan JM. Enhanced OsNLP4- OsNiR cascade confers nitrogen use efficiency by promoting tiller number in rice., 2021, 19(1): 167–176.
[77] Chen KE, Chen HY, Tseng CS, Tsay YF. Improving nitrogen use efficiency by manipulating nitrate remobilization in plants., 2020, (9): 1126–1135.
[78] Ranathunge K, El-Kereamy A, Gidda S, Bi YM, Rothstein SJ.transgenic rice plants with enhanced NH4+permeability show superior growth and higher yield under optimal and suboptimal NH4+conditions., 2014, 65(4): 965–979.
[79] Brauer EK, Rochon A, Bi YM, Bozzo GG, Rothstein SJ, Shelp BJ. Reappraisal of nitrogen use efficiency in rice overexpressing glutamine synthetase1., 2011, 141(4): 361–372.
[80] Sun HY, Qian Q, Wu K, Luo JJ, Wang SS, Zhang CW, Ma YF, Liu Q, Huang XZ, Yuan QB, Han RX, Zhao M, Dong GJ, Guo LB, Zhu XD, Gou ZH, Wang W, Wu YJ, Lin HX, Fu XD. Heterotrimeric G proteins regulate nitrogen-use efficiency in rice., 2014, 46(6): 652–656.
[81] Wang Q, Nian JQ, Xie XZ, Yu H, Zhang J, Bai JT, Dong GJ, Hu J, Bai B, Chen LC, Xie QJ, Feng J, Yang XL, Peng JL, Chen F, Qian Q, Li JY, Zuo JR. Genetic variations inmediate grain yield by modulating nitrogen utilization in rice., 2018, 9(1): 735.
Genetic improvement of nitrogen use efficiency in crops
Shan Li1, Yunzhi Huang1, Xueying Liu2, Xiangdong Fu2,3
Nitrogen (N) is an essential mineral nutrient for plant growth and development. N deficiency is the major factor limiting plant growth and crop production in most natural and agricultural soils. The green revolution of the 1960’s boosted crop yields through cultivation of semi-dwarf plant varieties. However, green revolution wheat and rice varieties have relatively poor nitrogen use efficiency (NUE), require a high N fertilizer supply to achieve maximum yield potential, and this leads to an increase in production costs and environmental problem. Therefore, a major challenge for sustainable agriculture is whether improvement of NUE through the reduction of N fertilizer supply can be achieved without yield penalty. In this review, we summarize the recent advances in understanding of molecular mechanisms underlying the regulation of N-responsive plant growth, utilization and possibility for improvements of NUE in crops, and new breeding strategies through modulation of N-responsive growth-metabolism coordination for future sustainable agriculture.
rice; green revolution; nitrogen use efficiency; sustainable agriculture

李姗,2012—2018年就读于中国科学院遗传与发育生物学研究所,在傅向东课题组攻读博士学位,目前任南京农业大学农学院教授,研究方向:不同植物激素互作调控水稻氮肥利用效率的分子机制。博士期间,主要研究了赤霉素信号转导途径调控水稻氮肥高效利用的分子机制,揭示了半矮秆基因导致作物氮肥利用效率降低的原因,阐明了DELLA-OsGRF4模块调控氮肥利用效率的分子机制,为提高“绿色革命”半矮秆品种氮肥利用效率提供了新策略。博士论文《提高水稻产量和氮肥利用效率的分子机制研究》获得2020年中国科学院优秀博士生论文。
2021-02-18;
2021-04-02
国家自然科学基金项目(编号:31830082, 91935301)和江苏省自然科学基金项目(编号:BK20200540)资助[Supported by the National Natural Science Foundation of China (Nos. 31830082, 91935301) and the Natural Science Foundation of Jiangsu Province (No. BK20200540)]
李姗,博士,教授,研究方向:植物激素调控水稻养分高效利用。E-mail: shanli@njau.edu.cn
黄允智,在读博士研究生,研究方向:植物激素调控水稻养分高效利用。E-mail: 2020201051@stu.njau.edu.cn
李姗和黄允智并列第一作者。
傅向东,研究员,博士生导师,研究方向:植物发育和环境适应的激素调控机理。E-mail: xdfu@genetics.ac.cn
10.16288/j.yczz.21-064
2021/4/26 15:01:23
URI: https://kns.cnki.net/kcms/detail/11.1913.R.20210425.1639.002.html
(责任编委: 储成才)
