人工DNA合成技术:DNA数据存储的基石
2021-07-21黄小罗戴俊彪
黄小罗,戴俊彪
(中国科学院深圳先进技术研究院,深圳合成生物学创新研究院,广东省合成基因组学重点实验室,深圳市合成基因组学重点实验室,广东 深圳 518055)

互联网、人工智能、大数据领域的发展催生了全球数据的指数级增长。以目前的发展趋势,传统的数据存储介质,比如硬盘、磁带、光盘,将无法应对不断增长的数据需求。DNA作为一种存储介质,具有存储密度高、存储时间长、占地面积小、维护成本低等诸多优点,是解决未来数据存储危机最有潜力的介质之一[1-2]。自2012年Church等[3]发表基于DNA介质的下一代数据存储研究开始,越来越多的研究工作开始聚焦在这一领域。目前已发表的DNA数据存储工作中[3-6],其流程大致包括如下几个主要的步骤:①数据编码,从存储在计算机上的数据中提取二进制0/1信息,并通过设计的0/1二进制信息到A/T/C/G序列的映射关系,将0/1二进制信息转换为A/T/C/G碱基序列;②DNA合成,利用人工DNA合成技术将碱基序列合成为可以被保存的DNA多聚物分子;③DNA测序,利用测序技术读取合成的DNA多聚分子的碱基序列;④数据恢复,利用步骤①中设计的0/1二进制信息到A/T/C/G序列的映射关系,将步骤③中获得的DNA序列转换为0/1二进制信息,并进一步转换成为存储的数据。人工DNA合成技术作为DNA存储流程中的核心技术,是DNA数据存储从概念走向应用的关键。
人工DNA合成技术,是在不依赖DNA模板的情况下,根据人为设计的任意序列进行DNA合成的技术。1953年,沃森和克里克[7]发现了DNA双螺旋结构,使得人们对DNA分子的认知实现了一个里程碑式的跨越。自此开始,一批化学家和生物学家们就开始了人工DNA合成的研究。目前的人工DNA合成技术,不仅能够合成几十到数百个碱基的寡聚核苷酸,而且能够通过化学合成、结合酶法拼接及微生物克隆等方法,合成Mb级别的微生物基因组。DNA合成也从实验室走向了商业化。GE Healthcare(美国)、Biolytic(美国)、BioAutomation(美国)等公司推出一系列不同通量的合成仪;提供DNA合成服务的公司也逐渐发展起来,包括IDT(美国)、GenScript(美国/中国)、GeneWiz(美国)、Twist Bioscience(美国)等。这些公司也在DNA合成技术开发、成本降低以及支撑DNA合成下游应用方面做出了重要贡献。
除DNA数据存储外,人工合成DNA在医药、农业、材料等多个合成生物学支撑的领域都发挥着重要的作用。在新冠肺炎[8]、乙肝[9]、埃博拉病毒病[10]等多种公共传染疾病的防控上,人工合成DNA探针被用作核酸检测试剂盒的关键原料。在医药开发上,人工合成DNA极大加速了生物抗体药改造[11]、基因治疗[12-13]、寡核苷酸药物[14]等开发过程,是这些药物应用不可或缺的技术手段。在农业育种上,人工合成DNA可以用于转基因农作物品种的改造,如将人工改造合成的来源于苏云金芽孢杆菌杀虫蛋白Cry的编码基因转化到农作物体内[15],产生了抗虫棉、抗虫稻等系列抗虫品种。在材料领域,基于人工合成DNA制备的DNA纳米机器人被报道成功地将凝血酶带到肿瘤细胞,杀死肿瘤细胞[16]。DNA人工合成技术对合成生物学领域的支撑作用,堪比测序技术对基因组学和精准医学领域的贡献,是合成生物学发展的关键技术。
本文总结了目前人工DNA合成的关键技术研究进展,包括寡核苷酸合成、基因合成、基因组合成以及新一代酶法DNA合成等。与此同时,本文进一步讨论了人工DNA合成技术在DNA数据存储中的应用。
1 寡核苷酸化学合成技术
在人工合成DNA中,单链寡核苷酸的应用形式最为广泛,比如PCR引物、NGS(next generation sequencing)捕获探针文库、寡核苷酸药物等。自从20世纪50年代,Todd等[17]合成出第1个二嘧啶核苷[d(TpT)和d(pTpT)],一系列寡核苷酸合成方法被开发出来,包括磷酸二酯法、磷酸三酯法、亚磷酸三酯法、亚磷酰胺法等[18-23]。其中,20世纪80年代发展起来的固相亚磷酰胺化学法[18-19,23]被广泛应用于各商业化的自动化合成仪开发中。目前大多使用的亚磷酰胺化学寡核苷酸合成法包括如下4个步骤(图1):
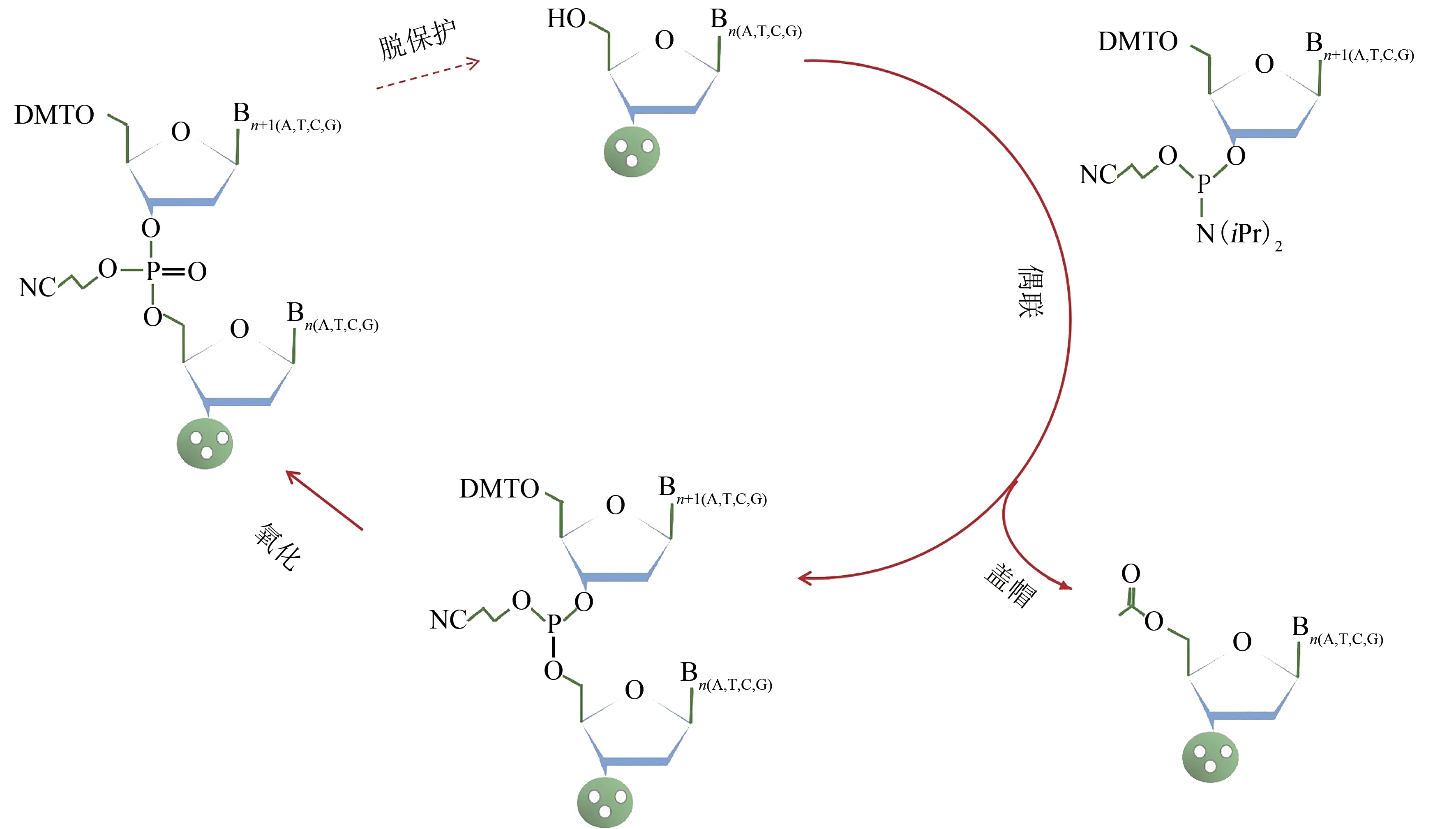
图1 亚磷酰胺四步化学寡核苷酸合成法[18-19,23,90]Fig.1 Oligonucleotides synthesis based on four-step"phosphoramide"method[18-19,23,90]
(1)脱保护:将连接在固相载体上的亚磷酰胺核苷上的保护基团DMT(dimethoxytrityl)基团,通过三氯乙酸的处理去除掉,获得游离的5′-羟基。
(2)偶联:新的DMT保护的亚磷酰胺核苷通过与四氮唑混合进行活化,得到活化的3′端,与上一个亚磷酰胺核苷的游离5′-羟基发生缩合反应。
(3)盖帽:步骤2中没有偶联成功的5′-羟基,通过加入乙酸酐和N-甲基咪唑进行乙酰化反应,避免与后续碱基的偶联反应,减少寡核苷酸合成过程中的删除错误。
(4)氧化:通过氧化剂碘的作用,将亚磷形式转化为稳定的五价磷形式。
通过以上4个步骤的循环,将与预定合成寡核苷酸序列一致的碱基,通过3′→5′的方式一个个延伸合成。根据实现方式的不同,寡核苷酸化学合成技术主要包括柱式寡核苷酸合成以及芯片寡核苷酸合成。
1.1 柱式寡核苷酸化学合成
柱式寡核苷酸化学合成利用一个带有反应腔的合成柱,装载用于寡核苷酸合成的固相载体,配合流体系统,来实现化学寡核苷酸合成的四步循环反应。目前常用固相载体为可修饰的多孔玻璃(controlled pore glass,CPG)载体[24-25],通过高分子聚乙烯等材料的颗粒包埋而成。而固相载体材料内部的孔腔,能够允许亚磷酰胺化学寡核苷酸合成的四步化学反应的试剂在其中流动,并依赖于CPG载体,将修饰的亚磷酰胺碱基一个个合成上去。寡核苷酸在合成柱上完成合成后,通过能破坏固相载体和寡核苷酸之间连接间臂的化学反应从合成柱上切割下来。根据合成柱体积以及其中带有的CPG载体量的不同,合成寡核苷酸的量也表现出很大的差别,从pmol到mmol级别不等。
柱式寡核苷酸化学合成是目前多款商用自动化合成仪采用的主要方法。目前的柱式寡核苷酸合成仪,比较成熟的有Biolytic公司开发的Dr.Oligo系列合成仪以及BioAutomation公司开发的Mermade系列合成仪。这些仪器能够实现48~1536个单合成柱的寡核苷酸并行合成。通过电磁阀,能够精确控制反应试剂流入合成柱,单个合成柱的产物规模通常在0.5~10 nmol。实验室经常使用的PCR引物、qPCR探针等,就是用这类合成仪进行合成。另外一种柱式合成仪单次合成寡核苷酸量相对较大,比如GE公司的Oligo-Plot系列,单柱的合成量多达30 mmol,主要用于寡核苷酸药物等大规模寡核苷酸原料的制备。
柱式寡核苷酸合成技术目前相对成熟,其应用也较为广泛。经过几十年的发展,通过仪器、软件、反应程序、反应步骤、纯化步骤等的优化,目前,脱盐纯化的单链PCR用寡核苷酸,单碱基市售价格能够低至0.3~0.6元/碱基。然而,柱式合成的寡核苷酸由于每轮合成反应效率通常低于99.5%,柱式合成120 nt以上、高纯度的引物非常困难,这一定程度上限制了柱式合成寡核苷酸的应用。因此,开发更长的单链柱式寡核苷酸合成技术对于进一步提升柱式寡核苷酸的应用将具有重要价值。
1.2 芯片寡核苷酸化学合成
不同于柱式合成,芯片合成中寡核苷酸的化学合成反应在修饰芯片载体上完成。从20世纪90年代Affymetrix的科学家开发出寡核苷酸芯片合成技术开始[26-27],芯片合成技术通过几十年的发展逐渐走向成熟。为了实现高通量并行的寡核苷酸化学合成,芯片合成技术需要保证在一个非常小的芯片位点上,能够不受干扰地单独完成每一轮的化学反应。为了实现这一目的,高通量光脱保护芯片合成技术[28-29]、电化学脱保护芯片合成技术[30]及喷墨打印合成技术[31-33]等被开发出来,这些技术同时被LC Science(美国)、CustomArray(美国)、Twist Bioscience(美国)进一步拓展及商业化。从设计思路上说,这些技术通过在芯片的点阵上,独立实现合成脱保护和偶联的过程,从而达到在芯片上高通量并行合成的目的;同时,因为芯片合成中单个反应体积小,从而极大减少试剂的消耗,实现低成本合成的目的。
高通量芯片寡核苷酸合成能够一次合成寡核苷酸多达数十万条,而成本仅是柱式合成的1/104到1/102。不同于柱式合成,合成出来的寡核苷酸是每条单独存在,高通量芯片合成的寡核苷酸通常以混合库的形式存在;同时,合成的混合库中单条寡核苷酸的量也远远低于柱式合成,从fmol到pmol不等。这一定程度上也限制了芯片寡核苷酸合成的应用。目前的芯片寡核苷酸合成主要用于突变体库构建、探针捕获文库、CRISPR文库构建等对合成量要求不高但序列种类复杂的领域。
尽管芯片合成寡核苷酸技术已经实现了一定程度上高通量、低成本的合成,然而,相比较高通量DNA测序技术,其通量要低4个数量级以上,单碱基成本高出5个数量级以上。这一定程度上制约了其在合成生物学领域的进一步大规模应用。因此,如何实现高通量寡核苷酸合成通量的进一步提升,将是这一领域技术发展面临的重要难题。
1.3 寡核苷酸的纯化方法
由于四步寡核苷酸化学合成法的每一轮化学合成的反应效率无法保证100%,同时合成过程中也伴随有一些副反应的发生,合成的产物中掺杂有比目的寡核苷酸序列短的寡核苷酸产物以及其他化学反应副产物。因此合成的寡核苷酸产物,通常还需要进一步纯化,以除去化学反应中的短的寡核苷酸和化学反应副产物。
常用的纯化方法包括直接脱盐纯化、OPC(oligonucleotide purification cartridge)柱纯化、PAGE(polyacrylamide gel electrophoresis)纯化、HPLC(high performance liquid chromatography)纯化等。
直接脱盐纯化,是将从固相载体上切割下来的引物,通过反复地溶剂洗脱,去除合成引物化学反应过程中产生的各种盐类,获得合成的寡核苷酸混合物。这种纯化方式,几乎无法有效地去除寡核苷酸中短链的产物,因此多用于对纯度要求不高的寡核苷酸纯化。
OPC柱是一种填有对带有5′-DMT基团寡核苷酸特异吸附能力的纯化柱。通过OPC柱对最后一个碱基带有5′-DMT基团(在合成步骤保留)寡核苷酸的特异吸附,去除截短的寡核苷酸。纯化的带5′-DMT基团的目的寡核苷酸最后通过酸处理除去DMT基团,获得目的寡核苷酸[34]。这种方法虽然在理论上能够很好地提高目的寡核苷酸的纯度,但是在实际应用中,要获得很好的纯化效果,需要充分的优化。
PAGE纯化是利用变性聚丙烯酰胺凝胶电泳对寡核苷酸进行纯化的方法[35]。优化的聚丙烯酰胺凝胶电泳能够很好地分辨不同长度的合成单链寡核苷酸。通过电泳后,将目的寡核苷酸条带切割下来,再通过溶剂将寡核苷酸从胶中释放出来,能够获得纯度较高的目的寡核苷酸。这种方法可以用于不同长度、不同应用场景的寡核苷酸纯化。
HPLC纯化是利用C18或者离子交换色谱柱,能够获得较其他方法更高的纯度[36]。分子生物学试验中常用的qPCR探针、NGS探针等多采用这种方法纯化。然而,HPLC纯化方法多适用于小于100个碱基的寡核苷酸纯化。
虽然目前的纯化方法能够实现不同应用场景的寡核苷酸纯化,但是该步骤通常耗费大量的人力,对于超过100个碱基的高纯度寡核苷酸纯化也比较困难,同时通量较低,因此需要开发更加高效的寡核苷酸纯化方法。另外,对于芯片寡核苷酸合成产物纯化,由于合成产物是混合物,而且单条寡核苷酸的量非常少,寡核苷酸产量也不均一,开发高纯度芯片寡核苷酸纯化技术也是高通量芯片寡核苷酸合成需要重点解决的问题。
值得提及的是,化学寡核苷酸合成常常伴随着较高的错误率。由于盖帽不充分、反应试剂纯度不够、反应环境湿度太高、酸处理时间过长、偶联时间不够等多方面原因[37-39],合成会出现碱基缺失、突变等多种错误。实验室优化的柱式化学合成的错误率通常在1/1000到1/500之间;芯片合成的错误率则通常在1/500到1/200之间。这一定程度上给化学合成寡核苷酸的应用造成了困扰,比如一些PCR引物克隆试验,出现了引入插入序列的错误问题。因此,未来的寡核苷酸化学合成技术可以关注更高保真度的合成纯化方法。
2 基因合成技术
不同于遗传学中“基因”的概念,合成基因中,“基因”主要是指体外合成的双链DNA片段或者克隆到质粒载体上的双链DNA,可以包含任意的长度。合成基因在酶工程定向进化、代谢工程改造、抗体工程等多个方向拥有很广泛的用途。最早的人工体外基因合成研究可以追溯到20世纪60~70年代,利用酶拼接实现从化学合成寡核苷酸到短的tRNA基因的合成[40-43]。从此之后,以单链寡核苷酸为基础原料,不同长度的基因合成拼接技术方法被开发出来。根据基因合成的技术步骤先后顺序,基因合成可以包括寡核苷酸拼装、基因合成纠错与克隆筛选以及大片段基因合成组装。
2.1 寡核苷酸拼装技术
如何从单链寡核苷酸拼接出双链DNA是基因合成的第一步。最早使用的寡核苷酸拼接方法,是连接酶依赖的寡核苷酸拼接方法。通过对单链寡核苷酸的5′末端进行磷酸化,然后依赖于连接酶,将完全互补配对的寡核苷酸拼接在一起。由于合成错误的碱基无法进行互补配对,这种方法能够实现较高保真度的寡核苷酸基因拼接。一系列的研究阐述了利用连接酶依赖的寡核苷酸拼接方法来实现基因组装[44-46]。最早的寡核苷酸的拼接,是一段段逐渐合成[40-43];然而直到“鸟枪”连接法的发明,使得多段DNA同时在一个反应混合物中组装成为可能[45-46]。连接酶链式反应(ligase chain reaction,LCR)技术的发明[47]进一步推进了基于连接酶的基因合成技术的应用[48]。根据连接酶的反应特点,片段与片段之间的连接效率是有限的,导致拼接的目的产物寡核苷酸片段产物相对较少。因此,通过连接酶拼接方法和PCR技术的结合,利用PCR技术对连接酶拼接的产物进行扩增,可以获得大量的寡核苷酸拼接产物[48-49]。
依赖于连接酶的基因合成组装技术常常需要合成正反两条完全互补的寡核苷酸链,才能完成基因合成,同时要求寡核苷酸5′端磷酸化,这常常造成合成成本的上升。基于重叠延伸PCR的寡核苷酸组装技术,聚合酶链式组装技术(polymerase chain assembly,PCA)能够一定程度解决这个问题。它通过合成包含碱基重叠区的基因正负链的寡核苷酸,利用优化的PCR程序和酶反应体系,将寡核苷酸拼接成一条完整的链。通过优化重叠区的碱基个数,比如仅使用15~25个碱基,能够减少一定量化学寡核苷酸碱基合成。为了提高PCR拼接的成功率,一系列优化的方法被发展出来,比如利用不对称PCR、系列PCR反应扩增等[50-53]。最近十几年,随着PCR仪器和PCR试剂价格的降低,PCA技术被广泛应用于工业化的基因合成应用中。
此外,林继伟等[54-56]发明了一种基于等温延伸的基因合成方法,利用独特设计的寡核苷酸,在等温聚合酶、限制性内切酶、外切核酸酶或者连接酶的复合物的共同作用下,将多条单链的发夹寡核苷酸,组装延伸成为一条双链的基因。其特殊设计的寡核苷酸上带有一个二类限制性内切酶的酶切位点,同时5′端带有一段与3′端互补配对的碱基,可以理论上形成带有几个3′端碱基悬挂的特殊发夹结构(图2)。这种方法,由于所有的反应在等温条件下完成,操作简便,可以用于大规模自动化的寡核苷酸基因合成拼装。

图2 一种单向等温的基因合成方法原理[54-55]Fig.2 A one-way isothermal gene synthesis method[54-55]
上述寡核苷酸拼装方法,结合柱式寡核苷酸合成被大量应用于工业化的基因合成生产中。然而,由于柱式合成寡核苷酸单碱基成本较高,使得基因合成的成本居高不下。而基于高通量芯片合成的寡核苷酸,其单条成本远低于以往的柱式合成寡核苷酸,在降低基因合成成本上表现出很好的潜力。2004年,Tian等[57]首次提出一种基于芯片寡核苷酸的组装方法。他们利用PCR扩增寡核苷酸池以提高用于基因拼装的寡核苷酸的量,然后用反相互补的寡核苷酸杂交筛选来降低用于基因拼装的寡核苷酸错误率,进一步地,利用一步酶组装反应将获得的寡核苷酸片段组装成多条基因。基于这种方法,他们成功合成组装了21条编码大肠杆菌30S核糖体亚基的基因。不同的研究在基因组装规模、组装长度、成本等多方面进行了优化。这些成果几乎都包括一个最核心的逻辑:通过设计的方法从一个混合文库中抓取一条目的基因所需要的寡核苷酸片段进行组装。比较有代表性的,2010年,Kosuri等[58]使用提前设计好的条形码标记用于一组特定基因组装的引物,通过先从合成文库中扩增出组装子库,再从子库中进一步扩增出用于基因组组装的寡核苷酸片段的方式,实现了47个基因,总长35 kb基因的组装。这个方法操作简便,且能够很好地进行扩展,具备工业生产的应用潜力。2011年,Quan等[32]利用喷墨打印合成技术,在一个芯片的微孔中,同时合成用于一个基因组装的多条寡核苷酸,通过原位扩增和组装,实现了多条基因在一个芯片上的合成。这项技术为开发工业化的高通量基因合成技术提供了很好的理论雏形,也促进了商业化技术平台比如Twsit Bioscience喷墨打印合成平台等的开发。另外,Kosuri领导的团队,在2018年以及2020年分别发表了DropSynth 1.0[59]和DropSynth 2.0技术[60],利用微磁珠对同一个基因的不同引物进行富集,然后包裹在一个微液滴的酶反应器内,通过扩增反应,实现基因的组装。这种方法目前成功率还较低,但是为未来进一步降低高通量基因合成成本提供了可选的技术方案。
值得提及的是,虽然不同的技术都能够实现寡核苷酸的拼接,但是在工业化生产中,如何通过工艺优化提升技术的稳定性,以及降低技术的成本是关键。通过技术方法间的组合设计,开发低成本、高效、稳定的工艺流程是一个可行的思路。另外,寡核苷酸的拼接常常面临高GC、高AT和高重复序列合成困难的问题,尤其是对于依赖于PCA组装或者包含PCA组装流程的寡核苷酸拼接技术。由于高温PCR扩增,涉及序列之间的退火再延伸的步骤,高GC/AT和高重复序列会增加不同寡核苷酸(或者DNA片段)之间的错配,从而造成组装失败;同时高温PCR扩增酶本身对于高GC或者高AT也会表现出一定程度的扩增困难。通过往PCR体系中添加GC扩增增强剂、优化PCR程序以及在寡核苷酸设计时避免将这些区域包裹在重叠区内等方式,能够一定程度解决这些问题[61-62]。
2.2 基因合成纠错与克隆筛选
用于基因合成拼装的合成寡核苷酸常常伴随着一定的错误,同时,基因合成拼接过程中,酶扩增或组装也会引入一定概率的碱基错误,因此,为了减少寡核苷酸拼接中的碱基错误,一方面,可以通过优化寡核苷酸拼接流程、程序以及选用高保真的扩增酶体系;另一方面,可以通过酶纠错技术降低基因合成拼接过程的错误率。基于酶的纠错技术,主要是通过利用双链DNA中错误序列和正确序列形成不匹配的区域,对错误序列进行识别或者切除。在基因合成过程中,由于酶扩增的产物是正确双链DNA产物和含有错误的双链DNA产物的混合物。通常需要先将产物进行变性再复性,然后通过错配识别酶或错配切割酶,将带有错配的双链复合物去除。MutS是一种常用的错配识别酶。在微生物体内,它能够识别并结合多种错误的碱基以及单链的小环。利用MutS与错配双链的结合,然后利用合适的方法去除蛋白-双链复合物,能够降低基因组装产物中的错误率[63-64]。将MutS固定在过滤柱上,也是一种能够去除组装基因中的错误链的办法。2020年,徐健团队[65]通过将MBP融合的MutS突变体固定在纤维素柱上,并利用该柱子识别并去除芯片寡核苷酸的错误,将基因合成组装的碱基准确率提升了37.6倍。另外一些酶,则具备错配识别并切断错误配对双链DNA的功能,包括T7 Endonuclease I、大肠杆菌Endonuclease V、米曲霉S1 Nuclease、芹菜CEL nuclease等[66]。其中一些酶被进一步拓展成为成熟的商业化产品,比如Transgenomic公司(美国)的Surveyor™内切核酸酶,以及Thermo Fisher Scientific公司(美国)的CorrectASE内切核酸酶(之前为美国Novici Biotech公司的ErrASE纠错试剂盒)等。在多个已报道的基因合成组装工作中,这些商品化的纠错酶都表现出了一定程度的纠错能力[32,58,67]。同时,关于不同的基因合成纠错酶的纠错能力比较分析表明,ErrASE能够将错误率降低到最低,而MutS能够很好地增加正确的基因合成组装数[68]。
尽管通过寡核苷酸拼接流程和体系优化以及酶纠错法能够在一定程度上降低基因合成组装的错误率,要获得100%序列正确的基因仍然需要进一步的技术流程。利用大肠杆菌克隆筛选是一种常用的方法。具体流程包括:首先将寡核苷酸拼接产物或者经过酶纠错后的寡核苷酸拼接产物克隆到质粒载体上,然后转化至大肠杆菌,对经抗性平板筛选的克隆进行培养并抽提质粒,最后利用测序验证筛选获得包含有100%序列正确的质粒。需要注意的是,在该步骤中,一些合成的基因克隆到质粒载体后,在大肠杆菌中会发生不稳定复制或产生毒性。这些基因常常因为克隆构建困难或在大肠杆菌中易产生突变而造成基因合成的失败。根据不同大肠杆菌菌株的遗传特性,更换不同的菌株能够在一定程度上缓解该问题。其中,Thermo Fisher Scientific公司推出了Stabl2和Stabl3大肠杆菌感受态系列,以及NEB公司推出了NEB Stable大肠杆菌感受态系列等用于解决基因克隆不稳定的问题。同时,利用低拷贝的载体,也能够实现更稳定的合成基因克隆。然而,在具体的基因合成应用中,更换不同菌株或者载体克隆策略常常费时费力,且仍未能够有效解决这类问题。因此,开发更加简单通用的方法,比如不依赖于大肠杆菌克隆的方法,对于工业基因合成仍然尤为重要。
2.3 大片段基因合成组装
由于寡核苷酸拼接组装中的碱基仍存在一定的错误率,为减少首次克隆筛选获得正确克隆的工作量,通常从寡核苷酸直接拼接组装的基因长度会控制在3 kb以内。对于更长的基因合成,则将首轮克隆筛选获得的正确的基因片段组装成更长的片段。一系列方法被应用其中,如Golden Gate组装[69-70]、Gibson组 装[71]、 循 环LCR[72-73]、 双 引 物TPA组装[74]、BioBrick组装[75]等。其中Golden Gate组装法和Gibson组装法,在大片段基因合成组装应用上相对成熟。基于Type IIS限制性内切酶的Golden Gate克隆技术,利用Type IIS限制性内切酶在识别序列下游位置切割DNA的特点,能够在任意需要相连的两个片段末端创造互补配对的黏性末端[69-70]。这种方法能够一次实现多个片段的组装,也能通过多级组装完成更多片段的组装[76]。该组装技术的优点在于,操作简便,能够一次实现较长基因的组装,同时对于包含各种特殊序列结构的基因也能够实现很好的组装效果。然而,如果人工合成的基因中有多个Golden Gate组装使用的限制性内切酶酶切位点,这个组装方法失败率较高。相比之下,Gibson组装方法没有酶切位点限制的问题,操作也很方便。利用片段之间互相重叠的同源区(通常20~150个碱基),在高温聚合酶、高温连接酶、外切核酸酶的作用下,能够实现多个片段在载体上的一步高效组装。根据Gibson等[71]的测试结果,其能够有效组装DNA长度达到数十万碱基。然而缺点是,当片段之间重叠区GC或者AT含量很高时,可能发生一定的错误。结合限制性内切酶切割,修改的Gibson组装方法能够一定程度解决这一问题[77]。另外,依赖于酵母同源重组系统,也能够很好地实现多个带有同源互补区设计的片段的一步组装[78-80]。对于一些在大肠杆菌组装克隆中有困难的基因合成,酵母同源重组组装是一个很好的选择。然而,由于目前常用的一些质粒载体不带有酵母的复制系统,且基于酵母体系的质粒制备成本相对较高,一定程度上限制了该方法的应用。
由于越大的质粒,在大肠杆菌中高拷贝复制时越不稳定。在大肠杆菌中组装的基因片段长度通常小于15 kb。对于更长的基因,可以利用特殊的克隆载体,比如包含单拷贝F质粒复制因子的BAC或者改造的BAC载体[81-82],来进行组装。然而,这类质粒克隆效率低下,规模化操作困难。因此,一些超大片段的组装可以通过转化至酵母载体上,在酵母中完成[78-80]。尽管目前的技术研究已经能够实现长度达到数十万碱基的片段组装[71],但目前大多商业化交付的基因合成仍然局限在15 kb以内。因此,如何建立更加高效、标准化的十万碱基级别的大片段基因组装方法及工艺,实现规模化的大片段基因合成交付,对于促进更广泛、深入的合成生物学研究及应用有着革命性的意义和价值。
3 基因组合成技术
合成组装一个生物体完整的基因组并使之有生命活性,一直是DNA合成领域努力的方向之一。2002年,Cello等[83]利用化学合成的寡核苷酸原料,从头合成了带T7 RNA聚合酶启动子的脊髓灰质炎病毒cDNA,并基于合成的cDNA,转录出病毒的RNA,同时在Hela细胞提取液中组装出有活性的病毒颗粒。它利用平均长度在69个碱基的合成寡核苷酸原料,首先通过末端互补方式,拼接成400~600个碱基的基因片段;然后将这些片段插入到质粒载体中,经测序验证后,通过逐步克隆的方式拼接成完整的病毒cDNA。紧接着,2003年,Venter实验室[84]利用优化的方法合成了一个5386 bp的细菌噬菌体ϕX174。该团队利用平均长度在42个碱基的寡核苷酸,首先进行PAGE纯化,然后利用T4多聚核苷酸激酶磷酸化,在Taq连接酶的作用下,将寡核苷酸连接成主要大小在700 bp左右的片段混合物;进一步地,通过PCA组装将这些片段混合物组装成完整的噬菌体DNA,经酶切环化后,转入大肠杆菌细胞中获得侵染能力的噬菌体。这两个工作开启了基因组合成的新纪元。
随着基因合成技术的进步,目前拼装一个完整的病毒或者噬菌体基因片段变得容易得多,甚至可以由CRO服务公司单独完成。2020年,Thao等[85]利用在第三方服务公司合成的基因片段,基于酵母组装平台,在T7 RNA聚合酶转录作用下,在一周内就完成了2020年新型冠状病毒SARS-CoV-2的合成。相对于几千碱基到数万碱基病毒或者噬菌体基因组,超过Mb水平的细菌基因组和酵母因组要大得多,因此,合成也困难得多。近十年来,多个研究成果实现了不同细菌基因组或者酵母染色体合成[86-92]。这些研究在基因组合成设计、基因组大片段拼接与分离、合成纠错调试、功能筛选与测试等多个关键技术环节都进行了不同程度的优化与测试。究其核心技术逻辑,可以大致概括为“一次从头合成”和“逐步替换从头合成”两种。其中,“一次从头合成”的代表工作为2010年,Gibson等[87]从寡核苷酸合成了丝状支原体Mycoplasma mycoides的基因组。在这个工作中,他们首先利用将从供应商获得的基于化学合成寡核苷酸组装的1078个1080 bp片段,利用酵母同源重组组装成109个10 080 bp的组装产物;然后基于这些10 080 bp的片段,利用酵母同源重组进一步组装获得11个100 kb的组装片段;进一步基于这些大片段,利用酵母组装成为1 077 947 bp完整的基因组。随后,完整的基因组合成被转移到另外一个支原体受体细胞中,在筛选标记的协助下获得有功能的细胞。“逐步替换从头合成”是“国际Sc2.0酵母基因组合成计划”采用的合成方法。其特点是,先利用常规的基因合成拼接技术,从寡核苷酸逐级拼装出10 kb的基因片段。然后将多个10 kb的基因片段经酵母同源重组系统拼接成30~60 kb的大片段,同时在筛选标记的协助下替换野生型基因组中对应序列的片段。通过30~60 kb大片段逐步替换的方式,实现人工酵母染色体的合成。目前,“国际Sc2.0酵母基因组合成计划”已经基于该方法,发表了6.5条染色体的合成工作[89-90]。相对于“一次从头合成”,这个方法的好处是,当出现设计的合成替换片段对酵母功能造成极大影响时,能够调整设计策略,进行及时的纠错修正。
尽管目前细菌、酵母染色体已合成成功,但对于更大的动植物细胞基因组合成仍然面临诸多挑战。比如,Mb级别的基因组片段的快速合成及其在动植物细胞基因组的高效替换被认为是动植物基因组合成的技术策略之一,然而也是需要进一步开发突破的关键技术瓶颈。同时,超大基因组的合成也面临着诸多问题,包括:①合成基因组大片段的碱基突变、缺失、移位等,需要大量的纠错测试工作;②部分大片段的质粒转化及组装可能面临失败,需要返工;③保证合成的基因组产生功能,需要一定的前期调研与设计工作。生命体的复杂性,使得生命再造的过程充满了艺术性和不确定性;然而,正是因为这种不确定性,每一步科学上的跨越才会无比珍贵。随着合成和组装DNA技术的不断进步,自动化仪器平台商业化的日趋成熟,我们有理由相信未来实现一个基因组的合成和今天的基因组测序一样简单、高效。
4 新一代酶法DNA合成技术
合成生物学的高速发展催生了大量的基因合成需求。传统的化学寡核苷酸合成法逐渐凸显出其缺点:①合成长度太短,且碱基错误率高,导致合成拼装过程耗时耗力;②其工艺过程要求高,通常需要控制在一个无水无氧的反应环境内;③合成过程中产生大量的污染性有机化学废弃物,对环境不友好。因此,基于生物酶催化的DNA合成技术给合成生物学家们带来了新的曙光。
基于模板DNA的聚合酶延伸反应技术已经被广泛应用于DNA的复制与扩增,如PCR技术。利用可逆化学修饰碱基的聚合酶延伸反应[93],“边合成边测序技术”(SBS,sequencing by synthesis)已经很好地应用于高通量DNA测序。这给生物酶催化的DNA合成提供了重要的理论启示。近十几年来,基于末端转移酶(terminal deoxyribonucleotidyl transferase,TdT)的新一代酶法DNA合成技术越来越受到重视。1959年,Bollum[94-95]首次阐述末端转移酶TdT可以用于不依赖于模板的DNA合成。1962年,Bollum发现将起始碱基的3′-OH基团封闭掉,能够阻止末端转移酶的碱基聚合反应,证实了dNTP是加到起始碱基的3′-OH端。与此同时,Bollum[96]也概念性地提出,能够利用封闭3′-OH端的碱基单体,基于TdT末端转移酶,合成核苷酸多聚体。在分类学上,TdT末端转移酶隶属于聚合酶X家族的一部分。在生物体内,它的主要功能是通过随机的碱基插入,增加抗原受体多样性[97-98]。2008年,Ud-Dean等[99]进一步完善了基于TdT末端转移酶的理论模型:利用不依赖于模板的TdT末端转移酶碱基聚合反应,基于可逆化学修饰碱基,根据任意设计的序列DNA,能够实现不依赖于模板的长片段人工DNA的合成。
基于TdT酶法DNA合成技术,从理论走向实际应用的一个关键点是,需要获得一个高效的TdT酶和可逆修饰碱基组合。由于TdT酶可以一次延伸多个连续的碱基,为了保证TdT酶依赖的DNA合成按照预定的序列一个个延伸,需要对碱基单体进行修饰,以保证第n+1个碱基合成完成后,反应能够被终止,而不会继续进行n+2个碱基的反应。同时碱基的修饰基团能够通过化学、物理或者生物的方法切除,以继续下一个碱基的合成。3′-O修饰的可逆dNTPs是TdT酶依赖的单链DNA合成反应很好的底物(图3)。2016年Mathews[100]报道了合成的NB-dNTPs[3′-O(-2-nitrobenzyl)-2′-deoxyribonu‐cleoside triphosphates] 以 及DMNB-dNTPs[3′-O-(4,5-dimethoxy-2-nitrobenzyl)-2′-deoxyribonucleoside triphosphates]能够被TdT酶催化利用,同时能够阻断TdT酶的第n+2延伸反应。同时,NB基团及DMNB基团能够在紫外线的作用下实现完全的降解。此外,可逆的修饰基团也可以加在碱基上面[101],但是实际的应用效果未见充分的报道。

图3 基于3′-O修饰的可逆dNTPs TdT酶法DNA合成示例[99-100]Fig.3 Illustration of TdTenzymatic DNA synthesis based on 3′-O modified reversible dNTPs[99-100]
另外,2018年,Keasling实验室[102]报道了一种新的策略,将dNTPs利用一个可以被切割的间臂偶联在TdT酶催化中心附近的位置,当TdT酶催化dNTPs进入单链寡核苷酸以后,TdT酶仍然结合在单链寡核苷酸上,阻止下一个TdT-dNTPs进入反应。通过切割间臂,在释放TdT酶后,新的TdT-dNTPs可以继续进入反应。虽然TdT-dNTPs目前使用成本价格较高,但是该方法反应速度快,同时具备很好的优化升级潜力,为TdT酶介导的DNA合成技术开发提供了很好的可选策略。
尽管目前的研究表明TdT酶能够很好地应用于
单链DNA的合成,然而开发可大规模交付且成熟的单链DNA合成技术仍需投入大量的研究,包括:通过TdT酶工程改造,提升其针对可逆修饰碱基的活性;整体优化酶循环反应工艺,提升每轮反应的碱基合成效率等。目前,至少有4家公司专注于这个方面的技术开发,包括DNAScript(法国)、Nuclera(英国)、Molecular Assemblies(美国)和Ansa Biotechnologies(美国)等。这些公司已获得了大量的资金支持,同时,已经基于生物酶法实现了一定长度的单链寡核苷酸的合成,甚至开发出了基于酶合成法的合成仪原型。另外,值得提及的是,在技术未成熟之前,尝试更多的方案对于实现生物酶法DNA合成技术的工业化应用仍然具有重要价值,比如不依赖模板的其他聚合酶[103-104]、RNA连接酶[105-106]等。2019年,Halpain等[104]就报道了一种方法,基于DNA聚合酶,借助于瞬时的寡核苷酸杂交,也能够实现单链DNA的合成。
5 DNA合成与DNA数据存储
DNA合成作为DNA数据存储的关键技术基础,是DNA数据存储从理论走向应用的基石。2012年,Church等[3]利用合成的54 898条159 nt寡核苷酸库存储了53 426个单词、11张JPG图片和一个JavaScript程序。每条寡核苷酸包含了96 nt的数据区,左右扩增引物区和测序引物区各22 nt以及一个19 nt的信息地址(索引)区。2017年,Erlich等[4]利用72 000条200 nt的寡核苷酸库,存储了2 146 816 bytes的信息。每一条寡核苷酸包含有128 nt的数据区,16 nt的种子区(类似于索引碱基功能),8 nt的纠错区,以及24 nt的左右扩增引物区。2018年,Organick等[5]利用9个合成的寡核苷酸文库,总计约1300万条寡核苷酸,存储了200 MB的数据信息。此外,也有研究工作利用寡核苷酸拼装的基因片段[107]、克隆的质粒[108-109]等实现数据信息在DNA中的存储。然而,鉴于成本以及可扩展性等原因,目前DNA数据存储技术主要依赖合成的芯片寡核苷酸文库。DNA合成的长度、成本及速度是影响DNA数据存储应用的关键因素。
存储数据信息的DNA长度直接决定存储信息碱基利用率。由于芯片寡核苷酸合成长度有限,在存储数据信息时,需要将二进制信息编码的寡核苷酸序列,拆分成一系列的序列片段。为了在测序解读时,能够将这些序列片段拼接成完整的存有二进制比特信息的DNA序列,需要在这些序列片段上添加索引序列(或者位置序列)[3,5],用来标识这些序列片段对应于存有二进制比特信息的DNA序列中的位置;比如用4个碱基代表256个序列片段的索引位置,AAAA=1,AAAT=2…GGGG=256。当拆分序列片段越多时候,用来标识片段位置的索引序列的长度就越长(通常,拆分序列的个数m≤4n,n为索引序列的碱基数)。另外,为了方便信息的随机读取和备份,还需要在存储信息的序列两端添加扩增引物[3-5]。甚至,还需要根据需求添加纠错码等[110]。在这种情况下,用于存储数据信息的寡核苷酸长度越长,存储信息碱基利用率就越高。例如,若采用Church等[3]的方法存储数据信息,当合成寡核苷酸长度达到600 nt时,存储信息碱基利用率能够从63%提升到接近90%的水平(图4)。尽管,寡核苷酸拼接的更长的基因片段也可以用于数据存储,然而由于单碱基的成本将增加10~100倍,推广使用困难。因此,未来的技术研究可以更加关注低成本长片段芯片寡核苷酸合成技术开发。

图4 存储信息碱基利用率与寡核苷酸合成长度之间的关系[本图根据Church等[3]使用的方法,左右引物各22 nt,索引(地址)序列19 nt,假定合成长度不同的情况下,计算存储信息碱基利用率]Fig.4 Relationship between base utilization of data storage and oligo length[Referring to methods used by George Church et al.,base utilization of data storage is calculated given that oligo length is different,while both the left and right flanking primers are 22 nt,and the index(address)sequence is 19 nt]
目前,DNA数据存储的主要成本来源于DNA合成。即便是最便宜的芯片合成寡核苷酸,其单碱基合成成本仍高于测序成本近5个数量级。按照当前DNA合成成本计算,仍需降低6~8个数量级才能使得DNA数据存储成本与目前硬盘存储的(约100元/TB)相近。然而,不同于其他DNA合成应用,用于DNA数据存储的合成DNA能够在一定程度上降低对保真度的要求。由于目前合成技术一次合成的DNA分子数通常大于fmol级别,使得即便在一定的碱基错误率情况下,合成DNA中针对一条序列也有不同的分子用于信息纠错。同时,经特殊设计的数据冗余及纠错码,能够进一步提升低保真合成DNA中存储数据信息的解读准确性[5-6,110]。DNA合成保真度要求的降低,一定程度降低了DNA合成工艺的要求,从而有利于开发出更低成本的DNA合成技术。如依赖未修饰dNTPs的TdT酶法合成已被验证其在DNA数据存储中的应用[111]。未来在该方向的研究将有望进一步开发适配于DNA数据存储的低成本合成技术。值得提及的是,利用通用合成的DNA片段,基于类似于“活字印刷”的原理来存储数据信息,也可能是一种非常有效的降成本方式。比如将英文的26个字母分别存储在通用合成DNA上,然后通过酶拼接或者其他方法,在存储信息时,进行自由组合,进而反复使用一次合成的DNA分子,能够潜在地降低成本,然而这些方法仍然需要进一步的开发。
此外,DNA合成的速度也决定了DNA信息存储的写入速度。目前的DNA合成技术依赖循环的化学反应或者酶催化反应,而每轮化学反应或者酶反应都需要较长的时间。如亚磷酰胺化学DNA合成,基于自动化合成仪的每轮化学合成反应耗时在数分钟到十几分钟。换言之,以合成200 nt的寡核苷酸为例,其耗时将达到几十个小时。相比于硬盘存储的快速写入,这个速度仍相距甚远。因此,除降低合成DNA的成本外,合成速度的提升也是DNA数据存储应用实现的关键。
同时,DNA合成作为DNA数据存储技术流程中的一个重要环节,可以与其他环节的技术工艺进行整合优化,以实现DNA数据存储整体成本的降低与效率的提升。如在信息的编码环节,通过提升从二进制信息编码获得的DNA序列的GC均一度,来提升DNA合成技术环节的成功率;通过开发适用于合成和测序错误率的纠错编码技术,提升在测序及后续数据解读过程中的准确性等。另外,为了实现更好的信息解读,还需要进一步地提升测序技术以及数据解读技术。比如,可以通过缩短DNA测序流程的建库时间以及整合快速的碱基序列读取技术,实现存储数据DNA的实时、快速读取;通过开发测序信号读取与解码一体化算法,加速A/T/C/G信息到0/1二进制信息的读取过程等。最终,通过开发基于数据编码、DNA合成、DNA测序、数据读取的一体的高效技术流程,实现DNA数据存储的大规模应用。
6 总结和展望
如果说DNA测序技术打开了人类对生命遗传规律的认知之门,那么人工DNA合成技术使人类进一步深度认知、改造甚至创造生命成为可能。DNA合成技术的发展使得生命科学从测序带来的可观测、可理解、可描述的数字化生命时代向可预测、可定量、可创造的合成生物学工程化时代迈进。
经过将近70年的发展,DNA合成技术已经从若干个寡核苷酸碱基的合成跨越到Mb级微生物基因组的合成(图5)。大规模的寡核苷酸、基因的合成已经实现了商品化交付,同时也很好地服务于科研及生物技术产业的发展。目前,高通量芯片DNA合成技术的单次合成碱基量超Mb,且成本仅为柱式合成的1/10 000到1/100。同时,基于芯片技术的更高通量的DNA合成技术,及其原位基因合成组装技术也正逐渐走向商业化。与此同时,新一代酶法DNA合成技术也为未来的更高通量及更低成本DNA合成带来了曙光。DNA合成仪器制造公司以及DNA合成服务公司也获得了众多产业化发展机遇。然而,相比于测序技术,DNA合成技术仍处于较早期水平。
目前DNA合成成本仍然较高,极大限制其在DNA数据存储等合成生物学领域的应用。因此,降低DNA合成成本仍是未来技术开发的关键。除开发核心技术流程外,适当降低用于DNA合成的化学和生物试剂原料的成本等也是影响DNA合成成本的重要因素。此外,人工成本也是DNA合成中的重要组成部分,尤其是高耗时耗力的基因和基因组合成、拼装过程。开发高集成的自动化平台对于降低人工成本、提高合成效率将起到重要的作用。
尽管目前已实现微生物基因组的从头合成,但对于102kb级以上大片段DNA的合成,其合成周期仍相对较长,达数月之久,且失败率较高。同时,目前基因组合成仍然停留在微生物水平,对于动植物基因组的合成仍需要突破众多技术瓶颈。这一定程度上限制了DNA合成在DNA数据存储、合成生物学生命再造等领域的应用。开发更高效的基因组水平的大片段DNA合成技术,将是DNA合成从生物体局部基因改造到大规模全局生命再造应用的关键。另外,尽管新一代酶法DNA合成技术通过最近十几年的发展,拥有一定的技术基础,距离工业规模的合成交付仍然有一定的距离,需投入更多的创新研究和持续的努力。
DNA合成技术的发展促进了代谢工程改造[112]、酶工程改造[113]、抗体工程[114]、IVD诊断[115]、寡核苷酸药物[14]、DNA数据存储[1,3-6]等多个合成生物学领域的发展。尤其是面向特定应用的DNA合成技术,将会给下游应用领域带来革命性的变革。比如通过建立DNA合成设计到特定应用的快速自动化合成平台,将加速有益于人类功能活性物质的生产或药物分子的菌株改造效率[116-120]。作为DNA数据存储流程的基础技术,人工DNA合成技术是DNA数据存储从概念走向大规模应用的关键。在全球数据大爆发的背景下,开发针对DNA数据存储的长片段、低成本、快写入的DNA合成技术(图5), 对于加速DNA数据存储的应用以及解决人类面临的数据危机尤为重要。
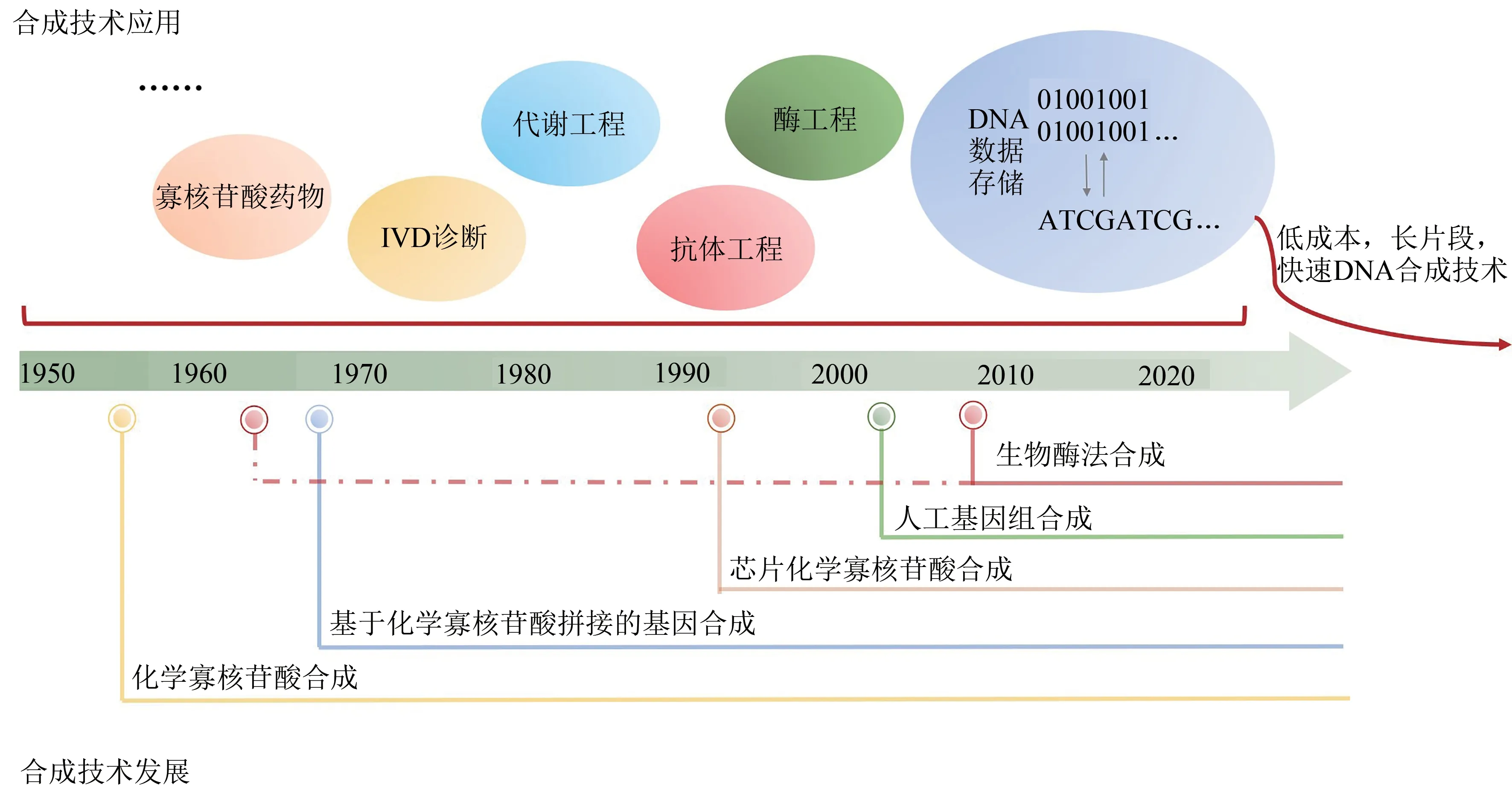
图5 DNA合成技术发展与应用Fig.5 Development and application of DNA synthesis technology
致谢:感谢中国科学院深圳先进技术研究院合成生物学研究所叶健文副研究员对本论文提出的修改建议。
