基于人工染色体的DNA信息存储前沿进展
2021-07-21杨洋樊春海
杨洋,樊春海,2
(1上海交通大学医学院附属仁济医院,分子医学研究院,上海 200127;2上海交通大学化学化工学院,上海 200240)
近二十年来人类在信息技术方面取得的进步,尤其是互联网和移动互联技术的发展,带来了前所未有的数据爆炸和信息存储危机。在科研与社会服务层面,更高分辨率的天文观测、医学成像以及交通监控正在不断产生大量的图像视频类数据。社交网络则是另一个重要场景,除了互动信息之外,个人化的照片与视频创造和传播呈现加速趋势,预计到2030年全球网民比例将从2017年的51%增长至近乎全覆盖,届时数据产生的速度也将翻倍。全球数字化的这一发展趋势使得数据量快速增长,据国际数据公司统计,2018年人类产生的数据总量达到了33 ZB(1 ZB≈109TB),而到2025年这一数字将增长为惊人的175 ZB。面对快速增长的海量数据,基于磁、光、电等的传统介质的存储技术面临功耗、体积以及使用寿命等限制,而DNA存储提供了应对数据存储发展挑战的新契机。
DNA因其极高的信息密度和非凡的稳定性成为存储系统的有力候选。首先,DNA的信息密度非常大,自然界中大部分生物的全部生命信息都存储在DNA中,以人类为例,人体大约可以产生40万种蛋白质,而它们需要在不同的发育阶段,以不同的数量和速度在不同的细胞中被表达、使用和代谢。所有这些蛋白及其程序控制相关的信息都被存储于仅仅23对染色体上。一个细胞中染色体所折叠的DNA全部拉直并连接虽然可以长达3 m,但其重量却仅有10-11g。一方面理论上,1 g DNA可以存储455 EB(1 EB≈106TB)的数据量,据此计算1亿部高清电影如果存储于DNA中,这些DNA只需要占据一块橡皮的大小,而利用2T的硬盘来存储的话则需要10万个硬盘。另一方面,DNA存储数据具有极高的稳定性,不但动植物化石中保存的DNA可以历经千年保持可读性,提纯的DNA经过浓缩与干燥,可在惰性气体保护下保存至少百年的时间。而如果把携载外部信息的DNA借由微生物进行保存,其拷贝数可以以指数形式大量扩增并代代相传,相比承载与维持服务器机组工作所需的巨大机房和空调系统,利用微生物携载DNA用于信息存储也是极其绿色节能的选择。因此,越来越多的国家已经开始将基于DNA的数据存储列为战略层面的发展方向,例如2021年1月,美国半导体产业协会(SIA)发布的《半导体10年计划》,将DNA数据存储列为未来海量数据存储的重要选项。我国科技部也早在2018年即开展了基于DNA的信息存储相关项目部署。在2021年3月份通过的国家“十四五”规划纲要的第九章中更明确提出“推动生物技术和信息技术融合创新”的目标,为大力发展合成生物学及DNA信息存储技术提供了政策引导。本文评述作者长期从事核酸分析、纳米技术以及DNA编码与计算相关的交叉学科研究并取得了显著成果,将在下文中简要综述DNA信息存储的历史发展,对近期基于酵母的人工染色体构建与DNA信息存储的进展工作稍作点评。
从1960年代苏联物理学家Mikhail Samiolvich Neiman首次提出关于DNA作为信息存储物质的设想[1],到1986年麻省理工学院的研究员Joe Davis将12个字母的词组转换为28个碱基对的DNA序列并插入大肠杆菌(E.coli)细胞中[2],DNA作为存储材料的潜力早已为人所知。但存储数据量和检索读出技术距离实用还有很远的距离。自1980年代以来不断进步的DNA固相合成技术快速发展,为大量数据的写入提供了基础,与此同时DNA测序技术的迭代升级使得信息的高效读取成为可能。近年来飞速发展的新一代测序技术(next generation sequencing,NGS)[3]提供了同时平行测序百万条短DNA序列的平台,利用NGS技术,一个人的基因组可以在一天内测序拼接完成,而传统的桑格尔(Sanger)测序法[4]在一台测序仪上完成这一工作则需要十年的时间。伴随着整个分子生物学的发展,我们终于可以编写、存储、检索和读取大量的DNA序列。同时,以DNA为媒介存储信息的工作也不断涌现,存储的数据量和数据类型都不断增加。1999年,纽约大学Risca等[5]利用69个碱基对成功编码和检索了含有22个字母、数字和字符的消息。2012年,哈佛大学Church课题组在《科学》杂志发表论文,详细介绍了如何使用DNA来存储一本含由53 426个词的书[6]。在存储算法研究方面,简单的二进制向四进制的转换并不能最大程度地利用DNA的存储能力,随机产生的特殊的序列组成(例如连续的G序列或C序列)有可能给合成与测序带来错误概率的积累,因检索与纠错需求带来的信息冗余又会使得存储密度大打折扣。因此,信息的编码算法起到举足轻重的作用。2017年Yaniv Erlich和Dina Zielinski开发了一种新型的“喷泉“码算法,可以将净信息密度提高到1.57 bit/bp,将DNA的实际容量提高到86%[7]。美国微软公司(Microsoft)在DNA存储领域一直推进技术革新,2016年他们与华盛顿大学合作发表的一篇有关DNA数据存储前景的文章描述了如何利用合成的DNA编写和检索三幅图像[8];在2019年,他们进一步开发了一套全自动的DNA存储与读取设备[9];同年,他们又利用纳米孔技术实现了1.67 MB的信息读取[10]。从这一领域快速增长的文章(2018年、2019年每年在PubMed上统计DNA data/information storage的相关文章超过1000篇)和专利数量(WIPO关于DNA数据存储的国际专利申请超过1 700余件)可以判断,国际上关于DNA信息存储的竞争在未来十几年中还将持续白热化。
2021年2月12日,天津大学元英进教授团队带领的跨学科团队于National Science Review上在线发表了以“An artificial chromosome for data storage”为题的研究论文(天津大学微电子学院青年教师陈为刚副教授、化工学院博士研究生韩明哲以及助理研究员周见庭为论文共同第一作者)。该工作中,研究者从头编码设计合成了一条长度为254 886 bp,专用于数据存储的酵母人工染色体,存储了两张图片及一段视频,编码覆盖率超过95%,并实现了数据的稳定复制与快速可靠读出(图1)。
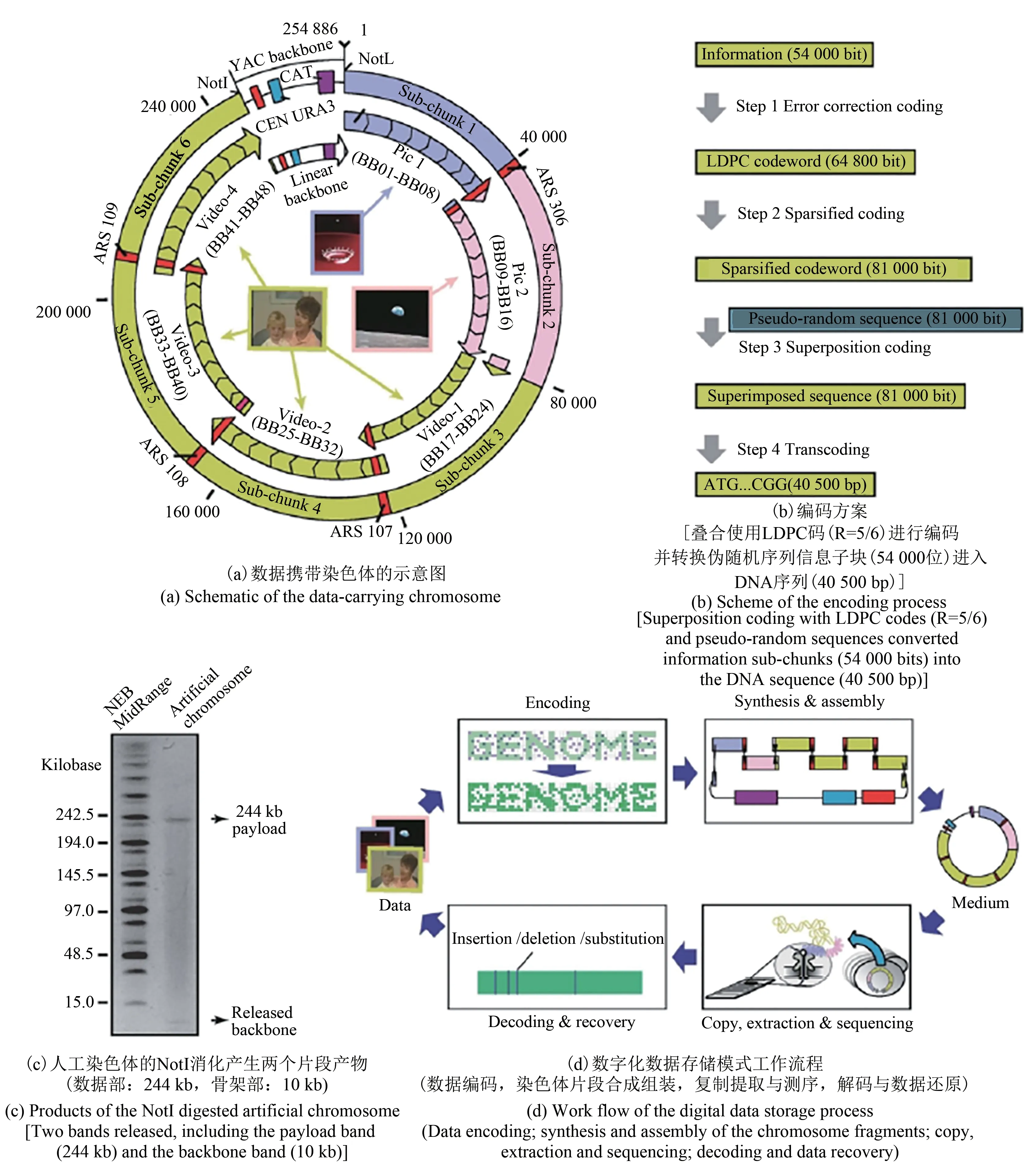
图1 设计与组装携载信息的人工染色体Fig.1 Design and assembly of the artificial chromosome that carriesdigital information
在存储环节,一方面该研究借助叠加伪随机序列应对三代测序的插入/删除(insertion/deletion)错误,采用现代通信中常用的低密度奇偶校验(low-density parity-check,LDPC)码纠正替代错误,实现了在高达10%错误率时的数据可靠恢复。另一方面,该染色体设计中,插入一定数量的酵母自主复制序列(autonomously replicating sequence,ARS),提升了染色体的稳定性,保障了其高效组装和稳定复制(>100代复制仍可读出)。该存储模式中,数据逻辑密度(包含载体)为1.19 bit/bp,与目前文献中指标最高的四进制编码DNA喷泉方案相当[8]。
在数据读取环节,该工作利用三代纳米孔测序技术在大约10 min时间内获得足够的原始读段后,结合自主设计的生物信息学与纠错译码混合流程,便可实现数据可靠恢复,所需测序覆盖度仅为16.8×。相比纯粹利用合成DNA存储再利用聚合酶链反应(PCR)技术进行备份的传统做法,利用酵母菌存储信息可以实现一次写入,多次读出,体现了极好的低成本与便携性优势。
这一最新研究成果为DNA存储技术提供了新颖的角度与方案,可以期待的是,通过进一步降低合成成本和构建多条人工染色体,人们能够在酵母菌中存储更多数据。随着合成生物学领域的更多技术进步,利用DNA和生命系统存储与利用信息将会成为大势所趋,然而相比于以硅基硬盘为基础的电子化信息存储,核酸/微生物硬盘的广泛应用还有赖于存储密度的进一步提高,合成组装操作的进一步简化,存、检、读方案的全面整合以及全流程的自动化集成。以DNA存储为核心的上述全链条的技术研发有望引领多学科的交互发展与共同进步。
