DNA数据存储:保存策略与数据加密
2021-07-21周廷尧罗源蒋兴宇
周廷尧,罗源,蒋兴宇
(南方科技大学生物医学工程系,广东 深圳 518055)

随着互联网和人工智能等信息技术的高速发展,人类产生的数据信息呈爆炸式增长。据国际数据公司(IDC)发布最新全球DataSphere显示,2020年创建和使用的数据量高达5.9×1022字节,并且今后每年数据量将以26%的速度增长。现有的数据存储体系主要有磁性存储(磁盘、磁带和机械硬盘)、光学存储(CD、DVD)和固体存储(闪存芯片、DRAM芯片)[1]。这些存储体系虽然近些年来得到了较大的发展,但其劣势日益凸显,如存储密度有限、有效存储时间短、生产设备能耗高、原材料硅的供应量有限且易污染环境等,已无法满足当前海量数据爆炸式增长的需求[2-3]。因此,迫切需要发展一种具有更好存储性能的新技术与新方法。
DNA是一种古老的存储介质,储存着从微生物到人类亿万生命的海量遗传信息。自20世纪60年代以来,因DNA分子的存储密度高、耗能低、寿命长、无磨损等潜在优势,人们曾就DNA分子作为存储媒介的可行性展开讨论[4-8]。近年来,随着DNA合成技术和新一代测序技术的突破性发展,DNA数据存储已成为当前全球数据信息存储技术的研究热点。2012年,哈佛医学院和约翰·霍普金斯大学合作利用DNA编码了一整本6.58×105字节的巨著,使DNA存储技术迈进了一大步[9]。2013年,欧洲生物信息研究所科研团队[10]将包含5种类型数据7.39×105字节的计算机文件(文本、PDF、照片、MP3和霍夫曼编码)编码到肉眼看不到DNA序列中,为大规模、长期且不经常访问的数字档案信息提供一种实用的存储技术。2016年,微软研究院和华盛顿大学合作将超过2.0×108字节的数据信息编码到DNA分子中,同时,微软公司已计划于2020年建立基于DNA分子的数据存储系统[11]。2017年,纽约基因组中心和哥伦比亚大学联合开发了一种高度可靠的DNA喷泉算法,该方法可接近每个核苷酸存储的信息的理论最大值[12]。与此同时,国内外一些企业也陆续推出基于DNA数据存储的商业化服务。
本文以DNA数据存储技术为主线,阐述DNA数据存储的基本理论和工作流程,重点介绍DNA保存的方法与策略、信息安全与数据加密的研究进展,最后讨论DNA数据存储现阶段面临的主要挑战及发展趋势。
1 DNA数据存储的基本理论和工作流程
在自然界中,DNA是由4种碱基——腺嘌呤(A)、胞嘧啶(C)、胸腺嘧啶(T)和鸟嘌呤(G)按照特定顺序键合而成的大分子聚合物。通过A与T、C与G碱基互补配体的原则,可形成双螺旋的DNA双链分子结构,承载着亿万生命的海量遗传信息。DNA存储技术即是以人工合成的DNA为存储介质,按照一定编码规则将文本文档、图片和声音文件等数据转化为DNA序列,进行存储并完整读取的技术[13-14]。与传统的存储介质相比,DNA数据存储具有如下优势。首先,存储密度高。根据Shannon信息的定义,单个核苷酸可存储最大容量为0.25字节;通过换算为物理密度可知,1 g DNA可理论上存储4.6×1020字节的数据量,存储密度比目前主流的存储介质(如磁带、HDD或固态存储)高出多个数量级[15]。2019年,华盛顿大学报道文件存储密度可达1.7×1019字节/g。以此推测,大约10 kg DNA分子就可满足2025年全球数据总量存储要求(预计数据量为1.75×1023字节),所占体积与篮球大小相似[16]。其次,DNA数据存储易复制。虽然DNA分子存储的数据不需要经常复制保存,如果需要,亦可轻松完成。DNA可利用聚合酶链式反应(PCR)进行指数化复制,显著提高了数据的复制效率[17]。此外,与当前存储在HDD上在线数据不同,DNA数据存储不需要电力供应,耗能低,即可长时间稳定地保存数据信息[18]。
DNA数据存储的基本工作流程主要包括将数据信息编码为DNA序列(编码),根据序列合成DNA分子(写入),组织这些DNA分子进行长期保存和数据加密(保存和加密),检索和有选择地访问(随机访问),读取DNA序列(读出),将其转换为数字信息(解码)[19],如图1所示。
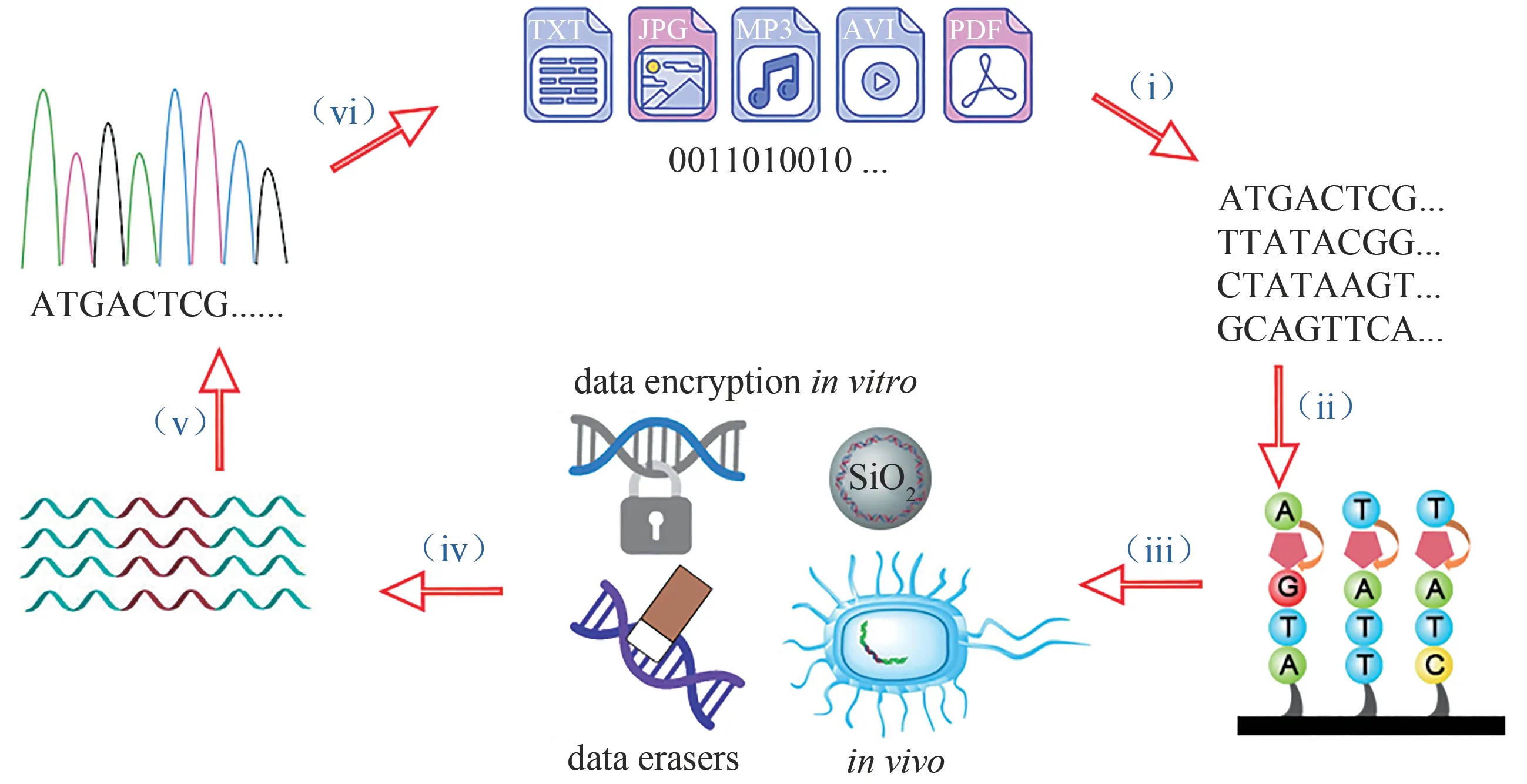
图1 DNA数据存储的基本工作流程[主要包括编码(i)、写入(ii)、保存和加密(iii)、随机访问(iv)、读出(v)与解码(vi)]Fig.1 Major steps of DNA data storage[including encode(i),write(ii),store and encrypt(iii),random access(iv),read(v)and decode(vi)]
1.1 编码
计算机中文本、图片、音频和视频等信息文件的保存与处理均采用以0、1为基元的二进制数,需按照一定的编码规则将其映射成以A、T、C、G四字符编码的DNA序列。虽然每种类型信息所用的模型有所差别,但其编码过程大致相似,一般包括数据压缩、引入纠错和转换DNA序列三个过程。
1.2 写入
根据编码的DNA序列完整无误地合成出来,制备一系列人工DNA分子。目前,合成DNA分子的主要方法有芯片法[20]、PCR法[21]、柱式合成法[22]和酶促合成法[23]。
1.3 保存和加密
选择合适的载体将合成DNA分子进行保存。目前合成DNA保存方法主要是细胞外保存法和细胞内保存法。将DNA分子包埋在特殊的基质中隔绝空气和水分,可有效延长其保存时间[24]。在延长DNA保存时间的同时,增强信息存储的安全性,也是DNA数据存储重要的研究方向。本文将在后续部分重点介绍DNA保存方法的研究进展,并简要总结信息安全与数据加密的最新研究成果。
1.4 随机访问
随机访问是用于计算机科学中选定信息的读取,是DNA数据存储可行性的一个关键特征。为了避免读取整个存储系统,需要设计索引方法来提取特定的DNA序列。目前索引方法主要是基于PCR的引物扩增[25]和基于修饰特定DNA序列的磁珠法[26]。
1.5 读出与解码
通过DNA测序仪器读取DNA序列。DNA测序法主要有“大规模并行合成测序法”[27]“DNA纳米球测序法”[28]和“纳米孔测序法”[29]。前者技术成熟,错误率低,但周期较长;后者技术在发展中,错误率相对较高,但能够实时读出。根据测定的DNA序列,设计解码程序,转换为二进制数据,最后还原成计算机文件。
2 DNA保存的方法与策略
与其他存储介质相比,DNA数据存储一个巨大的优势在于可以长久保存数据。近期考古学研究还可以对来自30万年前熊和人类的线粒体DNA测序[30-31]。然而,天然未受保护的DNA是一种非常脆弱的生物分子,易水解或者被氧化,有着特征性的半衰期[32-33]。它们的半衰期与存储温度和DNA链长密切相关,低温和防水环境可显著提高其稳定性,如常温下30个碱基DNA半衰期有500年,而-5℃存储DNA化石的半衰期可延长至15.8万年[34]。为了更有效地进行数据长期存储,国内外科研工作者发展了多种DNA保存的方法与策略,主要分为细胞外保存法和细胞内保存法。
2.1 细胞外保存法
DNA溶液通常在室温下可以稳定3~6个月,4℃可以稳定大约1年,而-20℃冷冻可以延长至2年[35]。将DNA分子保存在溶液中的最大优势是可以随机索引,可支持多次信息读取[25,36-37]。为防止样品在运输、存储、处理过程中发生降解,人们通常把DNA分子进行冻干处理进行保存。这样不但适用于长期稳定保存样本,并且能快速完整地回收样本。通过评估商品化Biomatrica、自制海藻糖和聚乙烯醇3种材质塑料孔板对DNA分子的稳定效果,发现Biomatrica塑料孔板在室温和56℃对DNA保护效果最佳;海藻糖孔板更适合对DNA短期56℃条件保存[38]。华盛顿大学科研人员[39]将脱水DNA斑点保存在玻璃上,研发一种基于数字微流控的可扩展DNA数据存储的方法(图2)。脱水DNA的斑点可以密集地排列在微流控设备上,回收过程不受邻近斑点影响。他们还将1.0×1012字节的数据存储在DNA的单个斑点中,并使用此方法成功实现了检索。另外,DNA还可以吸附存储在一种特殊材质滤纸中,可保护DNA分子在36个月内不被降解[40]。最近,瑞士联邦理工学院科学家发现碱土金属盐可增强干粉状DNA的稳定性,即使在相对湿度为50%、高DNA负载量(质量分数大于30%)情况下也具有显著稳定DNA的功能,可方便地进行数据的随机访问和信息读取[41]。
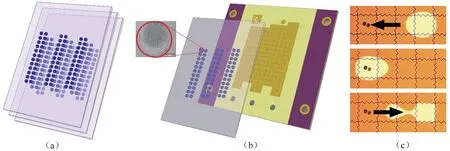
图2 基于数字微流控的高密度干粉DNA数据存储[(a)干粉DNA保存在玻璃板上;(b)将玻璃板置于数字微流控装备上便于检索数据,插图为一个斑点成像图,比例尺为275μm;(c)夹在玻璃板与电极间的水滴被激活,并移动到斑点DNA下进行补水[39]]Fig.2 High density dehydrated DNA data storage with digital microfluidic retrieval[(a)Dehydrated DNA stored on glass cartridges;(b)Cartridge was loaded onto digital microfluidic device to retrieve data,and inset showed photo‐graph of an actual magnified spot,scale bar:275μm;(c)A water droplet sandwiched between cartridge and electrodes was actuated to move under spotted DNA for rehydration[39]]
DNA分子可以在骨骼残骸或者沉积物中保存长达数十万年之久,主要得益于外层密集的骨骼残骸或者沉积物将DNA与环境中的水分和活性氧隔绝开。受此启发,国内外科研人员先后报道了一系列封装DNA分子的方法,使DNA分子得到更好的保护。不同介质保存DNA的情况见表1。DNA可包裹在二氧化硅颗粒中,模仿化石保护DNA免受侵蚀性环境(200℃、高浓度自由基)的影响,并利用氢氟酸对二氧化硅颗粒的刻蚀作用,可将DNA释放出来进行信息读取[42]。此外,借助同样的方法将DNA封装在二氧化硅颗粒,并采用纠错代码来纠正与存储相关的错误。由于DNA对热非常敏感,根据DNA衰败激活能(120~155 kJ/mol)[32,44]和70℃下DNA衰减曲线,可推断DNA保存在20℃的半衰期。通过加速老化试验(70℃和相对湿度50%)推测,数据信息可以在多种环境条件下保存DNA数千年。即使在70℃存放一周,原始信息可以无差错地恢复[24]。但是这些方法存在着DNA负载量低的缺陷,只有0.7%,降低了DNA数据存储密度。为了增加存储密度,瑞士联邦理工学院科研人员利用层层自组装技术,将DNA和阳离子聚合物聚乙烯亚胺(PEI)交替包覆在磁性颗粒表面,并在最外层包裹一层硅壳,制备了一种高DNA负载的磁性纳米颗粒。该方法大幅度提升了DNA存储密度,密度最高可达155 ng/cm,质量负载率为7.8%(不包裹硅)和3.4%(包裹硅)[43]。最近,瑞士联邦理工学院与以色列科学家联合开发了一种信息存储结构,可用于创建具有嵌入式存储DNA信息编码的材料[45]。他们设计了“万物DNA”框架体系,将用于3D打印兔子的信息编码到DNA分子,封装在160 nm硅球内部,并进一步分散在热塑性聚酯中,用于兔子的3D打印。通过剪下兔子一小块材料,提取、扩增和测序,就可以获取兔子3D打印信息,完美地复制五代兔子。这些包裹在二氧化硅颗粒中保存DNA的方法,通常不支持可逆的信息释放与封装,以及多次的信息读取。除了二氧化硅纳米颗粒,还利用其他材料如磷酸钙和聚合物等进行封装保护[46-47],并且对DNA自组装纳米结构可以实现精准生物矿化[48-51]。例如,如图3所示,上海交通大学樊春海院士团队[51]利用核酸框架结构为模板和静电吸附作用为驱动力,成功地制备出几何形状高度可控的磷酸钙纳米晶体。由于外层磷酸钙的隔绝保护作用,DNA的稳定性大大增强,同时保留核酸框架的结构信息,有效增加其在细胞内的转运效率,进一步拓宽了DNA框架结构的应用范围。与细胞内保存法相比,细胞外保存法因成本低、耐久性好、可延展性等优势更具有实用性。

图3 DNA框架结构诱导合成磷酸钙[51]Fig.3 Schematic Illustration for preparation of CaP templated by DNA-framework[51]

表1 不同DNA保存介质的比较Tab.1 Comparison of different DNA storage media
2.2 细胞内保存法
与体外DNA保存法相比,细胞内保存法可利用细胞内高效的DNA复制、校对和长链DNA修复机制,提供高效的随机访问路径和实时记录生物事件[25,52]。天然和工程化的DNA靶向和修饰酶常被用作DNA数据存储的写入工具。例如,天津大学元英进教授团队[53]利用酿酒酵母体内组装系统对长序列数据信息进行组装,可实现生物信息的低成本、高保真存储。天津大学齐浩教授团队[54]发现携带大量寡核苷酸池的细菌混合培养物是一种稳定的信息存储媒介,能够将4.45×105字节的数据文件存储在细菌内。根据写入机制不同,目前报道的研究工作大致分为两类。一类是利用重组酶进行细胞内数据存储。借助重组酶,信息被储存在一个特定的基因组位置[55-56]。例如,利用不同的重组酶在大肠杆菌内存储了1.375字节的记忆阵列,证实了重组酶可以分层并用于永久记录转录逻辑门的瞬时状态[57]。通过控制重组酶方向性,将可重写的信息数据存储在活细胞中[58]。需要说明的是,对于基于重组酶的DNA数据存储,在特定的位置需要一个特定的重组酶,因此,数据存储的密度不高,且与宿主基因组的接口有限,最终导致数据写入效率比较低。另外一类是利用CRISPR-Cas体系进行DNA数据存储[59-60]。其中,最受关注的是Cas9蛋白[61],它是一种可编程DNA切割酶,被用于定位由多个相同靶点组成的DNA地址[62-63]。这种方法除大规模记录细胞谱系信息外,还可通过将Cas9表达与细胞信号耦合来记录模拟信号,如将非二进制的基因突变(数据信息)记录到DNA中[64]。各种其他核酸酶也具有类似的功能,如CRISPR相关的核酸内切酶Cpf1、锌指蛋白核酸酶(ZFNs)以及类转录激活因子效应物核酸酶(TALENs)[65-69]。与上面介绍的基于重组酶的体系相比,此功能使模拟数据的存储密度更高,同时具有更直接的与宿主生理学接口。目前细胞内保存法还不能充分利用细胞基因组,而且还存在着由于遗传不稳定性而造成信息丢失的风险[70-71]。为了减少这种遗传不稳定性带来错误信息富集,德国汉堡工业大学[72]提出了一种用于活细胞正交信息编码、具有错误自我检测功能的三基块编码方案(SED3B)。SED3B采用一种全新的方法在小数据基块中添加错误检测的碱基,可与DNA分子固有的冗余部分相结合,进行有效的纠错。试验结果表明,SED3B在大肠杆菌中编码信息可连续复制超过12 000年,仍能提供可靠的数据结果。
3 信息安全与数据加密
3.1 数据加密
在大数据时代,信息数据安全扮演着尤为重要的角色。信息加密技术可用于防范未经授权的非法访问。密码术和隐写术是信息加密常用的两种策略,前者是使信息无法被外人理解,后者则是隐藏信息的存在[73-74]。传统的密码术和隐写术由于当今数学和计算机技术的介入很容易被破解,失去原有的信息加密效果[75-76]。随着生物学和信息学的发展,人们开始利用生物分子寻求新型加密技术[77],如蛋白质、适配体、细菌被用来保护信息安全[78-81]。然而,在这些研究中,信息安全很大程度上依赖于固定的生物分子反应模式,一旦对手发现相关的解密方式,其安全性将受到严重的威胁。发展新型安全可靠的密码体制,是信息安全和数据加密的研究重点和难点。
3.2 DNA密码
1994年,美国南加州大学利用DNA计算来解决一个NP完全问题的试验,标志着信息时代进入一个新阶段[82]。DNA密码是随DNA计算发展而产生的一项新兴技术[83-84],它主要是利用现代生物技术,以DNA分子为载体,充分发挥DNA固有的高存储密度和高并行性等优势,从而实现加密、认证和隐写等密码学功能[85-88]。DNA密码体系主要有3种方式。
(1)一次一密。1999年,美国杜克大学[89]利用映射替代和异或的方法提出了一次一密的密码体系。映射替代法是根据定义的映射关系将一定长度的DNA明文序列替换为对应的DNA密文序列;而异或法是利用光刻技术和荧光标记技术进行DNA明文和密文序列的异或运算。
(2)DNA隐写术。它的原理是利用大量的无关信息隐藏加密后的DNA信息,只有根据事前双方约定的信息,才能找到正确的DNA链,并获取隐藏的信息[87]。1999年,美国纽约西奈山医学院[7]发明了一种基于DNA的密码术策略来隐藏秘密消息,他们将第二次世界大战中著名的一条信息隐写在DNA微点中,并利用PCR技术成功地将其提取出来。最近,上海交通大学樊春海院士研究团队[90]开发了一套基于DNA折纸的分子加密系统。如图4所示,发送者Alice首先将文本信息HEY按字母顺序编码成盲文图案,然后进一步加密为杂交若干个生物素修饰短链的骨架链。接收者Bob通过加入订书链,将骨架链折叠为含有生物素化图案的DNA纳米结构。再加入链酶亲和素,使盲文图案在原子力显微镜(AFM)下可识别读出,最终获得文本信息。该方法实现了加密术与隐写术的完美整合,大大超越当前基于计算问题加密协议的限制。同时,还可通过对DNA折纸不同区域位点的定义以及DNA折纸间的特异性识别,实现完整性保护和访问控制的功能。

图4 基于DNA折纸的分子加密系统工作流程[90]Fig.4 Workflow of DNA origamicryptography for secure information communication[90]
(3)PCR引物作为密钥。该方法是基于DNA二元串对数据信息进行编码,然后混入大量相似DNA二元串,只有悉知PCR反应中引物序列的接收方,才能提取正确的消息[88]。由于引物信息的泄露可能导致消息不安全,中国科学院上海生命科学研究院[91]最近通过将特定引物(真实密钥)与非特定引物(假密钥)混合或将真实密钥与3′-端冗余序列连接来开发预密钥,然后利用CRISPR/Cas12a技术切割假密钥或去除3′-端冗余序列,从而产生用于信息提取的真实密钥,可更好地保护DNA编码数据的存储和传输。此外,上海交通大学左小磊教授团队[92]将特异序列DNA包裹在碳纳米管表面,研发了一种新型管状核酸,利用形成的特征高度和距离的不同模式对碳纳米管进行二维编码,并且可利用金纳米颗粒进行视觉解码。
3.3 数据清除
数据清除是保障信息安全重要的一环,将数据快速从存储设备中擦除,达到保护机密信息数据的目的。然而,DNA分子的良好化学稳定性对DNA存储中高度机密的数据清除提出了新的挑战。
破坏DNA分子的传统方法主要有使用紫外线照射[93]、DNase I[94]、>200℃的高温[95-96]以及氧化剂等[97]。这些销毁DNA分子方法各不相同,但一般难以在没有专门设备以及在合理的时间内完成。美国莱斯大学研究人员[98]报道了一种基于亚稳定杂交的DNA数据存储系统,可通过简单的加热过程快速永久擦除数据信息。如图5所示,在该存储系统中,每个文件地址都包含一个真实消息和至少一条错误消息,并且真实信息通过与“真实标记”寡核苷酸的杂交来区分。DNA杂交体的稳定性对温度非常敏感,只要温度高于其解链温度,DNA杂交体立即解离。原始真实信息通过加热解离(95℃加热5 min),将永远无法恢复。最近,美国北卡罗莱纳州立大学[99]报道了一种由T7启动子和单链突出域(ss-dsDNA)组成的动态DNA数据存储系统。该ss-dsDNA系统可通过从DNA转录信息而不破坏它来实现可重复的信息访问,同时,还可以进行DNA数据存储中文件的锁定与解锁、重命名以及删除等操作,为具有多种功能的信息存储奠定了坚实的基础。
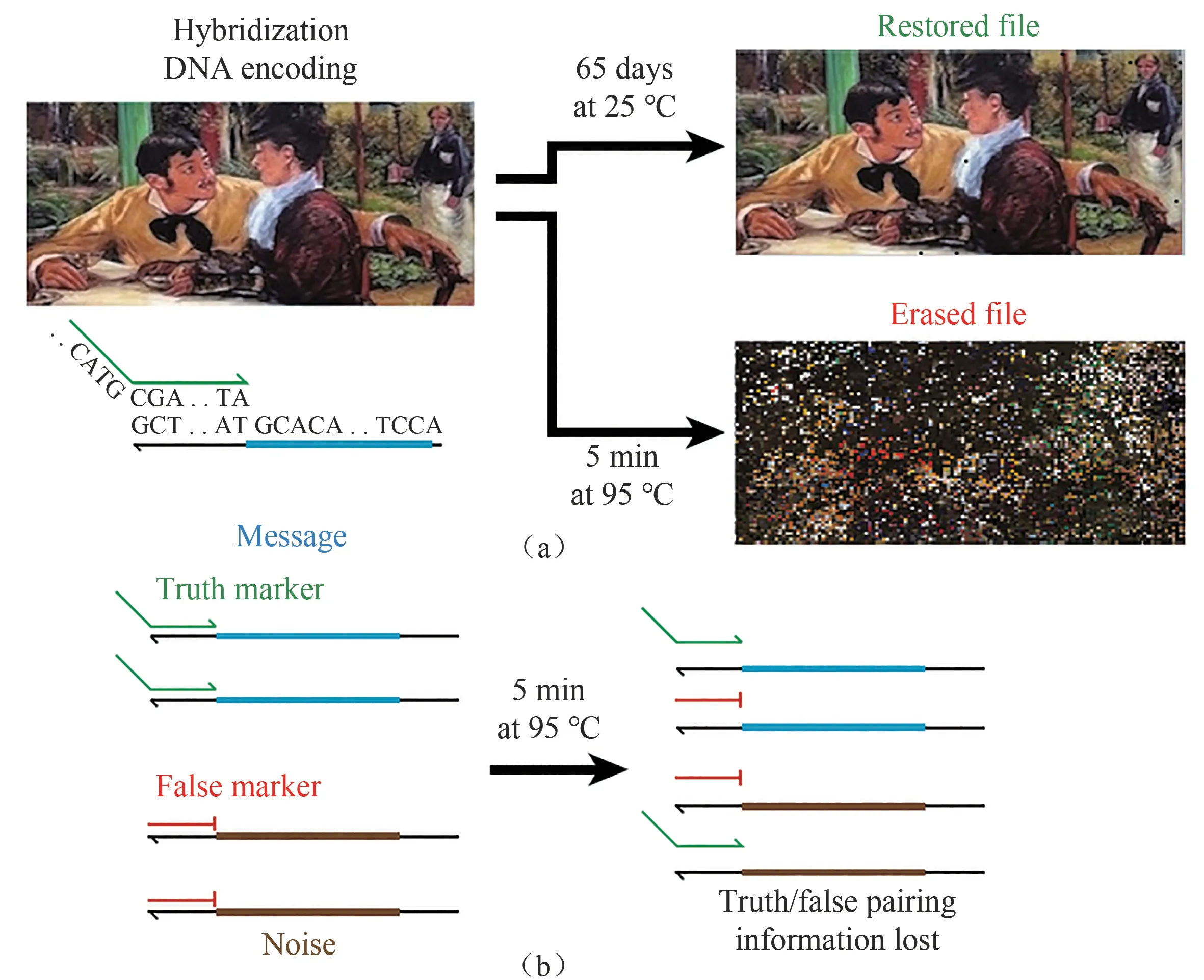
图5 基于亚稳定杂交的DNA数据存储系统的原理[(a)图片文件被编码为DNA序列,可以在室温下稳定地长时间保存,但是在暴露于95℃时会被永久快速擦除;(b)基于DNA杂交的真实信息编码[98]]Fig.5 Metastable hybridization-based DNA data storage[(a)Image file was encoded as DNA sequences,which could be stored steadily at room temperature for long periods of time,but was permanently and quickly erased when exposed to 95℃.(b)Truthful information encoding based on DNA hybridization[98]]
4 展望
以互联网与全球化普及为重要标志的信息革命正逐步改变人与数据间的相处方式,使人类社会步入了大数据时代。爆炸式增长的信息数据量与存储空间和技术不足的矛盾也日益突出。因其存储密度大、出错率低、能耗低等优势,DNA作为极具发展潜力的新一代存储介质,有望替代当今磁盘和光盘等主流存储方式。近些年,DNA数据存储技术的研发已取得较大进展。本文以DNA数据存储为主线,阐述了DNA数据存储的基本理论和工作流程,重点综述DNA保存方法与策略最新研究成果和信息安全与数据加密的研究进展。如图6所示,DNA数据存储除了用于文件存档和数据加密外,还被扩展用于验证物品真实性的分子标签和生物计算[100]。如国际上多个研究小组开发了一种基于DNA分子鉴定的方法,用于油品条形码的标签[101],还应用于含水层的环境示踪[102]。
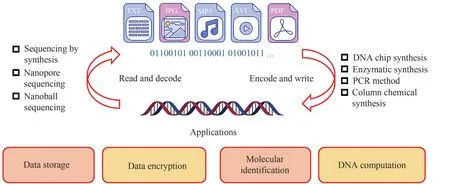
图6 DNA数据存储的主要工作流程及其应用Fig.6 Process overview and applications of DNA Data Storage
目前,诸多研究进展仍停留在实验室水平,主要原因如下:
首先,信息的写入和读取成本仍然很高,存储数据的效率太低。近期的研究发现创建存储1.0×106字节的数据大约需要花费3500美元[10,103-104]。尽管在过去10年中,DNA合成和测序成本下降了几个数量级,但DNA合成与测序技术本身存在的瓶颈问题,导致成本降低的步伐有所放缓。此外,先进的编码和解码算法同样需要提升DNA合成与测序方面技术要求,才能实现产业级的DNA数据存储。
其次,随机访问是信息存储另一个必需的功能。如何高效快速地从整个存储系统中读取某一数据文件是一个挑战。通常使用特异性引物进行PCR,以选择性访问存储在DNA中的某一特定的信息。美国华盛顿大学[25]利用强大的纠错码和算法,可以独立检测35个文件,而不相互干扰和产生错误。尽管这些技术复杂、缓慢且昂贵,但这些技术还是迈向DNA数据存储随机访问的第一步。
最后,虽然细胞外保存法和细胞内保存法都取得了一定的研究进展,但它仍然离便捷的实际应用还有很长的距离。兼顾存储持久性与简易读取数据,擦除数据与信息重写,自动化集成化写入、储存、读取数据等方面,面临着诸多挑战,还有很多技术难点有待突破。特别是,提升DNA数据存储效率、存储读取高度集成自动化以及数据加密新策略等将是今后DNA数据存储研究的重要发展方向。DNA数据存储凭借自身的独特优势受到国内外越来越多的科研工作者关注和重视,虽然现阶段仍存在着一些难点和挑战,但大量科学试验表明DNA作为一种新型的数据存储介质,无论是在存储容量、持久性上,还是在可扩展性上,都远胜于现有的存储介质。相信随着合成生物学的不断发展,这些挑战和技术难题将逐步得到解决,DNA数据存储将成为未来最有应用潜力的新型存储方式,引领人类社会进入更便捷、更智能的新时代。
