基于转录组的非洲菊PLC基因家族鉴定与进化分析
2021-07-20王秀美倪珊珊李乾玉李小芳林争春陈裕坤林玉玲赖钟雄杜迎刚
王秀美 倪珊珊 李乾玉 李小芳 林争春 陈裕坤 林玉玲 赖钟雄 杜迎刚
摘 要:磷脂酶C(phospholipase C,PLC)广泛参与植物的生命活动和代谢过程,在抵御真菌感染的过程中发挥重要作用。本研究基于非洲菊转录组测序数据,鉴定出13个非洲菊PLC基因家族成员,其中NPC有3个,PI-PLC有10个。非洲菊PLC的蛋白序列长度在98~594 aa之间,蛋白理论分子量为11 410.25~67 998.85 kDa,等电点为4.74~9.59,均为不稳定亲水蛋白。亚细胞定位预测结果显示,13个成员分别定位于细胞壁、细胞膜、细胞质、叶绿体和细胞核中,表明不同成员功能上可能存在多样性。进化树分析表明,13个非洲菊PLC蛋白可分为2个亚组,进化关系较近的蛋白结构组成相似。非洲菊PLC家族成员在拟南芥中的同源蛋白PLC2、PLC4、AT2G40116可相互作用。非洲菊转录组的FPKM值分析表明,GjPLC2、GjPLC3、GjPLC8、GjPLC10在非洲菊中表达量高,感病植株中变化大,可能在抵御真菌感染过程中发挥重要作用。研究结果可为进一步研究非洲菊PLC基因的生物学功能提供依据。
关键词:非洲菊;磷脂酶C;基因家族;进化分析
中图分类号:S682.1 文献标识码:A
Abstract: Phospholipase C (PLC) is widely involved in the life activities and metabolism processes of plants, and plays an important role in the process of resisting fungal infection. Based on the results of the transcriptome sequencing in Gerbera jamesonii, 13 members of G. jamesonii PLC gene family, including 3 NPC and 10 PI-PLC were identified in the study. The sequences of G. jamesonii PLC proteins ranged from 98 to 594 aa in length, theoretical molecular weight varied from 11 410.25 to 67 998.85 kDa, isoelectric point in the range of 4.74 to 9.59, and all were unstable hydrophilic proteins. The prediction results of subcellular localization showed that the 13 members were located in the cell wall, cell membrane, cytoplasm, chloroplast and nucleus, indicating that there might be diversity in functions of different mem-bers. Evolutionary trees analysis showed that 13 PLC proteins of G. jamesonii were divided into two subgroups, the structure and composition of proteins with relatively close evolutionary relationship were similar. G. jamesoniis homo-logous protein PLC2, PLC4 and AT2G40116 in Arabidopsis thaliana can interact with each other. The FPKM value analysis of G. jamesonii transcription showed that GjPLC2, GjPLC3, GjPLC8 and GjPLC10 were highly expressed in G. jamesonii and changed greatly in infected plants, which may play an important role in preventing fungal infection. These results could provide the basis for further study of the biological function of G. jamesonii PLC genes.
Keywords: Gerbera jamesonii Bolus; Phospholipase C; gene family; evolutionary analysis
DOI: 10.3969/j.issn.1000-2561.2021.05.010
磷脂酶C(phospholipase C,PLC)是一種普遍存在于动物、植物和微生物细胞中的脂质水解酶,根据作用底物的不同可主要分为磷脂酰肌醇特异性磷脂酶C(phosphoinositide-specific PLC,PI-PLC)和非特异性磷脂酶C(non-specific PLC,NPC)[1-2]。PI-PLC蛋白由N端的EF手型结构域、C端的C2结构域及中间的X、Y结构域组成[2],主要水解磷脂酰肌醇4,5-二磷酸(phosphatidyfinositol4, 5-bisphosphate,PIP2),生成水溶性三磷酸肌醇(inositol 1,4,5-trisphosphate,IP3)和脂溶性二酰甘油(diacylglycerol,DAG)[3]。而NPC蛋白只包含一个具有磷脂酶活性的磷酸酯酶区,主要水解磷脂酰乙醇胺(phosphatidylethanolamine,PE)、磷脂酰胆碱(phosphatidylcholine,PC)和磷脂酰甘油(phosphatidylglycerol,PG),生成DAG和相应磷酸盐[4]。
研究表明,由PI-PLC蛋白水解产生的IP3进入细胞液后能触发细胞内的Ca2+存储库释放Ca2+,进而诱发一系列生理生化反应;进入细胞质后则会经过多磷酸肌醇激酶(Inositol polyphosphate kinase,IPK2/IPMK)逐级磷酸化形成六磷酸肌醇(inositol hexaphosphate,IP6),IP6作为信号分子促进Ca2+释放,参与植物对病原菌胁迫的应答[5]。由PI-PLC和NPC蛋白水解产生的DAG在甘油二酯激酶(diacylglycerol Kinase,DGK)的催化作用下迅速磷酸化生成磷脂酸(phosphatidicacid,PA),并通过与一些蛋白激酶、脂激酶和NADPH氧化酶等之间的相互作用,调节植物细胞生长、非生物胁迫和细胞死亡等生理过程[6]。因此,PLC基因可以参与植物细胞的分裂、生长和分化以及细胞的免疫等诸多细胞信号转导过程。例如,在转基因烟草和拟南芥中过量表达AtPLC5会引起拟南芥和烟草叶片早衰[7];下调番茄SlPLC4和SlPLC2表达水平则损害其对叶霉病菌[8]、灰霉病[9]以及真菌诱导的木聚糖酶[10]的免疫功能。
非洲菊(Gerbera jamesonii Bolus)是世界五大鲜切花之一[11],原产于非洲南部的德兰士瓦,具有花朵大、色彩艳丽、花期长、切花产量高等优点,被广泛用作切花和盆栽观赏。但根腐病的泛滥严重限制了非洲菊产业的健康发展,据不完全统计,由隐地疫霉(Phytophthora cryptogea)引起的根腐病一般情况下的发病率为20%~30%,严重时可达80%以上[12],因此,选育优良的非洲菊抗病品种是生产上亟待解决的问题。目前,关于非洲菊的PLC基因研究还未见报道,本研究基于非洲菊健康植株和感染根腐病病原菌植株的转录组测序数据,在转录组水平上对PLC基因家族进行鉴定和生物信息学分析,同时研究非洲菊PLC基因家族在健康和感病植株中的差异表达情况,为进一步研究非洲菊PLC基因的生物学功能提供基础数据和参考。
1 材料与方法
1.1 材料
非洲菊(健康植株和感染根腐病病原菌植株)转录组数据由本实验室课题组测序获得(SRA:SRR9937065),模式植物拟南芥(Arabidopsis thaliana)的PLC家族蛋白序列下载自TAIR数据库(https://www.arabidopsis.org),水稻(Oryza sative L.)的PLC家族蛋白序列下载自RiceData数据库(http://www.ricedata.cn/gene)和NCBI的GeneBank数据库(http://www.ncbi.nlm.nih.gov/)。
1.2 方法
1.2.1 非洲菊PLC基因家族的鉴定 利用NCBI的BLAST(https://blast.ncbi.nlm.nih. gov/Blast.cgi)将非洲菊转录组测序数据进行初步筛选,得到PLC家族相关蛋白序列。利用在线软件HMMER(https://www.ebi.ac.uk/Tools/hmmer/)和SMART(http://smart. embl-heidelberg.de/)对其蛋白序列保守结构域逐个进行鉴定分析,去除掉重复和不含有PLC家族特征结构域的序列。
1.2.2 非洲菊PLC家族蛋白序列分析 利用在线软件ProtParam(http://web.expasy. org/protparam/)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_ automat.pl)、Plant- mPLocServer(http://www.Csbio. sjtu.edu.cn/bioin- f/plant-multi)分析预测非洲菊PLC蛋白序列的理化性质、二级结构进行及亚细胞定位。利用在线软件MEME Suite 5.1.1(http:// meme-suite.org/tools/meme)分析非洲菊PLC家族蛋白的保守基序,设置基序数目为30。
1.2.3 非洲菊PLC家族蛋白序列比对及进化树构建 利用多序列对比软件Clustalx对鉴定得到的非洲菊PLC蛋白序列与拟南芥、水稻的PLC蛋白进行多序列比对,使用MEGA 7.0软件,采用邻接法,Bootstrap设为1000,其他参数为默认值下建系统发育树,并使用在线软件iTOL(https:// itol.embl.de/upload.cgi)对进化树进行美化。
1.2.4 非洲菊PLC家族蛋白互作分析 将非洲菊PLC蛋白序列导入在线软件STRING(https:// string-db.org/),以模式植物拟南芥为参考序列构建蛋白互作网络,预测非洲菊PLC家族成员在拟南芥中的同源蛋白的互作关系。
1.2.5 非洲菊PLC基因家族在健康和感病植株中差異表达的FPKM值分析 利用非洲菊转录组测序数据,分析PLC基因在健康和感病植株中的差异表达。以非洲菊健康植株和感病植株每千个碱基的转录每百万映射读取的reads(reads per ki-lobase of exon model per million mapped reads,FPKM)为基因转录丰度,利用Heml软件做热图。
2 结果与分析
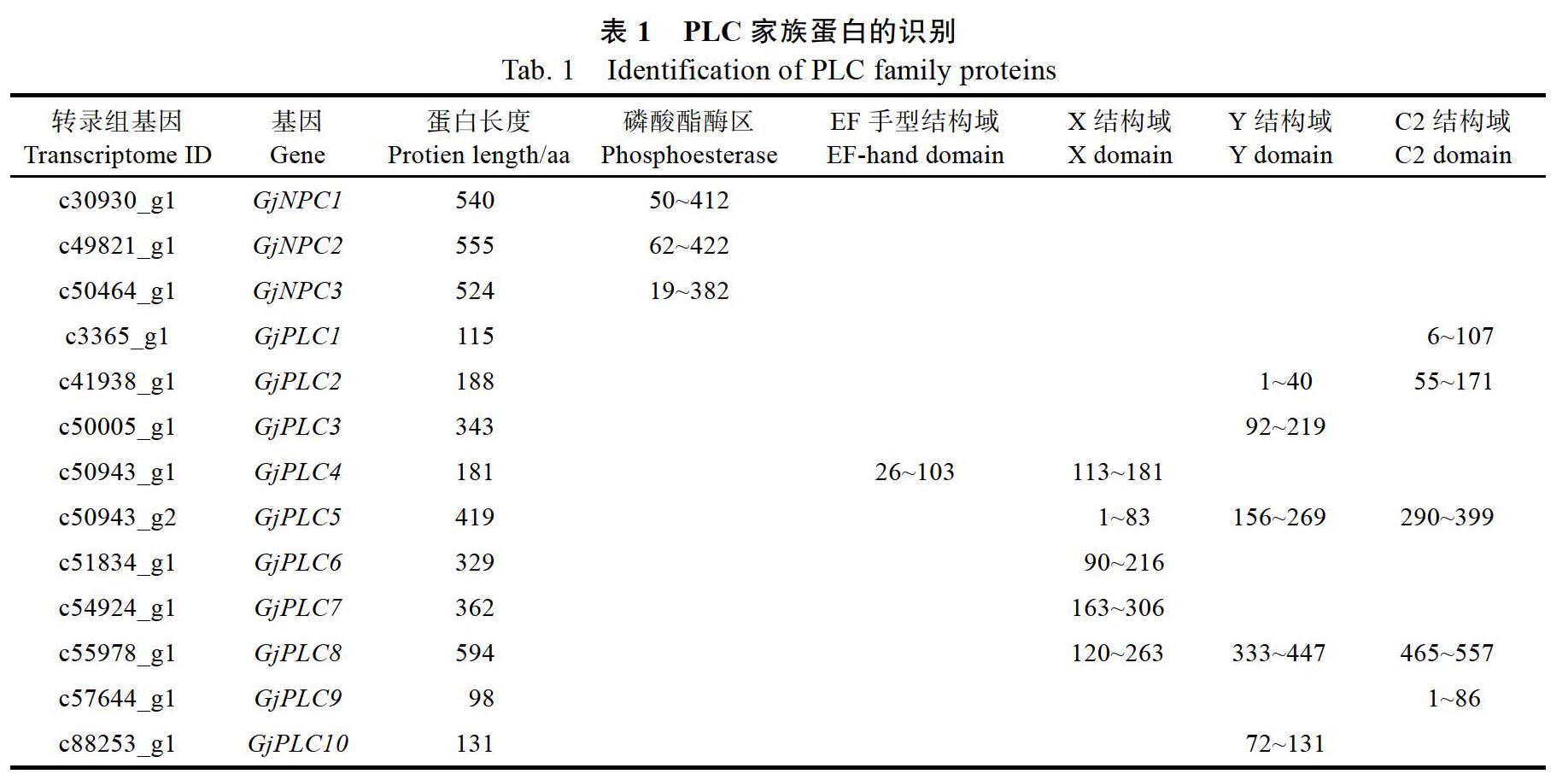
2.1 非洲菊PLC家族基因的鉴定
基于本课题组前期获得的非洲菊转录组测序数据,利用NCBI的BLAST初步筛选得到23条PLC家族相关的蛋白序列。PI-PLC蛋白包含EF手型结构域、X结构域、Y结构域和C2结构域,而NPC蛋白中只含有一个磷酸酯酶区,通过HMMER和SMART对其蛋白序列保守结构域进行筛选,去除掉重复和不含有PLC家族特征结构域的序列,最终得到13条非洲菊PLC蛋白序列(表1)。其中NPC有3条,PI-PLC有10条。为方便研究,根据PLC基因的种类和转录组基因ID的大小统一非洲菊PLC基因编号(GjNPC1~ GjNPC3,GjPLC1~GjPLC10)。
2.2 非洲菊PLC家族蛋白序列分析
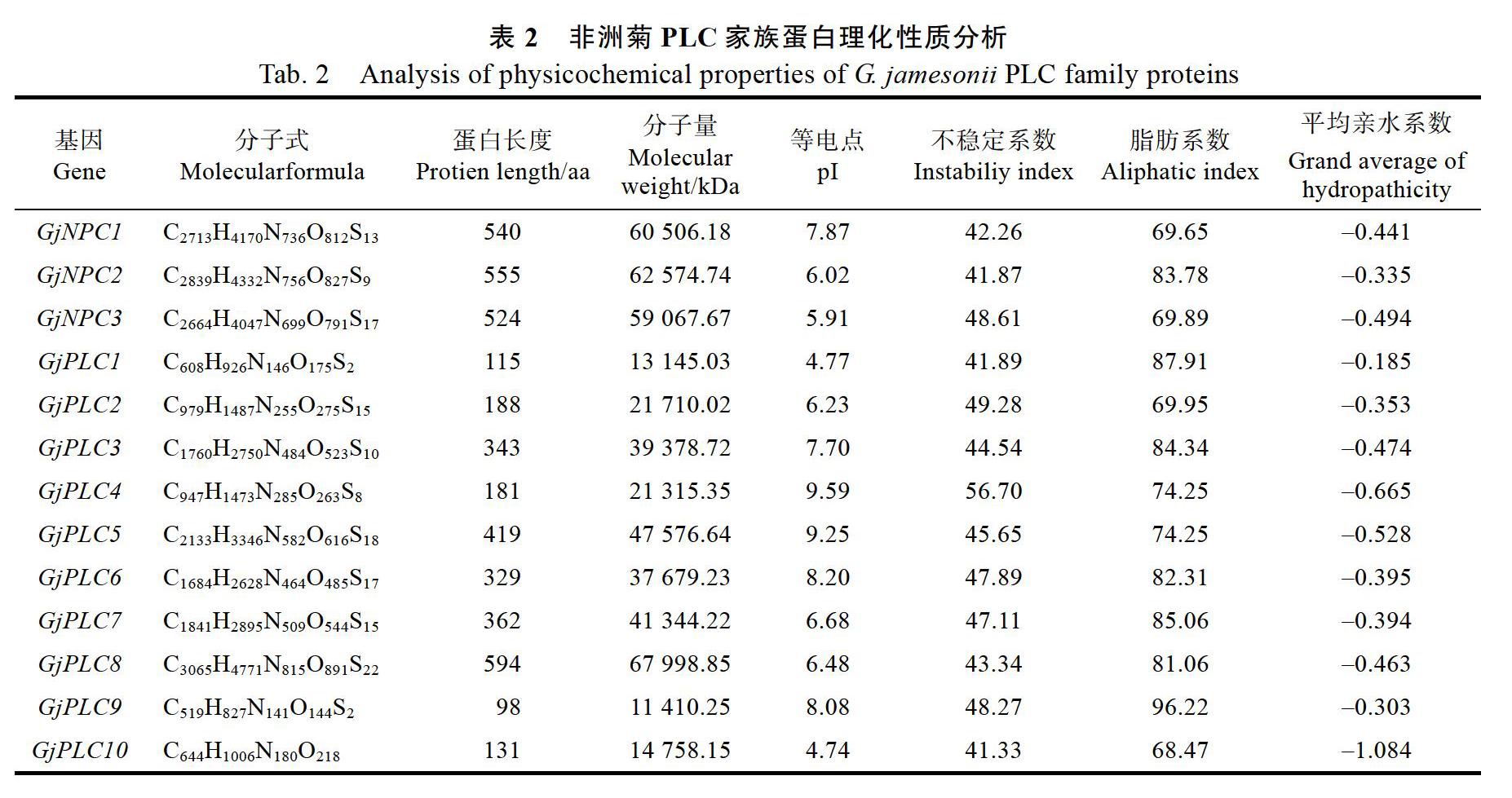
通过PLC蛋白序列的理化性质分析(表2),发现非洲菊PLC蛋白序列的长度在98~594 aa之间,预测蛋白理论分子量为11 410.25~ 67 998.85 kDa,等电点为4.74~9.59。预测理论等电点小于7的酸性蛋白有7个,大于7的碱性蛋白有6个。所有蛋白的不稳定系数都大于40,表明它们均为不稳定蛋白;平均亲水系数都为负值,表明他们均为亲水蛋白。
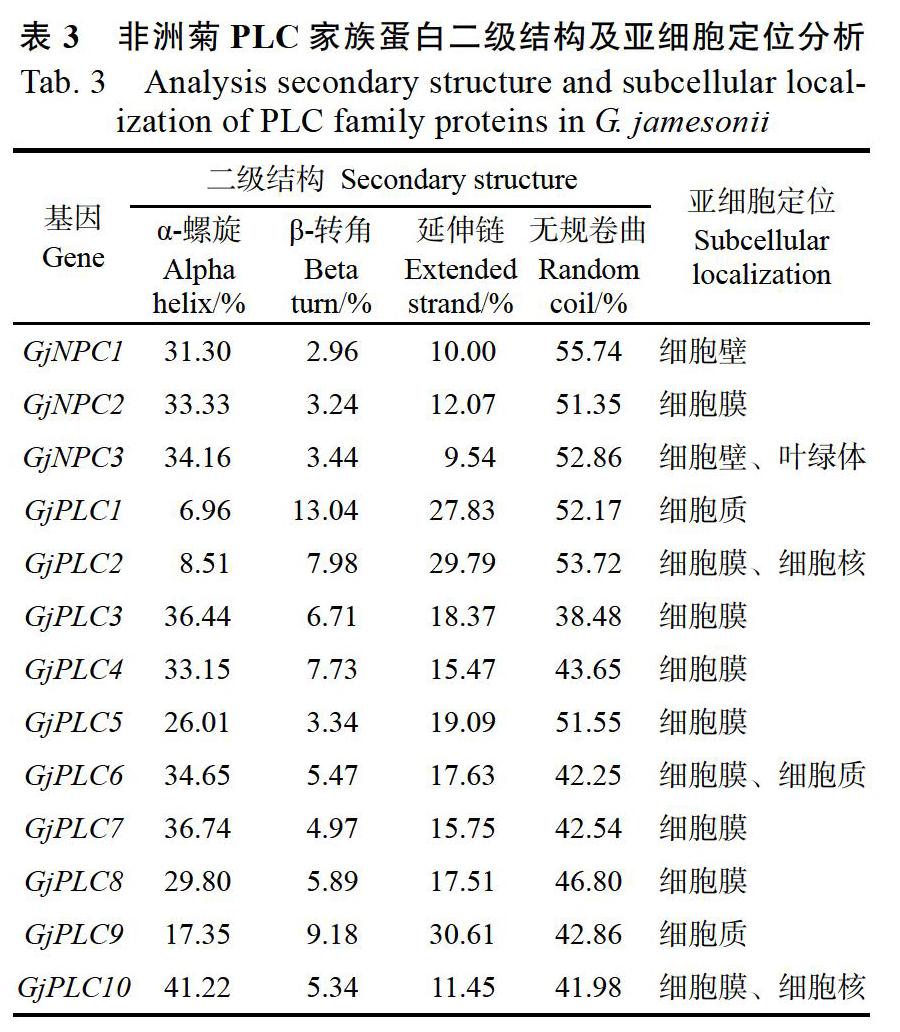
通过在线软件SOPMA分析预测非洲菊PLC蛋白序列的二级结构(表3),发现非洲菊PLC家族蛋白中无规则卷曲结构所占比例最高,除了GPLC1,β-转角结构所占比例最低。在NPC中所有α-螺旋结构所占比例都高于延伸链结构,PI-PLC中则有7个α-螺旋结构所占比例高于延伸链结构。
通过在线软件Plant-mPLoc Server分析预测非洲菊PLC蛋白序列的亚细胞定位(表3),發现13个非洲菊PLC蛋白在细胞壁、细胞膜、细胞质、叶绿体和细胞核中均有定位。其中,GjNPC1定位于细胞壁,GjNPC2、GjPLC3、GjPLC4、GjPLC5、GjPLC7、GjPLC8定位于细胞膜,GjNPC3定位于细胞壁和叶绿体,GjPLC1与GjPLC9定位于细胞质,GjPLC2与GjPLC10定位于细胞膜和细胞核,GjPLC6定位于细胞膜和细胞质。
利用在线软件MEME对非洲菊的PLC家族蛋白保守基序进行分析,本研究共预测分析了30个保守基序(图1)。发现在13个非洲菊PLC蛋白序列中,motif4、motif9、motif13、motif1、motif17相连的基序可能为磷酸酯酶区,motif21、motif25、motif23、motif3、motif14、motif5、motif16和motif27、motif30、motif26相连的基序可能为X结构域,motif12、motif11、motif2、motif15相连的基序可能为Y结构域,motif8、motif10、motif6相连的基序可能为C2结构域。
上述结果表明,非洲菊PLC蛋白无论在理化性质、二级结构、还是亚细胞定位方面都有较大的差异,暗示非洲菊PLC基因具有不同生物学特征。
2.3 非洲菊PLC家族蛋白的进化分析
由图2可知,13个非洲菊PLC蛋白成员可以分为GroupⅠ、GroupⅡ2个亚组,其中3个NPC蛋白分布在GroupI中,10个PI-PLC蛋白分布在GroupⅡ中。GroupⅡ分为a、b、c 3部分,GroupⅡa中有2个非洲菊PLC成员(GjPLC1、GjPLC9)和3个水稻PLC成员(OsPLC1、OsPLC2、OsPLC3);GroupⅡb中有4个非洲菊PLC成员(GjPLC2、GjPLC5、GjPLC8、GjPLC10)、4个拟南芥PLC成员(AtPLC1、AtPLC2、AtPLC3、AtPLC7)和1个水稻PLC成员(OsPLC4);Group IIc中有4个非洲菊PLC成员(GjPLC3、GjPLC4、GjPLC6、GjPLC7)和4个拟南芥PLC成员(AtPLC4、AtPLC5、AtPLC8、AtPLC9)。非洲菊PLC蛋白与双子叶植物拟南芥在进化上亲缘较关系较近,与单子叶植物水稻关系较远,且进化关系较近的非洲菊PLC蛋白结构组成相似,可能具有相似的生物学功能。
2.4 非洲菊PLC家族蛋白互作分析
非洲菊PLC家族成员在拟南芥中的同源蛋白的互作关系预测分析显示(表4、图3),13个非洲菊PLC家族成员在拟南芥中同源性较高的蛋白有7个,分别为NPC1、NPC3、NPC6、PLC2、PLC4、AT2G40116、AT4G38690。其中,NPC1、NPC3、NPC6可以共存并相互结合,NPC1、PLC2可以共同表达,PLC2、PLC4、AT2G40116可以相互结合、催化并产生反应。表明拟南芥的PLC2、PLC4、AT2G40116蛋白家族成员可相互作用,而与其同源性较高的非洲菊蛋白家族成员GjPLC1、GjPLC2、GjPLC4、GjPLC5、GjPLC7、GjPLC8、GjPLC9、GjPLC10是否可以相互作用还需进一步验证。
2.5 非洲菊PLC基因家族在健康和感病植株中差异表达的FPKM值分析
从图4可知,在非洲菊健康和感病植株中,GjPLC2、GjPLC3、GjPLC8、GjPLC10的表达量较高,GjNPC1、GjNPC3、GjPLC1、GjPLC6、GjPLC7、GjPLC9表达量较低。与健康植株相比,GjNPC2、GjPLC1、GjPLC2、GjPLC6、GjPLC10在感病植株中上调,其中GjPLC2、GjPLC10上调量较高;GjNPC1、GjNPC3、GjPLC3、GjPLC4、GjPLC5、GjPLC7、GjPLC8、GjPLC9在感病植株中下调,其中GjPLC3、GjPLC4、GjPLC5、GjPLC8下调量较高。GjPLC2、GjPLC3、GjPLC8、GjPLC10在非洲菊中表达量高,且与健康植株相比,感病植株中的表达量变化大,推测其在抵御真菌感染过程中发挥重要的生物学作用。
3 讨论
3.1 非洲菊PLC基因家族成员多且可能具有不同的生物学功能
自1980年Irvine等[13]得到一种能分解磷脂酰肌醇的PLC后,水稻和拟南芥等模式植物中PLC相继被人们克隆了出来,近年来随着生物技术的发展,越来越多的PLC基因家族在多种不同的植物中被预测或鉴定出来,其中,拟南芥[14]中有14个(9个PI-PLC,5个NPC),水稻[15]中有9个(4个PI-PLC,5个NPC),棉花[16]中有15个(9个PI-PLC,6个NPC),亚麻芥[17]中有10个,玉米[18]中有9个、蓖麻[19]中有6个;杨树[20]、兰花[21]、桃[22]中只鉴定出PI-PLC,分别有7个、3个、5个。本研究基于实验室课题组获得的非洲菊转录组测序数据,鉴定出13个非洲菊PLC基因,其中NPC有3个,PI-PLC有10个。
本研究对非洲菊PLC蛋白序列的亚细胞定位分析,发现其在细胞壁、细胞膜、细胞质、叶绿体和细胞核中均有定位,且大部分位于细胞膜,这正好符合了Hong等[2]证实的大部分PLC定位在细胞膜上的结论。对非洲菊PLC家族的蛋白结构进行分析发现,NPC只包含一个磷酸酯酶区,PI-PLC则含有EF手型结构域、X结构域、Y结构域和C2结构域4个结构域,这与模式植物拟南芥[14]和水稻[15]的保守结构相同。对非洲菊分子进化树分析,发现非洲菊PLC蛋白可分为2个大亚组,GroupⅠ为NPC,GroupⅡ为PI-PLC,其中GroupⅡ分为a、b、c 3个小亚组,进化关系较近的非洲菊PLC蛋白结构组成相似,可能具有相似的生物学功能。另外,对非洲菊PLC蛋白序列的分析,发现这13个蛋白在理化性质、二级结构等方面有较大的差异,表明非洲菊PLC基因可能具有不同生物学特性和功能。
3.2 非洲菊PLC基因家族可能参与植物免疫反应
越來越多的研究表明,磷脂酶C在植物先天免疫反应中起核心作用,可参与植物生物胁迫的适应过程[23],例如水稻[24-25]中的OsPI-PLC1在病原菌胁迫下,可通过Ca2+信号通路参与植物抗病反应;拟南芥[26]感染病菌后AtPLC5蛋白可以参与信号胁迫传导过程,增强抵御病原菌入侵能力;烟草[27]中的NtPLC4过量表达后,烟草植株抗病性显著提高等。本研究利用非洲菊转录组测序数据中的FPKM值,分析非洲菊PLC基因家族在健康和感病植株中的差异表达发现,与健康植株相比,感病植株中有5个基因上调,8个基因下调。其中,GjPLC2、GjPLC3、GjPLC8、GjPLC10的表达量高,表达量变化大,推测其在抵御真菌感染过程中发挥重要的生物学作用。
参考文献
[1] 黄 冬, 吴 燕. 植物磷脂酶C的功能研究进展[J]. 生命科学, 2017(6): 59-65.
[2] Hong Y, Zhao J, Guo L, et al. Plant phospholipases D and C and their diverse functions in stress responses[J]. Progress in Lipid Research, 2016, 62: 55-74.
[3] Rupwate S D, Rajasekharan R. Plant phosphoinosi-tide-specific phospholipase C: an insight[J]. Plant Signaling and Behavior, 2012, 7(10): 1281-1283.
[4] Pokotylo I, Pejchar P, Potock? M, et al. The plant non-specific phospholipase C gene family. Novel competitors in lipid signalling[J]. Progress in Lipid Research, 2013, 52(1): 62-79.
[5] Lemtiri-Chlieh F, Macrobbie E A, Webb A A, et al. Inositol hexakisphosphate mobilizes an endomembrane store of calcium in guard cells[J]. Proceedings of the National Academy of Sciences of the United States of America, 2003, 100(17): 10091-10095.
[6] Wang X, Devaiah S P, Zhang W, et al. Signaling functions of phosphatidic acid[J]. Progress in Lipid Research, 2006, 45(3): 250-278.
[7] Zhang J W, Xia K K, Yang Y M, et al. Overexpression of Arabidopsis phosphoinositide-specific phospholipase C5 induces leaf senescence[J]. Plant Cell, Tissue and Organ Culture, 2015, 120(2): 585-595.
[8] Vossen J H, Abd-El-Haliem A, Fradin E F, et al. Identifica-tion of tomato phosphatidylinositol-specific phospholipase-C (PI-PLC) family members and the role of PLC4 and PLC6 in HR and disease resistance[J]. The Plant Journal, 2010, 62(2): 224-239.
[9] Gonorazky G, Guzzo M C, Abd-El-Haliem A M, et al. Si-lencing of the tomato phosphatidylinositol-phospholipase C2 (SlPLC2) reduces plant susceptibility to Botrytis cinerea[J]. Molecular Plant Pathology, 2016, 17(9): 1354-1363.
[10] Gonorazky G, Ramirez L, Abdelhaliem A, et al. The tomato phosphatidylinositol-phospholipase C2 (SlPLC2) is required for defense gene induction by the fungal elicitor xylanase[J]. Journal of Plant Physiology, 2014, 171(11): 959-965.
[11] 李有清, 吕长平, 胡春梅. 非洲菊种质资源研究进展[J]. 湖南农业科学, 2014(5): 53-55.
[12] 郝向阳, 林 觅, 孙雪丽, 等. 福建非洲菊产区根腐病病原菌的分离与鉴定[J]. 福建农业学报, 2018, 33(4): 391-395.
[13] Irvine R F, Letcher A J, Dawson R M. Phosphatidylinositol phosphodiesterase in higher plants[J]. Biochemical Journal, 1980, 192(1): 279-283.
[14] Mueller-Roeber B, Pical C. Inositol phospholipid metabol-ism in Arabidopsis. Characterized and putative isoforms of inositol phospholipid kinase and phosphoinositide-specific phospholipase C[J]. Plant physiology, 2002, 130(1): 22-46.
[15] Singh A, Kanwar P, Pandey A, et al. Comprehensive ge-nomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in rice[J]. PLoS One, 2013, 8(4): e62494.
[16] Zhang B, Wang Y, Liu J Y. Genome-wide identification and characterization of phospholipase C gene family in cotton (Gossypium spp.)[J]. Science China Life sciences, 2018, 61(1): 88-99.
[17] 李晓薇, 戢舒涵, 赵 旭, 等. 亚麻芥未成熟胚中磷脂酶基因家族的转录组学分析[J]. 中国油料作物学报, 2018, 40(6): 793-800.
[18] 董洁静, 徐晶宇, 林俊俊, 等. 玉米PLCs基因家族鉴定及表达谱分析[J]. 植物生理学报, 2018, 54(6): 1018-1028.
[19] 李麗丽, 赵 永, 常如慧, 等. Lm型蓖麻花序中PLC基因家族的表达量及生物信息学分析[J]. 分子植物育种, 2018, 16(20): 6604-6615.
[20] 张杰伟, 丁莉萍, 陈亚娟, 等. 杨树磷酸肌醇特异性磷脂酶C基因家族鉴定与分析[J]. 福建农业学报, 2016, 31(11): 1181-1186.
[21] 胡广隆, 陈亚娟, 魏建华, 等. 兰花磷酸肌醇特异性磷脂酶C基因家族生物信息学分析[J]. 西南农业学报, 2017, 30(10): 2218-2223.
[22] 张杰伟, 任 飞, 张中保, 等. 桃磷酸肌醇特异性磷脂酶C基因家族鉴定与分析[J]. 江苏农业学报, 2017, 33(1): 185-190.
[23] Abd-El-Haliem A M, Joosten M H. Plant phosphatidylinositol-specific phospholipase C at the center of plant innate immunity[J]. Journal of Integrative Plant Biology, 2017, 59(3): 164-179.
[24] 张卫东. 水稻甘油二酯激酶基因OsDAGK1以及磷脂酶C/甘油二酯激酶途径在抗病反应中的作用[D]. 杭州: 浙江大学, 2004.
[25] Song F. Molecular cloning and characterization of a rice phosphoinositide-specific phospholipase C gene, OsPI-PLC1, that is activated in systemic acquired resistance[J]. Physiological and Molecular Plant Pathology, 2002, 61(1): 31-40.
[26] 马文文. 拟南芥AtPLC5在植株对病原菌胁迫响应中的作用[D]. 北京: 中央民族大学, 2006.
[27] 王 蕾. 烟草磷脂酶C基因的表达模式和过表达NtPLC4转基因烟草提高抗病性研究[D]. 泰安: 山东农业大学, 2011.
责任编辑:白 净
