生防菌株BacillusvelezensisZ全基因组测序分析
2021-07-20高圣风徐毕爽陆大倩刘爱勤苟亚峰孙世伟王政孟倩倩
高圣风 徐毕爽 陆大倩 刘爱勤 苟亚峰 孙世伟 王政 孟倩倩

摘 要:生防菌株Bacillus velezensis Z对胡椒瘟病等多种植物病害具有良好的生防效果;全基因组测序能够为其分子机理研究和开发应用提供信息基础。本研究开展该菌株全基因组测序,并进行比较基因组学和抑菌次生代谢产物合成基因簇预测研究。结果表明:B. velezensis Z菌株的基因组中含有1条4 054 780 bp大小的环形染色体DNA和1个17 122 bp大小的环形质粒,总基因组的GC含量为46.24%,共编码基因4173个;包含27个rRNA,86个tRNA基因,34个sRNA;含有串联重复序列179个,其中13个微卫星DNA,138个小卫星DNA;通过比较基因组学分析,结果发现该菌株与贝莱斯芽孢杆菌模式菌株FZB42同源性极高,与枯草芽孢杆菌模式菌株168之间具有一定的遗传距离;同时发现B. velezensis Z基因组中共编码次生代谢产物合成基因簇13个,其中8个与表面活性素(surfactin)、泛革素(fengycin)、溶杆菌素(bacilysin)、macrolactin H、bacillaene、difficidin、plantazolicin、amylocyclicin等已知基因簇完全相似或高度相似,其他5个基因簇皆功能未知。总之,本研究揭示了B. velezensis Z的全基因组遗传信息,明确其与贝莱斯芽孢杆菌和枯草芽孢杆菌的比较基因组学关系,预测了抑菌产物合成编码基因簇,为该生防菌株及其抑菌产物的机理研究和开发应用奠定基础。
关键词:贝莱斯芽孢杆菌;全基因组测序;比较基因组学;次生代谢产物;抑菌
中图分类号:S435.73 文献标识码:A
Abstract: Bio-control strain Bacillus velezensis Z shows good effects on the control of various diseases, especially good for the black pepper Phytophtora foot rot disease. The whole genome sequencing is an important basis to further study the molecular mechanism. In this study, the whole genome of the strain was completely sequenced using both Illumina HiSeq 4000 and PacBio RS II, and the gene function annotation, analyses of comparative genomics and secondary metabolite biosynthetic gene clusters were performed using blast alignment tools. The complete genome sequence of B. velezensis Z was consisted of a circular 4 054 780 bp chromosome and a 17 122 bp plasmid with GC content of 46.24%, 4173 identified genes, 27 rRNA, 86 tRNA, 34 sRNA, 179 tandem repeat regions including 138 minisatellite DNA and 13 microsatellite DNA. The genome showed highly homologous with the model strain B. velezensis Z FZB42 by the analyses of core/pan gene and gene family. B. velezensis Z harbored 13 secondary metabolite biosynthetic gene clusters, including 8 clusters involved in the synthesis of surfactin, plantazolicin, macrolactin H, bacilysin, fengycin, difficidin, amylocyclicin, bacillaene, and others functionally unknown. In conclusion, the genetic information, comparative genomics and antibiotics synthesis gene clusters of the B. velezensis Z were revealed in this study, and it would be useful for further research and utilization of the strain.
Keywords: Bacillus velezensis; whole genome sequencing; comparative genomics; secondary metabolite; antibiotic
DOI: 10.3969/j.issn.1000-2561.2021.05.003
根際促生细菌(plant growth-promoting rhi-zobacteria,PGPR)天然生存于植物根际,具有拮抗病原菌、促进植株生长、诱导植株抗病、修复根际环境等多种功效,是当前实施“减肥、减药”的有效支撑手段[1-2]。芽孢杆菌(Bacillus spp.)具有广普高效、容易培养、耐逆境、耐储藏等特点,是国内外研究和应用最多的生防菌类群。次生代谢产物是芽孢杆菌发挥生防功效的重要手段。目前已发现芽孢杆菌能够产生多种对植物有益的次生代谢产物,包括非核糖体途径(non-ribosomal peptide synthesis,NRPS)合成的脂肽类化合物[3-4]、聚酮合酶合成途径(polyketide synthase,PKS)合成的聚酮类化合物[5-7],以及核糖体途径(ribosomal peptide synthesis,RPS)合成的含噻唑(啉)线性肽类物质(linear azol(In)E-containing peptides,LAP)[8]、细菌素(bacteriocin)[9]、硫肽类物质(thiopeptide)[10]、萜烯类物质(terpene)[11]等。芽孢杆菌分泌的有益次生代谢产物种类繁多,全基因组测序是认识和利用生防菌株的重要基础。
生防菌株B. velezensis Z由本研究室从胡椒叶片中分离获得,在盆栽试验中对胡椒瘟病的防效达83%[12],具有良好的生防应用前景。该菌株曾被认为是枯草芽孢杆菌[13]。本研究对B. velezensis Z进行全基因组测序和精准鉴定,并基于全基因组序列开展比较基因组学分析及抑菌产物合成基因簇研究,为该生防菌株的后续研究和利用提供基础。
1 材料与方法
1.1 材料
生防菌株B. velezensis Z由本研究室从感染胡椒瘟病的胡椒叶片组织中分离获得,保存于中国微生物保藏管理委员会普通微生物中心(China General Microbiological Culture Col-lection Center, CGMCC),菌株保藏编号为No. 4844。
1.2 方法
1.2.1 全基因组测序 生防菌株B. velezensis Z在LB培养基中于37 ℃ 200 r/min条件下培养12 h 后,1000?g离心收集菌体,提取基因组总DNA,DNA纯度检测合格后,委托深圳华大基因股份有限公司进行de novo测序分析。测序采用第三代PacBio平台与第二代Illumina平臺相结合的测序技术,测序结果与COG(cluster of ortho?logous groups of proteins)、GO(gene ontology)等12个数据库比对进行基因功能注释[14]。
1.2.2 比较基因组学分析 从NCBI Genome上下载B.velezensis FZB42、B. subtilisW23、B. subtilis168等模式菌株的全基因组序列,与B. velezensis Z全基因组序列(包含质粒)一起进行以下比较基因组学分析。
(1)共有基因(Core Gene)和特有基因(Specific Gene)分析:参照Fu等[15]的方法,将生防菌B. velezensis Z的蛋白基因集与其他3个模式菌株的蛋白基因集一起采用CD-HIT 4.6.6软件进行聚类分析,将聚类获得的所有非冗余的基因集定为Pan Gene,提取聚类结果中4个菌株共有的基因集为Core Gene,单个菌株中特有的基因集为Specific Gene,Pan Gene中去除Core Gene为非共有基因集(Dispensable Gene)。根据各基因集在4个菌株基因组中的分布情况,绘制Dispensable Gene热图和Pan Gene文氏图以展现菌株间的聚类情况。参照Tannistha等[16]的方法,将Core/Pan分析结果用TreeBeST 1.9.2软件采用最大似然法(PHYML)计算,bootstraps参数设置为1 000次,构建系统进化树。
(2)基因家族(Gene Family)分析:基于4个菌株的全部基因,对蛋白序列进行BLAST比对分析去除冗余,然后将非冗余结果进行TreeFam聚类处理,再将聚类的基因家族进行多序列比对后转化成CDS区域的氨基酸多序列比对数据,最后用TreeBeST 1.9.2软件采用NJ法构建系统发育树[16-18]。
1.2.3 次生代谢产物合成基因簇分析 生防菌株B. velezensis Z的抑菌次生代谢产物合成基因簇分析方法与前人[14]报道相同,采用antiSMASH软件(https://antismash.secondarymetabolites.org/#!/ start)[19]在线预测。并结合NCBI BLAST比对分析结果,对预测的次级代谢产物合成基因簇进行分析。
2 结果与分析
2.1 全基因组测序结果
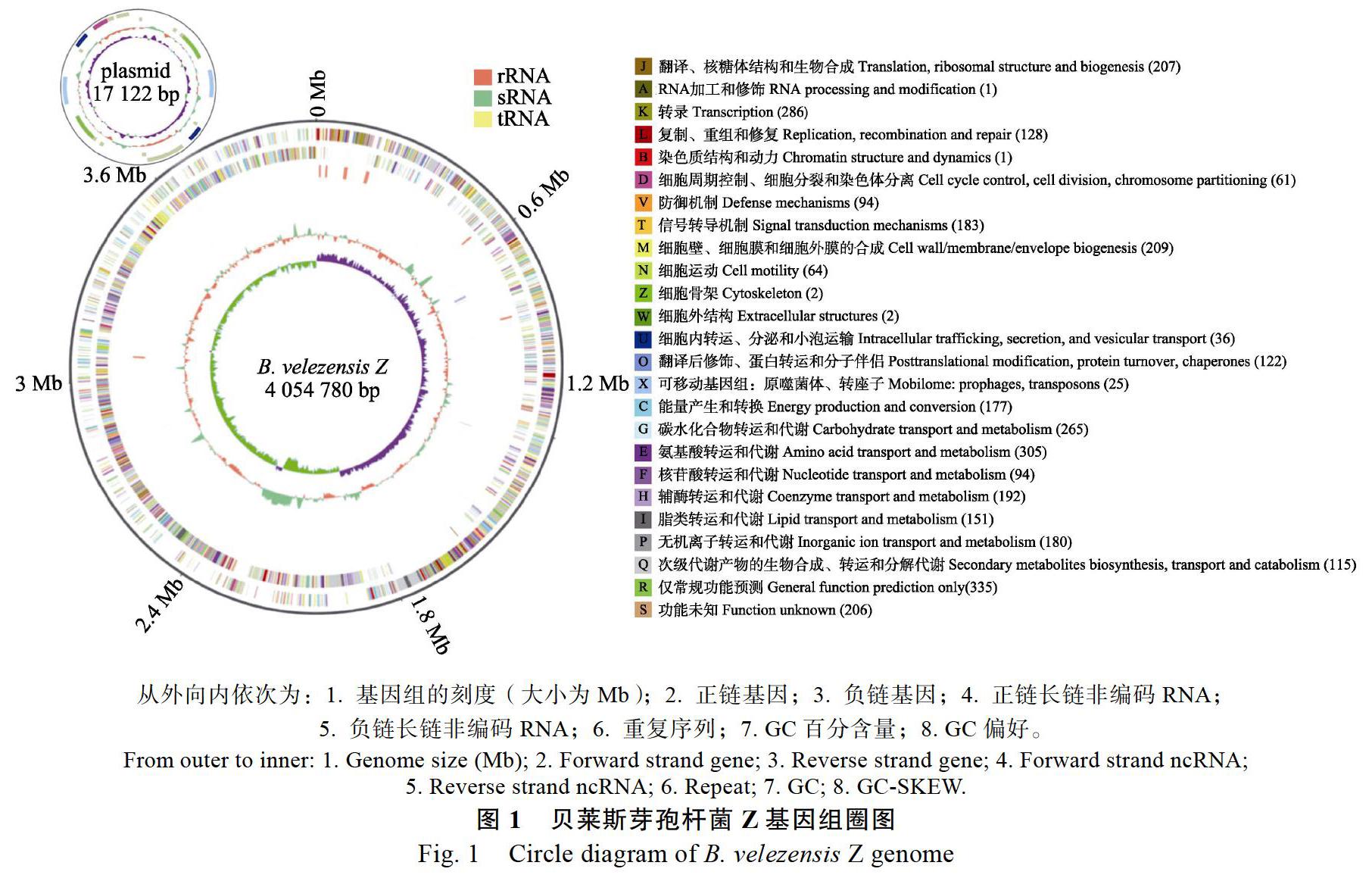
测序结果分析发现,生防菌株B. velezensis Z含有1条4 054 780 bp大小的染色体和1个17 122 bp大小的质粒,总基因组的GC含量为46.24%,共编码4173个基因,基因注释率为98.39%;其中染色体的GC含量为46.27%,编码基因4155个,含有27个rRNA、86个tRNA基因、34个sRNA;含有串联重复序列177个,其中13个微卫星DNA,138个小卫星DNA。另外,质粒DNA的GC含量为39.84%,编码基因18个,含有sRNA和串联重复序列各2个。
基于COG数据库注释结果构建基因组圈图(图1)。在预测的4173个基因中,(1)遗传信息类别:染色质结构与动力相关基因1个,RNA加工与修饰相关基因1个,核糖体结构、翻译和生物合成相关基因231个,转录相关基因286个,复制、重组和修复相关基因128个;(2)细胞相关类别:细胞周期控制、染色体分离和细胞分裂相关基因61个,细胞膜、细胞外膜和细胞壁的合成相关基因209个,细胞骨架相关基因2个,细胞外结构相关基因2个,细胞内转运、胞外分泌和小泡运输相关基因36个,细胞运动相关基因64个,防御机制相关基因94个,信号转导机制相关基因183个;(3)新陈代谢类别:翻译后修饰、分子伴侣和蛋白转运相关基因122个,可移动基因组序列(转座子、原噬菌体)相关基因25个,能量产生与转换相关基因177个,氨基酸转运与代谢相关基因305个,碳水化合物转运与代谢相关基因265个,脂类转运与代谢相关基因151个,核苷酸转运与代谢相关基因94个,辅酶转运与代谢相关基因192个,无机离子转运与代谢相关基因180个,次级代谢产物合成、转运与代谢相关基因115个;未知(Poorly)类别:仅常规功能预测的基因335个,功能未知基因206个。
2.2 比较基因组学分析
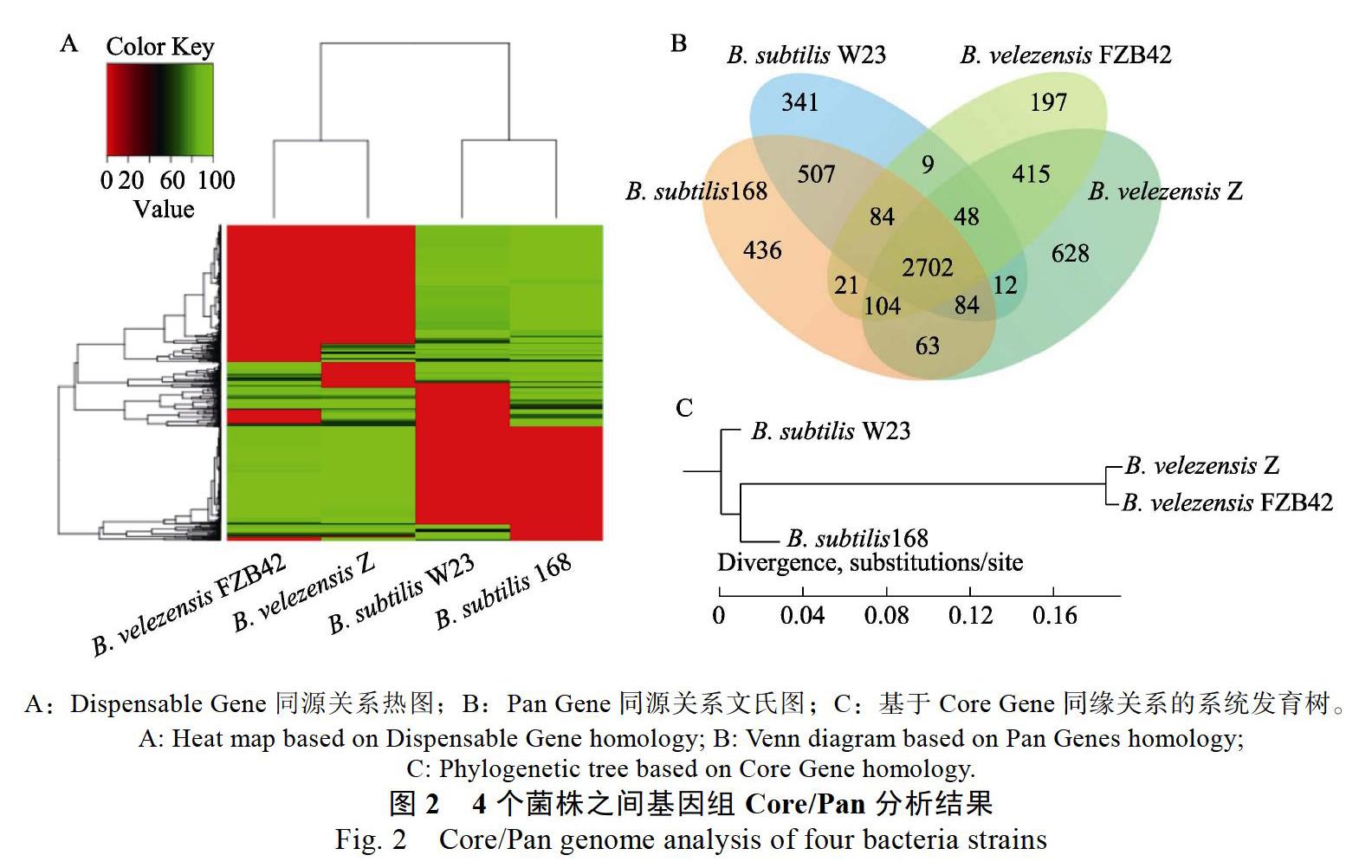
2.2.1 基于4个菌株全基因组序列的Core/Pan分析 通过Core/Pan分析,发现B. velezensis Z与3个模式菌株的所有非冗余的Pan Gene数量为5651个,长度为1 578 662 bp;其中共有的Core Gene数量为2702个,长度为823 203 bp;非共有的Dispensable Gene数量为1347个,长度为45967 bp;各菌株特有Specific Gene数量分别为628个、436个、341个和197个。4个菌株中,以B. velezensis Z的Specific Gene数目最多,散布于基因组中,部分基因功能预测与外排转运、核糖体、脂类转运代谢、膜蛋白、DNA修饰、次生代谢产物等相关,大部分基因功能未知(图2)。通过Dispensable Gene同源关系热图(图2A)、Pan Gene同源关系文氏图(图2B)和Core Gene系统发育树(图2C)可看出,B. velezensis Z的基因组与贝莱斯芽孢杆菌模式菌株FZB42最为相似,与另外2株枯草芽孢杆菌模式菌株168具有明显的遗传距离。
2.2.2 基于4个菌株全基因组序列的Gene Family分析 通过Gene Family分析,发现B. velezensis Z、B. velezensis FZB42、B. subtilis 168、B. subtilis W23等4个菌株能够聚类到Gene Family的基因数量分别为3685个、3907个、3706个和3565个,Gene Family数量分别为2710个、2816个、2715个和2601个;其中共有的Gene Family数量为2777个,各菌株特有的Gene Family数量分别为6个、11个、7个和3个(图3A、图3B)。通过Gene Family系统发育树可看出,B. velezensis Z菌株与B. velezensis FZB42菌株聚类到同一分支,与另外2株枯草芽孢杆菌之间的遗传距离约为0.20(图3C)。
2.3 基于全基因组序列的次生代谢产物预测分析
通过antiSMASH在线预测和NCBI BLAST比对分析,结果发现B. velezensis Z菌株共编码13个次生代谢产物合成基因簇,其中8个基因簇能够找到完全相似或相似度极高的已鉴定基因簇,另有5个基因簇未能找到已鉴定基因簇(表1)。由表1可看出,B. velezensis Z菌株编码的次生代谢产物有非核糖体途径的表面活性素(surfactin)、溶杆菌素(bacilysin)和泛革素(fengycin);聚酮合酶合成途径的bacillaene、macrolactin和difficidin;以及核糖体途径中Linear azol(in)e-containing peptides(LAP)类的plantazolicin和Bacteriocin类的amylocyclicin。在与已知基因簇比对结果中,表面活性素和Plantazolicin编码基因簇的相似度分别为82%和91%,其他基因簇的相似度均为100%。此外,B. velezensis Z菌株还可能编码5种未知的次生代谢产物,初步预测为2种聚酮类物质、2种萜烯类物质和1种硫肽类物质。在与其他菌株基因组比对结果中,仅有1个基因簇的相似度高达97%,其他基因簇均不高于70%。
3 讨论
细菌de novo测序是其分子机理研究的重要工具,广泛用于遗传进化关系研究、功能相关基因鉴定、工程菌改造等领域,能够为生防菌的分子机理研究及开发利用提供信息基础。本研究对生防菌株B. velezensis Z进行de novo全基因组测序,虽然该菌株的基因组(包括质粒)大小仅有3.88 Mb,但其各种功能元件非常丰富,编码基因长度占总基因组的88.71%。此外,B. velezensis Z中还携带一个大小为16.72 kb的质粒,共有22个ORF阅读框,其中ORF2~ORF9等8个ORF阅读框与ORF12~ORF19等阅读框的同源性高达100%,每组ORF阅读框中均编码Rap(ORF5/ ORF15)和Rep蛋白(ORF7/ORF17),表明该质粒的功能可能与细菌群体感应的Rap–Phr双组分调控系统相关[24],为研究和利用生防菌株的群体感应提供了材料和基础。
B. velezensis的種名和分类地位曾经历多次定名、改名、分类地位失效等波折,直至2016年才正式确立[25-27]。之前,B. velezensis Z曾被鉴定为枯草芽孢杆菌[12],2016年后被重新鉴定为贝莱斯芽孢杆菌(结果未发表)。本研究基于全基因组序列开展比较基因组学分析,GeneFamily分析和Core/Pan分析结果均表明,B. velezensis Z与贝莱斯芽孢杆菌模式菌株FZB42同源性极高,与2株枯草芽孢杆菌模式菌株168和W23的遗传距离均在0.16以上,再次证明该菌株是贝莱斯芽孢杆菌。
芽孢杆菌能够分泌丰富多样的次生代谢产物,是其发挥防病、促生长等对植物有益效果的重要前提。王晓宇等[22]发现B. subtilisBs-916菌株的全基因组中包含8个NRPS/PKS基因簇,可以合成fengycin、surfactin、bacillomycin L等物质,具有抑制细菌、真菌、形成生物膜、累积铁离子等作用。Wu等[23]发现B. velezensis NAU-B3的基因组中包含bacillomycin D、bacilysin、bacillaene等8个功能已知的次生代谢产物编码基因簇和1个功能未知基因簇,其长度占基因组总长度的8.5%,合成途径涵盖NPRS途径、PKS途径、PRS等途径。本研究发现生防菌B. velezensis Z编码的fengycin、bacilysin、macrolactin、bacillaene、difficidin、amylocyclicin等6种抑菌产物合成基因簇与已鉴定基因簇完全相同,surfactin和plantazolicin合成基因簇与已鉴定基因簇的相似度也高达82%和91%。这些次生代谢产物被报道具有拮抗植物病原细菌、真菌、病毒,诱导植株抗病性,积累铁离子,生物膜形成等有益功效[3-11],说明生防菌B. velezensis Z具有良好的应用价值。此外,从B. velezensis Z菌株的基因组中还预测出5种未知的次生代谢产物合成基因簇,为今后深度挖掘B. velezensis Z生防潜力和发现新型抑菌物质提供基础。
参考文献
[1] Cao Y, Pi H, Chandrangsu P, et al. Antagonism of two plant-growth promoting Bacillus velezensis isolates against Ralstonia solanacearum and Fusarium oxysporum[J]. Scien-tific Reports, 2018, 8(1): 4360.
[2] Venance C M, Alvina H R, Ayaz F, et al. Volatile organic compounds of endophytic Bacillus spp. have biocontrol ac-tivity against Sclerotinia sclerotiorum[J]. Phytopathology, 2018, 108(12): 1373-1385.
[3] Koumoutsi A, Chen X, Henne A, et al. Structural and func-tional characterization of gene clusters directing nonribo-somal synthesis of bioactive cyclic lipopeptides in Bacillus amyloliquefaciens strain FZB42[J]. Journal of Bacteriology, 2004, 186(4): 1084-1096.
[4] Rajesh R, Shen B, Yu C, et al. Molecular and biochemical detection of fengycin- and bacillomycin D-producing Bacil-lus, spp. antagonistic to fungal pathogens of canola and wheat[J]. Canadian Journal of Microbiology, 2007, 53(7): 901-911.
[5] Schneider K, Chen X, Vater J, et al. Macrolactin is the poly-ketide biosynthesis product of the pks2 cluster of Bacillus amyloliquefaciens FZB42[J]. Journal of Natural Products, 2007, 70(9): 1417-1423.
[6] Moldenhauer J, Daniel C D, Albert C, et al. The final steps of bacillaene biosynthesis in Bacillus amyloliquefaciens FZB42: direct evidence for beta, gamma dehydration by a trans-acyltransferase polyketide synthase[J]. Angewandte Chemie International edtion. in English, 2010, 49(8): 1465-1477.
[7] Wu L M, Wu H J, Chen L N, et al. Difficidin and bacilysin from Bacillus amyloliquefaciens FZB42 have antibacterial activity against Xanthomonas oryzae rice pathogens[J]. Scientific Reports, 2015, 5(1): 463-471.
[8] Scholz R, Molohon K J, Nachtigall J, et al. Plantazolicin, a novel microcin B17 streptolysin S-like natural product from Bacillus amyloliquefaciens FZB42[J]. Journal of Bacteriology, 2011, 193(1): 215-224.
[9] Scholz R , Vater J , Budiharjo A , et al. Amylocyclicin, a novel circular bacteriocin produced by Bacillus amylolique-faciens FZB42[J]. Journal of Bacteriology, 2014, 196(10): 1842-1852.
[10] Roessler C, Arndt H D. A master switch in thiopeptide biosynthesis[J]. Cell Chemical Biology, 2018, 25(2): 121-123.
[11] Sato T , Yamaga H, Kashima S, et al. Identification of novel sesterterpene/triterpene synthase from Bacillus clausii[J]. Chembiochem Communications, 2013, 14(7): 822-825.
[12] 桑利伟, 刘爱勤, 高圣风, 等. 防治胡椒瘟病生物农药筛选及其盆栽药效试验[J]. 热带作物学报, 2017, 38(5): 915-918.
[13] 桑利伟, 刘爱勤, 谭乐和, 等. 一种内生枯草芽孢杆菌: CN102250816A[P]. 2011-11-23.
[14] 高圣风, 刘爱勤, 桑利伟, 等. 胡椒瘟病生防菌Bacillus subtilis VD18R19全基因組测序及比较基因组学分析[J]. 热带作物学报, 2018, 39(10): 2021-2027.
[15] Fu L M, Niu B F, Zhu Z W, et al. CD-HIT: accelerated for clustering the next-generation sequencing data[J]. Bioinfor-matics, 2012, 28(23): 3150-3152.
[16] Tannistha N, Catherine O, Arvind P S, et al. A genomic survey of positive selection in Burkholderia pseudomallei provides insights into the evolution of accidental virulence[J]. PLoS Pathogens, 2010, 6(4): 1-15.
[17] Edgar R C. MUSCLE: multiple sequence alignment with high accuracy and high throughput[J]. Nucleic Acids Re-search. 2004, 32(5): 1792-1797.
[18] Edgar R C. MUSCLE: a multiple sequence alignment me-thod with reduced time and space complexity[J]. BMC Bio-informatics, 2004, 5(1): 113.
[19] Kai B, Simon S, Katharina S, et al. antiSMASH 5.0: updates to the secondary metabolite genome mining pipeline[J]. Nucleic Acids Research, 2019, 47(W1): 81-87.
[20] Jung J Y, Chun B H, Moon J Y, et al. Complete genome sequence of Bacillus methylotrophicus JJ-D34 isolated from deonjang, a Korean traditional fermented soybean paste[J]. Journal of Biotechnology, 2016, 219: 36-37.
[21] Qin Y, Han Y, Yu Y, et al. Complete genome sequence of Bacillus amyloliquefaciens L-S60, a plant growth-promoting and antifungal bacterium[J]. Journal of Biotechnology, 2015, 212: 67-68.
[22] 王晓宇, 罗楚平, 陈志谊, 等. 枯草芽胞杆菌Bs-916的全基因组分析[J]. 中国农业科学, 2011, 44(23): 4807-4814.
[23] Wu H, Qiao J, Blom J, et al. The rhizobacterium Bacillus amyloliquefaciens subsp. plantarum NAU-B3 contains a large inversion within the central portion of the genome[J]. Genome Announcements, 2013, 1(6): e00941.
[24] Qiao J, Tian D, Huo R, et al. Functional analysis and appli-cation of the cryptic plasmid pBSG3 harboring the RapQ–PhrQ system in Bacillus amyloliquefaciens B3[J]. Plasmid, 2010, 65(2): 141-149.
[25] Wang L, Lee F, Tai C, et al. Bacillus velezensis is a later heterotypic synonym of Bacillus amyloliquefaciens[J]. In-ternational Journal of Systematic and Evolutionary Microbiology, 2008, 58(3): 671-675.
[26] Dunlap C A, Kim S J, Kwon S W, et al. Phylogenomic anal-ysis shows that Bacillus amyloliquefaciens subsp. plan-tarum is a later heterotypic synonym of Bacillus methylotrophicus[J]. International Journal of Systematic and Evolutionary Microbiology, 2015, 65(7): 2104-2109.
[27] Dunlap C A, Kim S J, Kwon S W, et al. Bacillus velezensis is not a later heterotypic synonym of Bacillus amyloliquefa-ciens; Bacillus methylotrophicus, Bacillus amyloliquefaciens subsp. plantarum and ‘Bacillus oryzicola are later hetero-typic synonyms of Bacillus velezensis based on phyloge-nomics[J]. International Journal of Systematic and Evolutionary Microbiology, 2016, 66(3): 1212-1217.
責任编辑:黄东杰
