基于线粒体Cyt b基因序列的长江中上游草鱼野生和养殖群体遗传多样性比较研究
2021-07-15欧阳美张晓宇张富铁刘焕章
欧阳美,张晓宇,张富铁,刘焕章
(1.中国科学院水生生物研究所,中国科学院水生生物多样性与保护重点实验室,武汉 430072;2.中国科学院大学,北京 100049)
草鱼(Ctenopharyngodonidellus),俗称草鲩、草棒,属鲤形目(Cypriniformes)鲤科(Cyprinidae)雅罗鱼亚科(Leuciscinae)草鱼属(Ctenopharyngodon),与青鱼(Mylopharyngodonpiceus)、鲢(Hypophthalmictuthysmolitrix)、鳙(Aristichthysnobilis)并称为我国的“四大家鱼”。草鱼在我国其广泛分布于长江中下游江河湖泊水域,是我国重要的的淡水鱼类资源[1]。
在水利工程建设、环境污染、过度捕捞等多重作用下,草鱼自然资源逐渐衰退,主要表现在性早熟、成熟个体趋小、生长过缓、抗病抗逆性差等方面,因此其种质资源保护备受人们重视[2]。物种种质资源的保护需要评估其种质现状,最重要的是了解物种种群的遗传多样性水平。遗传多样性是物种繁衍、抵抗疾病和适应环境变化的物质基础;遗传结构反映了物种遗传变异的时空分布格局[3]。目前已有部分学者对草鱼群体遗传学方面做了相关的研究,例如汪焕等[4]和季晓芬等[5]利用微卫星标记对长江草鱼进行分析,发现长江草鱼群体的遗传多样性水平总体较高;汪焕等[4]发现4个养殖场的草鱼均存在近亲繁殖现象。
近年来,线粒体DNA Cytb基因,其进化速度适中、变异率相对较高,已作为探讨种间、种内遗传和分化变异的优良标记[6],被广泛应用于鱼类等动物的种群分化、系统发育等方面[7]。朱叶[2]、李树华等[8]、朱秀芳[9]以及翟东东等[10]通过线粒体Cytb基因序列对长江流域多个地点草鱼遗传多样性和遗传结构进行分析,结果均显示草鱼的遗传多样性较低,且群体间无差异;李思发等[11]同样利用线粒体基因标记分析长江流域四大家鱼遗传多样性,显示草鱼遗传多样性水平较低;而马琴[12]则基于线粒体Cytb基因序列分析三峡库区草鱼种群遗传多样性,发现库区草鱼种群遗传多样性较丰富。
已有研究存在采样点单一、采样范围小,群体数量少等问题,不能有效反映草鱼在长江中上游主要区域种质变化情况,同时缺乏对养殖群体的广泛研究。因此,本研究对长江中上游草鱼主要养殖地进行了全面的本底调查研究,旨从分子水平上探讨草鱼野生和养殖群体遗传差异,全面掌握草鱼野生和养殖群体的遗传多样性现状;评估草鱼群体种质资源状况,分析养殖群体种质衰退情况,为草鱼种质资源评价、开发利用及良种选育提供更为全面的遗传资料和技术支持。
1 材料与方法
1.1 材料
本研究共采集到长江中上游地区(图1)14个地点22个群体的草鱼样本(包括亲鱼鳍条、肌肉和鱼苗),地点从西往东依次是木洞(MD)、万州(WZ)、荆州窑湾(YW)、石首原种场(SS)、荆州锣场(LC)、蔡甸康祥(CD)、涨渡湖(ZDH)、保安湖(BAH)、黄州幸福渔业合作社(HZ)、阳新百容良种有限公司(YX)、浠水水产良种场(XS)、蕲春正大水产(QC)、瑞昌原种场(RC)、鄱阳湖(PYH)。采集的草鱼样品置于95%乙醇中保存,带回实验室提取基因组 DNA,样品来源及数量情况见表1。

表1 长江中上游草鱼采样情况

图1 长江中上游草鱼采样点
1.2 基因组DNA的提取和检测
本实验采用高盐抽提法[13]提取DNA。DNA经1%琼脂糖凝胶电泳和紫外凝胶成像系统拍照确认质量后,于-20 ℃冰箱中保存备用。
1.3 PCR扩增及序列测定
Cytb扩增和测序的引物为通用引物L14724:5′-GACTTGAAAAACCACCGTTG-3′和H15915:5′-CTCCGATCTCCGGATTACAAGAC-3′[14],引物由武汉天一辉远生物有限公司合成。扩增反应在25 μL的体系下进行:模板DNA 50 ng;2×PCR Mix 12.5 μL(包含Taq酶2.5 U,dNTPs 10 μmol,MgCl20.1 mmol),上、下游引物(10 μmol /L)各1 μL,其余体积用双蒸水补足。反应程序:94 ℃预变性5 min,94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,经35个循环后再72 ℃延伸7 min。PCR产物经琼脂糖电泳验证后,将目的条带准确清晰扩增出来的样品送生工生物工程股份有限公司,利用正反引物进行双向测序。
1.4 DNA序列数据的整理与分析
参照测序峰图,对序列进行编辑和校正,查验无错漏后,经NCBI BLAST程序确认为草鱼序列后,再使用MEGA7[15]软件进行多重序列比对;同时计算碱基含量、转换/颠换比率,分析序列变异位点等序列特征;同时基于Tajima-Nei模型计算群体间的遗传距离,构建UPGMA(Unweighted pair group method using arithmetic average)系统发育树,利用Kimura双参数距离模型以邻接法(Neighbor Joining,NJ)构建单倍型间系统发生关系树,所有的计算均采用了1 000次的重复。使用DnaSP5.1[16]软件计算各地理种群的多态位点(S)、单倍型数目(H)、单倍型多样性指数(Hd)、核苷酸多样性指数(Pi)等。通过Network软件[17]中的Median Joining法构建单倍型网络图。使用Arlequin3.1软件[18]计算遗传分化系数Fst,同时进行中性检验来验证群体在历史上是否发生过种群扩张。
2 结果
2.1 草鱼群体细胞色素b基因序列变异分析
草鱼677个个体均获得长度为1 008 bp的同源序列,养殖和野生群体的碱基组成无差异,其中碱基 C 的含量显著低于其他碱基的含量,两者的A+T和G+C含量一样,且A+T(58.5%)明显大于G+C(41.5%),存在一定的碱基组成偏倚性,这与线粒体蛋白质编码基因的特点相符。养殖和野生群体检测到多态位点分别为27、22,其中单突变位点分别为16、12,简约信息位点分别为11、10,均无碱基插入或缺失,两者的转换颠换均未达饱和,转换数大于颠换数,其比值分别是3.67、1.45。
2.2 单倍型关系及系统发育分析
677条序列检测出35种单倍型(Hap_1-Hap_35),包括12个共享单倍型,23个独有单倍型。23个独有单倍型中,野生群体占5个,其余独有单倍型为养殖群体所有。在所有单倍型中,Hap_1和Hap_2两种单倍型在群体间分布最广,与其他单倍型关系紧密,为所有群体共享,其中频率最高的是Hap_1,出现290次,频率达0.428 4,其次是Hap_2,频率达0.344 2。草鱼各野生群体单倍型数在4~11之间,原种场和苗种场各养殖群体单倍型数在2~7(表2)。
基于Cytb基因序列的草鱼群体的单倍型数据,以草鱼(登录号:HM237990.1)为标准序列,以鲢(登录号:MH797122.1)、青鱼(登录号:MF687137.1)以及鳙(登录号:HM162839.1)作为外类群,采用邻接法构建单倍型间分子系统树,从图2可以看出野生和养殖群体各单倍型混杂在一起,没有形成明显的独立分支,并且与外类群分开。

图2 基于Cyt b序列单倍型构建的草鱼NJ树
2.3 单倍型网络图谱的构建
Network软件生成的单倍型网络图(图3)能够直观地展现出35种单倍型之间的关系。其中H_1和H_2单倍型位于网络关系图的扩展中心,两单倍型为所有草鱼群体所共享,为主体单倍型,与其他单倍型短支相连,可以推测H_1和H_2两种单倍型在这一单倍型组中相对较为原始,周围的单倍型由其衍生而来。草鱼群体的单倍型在各部分均有分布,没有产生明显的谱系分化。

图3 草鱼各单倍型间的进化网络图
2.4 草鱼野生和养殖群体遗传多样性分析
各群体遗传多样性参数见表2,草鱼野生群体单倍型多样性和核苷酸多样性均值(Hd:0.769;Pi:0.001 43)均高于原种场和苗种场养殖群体均值(Hd:0.766;Pi:0.001 13)/(Hd:0.649;Pi:0.000 84)。野生群体中,WZ群体的单倍型多样性和核苷酸多样性水平高于MD和PYH群体(Hd:0.857 vs 0.778 vs 0.717;Pi:0.002 05 vs 0.001 52vs 0.001 05),且其是所有检测群体中多样性水平最高的。QSS和QRC原种场养殖亲鱼单倍型和核苷酸多样性均高于其人工养殖鱼苗的多样性,略低于野生群体。

表2 基于Cyt b基因的草鱼群体遗传多样性分析
苗种场群体中,YX群体的单倍型多样性和核苷酸多样性(Hd:0.791;Pi:0.001 47)是所有苗种场检测群体中最高的,另外,HZ、ZDH、QC2以及QC3群体的多样性水平也较高,其余苗种场群体的多样性均低于苗种场群体总体多样性水平,其中最低的是CD1群体(Hd:0.247;Pi:0.000 24)。
苗种场大部分群体多样性水平偏低,尤其是CD三个批次鱼苗的遗传多样性均很低(Hd:0.247/0.468/0.443;Pi:0.000 24/0.000 56/0.000 44),且各批次差异较大;QC第一批鱼苗的遗传多样性(Hd:0.337/0.768/0.728/0.633;Pi:0.000 33/0.001 19 /0.000 98/0.000 76)明显低于其他批次的遗传多样性;BAH放流群体遗传多样性低于苗种场平均水平。
2.5 草鱼野生群体的历史动态分析
一般来说,中性检验的数值为负且P值显著则表示种群在其历史上经历了扩张[19]。草鱼野生群体的中性检验(表3)结果表明,PYH群体Tajima′s D与Fu′s Fs值都为负值,但P值均不显著(P>0.05);MD群体Tajima′s D值为负,但P值不显著(P>0.05)。WZ群体Tajima′s D与 Fu′s Fs的检验结果均为负值,且P值均显著(P<0.05),说明WZ群体经历过种群的扩张。若将草鱼野生群体作为一个整体来计算,其Tajima′s D值和Fu′s Fs值为负,且P值均显著(P<0.05),说明长江中上游草鱼野生群体经历过种群的扩张。

表3 草鱼野生群体中性检验
2.6 草鱼群体遗传结构分析
Arlequin软件计算各群体的遗传分化系数(表4),野生群体(MD、WZ和PYH)和原种场亲鱼(QSS和QRC)间无遗传分化存在(Fst在-0.030 30~0.061 44之间,P值均大于0.05),但这5个群体与除LC、QC2、QC3外的苗种场群体间有显著遗传分化(P<0.05),各苗种场群体两两间也大范围地存在显著遗传分化(Fst在0.042 32~0.582 62之间,P值均小于0.05)。

表4 草鱼群体间的遗传分化系数
MEGA7软件计算草鱼各群体间遗传距离(表5),群体间遗传距离最大的是WZ和HZ群体(0.001 914),最小的是CD1和CD3群体间(0.000 360)。根据Nei′s[20]遗传距离运用 UPGMA 法对草鱼群体进行聚类分析发现(图4),苗种场群体中,CD、XS、YW以及SS人繁群体与其他群体之间遗传距离较小,而YX和HZ群体与其他群体之间遗传距离较大,各个野生群体与其他群体遗传距离更大,尤其是WZ群体和其他群体之间。

表5 草鱼群体间的遗传距离
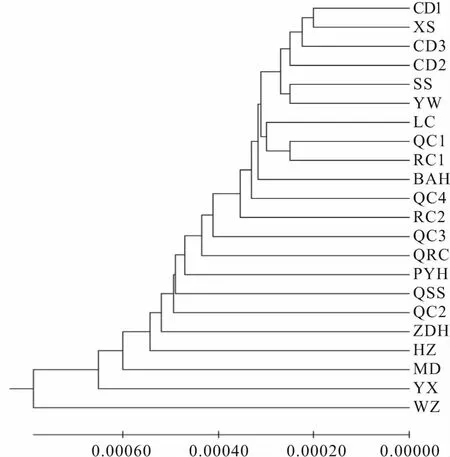
图4 基于Nei′s遗传距离构建的草鱼群体UPGMA树
3 讨论
物种的遗传多样性是其长期进化的产物,是其生存适应和发展进化的前提,物种遗传多样性越高,其对环境变化的适应能力就越强,反之种群适应能力降低。单倍型多样性(Hd)和核苷酸多样性(Pi)是衡量物种遗传多样性的两个重要指标[21]。Grant等[22]认为核苷酸多样性指数低于0.005时,表明该群体的遗传多样性较低。根据本研究草鱼野生群体以及养殖群体的核苷酸多样性情况可知(表2),无论是野生群体还是养殖群体,两者的Pi都远低于0.005,说明草鱼各群体核苷酸多样性处于较低水平,因此整体而言,长江中上游草鱼的遗传多样性较低。
3.1 草鱼野生群体的遗传多样性及历史动态分析
野生群体中,WZ群体的单倍型多样性和核苷酸多样性水平高于MD和PYH群体。比较基于不同线粒体基因标记的草鱼野生群体遗传研究,发现本研究草鱼野生群体遗传多样性水平与李树华等[8]、朱秀芳[9]、翟东东等[10]、谭书贞等[23]以及傅建军等[24]揭示的野生群体大致相当,均处于较低水平,且近十年来未出现下降情况。
本研究Tajima′s D与 Fu′s Fs中性检验显示只有WZ群达到显著变化的水平,符合种群扩张的解释,说明WZ草鱼野生群体在历史上经历了种群的扩张。这与傅建军等[24]利用线粒体控制区序列分析草鱼历史动态的结果一致。
Grant等[22]研究结果表明,当Hd≥0.5、Pi<0.005时,说明群体是受瓶颈效应后迅速扩张导致。本研究野生草鱼群体的单倍型多样性都挺高(Hd>0.5),但核苷酸多样性较低(Pi<0.005),说明野生草鱼群体确实是遭受了瓶颈效应之后迅速扩张而来。
3.2 草鱼养殖群体的遗传多样性分析
养殖和野生群体因为受环境或者人为选择压力的影响,遗传多样性会存在一定差异。本研究显示,草鱼群体多样性水平大小依次是,野生群体>原种场群体>苗种场群体>放流群体,野生群体多样性水平要高于养殖群体,分析其原因,可能是长时间的单一亲本繁殖、有效亲本数量有限、遗传漂变及相对封闭的养殖环境对养殖鱼类的影响等引起养殖鱼类遗传多样性下降。MD、WZ和PYH野生群体(Hd:0.769;Pi:0.001 43)、QSS和QRC原种场亲鱼群体(Hd:0.766;Pi:0.001 13)的遗传多样性水平相差不大,说明原种场养殖亲鱼是来源于野生江苗,多样性水平未出现明显变化。QSS和QRC原种场养殖亲鱼的遗传多样性均高于其人工养殖鱼苗的多样性,且存在原种场人工繁殖鱼苗比部分普通苗种场如YX、HZ人工养殖鱼苗遗传多样性水平还低的情况,说明原种场繁育亲本缺乏科学的配对管理,没有严格遵循管理要求进行筛选,从而导致人工养殖鱼苗群体多样性水平下降。
苗种场群体间的遗传多样性水平差异较大,如YX群体明显比CD和XS等群体遗传多样性水平高,说明YX群体有效亲本数量较多,种质优良,繁殖过程进行了较好的管理。同一苗种场多批次鱼苗的多样性水平也存在较大差异,比如QC1群体明显比QC其他批次多样性水平低,部分苗种场群体的多样性水平明显较低,尤其是CD三个群体、XS群体以及QC1群体,单倍型和核苷酸多样性指数都很低(Hd<0.5;Pi<0.005),这说明这些苗种场参与繁育亲本少、出现了近亲繁殖现象,遗传多样性水平有下降的趋势,反映出部分养殖场并未按照苗种场相关规定进行严格的亲本优选配对、交换利用和苗种培育管理等。总之,苗种场大部分群体的多样性水平都很低且明显低于野生群体,一般认为,鱼类人工养殖群体遗传多样性会有一定程度的下降,而导致鱼类人工养殖群体遗传变异性降低的主要因素是建立者效应或遗传漂变以及人工选择[25],这一点在其他鱼类研究中也有报道,Taniguchi等[26]用微卫星标记对香鱼群体研究结果说明人工养殖群体的遗传多样性确实会有一定程度的降低。
BAH放流群体遗传多样性水平低于野生、苗种场和原种场群体的遗传多样性水平,说明人工增殖放流时没有使用遗传多样性水平高的群体,放流过程缺乏鱼种质量管控环节。
3.3 草鱼群体遗传结构分析
野生群体(MD、WZ和PYH)和原种场亲鱼(QSS和QRC)间无遗传分化存在,但这5个群体与除LC、QC2、QC3外的苗种场群体间有显著遗传分化(P<0.05),各苗种场群体两两间也大范围地存在显著遗传分化(P<0.05)。野生地理群体间无分化,这与傅建军[24]等研究长江水系木洞与万州的结果相符,反映野生地理群体亲本量巨大,繁育自由交配,基因交流广泛,因此不存在遗传分化现象。苗种场间以及与野生来源的群体间存在大范围的显著遗传分化情况,说明苗种场在亲本量贮存或者更新方面出现不足,且在人工干预情况下,苗种场之间缺乏交流。
群体间的遗传距离能反映群体间的亲缘关系,值越大亲缘关系越疏远。MEGA7软件计算草鱼各群体间遗传距离(表5),群体间遗传距离在0.000 36~0.001 914之间,这明显低于Shaklee等[27]提出的0.05、0.30、0.90作为鱼类在种群、种以及属的三级水平上的遗传距离分界值,较小的遗传距离说明草鱼各群体间没有产生明显的分化。研究中草鱼野生群体间的遗传距离较小,显示草鱼地理群体间有很近的亲缘关系,与遗传分化研究结论对应,也与朱叶[2]的结果相符。WZ群体与其他群体距离相对较大,养殖群体间的遗传距离较小,中游养殖场群体多与PYH野生群体以及QSS和QRC原种场群体亲缘关系近,这从某个角度上说明,长江中游的养殖亲鱼多来源于中游野生群体,较少来源于上游野生资源。
草鱼35种单倍型中,单倍型Hap_1和Hap_2是各群体共享的主体单倍型,可以推断其是较原始的、对外界环境适应能力强的且能在草鱼群体中稳定存在的优势单倍型。从单倍型发育树来看,所有单倍型聚为一大支,表明群体的亲缘关系较近,各群体没有形成明显的地理格局。mtDNA遵循严格的母系遗传,一般不发生重组,后代能够完整地保存母本遗传信息,mtDNA一个单倍型即可代表一个母系集团[28]。根据本研究草鱼线粒体DNA的Cytb序列单倍型数量,我们推测3个野生群体的雌性亲鱼数量至少有12尾,16个苗种场养殖群体至少有22尾。遗传多样性低的群体往往对应的单倍型数量较少,如CD1和QC1群体的单倍型数量仅为2,表明这些养殖群体雌鱼亲本数量确实较少(表2)。
3.4 草鱼种质资源的保护建议
鱼类在人工繁殖过程中遗传多样性降低,进而影响鱼类生长、繁殖及适应能力,这是鱼类人工繁殖过程中出现“退化”现象的主要原因之一[29]。本研究中,草鱼部分养殖群体遗传多样性显著降低,说明养殖草鱼已经出现了种质退化。此外,近年来长江水域因为污染、过度捕捞等原因,鱼类栖息地丧失,草鱼的野生资源量减少[10];水电开发和江湖阻隔等使草鱼群体出现隔离,部分地方如赣江等草鱼群体栖息地出现片段化情况,这些都会降低草鱼的遗传多样性,加速草鱼的种质衰退。因此,对于野生草鱼资源,必须加强各水系草鱼群体遗传多样性的系统研究和维护,进行原种资源的保存、管理和有序利用。对养殖草鱼群体,须严格管理原种场、苗种场,保证草鱼繁育亲本的质量和有效繁育群体足够大,加强苗种培育管理,防止近交和混杂[1,11]。
致谢:
衷心感谢各原种场和苗种场等无私提供了实验样本,感谢中国科学院水生生物研究所鱼类生态学与资源保护学科组各位老师和研究生们在采样和数据分析上给予的帮助!
