酸面团中微生物基因组DNA提取方法的优化
2021-07-13李海峰符加珂李志建
胡 双,李海峰*,符加珂,李志建
1.河南工业大学 生物工程学院,河南 郑州 450001 2.河南工业大学 粮油食品学院,河南 郑州 450001
酸面团由面粉、水和微生物(主要是酵母菌和乳酸菌)经混合后发酵而成[1],在面制食品制作中的应用有非常久远的历史。利用酸面团进行发酵不仅可以改善面团的流变特性、增强风味,而且还能改善面制品的比容、质构特性,利于货架期的延长[2-4]。酸面团中微生物的种类繁多,各种微生物在发酵代谢过程中能够产生大量的酯、醇、醛和酮等风味化合物,赋予面团独特的风味[4-6]。
酸面团中的微生物生态系统对面制品的品质也有着极其重要的影响[7-8]。首先,酵母菌能在发酵时产生大量气体,使面制品体积膨大且松软;其次,乳酸菌能酸化面团,产生多种有机酸等风味成分[9];再者,有研究发现乳酸菌代谢能够产生胞外多糖和抑菌物质,不仅可以延缓面制品老化,还可以抑制有害菌的生长,延长面制品保质期[10];此外,由于一些霉菌的存在使面制品的品质更佳,如根霉菌,不仅可以分泌淀粉酶和糖化酶将淀粉质原料糖化,还能产生乳酸、琥珀酸等有机酸和酯类物质,有效提高了面制品的口感和风味[11-12]。另外,研究发现传统发酵食品中的一些菌种在发酵过程中会产生亚硝酸盐[13-14]、生物胺[15]等有害物质,给食用者带来安全隐患。
因此,有效提取酸面团中微生物的基因组DNA,达到高通量测序等方法所需的浓度与纯度,然后分析面团样品中微生物的群落结构组成、演替过程及功能特性,能够极大地推动在分子水平上对酸面团组成与功能特性的研究。同时,也能够让酸面团更好地应用于发酵面制食品行业中,提高面制品的质量和风味。但是,目前虽然有很多用于各类环境样品DNA提取的试剂盒或方法,但酸面团中含有的大量淀粉、面筋蛋白、糖类和酚类等物质[16-17],在发酵过程中,面筋网络结构的扩展也比较充分,往往把微生物包裹在其中,进一步增加了DNA提取的难度[5]。利用已报道的DNA提取方法虽然能获取一部分酸面团基因组的DNA,但在浓度和纯度方面都很难满足测序要求,尤其是国内,专门针对酸面团中微生物基因组DNA提取方法的报道还相对较少。
作者采用3种样品前处理与4种不同的提取方法对酸面团中微生物基因组DNA进行提取,从提取DNA产物的浓度、纯度以及对基因组进行16S rRNA基因的PCR扩增产物效果加以比较与讨论,旨在找到一种快捷高效且高质量的酸面团中微生物基因组DNA提取方法,为利用非培养等分子生物学方法研究酸面团中微生物的种群多样性的关键步骤——DNA的提取,提供方法借鉴和参考。
1 材料与方法
1.1 材料
1.1.1 样品
老面:河北省张家口市;酵子:河南省商丘市。
1.1.2 主要试剂
EDTA、Tris、NaCl、无水乙醇、β-巯基乙醇:上海国药集团化学试剂有限公司;SDS:美国Amresco公司;琼脂糖:北京擎科新业生物技术有限公司;异戊醇、异丙醇:天津市富宇精细化工有限公司;氯仿:东营市隆兴化工有限公司;以上试剂均为分析纯。6×Loading Buffer、50×TAE缓冲液:北京索莱宝科技有限公司;DL 5 000 Marker、2×San Taq PCR Mix:上海生物工程有限公司。采用的细菌16S rRNA基因上下游引物分别为P0(GAGAGTTTGATCCTGGCTCAG)和P6(CTACGGCTACCTTGTTACGA),由上海生物工程有限公司合成。
1.1.3 试剂盒
D2100通用基因组DNA提取试剂盒:北京索莱宝科技有限公司;KG-203快速DNA提取检测试剂盒:北京天根生化科技有限公司。
1.1.4 DNA提取液
CTAB提取液:2% CTAB、1.4 mol/L NaCl、0.02 mol/L EDTA、0.1 mol/L Tris、2%β-巯基乙醇,pH8.0。STES提取液:0.5 mol/L NaCl、0.02 mol/L EDTA、0.2 mol/L Tris、0.1% SDS,pH7.6。
1.2 主要仪器与设备
GXZ型恒温光照培养箱:宁波江南仪器厂制造有限公司;TGL-18 M型台式高速冷冻离心机:上海卢湘仪离心机仪器有限公司;YXQ-LS型高压灭菌锅:上海博讯实业有限公司;ZKSY-6型智能水浴锅:郑州长城科工贸有限公司;BG-Power600型琼脂糖凝胶电泳仪:北京百晶生物技术有限公司;Universal Hood Ⅱ型紫外凝胶成像及分析系统:美国Bio-Rad公司;Nano Drop One型超微量分光光度计:美国Thermo Fisher Scientific公司;A200型基因扩增仪:杭州朗基科学仪器有限公司。
1.3 酸面团样品制备及前处理
1.3.1 酸面团样品的制备
将5 g酵子样品、100 g面粉、50 mL无菌水混合均匀,置于30 ℃、湿度80%的醒发箱中自然发酵24 h后备用。将10 g河北老面样品、100 g面粉、50 mL无菌水混合均匀,置于30 ℃、湿度80%的醒发箱中自然发酵24 h后备用。
1.3.2 酸面团样品的前处理
称取5 g发酵好的酸面团,采取以下3种方法进行前处理,用于后续微生物基因组DNA的提取。(1)烘干法:将样品掰碎,置于50 ℃恒温干燥箱内烘干1 h后取出,在室温下研磨成粉末状。(2)洗去面筋法:将样品与无菌水1∶ 1混合,用玻璃棒搅拌10 min,然后挑去面筋。(3)离心沉淀法:参照文献[18]的方法并稍加改进,向5 g样品中加入0.8%的生理盐水40 mL,制成匀浆,1 500 r/min离心5 min后,取上清液,5 000 r/min离心15 min,弃上清液后备用。
1.4 酸面团中微生物基因组DNA的提取方法
1.4.1 改良CTAB法
根据文献[19]的方法略加改进,向前处理过的酸面团样品中加入65 ℃预热的CTAB提取液0.5 mL,将其混合均匀后转移到无菌的1.5 mL离心管中,65 ℃恒温水浴1 h。取出,冷却至室温,4 ℃、12 000 r/min离心10 min,然后吸取上清液于另一无菌1.5 mL离心管中,同时加入等体积的氯仿-异戊醇(体积比24∶ 1,下同),反复倒置使其充分混匀,4 ℃、12 000 r/min离心5 min后吸取上清液于另一1.5 mL离心管中,同时加入等体积的异丙醇,混匀后将其放置在-20 ℃冰箱中1 h;然后4 ℃、12 000 r/min离心15 min后即获得DNA沉淀,用70%乙醇清洗2次,沉淀自然干燥至无酒精味;最后加入50 μL TE缓冲液溶解DNA,并贮存于4 ℃冰箱中。
1.4.2 STES法
向前处理过后的酸面团样品中加入0.5 mL STES缓冲液,并加入2粒玻璃珠与120 μL氯仿-异戊醇,间歇涡旋震荡5 min。将匀浆液转移至无菌的1.5 mL离心管中,4 ℃、12 000 r/min离心2 min。取上清液至另一无菌的1.5 mL离心管中,用等体积的氯仿-异戊醇抽提,在4 ℃、12 000 r/min条件下离心2 min,重复2次抽提。取上清液于另一1.5 mL离心管中,加入2倍体积无水乙醇,在-20 ℃条件下沉淀2 h后于4 ℃、12 000 r/min离心2 min,收集DNA沉淀。用70%的乙醇清洗2次,沉淀自然干燥至无酒精味;加入50 μL TE缓冲液溶解DNA,并贮存在4 ℃冰箱中。
1.4.3 通用基因组DNA提取试剂盒法
按照试剂盒说明书操作步骤进行。
1.4.4 快速DNA提取检测试剂盒法
按照试剂盒说明书操作步骤进行。
1.5 微生物基因组DNA的电泳检测
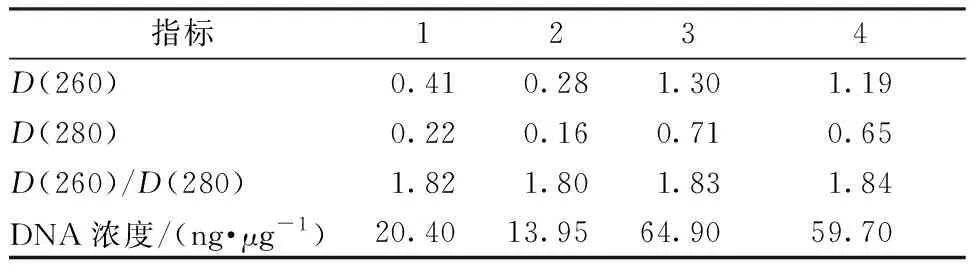
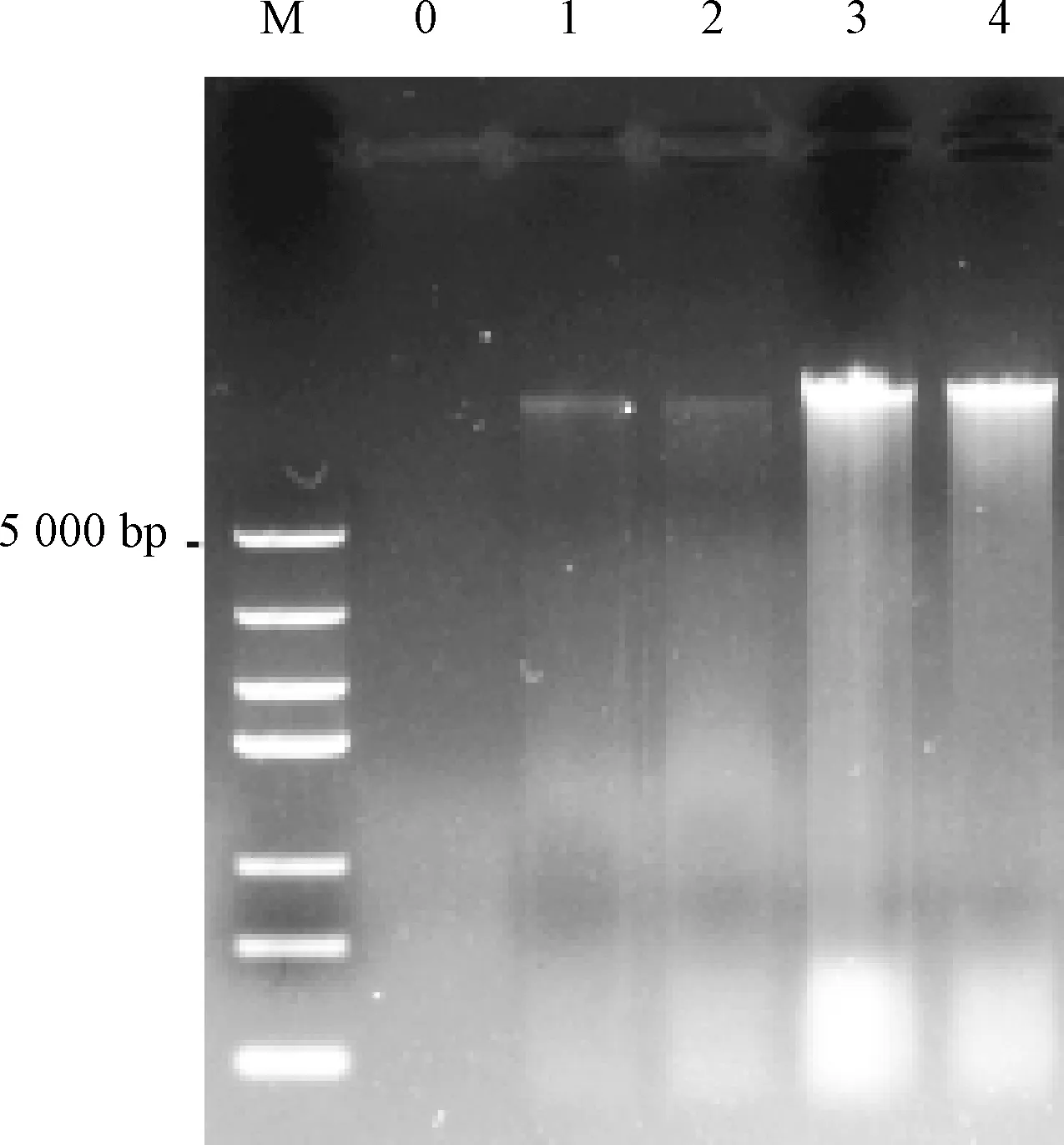
分别取5 μL DNA提取产物,1 μL的6×Loading Buffer上样缓冲液,进行0.8%琼脂糖凝胶电泳,用凝胶成像系统观察并拍照。使用Nano Drop One超微量分光光度计测定DNA产物的D(260)和D(280),以比较DNA的浓度及纯度。通常认为1.8 以4种方法提取的微生物基因组DNA为模板,分别进行16S rRNA基因的PCR扩增。采用25 μL扩增体系,反应体系包括:2×San Taq PCR Mix 12.5 μL、引物0.5 μL、模板DNA 1 μL、ddH2O补至25 μL。PCR反应程序:94 ℃预变性5 min;94 ℃变性30 s,55 ℃退火30 s,72 ℃延伸1 min,30个循环;72 ℃终延伸10 min;4 ℃维持。之后利用0.8%琼脂糖凝胶电泳PCR产物进行检测。 注:M为DL 5 000 Marker;0为空白对照;1为离心沉淀法;2为洗去面筋法;3为烘干法。图1 应用不同前处理方法获得酸面团中微生物基因组DNA的电泳图Fig.1 Electrophoretogram of microbial sourdough genomic DNA extracted by using different pre-treatments 不同前处理条件下,使用通用基因组DNA提取试剂盒提取酸面团中微生物基因组DNA的结果如图1所示。由表1可知,通过离心沉淀法前处理,提取到的DNA条带清晰,完整性较好,且纯度和浓度最高,D(260)/D(280)为1.80,5 g酸面团样品中提取的微生物基因组浓度达到80.10 ng/μg;利用洗去面筋法进行前处理,提取到的DNA片段完整性差,D(260)/D(280)为1.91,纯度尚可,但所得DNA的浓度低、质量差,可能是由于在前处理时酸面团中微生物的DNA并没有完全溶解在提取溶液中,同时还出现了一定程度的降解;利用烘干法进行前处理,提取的DNA片段无明显条带,且D(260)/D(280)大于2.0,表明DNA产物中的RNA含量较多,也可能在烘干过程中由于高温的影响出现了DNA的降解,因此,该前处理方法不适合酸面团中微生物DNA的提取。 表1 不同前处理方法处理后利用通用基因组DNA提取试剂盒所提取的酸面团中微生物基因组DNA纯度和浓度检测结果Table 1 Purity and concentration of microbial genomic DNA from sourdough extracted by the universal genomic DNA extraction kit under different pre-treatment methods 注:M为DL 5 000 Marker;0为空白对照;1为CTAB法;2为STES法;3为通用基因组DNA提取试剂盒;4为快速DNA提取检测试剂盒。图3同。图2 4种方法所提取的酸面团中微生物基因组DNA电泳图Fig.2 Electrophoretogram of microbial genomic DNA extracted from sourdough by 4 methods 由图2可知,利用改良CTAB法、STES法、通用基因组DNA提取试剂盒法提取得到的DNA片段完整性较好,且片段大小一致;利用快速DNA提取检测试剂盒法所提取的产物并没有得到明显的微生物基因组DNA条带,利用通用基因组DNA提取试剂盒法所提取的DNA片段有轻微拖尾现象;利用STES法提取的DNA片段亮度较弱。 DNA纯度检测结果表明:利用4种方法所提取的DNA产物的D(260)/D(280)从大到小依次为CTAB法、STES法、通用基因组DNA提取试剂盒法、快速DNA提取检测试剂盒法,其中利用CTAB法和STES法获得的DNA的D(260)/D(280)均大于1.9,表明采用这2种方法所提取的微生物基因组DNA中含有一定量的RNA,而利用快速DNA提取检测试剂盒法所提取的微生物基因组DNA的纯度较低(D(260)/D(280)<1.7),蛋白质和酚类物质等杂质含量较高,说明该试剂盒不适用于酸面团中微生物基因组DNA的提取。综合来看,利用通用基因组DNA提取试剂盒所提取的微生物基因组DNA浓度和纯度相对较高。4种DNA的提取方法中,STES法和CTAB法的操作步骤复杂,用时较长;而试剂盒法用时相对较短,操作简单,且减少了有毒有机试剂的使用,可以减轻对操作人员的健康损害。 以改良CTAB法、STES法、通用基因组DNA提取试剂盒法和快速DNA提取检测试剂盒法4种方法制得的酸面团中微生物基因组DNA为模板,进行16S rRNA基因PCR扩增反应。由图3可知,改良CTAB法、STES法与通用基因组DNA提取试剂盒法提取的酸面团中微生物基因组DNA均能扩增出目的条带,大小约1 500 bp,其中通用基因组DNA提取试剂盒法的条带最亮且杂带最少,说明该方法提取的基因组能更好地应用于后续实验。然而,快速DNA提取检测试剂盒法未能扩增出目的条带,进一步说明该方法不适合用于酸面团中微生物基因组DNA的提取。 图3 4种方法所提取的酸面团中微生物基因组DNA 16S rRNA基因PCR扩增Fig.3 PCR amplification of microbial genomic DNA 16S rRNA gene in sourdough extracted by 4 methods 在获得了具有较好质量的酸面团中微生物基因组DNA的基础上,把处理效果较好的离心沉淀前处理法与通用基因组DNA提取试剂盒提取法进行了结合,同时对河北老面以及酵子发酵的酸面团中微生物基因组DNA进行提取。结果表明,该组合方法能够有效提取不同酸面团样品中微生物的基因组DNA,且提取的基因组DNA电泳结果显示条带清晰明亮,完整性好(图4),DNA产物的纯度较高(1.8 表2 通用基因组DNA提取试剂盒法(离心沉淀法)提取的酸面团中微生物基因组DNA纯度和浓度检测结果Table 2 Purity and concentration of microbial genomic DNA extracted from sourdough by universal genomic DNA extraction Kit (centrifugal precipitation method) 注:M为DL 5 000 Marker;0为空白对照;1、2为河北老面;3、4为酵子酸面团。图4 通用基因组DNA提取试剂盒法(离心沉淀法)提取的酸面团中微生物基因组DNA电泳图Fig.4 Electrophoretogram of microbial genomic DNA from sourdough extracted with universal genomic DNA extraction kit (centrifugal precipitation method) 本研究比较了烘干法、洗去面筋法和离心 沉淀法3种前处理方法对酸面团中微生物基因组DNA提取结果的影响。结果表明:在3种前处理方法中,离心沉淀法进行前处理后提取到的DNA片段完整性较好,且纯度和浓度最高,最适合用于酸面团中微生物基因组DNA的提取;洗去面筋前处理法次之;而利用烘干处理法提取的DNA片段无明显条带,且DNA纯度太低,说明该前处理方法并不适合用于酸面团中微生物基因组DNA的提取。 从所提取的酸面团中微生物基因组DNA产物的纯度、浓度及作为模板进行16S rRNA的PCR扩增结果来看,利用通用基因组DNA提取试剂盒法提取的微生物基因组DNA纯度和浓度高,且PCR产物杂带最少,亮度较高;利用STES法和CTAB法提取的微生物基因组DNA效果次之;但用快速DNA提取检测试剂盒法提取的微生物基因组DNA纯度最低,DNA产物中蛋白质、淀粉等杂质含量较多,且PCR扩增未能扩增出目的条带,因此,该方法并不适用于酸面团中微生物DNA的提取。 综上所述,使用离心沉淀法进行前处理后,结合通用基因组DNA提取试剂盒法对酸面团中微生物基因组DNA进行提取,是获得高浓度、高纯度与高质量微生物基因组DNA的有效方法,本研究可为对酸面团在分子生物方面的研究提供方法借鉴和参考。1.6 微生物基因组DNA的PCR扩增检验
2 结果与分析
2.1 酸面团样品前处理的优化
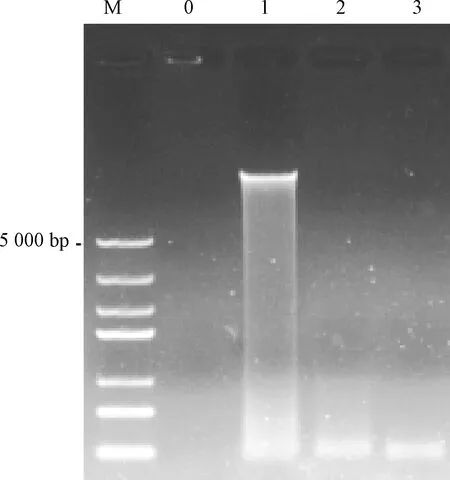

2.2 微生物基因组DNA提取产物的电泳检测及纯度、浓度比较
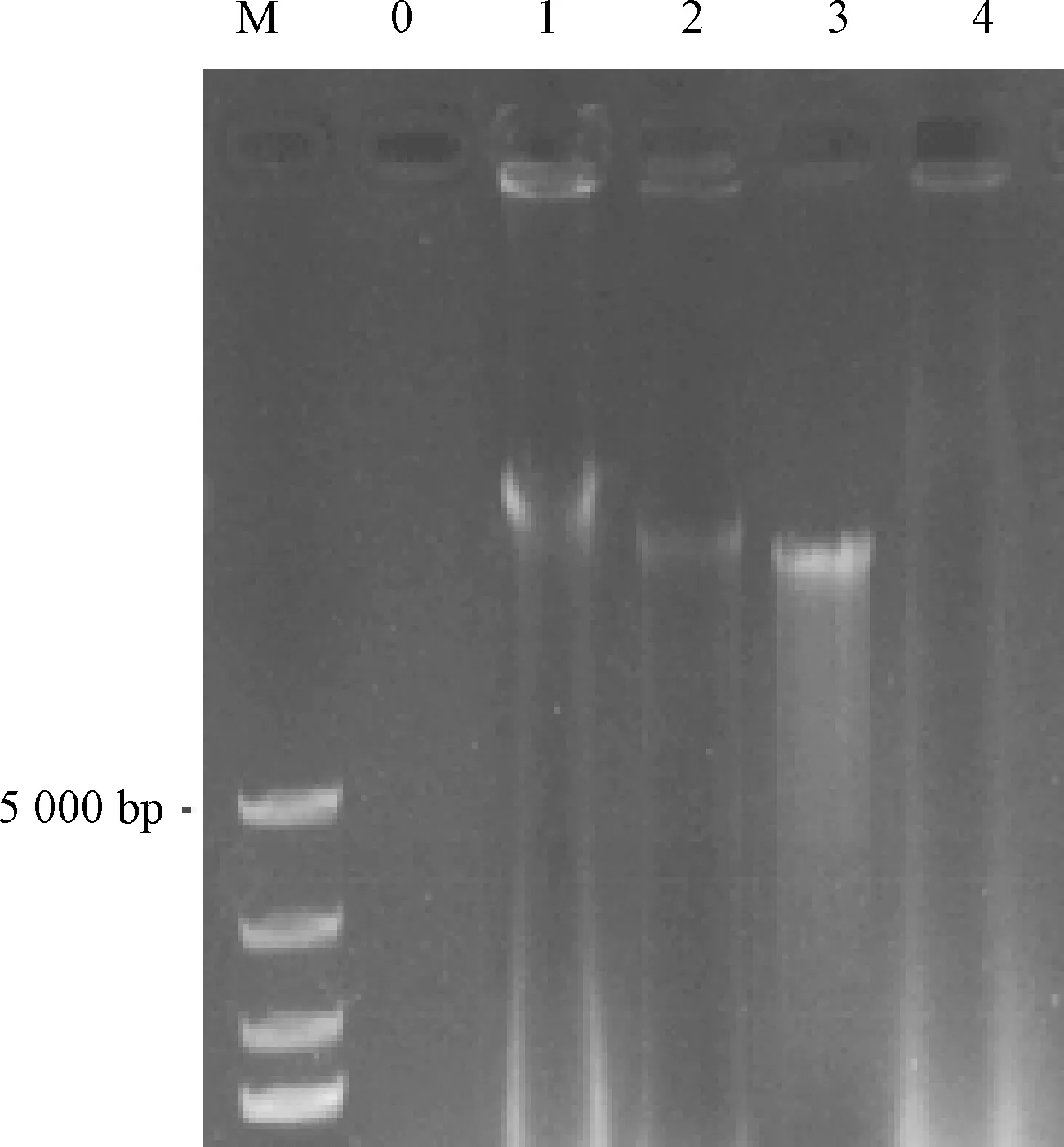
2.3 微生物基因组DNA提取产物的PCR扩增检验
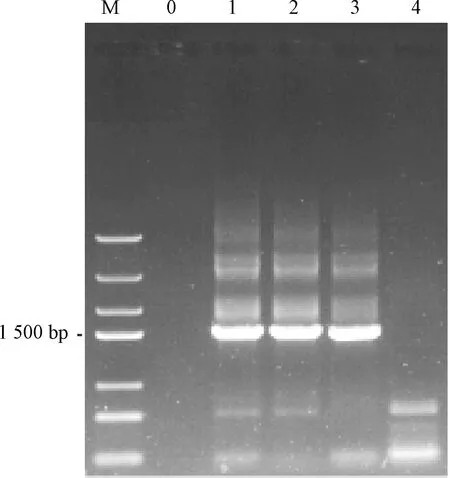
2.4 两种不同酸面团样品中微生物基因组DNA的提取
3 结论
