烟酰胺-N-甲基转移酶在胃癌中高表达并与预后负相关:基于生物信息学方法
2021-07-08文智慧梁炜祺钟依秀张庆玲2
文智慧,梁炜祺,3,钟依秀,3,孙 菲,3,张庆玲2,
1南方医科大学基础医学院病理学系,广东 广州 510515;2广东省人民医院//广东省医学科学院病理科,广东广州 510080;3南方医科大学第一临床医学院,广东 广州 510515
胃癌是消化道常见的恶性肿瘤之一,世界卫生组织最新公布数据显示,在中国癌症患者中,胃癌总发病率和死亡率均排名第3[1]。由于早期胃癌缺乏特异性症状,难以被发现,90%患者诊断时已经处于进展期[2]。虽然手术、辅助放化疗等治疗方法在一定程度上能够改善胃癌患者的生存期,但由于化疗耐药等原因胃癌患者的5年生存率仅为5%~15%[3]。寻找胃癌预后标志物,可能为胃癌患者生存和治疗提供相关指导。
利用公共数据库进行生物信息学分析为癌症分子机制的研究提供新的平台和思路,其具有高通量和全方位分析的特点,在探讨癌症发展过程中基因表达和调控等方面的优势明显[4]。本文通过GEO、Oncomine、TCGA3大数据库分析,筛选出胃癌组织中显著差异表达的基因——NNMT,烟酰胺-N-甲基转移酶(NNMT)是一种肿瘤代谢相关产物,通过调节胞内尼克酰胺水平及以S 腺苷蛋氨酸为甲基供体调节N-甲基化后的修饰,从而调控药物、毒素、激素和微量元素的代谢,参与多种疾病的发生和病理生理过程[5]。研究发现,NNMT在脑胶质瘤[6],皮肤癌[7],肠癌[8],胰腺癌[9],胃癌[10]等组织中异常表达。NNMT的较低表达预示了胃癌的预后更好[11];NNMT 可作为肠癌和卵巢癌的潜在预后和预测性生物标志物[12-13]。但NNMT在胃癌中的作用及发挥生物功能的分子机制尚未完全阐明,为进一步探讨NNMT 在胃癌中的作用机制,本研究首先利用临床样本对NNMT 的表达进行验证,在胃癌细胞中研究其生物学功能,并进一步结合STRING、KMplotter、GSEA、DAVID、Cytoscape 等多种工具对NNMT 相互作用蛋白及其相关信号通路进行分析和探讨,为胃癌发生发展的分子机制及诊疗研究提供新的思路。
1 资料和方法
1.1 GEO数据库
GEO数据库(http://www.ncbi.nlm.nih.gov/geo/)是由美国国立生物技术信息中心创建并维护的基因表达数据库。以“gastric cancer”为关键词进行搜索,界定物种为人类以及数据类型为转录组数据后进行筛选,共确定了20个数据集。纳入标准:①每个数据集包括胃癌样本组和对照样本组(健康组织、相邻的非癌组织、正常胃癌细胞系);②每组至少包含2个样本。排除标准:①重复数据集,是其他数据集的一部分;②数据集类型不是转录组数据;③数据集缺乏正常对照组样本。最后选择纳入GSE19826和GSE13861基因表达谱。使用R语言对数据进行归一化,筛选差异表达基因的同时对NNMT进行差异性分析。
1.2 Oncomine数据库
Oncomine 数据库(https://www.oncomine.org/resource/login.html)是由密歇根大学的医师、科学家和软件工程师所搭建的一个大型肿瘤基因芯片数据库。输入基因为“NNMT”;分析类型为“Differential Analysis”-“Cancer vs.Normal Analysis”;肿瘤类型为“Gastric Cancer”;数据类型为“mRNA”。经检索,共得到5 个研究机构的芯片分析数据。选择可信度较高的“Chen Gastric”,对NNMT基因进行差异性分析。
1.3 TCGA数据库数据
TCGA 数 据 库(http://www.cancergenome.nih.gov/)是美国国家癌症研究所(NCI)和美国国立人类基因组研究所(NHGRI)合作开发的一个项目。TCGA数据库收录的胃癌病例共有443 例(TCGA-STAD),其中转录组基因表达谱数据共有380 例。对TCGASTAD 分类中的HTSeq-Counts 数据类型进行下载整理,共得到407例患者组织样本信息。使用R语言中的“edgeR”包对其中375 例胃癌组织和32 例癌旁组织的研究数据进行归一化并进行差异基因的筛选。
1.4 目标基因的生物信息学分析
使用在线分析工具GEPIA[14]及UALCAN[15]在TCGA数据库中对NNMT 基因进行差异表达分析;使用KMplotter[16]对该基因进行生存分析;通过基因集分析工具GSEA3.0(Gene Set Enrichment Analysis)[17]对NNMT 进行单基因及相关基因集通路富集分析(KEGG)。使用David在线分析工具(https://david.ncifcrf.gov/)对目标基因进行基因本体(gene ontology,GO)功能注释。使用STRING(http://string-db.org/)对NNMT 及从TCGA-STAD 中筛选到的差异基因进行蛋白质-蛋白质相互作用(PPI)分析;使用Cytoscape3.7.1软件的CytoHubba[18]插件,采用12 种算法对TCGASTAD中筛选到的差异基因进行Hub分析,提取每种算法中排名前25 的基因,然后计算重复出现的基因个数,重复次数越多说明该基因重要性越高。
1.5 临床资料收集
收集2017年1~12月南方医院普外科28例胃癌患者的手术标本及对应的临床资料。其中男性16例,女性12例,年龄35~80岁,均为低分化腺癌,依据TNM分期(2018年AJCC胃癌TNM分期,第八版)其中Ⅱ期10例,Ⅲ期18例,收集患者肿瘤及癌旁组织。纳入标准:(1)均行根治性切除术并经病理确诊为胃癌;(2)术前未经放化疗治疗;(3)临床病理特征等资料完整。排除标准:(1)合并其他恶性肿瘤;(2)临床病理特征及术后随访资料不完整;(3)未接受手术治疗的胃癌患者;(4)失访者。本实验获得南方医科大学南方医院机构委员会批准,并获得患者知情同意。
1.6 标本选取与处理
选取癌组织及距肿瘤边缘>5 cm 处癌旁正常组织。所有组织标本离体1 h 内经生理盐水洗净后迅速置于液氮中,速冻后放入-80 ℃超低温冰箱保存,用于RNA提取。
1.7 主要试剂和仪器
RNA 提取试剂盒(爱思进);反转录试剂盒(Fermentas);SYBR-Green qPCR Mix试剂盒(Roche);PCR 引物(睿博兴科);NNMT抗体(Proteintech);山羊抗兔IgG(H+L)-HRP(弗德生物);RPMI 1640培养基(Gibco);胎牛血清(FBS;杭州四季青)。NNMTsiRNA(SANTA CRUZBIOTECHNOLOGY)。AGS、SGC7901、MGC803、BGC823、MKN45、HGC27、GES-1 细胞由南方医科大学病理实验室所提供。
1.8 qRT-PCR
依据Trizol试剂的说明书提取各组细胞总RNA,琼脂糖凝胶电泳检测RNA的完整性,紫光分光光度计测定A260/280的值和总RNA 浓度。将比值在1.8~2.0的RNA 样本进行逆转录并于-20 ℃保存。qRT-PCR 按SYBR Green试剂盒说明操作。
引物序列:
5'-ATCCACTGGCGTCTTCAC-3'(GAPDH,Forward)
5'-AGGCATTGCTGATGATCTTGA-3'(GAPDH,Reverse)
5'-GAATCAGGCTTCACCTCCAA-3'(NNMT,Forward)
5'-CCCAGGAGATTATGAAACACC-3'(NNMT,Reverse)
1.9 细胞培养与转染
人胃癌细胞均于含10%胎牛血清的RPMI 1640培养基,37 ℃、5%的CO2、95%湿度培养箱中孵育培养,待细胞密度达到80%时进行传代、冻存。转染前1 d将细胞接种于6孔板,待细胞贴壁后进行转染,将5 μLsiRNA/3 μg 质粒与250 μL Opti-MEM 混匀细胞转染,5 μL Lipo 2000与250 μL Opti-MEM混匀,室温放置5 min,后将两部分合并混匀静置15 min,将转染复合物逐滴、均匀加入6孔板中,48 h后进行实验。
1.10 Western blot
收集细胞并提取总蛋白,加入上样缓冲液后沸水煮10 min,待冰上冷却,进行SDS-PAGE电泳和转膜。5%脱脂奶粉中室温封闭1 h,PBST缓冲液洗膜后,加入抗GAPDH或抗NNMT抗体4 ℃孵育过夜。次日洗膜后,加入山羊抗兔/鼠二抗(1∶5000),并在摇床上室温2 h。再次洗膜后,加入ECL化学发光液,使用Bio-Rad化学发光仪进行曝光,显影。
1.11 CCK8增殖实验
将5×103/孔的细胞移植于96孔板,待完全贴壁后进行转染。分别在转染的第0、1、2、3、4、5天每孔各加入10 μLCCK-8试剂,培养箱中常规培养2 h。酶标仪检测A450nm处的光密度值。
1.12 划痕愈合实验
预先在6孔板底做3条标记线,标记8个观察区域,瞬时转染12 h后用10 μL移液器枪头轻轻地呈“I”划过板底,PBS清洗六孔板中的细胞2遍,换上2%的小牛血清培养基。在划痕后的0、24、48 h不同时间点拍照,动态观察细胞的迁移情况。设置3 个副孔,实验重复3次。迁移距离以划痕后0 h为参照,计算划痕两侧细胞迁移距离之和。
1.13 Transwell小室细胞运动实验
Transwell迁移实验上室加入用200 μL无血清培养基配制的2×105/mL 的细胞悬液,下室加入500 μL 含20%FBS的1640培养基。常规培养48 h后撤出小室,PBS洗去培养基,用湿棉棒擦去上室底部膜表面的细胞,0.1%结晶紫染色10 min,PBS冲洗并干燥后镜下随机选3个视野观察及计数。
1.14 平板克隆形成实验
细胞以500/孔移植于6孔板,每隔2~3 d更换培养基,2 周后PBS 清洗,4%多聚甲醛固定,结晶紫染色10 min后PBS清洗,待干燥后显微镜下观察拍照,进行计数并统计克隆团数制作图表。
1.15 数据处理
采用R-3.4.1进行芯片数据的预处理;采用Prism Graphpad 7.0软件对其结果进行统计学分析,计量资料以均数±标准差表示,组间比较采用t检验,以P<0.05为差异有统计学意义。
2 结果
2.1 基于公共数据库对NNMT在胃癌中的表达及生物学特征的探究
GSE19826表达谱中,12对配对样本显示NNMT在肿瘤组织中表达较癌旁组织显著上调(图1A,P=0.0466),对GSE13861表达谱中胃癌的多个亚型进行差异分析,再次显示NNMT在胃癌中高表达(图1B,P=0.016,P=0.0013,P<0.001,P<0.001)。利用Oncomine数据库进行分析,结果同样显示NNMT在弥漫型和混合型两个胃癌亚型中存在高表达现象(图2,P<0.001)。再利用在线分析工具GEPIA和UALCAN对TCGA数据库中胃癌芯片数据(TCGA-STAD)进行差异基因表达分析,结果显示NNMT不仅在癌组织和正常组织中存在差异表达(图3A,B),并且在除Ⅰ期外的不同临床分期的病变组织中的表达水平均升高,其中以Ⅳ期肿瘤组织中NNMT 升高最明显(图3C,P<0.001)。利用KMplotter工具对NNMT基因在胃癌样本中进行生存分析,结果显示NNMT 高表达患者的生存时间低于NNMT低表达的患者(图3D,P<0.001)。

图1 GEO数据库胃癌组织样本中的NNMT表达分析Fig.1 The expressions of NNMT in GEO database.A:The expression of NNMT in GSE19826 dataset;B:The expression of NNMT in GSE13861 dataset.

图2 NNMT在Oncomine数据库中的差异表达Fig.2 Differential expression of NNMT in Oncomine database.

图3 NNMT在TCGA-STAD数据库中的差异表达及生存分析Fig.3 Differential expression and survival analysis of NNMT in TCGA-STAD database.A:Differential expression analysis after R language normalization;B:Differential analysis of NNMT between cancer tissues and normal tissues by UALCAN;C:Differential analysis of NNMT in gastric cancer tissues in different stages by UALCAN;D:Online survival analysis with UALCAN.
使用GSEA软件进行NNMT的单基因KEGG通路富集分析以明确NNMT 的生物学功能(图4A)。NNMT 高表达表型通路分析结果显示:细胞外基质受体相互作用(图4B),造血细胞系(图4C),粘着斑(图4D),细胞因子受体相互作用(图4E),细胞黏附分子(图4F)。而在NNMT低表达表型组的通路富集结果显示胃癌中NNMT的低表达可能与以下生物学过程相关:碱基切除修复(图4G),错配修复(图4H),核黄素代谢(图4I)。

图4 NNMT单基因GSEA分析Fig.4 Single-gene GSEA analysis of NNMT.A:GSEA analysis report;B:ECM receptor interaction pathway;C:Hematopoietic cell lineage pathway;D:Focal adhesion pathway;E:Cytokine receptor interaction pathway;F:Cell adhesion mole-cules,CAMs pathway;G:Base excision repair pathway;H:Mismatch repair pathway;I:Riboflavin metabolism pathwa.
为进一步探究NNMT与TCGA-STAD中得到的差异表达基因之间的关系,将所有差异基因和NNMT通过STRING进行PPI(蛋白互作网络)分析,结果显示,NNMT与AURKA等16个蛋白存在互相作用关系,且与TAGLN、PTRF、AKAP12、IGF2BP2蛋白存在较高的共表达情况(相关系数绝对值>0.6,表1,图5)。最后,使用Cytoscape软件提供的12种算法,对用于PPI分析的前2000个差异基因(不包括NNMT)进行拓扑分析,并构建Hub基因网络(图6),然后提取在所有网络中高频次出现的10个基因:CDK1,BRCA1,RAD51,TOP2A,AURKA,MAPK3,JUN,EZH2,HSP90AA1,AR(表2)。
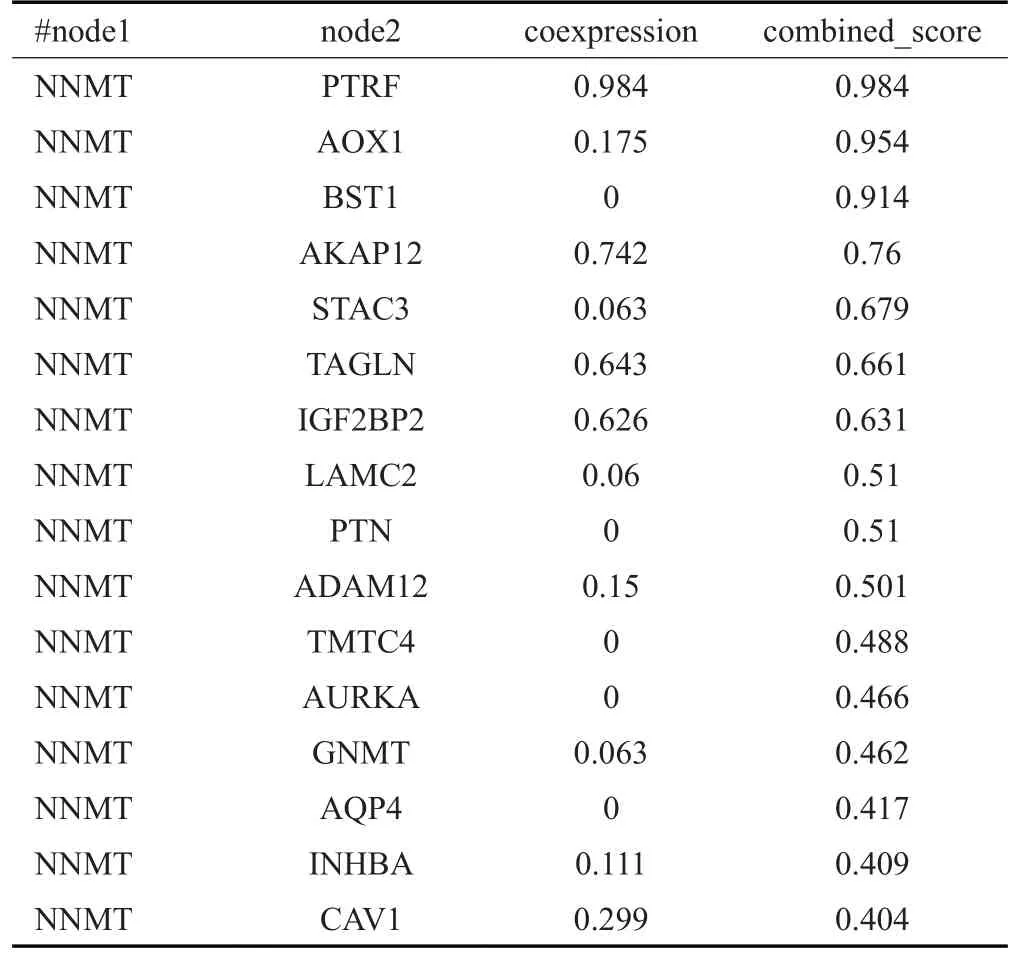
表1 NNMT与差异基因的PPITab.1 PPI of NNMT and differentially expressed genes

图5 NNMT与TCGA-STAD差异基因的蛋白互作网络分析Fig.5 PPI analysis of NNMT and TCGA-STAD differential genes.

图6 TCGA-STAD中的关键基因网络构建Fig.6 Construction of crucial genetic network in TCGA-STAD.
2.2 NNMT在胃癌组织样本中高表达
NNMT和甘油醛-3-磷酸脱氢酶(GAPDH)蛋白的熔解曲线均为单峰特异(图7A),引物特异性好,无引物二聚体产生,标准曲线的相关系数均为0.998,PCR扩增效率恒定且接近100%。qRT-PCR实验结果显示,癌组织中NNMT mRNA 相对表达量高于癌旁组织(P=0.0466,图7B)。

图7 NNMT 在胃癌组织样本中的表达分析Fig.7 NNMT expression in clinically derived gastric cancer samples.A:qRT-PCR dissolution curve;B:Relative expression of NNMT in samples.
2.3 NNMT过表达及干扰细胞株的构建
Western blot实验结果显示,SGC7901、MGC803和BGC823细胞株高表达NNMT,而MKN45、HGC27和AGS 细胞株低表达NNMT(图8A)。因MKN45 的GAPDH与其他蛋白差异较大,后续实验未使用该细胞株。其他蛋白表达结果与qRT-PCR的结果基本保持一致(图8B)。故选用SGC7901和MGC803细胞株构建NNMT敲除细胞株,HGC27和AGS细胞株构建NNMT过表达细胞株进行后续实验。成功构建了NNMT过表达质粒载体,并购买NNMT干扰片段进行细胞模型的构建。qRT-PCR和Western blot实验表明,过表达细胞株AGS中NNMT的mRNA和蛋白表达水平升高(P=0.0044);干扰NNMT 的SGC7901 细胞株则明显降低(P=0.01,图9B、C)。

图8 胃癌细胞中NNMT的表达Fig.8 NNMT expression in gastric cancer cells.A:Western blot;B:qRT-PCR experiment.

图9 NNMT过表达和干扰胃癌细胞株的构建Fig.9 Overexpression and knockdown of NNMT in gastric cancer cell lines.A:Western blot results.B:Expression of NNMT mRNA in AGS by qRT-PCR;C:Expression of NNMT mRNA in SGC7901 by qRT-PCR.
2.4 NNMT对胃癌细胞的生物学功能的影响
CCK8增殖实验结果显示,与对照组细胞相比,过表达NNMT 的AGS 细胞增殖能力增加;干扰NNMT的SGC7901细胞的增殖能力降低(P<0.001,图10C)。平板克隆形成实验也观察到相同的趋势(P<0.01,图10F~G)。划痕愈合实验结果显示,与对照组相比,过表达NNMT的AGS迁移能力增加(P<0.001,图10A),干扰NNMT 的SGC7901 细胞株迁移能力减弱(P<0.001,图10B)。与Transwell小室细胞运动实验分析结果相同(P<0.01,图10D~E)。

图10 NNMT 对胃癌细胞的生物学功能的影响Fig.10 Effect of NNMT expression modulation on biological function of gastric cancer cells.A:AGS cell migration test;B:7901cell migration test;C:Results of AGS and 7901cck8 determination;D:Transwell results of AGS;E:Transwell results of 7901;F:AGS cloning formation experiment;G:SGC7901 cloning formation experiment.
3 讨论
目前已知原癌基因的激活和抑癌基因的失活对胃癌的发生、发展有重要的作用[19-21]。关于NNMT与肿瘤的研究,有研究利用蛋白质组学的方法在高级别卵巢浆液性癌中发现在发生转移的组织间质中NNMT呈高表达状态[22],其通过调节DNA 和组蛋白甲基化以驱动CAF(肿瘤相关成纤维细胞)表型的变化,对于CAF标志物的表达、细胞因子及肿瘤相关性细胞外基质的分泌具有重要的作用;间质NNMT的表达会促进高级别卵巢浆液性癌的进展,与临床的不良预后相关。另有一些研究发现NNMT的下调导致膀胱癌侵袭和转移的能力增强[23];NNMT上调与口腔鳞癌淋巴结转移呈负相关,并与无进展生存时间和总体生存时间的延长有关[24]。本研究通过GEO、Oncomine和TCGA 3大肿瘤数据库分析筛选出NNMT,验证其在胃癌中高表达,具有原癌基因特性。与先前报道一致,本研究发现胃癌组织中NNMT的mRNA水平高于癌旁组织;与人正常胃黏膜上皮细胞相比胃癌细胞中NNMT的表达上调。我们还通过在线数据库分析发现,NNMT在除Ⅰ期外的不同临床分期的病变组织中的表达水平均升高,以Ⅳ期肿瘤组织中NNMT升高最明显,并且NNMT表达与癌症进展(Ⅰ~Ⅳ期)显着正相关。这提示NNMT高表达可作为胃癌进展的预测指标。进一步利用TCGA-STAD进行KMplotter生存分析可以发现NNMT高表达的胃癌患者生存期更短,提示NNMT高表达患者预后更差。

NNMT影响多种癌症细胞的增殖,迁移,侵袭和分化能力[25]。有文献表明,PI3K/Akt通路在NNMT诱导的细胞侵袭和MMP-2激活中起着至关重要的作用[26]。NNMT通过稳定SIRT1的活性来增强前列腺癌细胞的侵袭和转移[27];SIRT1 的活性降低削弱了乳腺癌中NNMT对阿霉素和紫杉醇的抗性作用[28]。但是NNMT在胃癌中的功能机制和相互作用蛋白仍然有待探索。本研究利用GSEA 单基因富集分析的方法对TCGASTAD中筛选出胃癌相关的数据进行分析,我们观察到NNMT的表达上调与细胞外基质受体相互作用、造血细胞系、粘着斑、细胞因子受体相互作用、细胞黏附分子等信号通路存在潜在的相关性,这些通路对上皮间质转化[29],金属离子激活等起着重要作用[30],并参与肿瘤的侵袭转移进程。而NNMT表达下调则可能与碱基切除修复、错配修复、核黄素代谢相关,这些生物过程参与肿瘤细胞的增殖和分化[31-33]。可见,胃癌的发生发展是一个多基因,多步骤参与的复杂过程,以上发现有助于揭示NNMT在胃癌中的作用和加强对胃癌发生发展机制的了解。
蛋白间相互作用网络分析能够层次清晰地展示基因间的相互关系,为发现关键作用基因及其调控关系网络提供重要预测价值。因此,我们将NNMT与TCGASTAD中得到的差异基因进行PPI分析,共得到16个与NNMT 存在互相作用关系的基因,且发现TAGLN、PTRF、AKAP12、IGF2BP2 基因与NNMT 存在较强的共表达关系。同时,我们使用Cytoscape对PPI网络进行更深层次的Hub基因挖掘,进一步得到高频次出现的10个基因:CDK1,BRCA1,RAD51,TOP2A,AURKA,MAPK3,JUN,EZH2,HSP90AA1和AR。这些基因的作用主要体现在细胞周期、细胞凋亡、DNA损伤、DNA合成、细胞信号传导等方面。两次分析结果交叉比对显示AURKA是唯一重复出现的基因,提示AURKA是与NNMT具有潜在联系的重要基因。AURKA编码的蛋白质是调节细胞周期的激酶,在染色体分离过程中参与微管的形成和纺锤体极点的稳定。AURKA在包括胃癌在内的多种癌症中过表达,并且促进癌症的进展[34-35]。目前在胃癌中尚未有这两个基因互作关系的相关报道,需要进一步的实验来确认NNMT与AURKA在胃癌中的互作机制。
综上所述,NNMT可能在预测胃癌的进展和预后方法有较大价值,但是否是一个候选的胃癌预后标志物,有待更进一步的研究和验证。
