基于深度学习构建大肠埃希菌和肺炎克雷伯菌的快速诊断模型
2021-07-02赖丽莎邓任堂张露揭育帮丘文峰徐军发付文金广东医科大学检验医学研究所生物医学工程学院广东东莞53808广东医科大学附属厚街医院检验科广东东莞53945
赖丽莎,邓任堂,张露,揭育帮,丘文峰,徐军发,付文金(1.广东医科大学 a.检验医学研究所,b.生物医学工程学院,广东东莞 53808;.广东医科大学附属厚街医院检验科,广东东莞 53945)
泌尿系感染(urinary tract infection,UTI)是泌尿系受到细菌侵袭而出现的炎症,严重影响患者的生活质量,一旦扩散,将造成全身性感染[1]。目前,明确UTI病原菌的金标准是进行尿培养[2]。然而,尿培养所需时间长,可能耽误感染患者的及时治疗。因此,寻找一种快速可靠且易于分辨UTI病原菌的方法迫在眉睫。
随着人工智能技术的推广,深度学习越来越普遍应用于医学[3-5]。深度学习是机器学习的分支,是一种以人工神经网络为计算架构,对数据进行表征学习的方法。卷积神经网络(convolutional neural network,CNN)作为深度学习最有效的神经网络方法,已在医学显微图像分类中有较多应用,例如:细菌性阴道病图像分类[6],糖尿病眼底视网膜病变图像分型[7],乳腺活检样本组织学切片中的癌症标志物检测[8-9],血细胞形态识别[10-12]等。然而将深度学习技术与UTI革兰染色图像分析相结合,判断中段尿沉渣中的病原菌,鲜有研究。本研究通过构建快速诊断模型EKNet,对引起UTI常见的革兰阴性杆菌大肠埃希菌和肺炎克雷伯菌[13]进行分类,以实现对UTI病原菌的快速诊断。
1 资料与方法
1.1一般资料 选取2020年5月至12月在广东医科大学附属厚街医院确诊UTI(未用抗菌药物)病原菌为大肠埃希菌、肺炎克雷伯菌患者的中段尿标本,其中大肠埃希菌368例,肺炎克雷伯菌292例,采用ARIS 2X微生物系统鉴定病原菌。
1.2仪器与试剂 革兰染色液(珠海贝索公司),BX51显微镜(奥林巴斯公司),TK-C9501EC高清彩色摄像机(日本JVC公司),ARIS 2X微生物鉴定系统(美国赛默飞公司),电热恒温培养箱(德国BINDER公司)。
1.3方法
1.3.1样本收集、处理与图像采集 留取UTI患者清洁中段尿5 mL,离心后吸取尿沉渣涂片,自然干燥,革兰染色。收集经ARIS 2X微生物系统鉴定病原菌为大肠埃希菌或肺炎克雷伯菌的尿沉渣标本涂片,采集细菌显微图像。见图1。为保持数据来源的多样性和真实性,每份标本仅选取1张图像。显微图像于正常临床检查过程中收集,为达到真实性,没有对深度学习的显微图像的生物丰度或染色质量进行预筛选,图像在染色强度、染色伪影和样本分布方面具有典型的可变性。
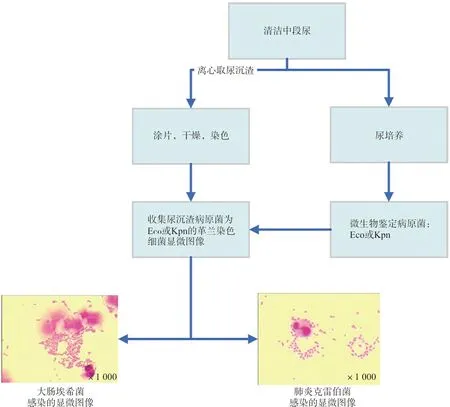
图1 显微图像采集步骤(革兰染色)
1.3.2显微图像数据集的划分 为验证学习模型的训练有效性,将760像素×570像素的显微图像数据按照8∶1∶1的比例随机划分为训练集、验证集和测试集3个部分。训练集用于训练模型,调整参数;验证集用于检验模型的效果;测试集用于检验最优模型的性能和分类能力。见表1。

表1 各类样本图像数目
1.3.3细菌显微图像的预处理 为减少细菌图像特征之间的相关性,同时保证模型的泛化能力,对原始图像进行归一化,对训练集图像以随机水平翻转的方式进行增强操作,将数据增强后的细菌图像每批8张图片进行归一化后传入网络。
1.3.4基于深度学习方法的网络训练 细菌图像分类的深度学习方法是一种改进的轻量级残差网络,暂命名为EKNet(取Eco和Kpn英文缩写的首字母),网络参数个数为2 791 826,训练优化器使用Adam,学习率为0.000 1,将预处理后的细菌图像输入残差网络模型,计算交叉熵损失,通过反向传播算法进行参数优化,获得预训练模型EKNet。使用基于PyTorch的开源深度学习框架LisaDPF(开源地址:https://gitee.com/summit/lisadpf),可以让使用者针对应用场景快速生成训练模型。该模型基于残差网络ResNet18进行改进,网络由残差前的卷积层(conv)、池化层(pool)、残差块层(Bottleneck)和全连接层(fc)组成,EKNet的网络结构见图2。

注:网络输入显微细菌图像,输出所属细菌分类的概率。框中的数字代表每个模块输出的通道数。conv,卷积层;pool,最大池化层;bottleneck,残差块层;avg pool,平均池化层;fc,全连接层。
1.3.5模型比对 以图像分类常用的ResNet50模型和AlexNet模型作为对照,对表1训练集和验证集的显微图像进行迭代训练和验证,并对模型进行比对,评估EKNet模型的实用性。
1.3.6临床测试 为评估EKNet模型的性能,使用UTI患者尿沉渣显微图像作测试集(病原菌为未知的革兰阴性杆菌)对模型进行测试,输出大肠埃希菌和肺炎克雷伯菌2种细菌类别中的每一种的相对概率,以概率高的细菌作为测试集的输出结果,测试集的正确性以尿培养的结果作为评判。通过对测试集的准确性进行量化,初步评估模型的泛化能力。
2 结果
2.1深度学习模型的建立 本研究模型在残差网络ResNet18基础上进行改进。与ResNet网络模型相比,当前EKNet 4个残差块层的通道数均为ResNet18的一半,在不降低精度的同时,EKNet模型的参数量大幅度减少,以降低模型的复杂度,提高其泛化性,并减少推理时间。该模型测试单个样本的平均用时是0.165 s,优于ResNet模型的0.38 s和AlexNet模型的0.66 s。使用不同的模型对表1训练集和验证集的显微图像进行迭代训练和验证,结果表明,EKNet模型能对2种不同的细菌进行精确的分类。EKNet模型实验结果的验证集准确率达到98.8%,优于ResNet50模型的96.4%和AlexNet模型的78.6%。见表2。EKNet模型的训练损失和验证损失均能保持一致下降,表明训练过程无过拟合迹象,见图3A。相对于EKNet模型,ResNet50模型的训练集与验证集的损失函数在纵向存在一定距离,即存在一定的过拟合现象,见图3B。与EKNet模型相比,AlexNet模型的训练无法正常收敛,存在明显过拟合现象,见图3C。

表2 不同模型的训练结果对比
2.2临床测试结果 104例测试集包含大肠埃希菌56例,肺炎克雷伯菌48例,输出结果与尿培养结果完全吻合,正确率为100%。该模型测试样本的平均用时为0.165 s。

注:A,EKNet模型;B,ResNet50模型;C,AlexNet模型。
3 讨论
UTI是常见的感染性疾病,革兰染色法能在较短时间内对感染的细菌进行识别[14],但革兰染色结果只能提供细菌形态学依据,不能定位病原学种类。本研究选择使用CNN,是因为其在图像分析任务中具有出色的能力,除图像采集之外无需人工干预。本研究拟建立EKNet模型对大肠埃希菌和肺炎克雷伯菌这2种细菌图像进行快速判读分析,快速可靠地分辨UTI病原菌,为临床早期诊断提供重要的指导意义。
ResNet18是针对ImageNet自然场景数据集的分类,由于细菌显微图像与通过摄像机采集的自然场景图像在采集方式、数据源上不一致,使用原模型对细菌进行分类可能会失效,而且网络模型深度增加容易出现参数量过多、过拟合以及难以训练等问题[15]。因此,本研究在残差网络ResNet18基础上进行改进:引入残差块、进行数据增强和归一化。引入的残差块有利于网络在梯度下降时的快速收敛,提高模型的表达能力;数据增强能有效地缓解训练数据不足导致的深度学习模型过拟合,进一步提高模型的精度和泛化能力;归一化处理能消除采集数据过程中存在的噪音,使采集的数据统一到一个特征维度,有利于网络训练在反向传播期间更好地收敛。与ResNet18网络模型相比,EKNet模型的特征通道数量减少,通过减少模型参数以降低模型的复杂度,对图像的识别准确率达98.8%。在临床测试中,EKNet模型的输出结果与尿培养结果完全吻合,显示该模型具有较好的泛化能力,在大肠埃希菌和肺炎克雷伯菌这2种细菌之间具有强大的区分能力。由此可见,本研究构建的EKNet模型实现了对泌尿系统感染常见细菌的快速诊断。
本研究也存在一定的局限性。该模型目前仅包含大肠埃希菌和肺炎克雷伯菌2种病原体,缺乏抗菌药物作用下的细菌L型和其他革兰阴性杆菌。课题组正在添加样本的种类与数量,以期区分更多不同类别的细菌。我们相信,提供深度学习图像分类的结果,可以为微生物的诊断提供广阔的前景。
综上所述,本研究成功构建的大肠埃希菌和肺炎克雷伯菌的图像诊断模型,具有识别准确率高、速度快、操作简便、泛化强等优点,为临床UTI的快速诊断提供参考。
