新型冠状病毒相关TMPRSS2蛋白结构特征和抗原表位分析
2021-06-30戴姿薇
戴姿薇, 唐 标
(湖南中医药大学 医学院,湖南 长沙 410208)
由新型冠状病毒(Severe acute respiratory syndrome coronavirus 2, SARS-CoV-2)引起的新型冠状病毒肺炎给全球公共卫生带来了巨大的威胁,且因其蔓延所造成的新型冠状病毒肺炎疫情成为国际关注的突发公共卫生事件[1]。SARS-CoV-2是一种单链正股非片段性且隶属于β类RNA冠状病毒组的新型病毒,其核心区被包裹于自身刺突蛋白(Spike Protein, S蛋白)、膜糖蛋白、核衣壳蛋白、包膜蛋白组成的外围包膜中。S蛋白由S1亚基和S2亚基组成,S1亚基负责与受体结合,S2亚基负责膜融合[2]。当SARS-CoV-2感染宿主时,S蛋白通过与宿主同源血管紧张素转换酶2(Angiotensin-Converting Enzyme 2,ACE2)受体或CD147跨膜糖蛋白的结合而进入宿主细胞,由跨膜蛋白酶丝氨酸2(transmembrane protease serine 2, TMPRSS2)切割并激活,然后S2亚基和TMPRSS2联合触发病毒包膜与宿主包膜的融合,随后 SARS-CoV-2将其基因组释放至宿主细胞质中,在非结构蛋白的帮助下组装子代病毒释放到胞外[3]。在SARA-CoV-2感染人体的过程中,TMPRSS2是S蛋白发挥作用的枢纽,是SARS-CoV-2进入宿主并发挥其传染致病性的关键环节,根据TMPRSS2的结构和功能特性研制抗新型冠状病毒肺炎药物,可能为当前新型冠状病毒疫情的防控提供重要方向[4-6]。本研究采用生物信息学方法,利用ProtParam、ProtScale服务器对TMPRSS2进行一级结构如氨基酸理化性质、疏水性的分析预测;利用COILS Server、SignalP、TMPred、TargetP Server、NetPhos Server、NetNGlyc Server服务器对TMPRSS2功能结构如卷曲螺旋区、信号肽、跨膜结构域、亚细胞定位、磷酸化位点、糖基化位点的分析预测;利用SOPMA、Pfam、SWISS-MODEL服务器对TMPRSS2进行二级结构、结构域、三级结构的分析预测;利用IEBD对TMPRSS2进行B细胞表位、T细胞表位的预测分析。从TMPRSS2的理化特性、结构特征、抗原表位等方面较全面地分析其蛋白特质,为基于TMPRSS2研发抗SARS-CoV-2的药物实验提供参考。
1 材料与方法
1.1 材料
1.1.1 数据来源 登录美国国家生物信息中心NCBI网站(NCBI网址:http://www.ncbi.nlm.nih.gov/)获取TMPRSS2蛋白酶氨基酸序列信息,并下载对应FASTA格式的氨基酸序列(ACCESSION: O15393)。
1.1.2 主要数据库 利用ProtParam分析预测TMPRSS2蛋白酶氨基酸组成、理论等电点、分子质量等理化性质;利用ProtScale分析预测TMPRSS2蛋白酶疏水性;利用COILS Server分析预测TMPRSS2蛋白酶卷曲螺旋区;利用SignalP分析预测TMPRSS2蛋白酶信号肽区域;利用TMPred分析预测TMPRSS2蛋白酶跨膜结构域;利用TargetP Server分析预测TMPRSS2蛋白酶亚细胞定位;利用NetPhos Server分析预测TMPRSS2蛋白酶磷酸化位点;利用NetNGlyc Server分析预测TMPRSS2蛋白酶糖基化位点;利用SOPMA分析预测TMPRSS2蛋白酶二级结构;利用Pfam分析预测TMPRSS2蛋白酶结构域;利用SWISS-MODEL同级建模TMPRSS2蛋白酶三级结构;利用IEBD分析预测TMPRSS2蛋白酶B细胞、T细胞表位。
1.2 方法
1.2.1 TMPRSS2蛋白酶一级结构分析预测 于ProtParam网页上将TMPRSS2蛋白酶FASTA格式氨基酸序列输入,选择“Compute parameters”输出,呈现TMPRSS2蛋白酶氨基酸数量、分子质量、理论等电点、氨基酸组成、正负电荷残基总数、原子组成、分子式、脂肪系数、总平均亲水性等理化性质分析预测结果;于ProtScale网页上将TMPRSS2蛋白酶FASTA格式氨基酸序列输入,选择“linear”权重变化模型与“Hphob./Kyte&Doolittle”计算方法,选择“Submit”输出,呈现TMPRSS2蛋白酶疏水性分析预测结果[7-8]。
1.2.2 TMPRSS2蛋白酶功能结构分析预测 于COILS Server网页上将TMPRSS2蛋白酶FASTA格式氨基酸序列输入,选择默认矩阵MTIDK,赋予无权重选项后呈现TMPRSS2蛋白酶卷曲螺旋区分析预测结果;于SignalP网页上选择“Eukaryotes”数据库训练集,D-cutoff values选择“Default”默认值,Method选择“Input sequences may include TM regions”,以“standard”模式输出,呈现TMPRSS2蛋白酶信号肽分析预测结果;于TMPred网页上选择以17~33个氨基酸作为跨膜螺旋疏水部分要求长度后输入TMPRSS2蛋白酶FASTA格式氨基酸,选择“Run TMPred”输出,呈现TMPRSS2蛋白酶跨膜结构域分析预测结果;于TargetP Server网页上选择“non-plant”的生物类别,以“Long output”形式输出,呈现TMPRSS2蛋白酶亚细胞定位分析预测结果;于NetPhos Server网页上选择“all three”的预测范围和“classic”的输出形式,呈现TMPRSS2蛋白酶磷酸化位点分析预测结果;于NetNGlyc Server网页上将TMPRSS2蛋白酶FASTA格式氨基酸序列输入,选择“Generate graphics”,选择“Submit”输出,呈现TMPRSS2蛋白酶糖基化位点分析预测结果[9-13]。
1.2.3 TMPRSS2蛋白酶高级结构分析预测 于SOPMA网页上将TMPRSS2蛋白酶FASTA格式氨基酸序列输入,选择“4(Helix,Sheet,Turn,Coil)”的预测范围,Similarity threshold填入“8”,选择“SUBMIT”输出,呈现TMPRSS2蛋白酶二级结构分析预测结果;于Pfam网页上选择“SEQUENCE SEARCH”输入TMPRSS2蛋白酶FASTA格式的氨基酸序列,选择“GO”输出,呈现TMPRSS2蛋白酶结构域分析预测结果;于SWISS-MODEL网页上将TMPRSS2蛋白酶FASTA格式氨基酸序列输入,选择“Build Model”同源建模,根据模型评分成功选择模型后即呈现TMPRSS2蛋白酶三级结构分析预测结果;于CHPModels服务器输入TMPRSS2蛋白酶氨基酸FASTA格式,点击“submit”输出TMPRSS2三级结构同源模体预测结果[14-16]。
1.2.4 TMPRSS2蛋白酶B细胞表位和T细胞表位分析预测 于IEBD网页上选择“B Cell Epitope Prediction”后点击“Prediction of linear epitopes from protein sequence”,并输入TMPRSS2的FASTA格式的氨基酸序列,选择“Bepipred Linear Epitope Prediction”模式输出,呈现TMPRSS2蛋白酶的B细胞表位预测结果;于IEBD网页上选择“T Cell Epitope Prediction”中的“MHC Ⅰ Binding”输出选项,呈现TMPRSS2蛋白酶的T细胞表位预测结果。
2 结果与分析
2.1 TMPRSS2蛋白酶氨基酸理化性质预测分析
利用ProtParam服务器在线预测分析TMPRSS2蛋白酶氨基酸理化性质,结果如表1所示。TMPRSS2蛋白酶由492个氨基酸组成,其中所占比例前四位依次为丝氨酸(Ser)、甘氨酸(Gly)、缬氨酸(Val)、脯氨酸(Pro),不存在吡咯赖氨酸(Pyl)和硒代胱氨酸(Sec);正电荷残基数39个,负电荷残基数35个;理论等电点8.12;分子质量53 859.18;不稳定系数41.94;脂肪系数72.70;总平均亲水性-0.248。
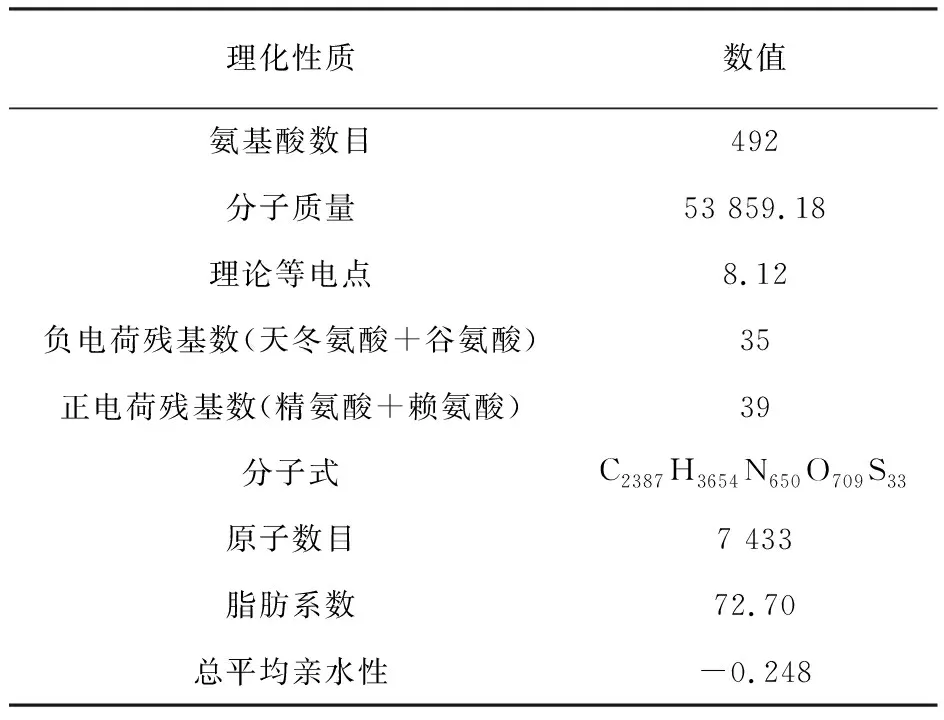
表1 TMPRSS2氨基酸理化性质部分分析结果
2.2 TMPRSS2蛋白酶疏水性预测分析
利用ProtScale服务器在线预测分析TMPRSS2蛋白酶疏水性,结果如图1所示。其中评分最高的氨基酸依次为第98位的丙氨酸、第97位的甘氨酸、第423位的丙氨酸、第88位的苏氨酸,表明此四处位置氨基酸疏水性最强;评分最低的氨基酸为第340位的天冬氨酸、第341位的丝氨酸,分数均为-2.733,表明此两处位置氨基酸亲水性最强。

图1 ProtScale预测TMPRSS2疏水性Fig.1 Hydrophobic characteristics analysis results of TMPRSS2 via ProtScale
2.3 TMPRSS2蛋白酶卷曲螺旋区预测分析
利用COILS Server服务器在线预测分析TMPRSS2卷曲螺旋结构,以0.5为阈值筛选结果,并未预测到TMPRSS2具有卷曲螺旋结构。
2.4 TMPRSS2蛋白酶信号肽预测分析
利用SignalP服务器在线预测分析TMPRSS2信号肽区段,结果如图2所示。C分值代表预测剪切点分值,S分值代表信号肽预测分值,Y分值代表综合剪切点分值,总体来看TMPRSS2可能不具有信号肽特点。
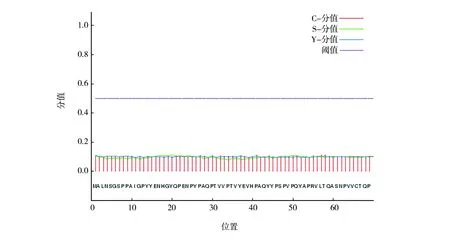
图2 SignalP预测TMPRSS2信号肽区段Fig.2 Signal peptide analysis results of TMPRSS2 via SignalP
2.5 TMPRSS2蛋白酶跨膜结构区预测分析
利用TMPred服务器在线预测分析TMPRSS2跨膜结构区,结果如图3所示。结果分为两个有意义的跨膜结构区:跨膜方向为由膜内向外的区段和由膜外向内的区段。经预测可知,跨膜方向由膜内向外的预测结果为5个区域,分数由高到低依次为84~102位、275~292位、310~326位、416~432位、450~467位;跨膜方向由膜外向膜内的预测结果为5个区域,分数由高到低依次为87~106位、230~251位、277~293位、308~324位、415~432位。

图3 TMPred预测TMPRSS2跨膜结构区Fig.3 Transmembrane domain analysis results of TMPRSS2 via SignalP
2.6 TMPRSS2蛋白酶亚细胞定位预测分析
利用TargetP Server服务器在线预测分析TMPRSS2亚细胞定位,结果如表2所示。该序列定位于线粒体分值为0.231,信号肽存在可能性分值为0.059,可靠性等级为3,因此TMPRSS2可能并非定位于线粒体且不具备分泌途径即信号肽的特点。
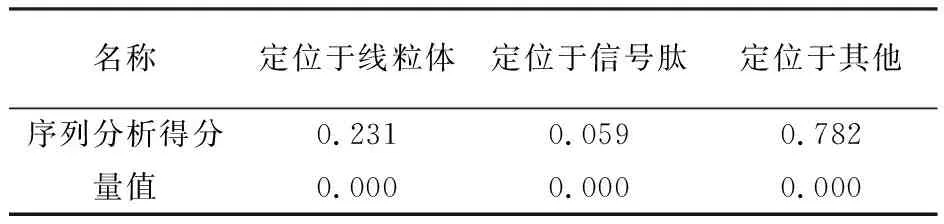
表2 TargetP Server预测TMPRSS2亚细胞定位
2.7 TMPRSS2蛋白酶磷酸化位点预测分析
利用NetPhos Server服务器在线预测分析TMPRSS2磷酸化位点,结果如图4所示。以0.5为阈值,丝氨酸磷酸化位点预测为1个,位于第18位;苏氨酸磷酸化位点预测为1个,位于第7位;酪氨酸磷酸化位点预测为2个,位于第19和23位。

图4 NetPhos Server预测TMPRSS2磷酸化位点Fig.4 Phosphorylation sites analysis results of TMPRSS2 via NetPhos Server
2.8 TMPRSS2蛋白酶糖基化位点预测分析
利用NetNGlyc Server服务器在线预测分析TMPRSS2的糖基化位点,结果如图5所示。以0.5为阈值,得到分别位于第128、213、249位置的氨基酸区域具有糖基化修饰的可能性,可能性分值分别为0.606 9、0.527 4、0.633 8。
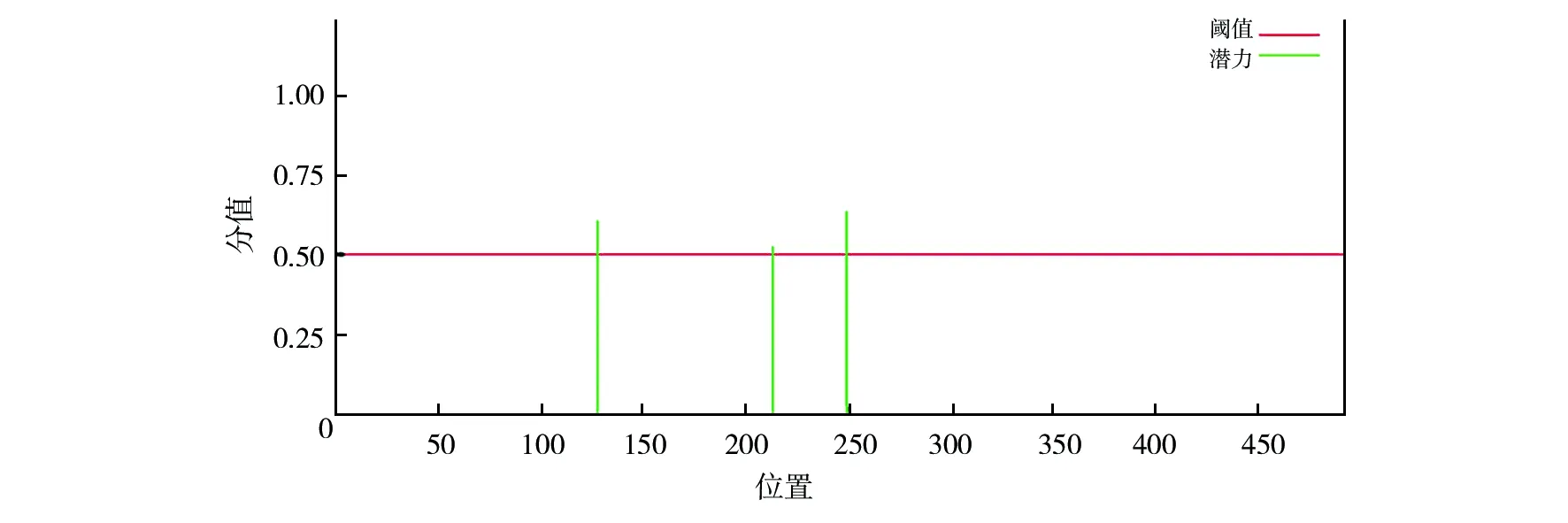
图5 NetNGlyc Server预测TMPRSS2糖基化位点Fig.5 Glycosylation sites analysis results of TMPRSS2 via NetNGlyc Server
2.9 TMPRSS2蛋白酶二级结构预测分析
利用SOPMA服务器在线预测TMPRSS2蛋白酶二级结构,结果如图6所示。α-螺旋结构占比15.45%,β-折叠结构占比20.53%,β-转角结构占比8.74%,无规则卷曲结构占比55.28%。

图6 SOPMA预测TMPRSS2二级结构Fig.6 Secondary structure prediction of TMPRSS2 via SOPMA
2.10 TMPRSS2蛋白酶结构域预测分析
利用Pfam服务器在线预测TMPRSS2蛋白酶保守结构域,如表3所示。根据UniProtKB数据库建立的来自基础序列数据库Pfamseq的Pfam-A预测到2个保守结构域:Trypsin和Scavenger receptor cysteine-rich domain;由ADDA数据中非冗余cluster自动生成补充数据库的Pfam-B预测到1个保守结构域:Low-density lipoprotein receptor domain class A。
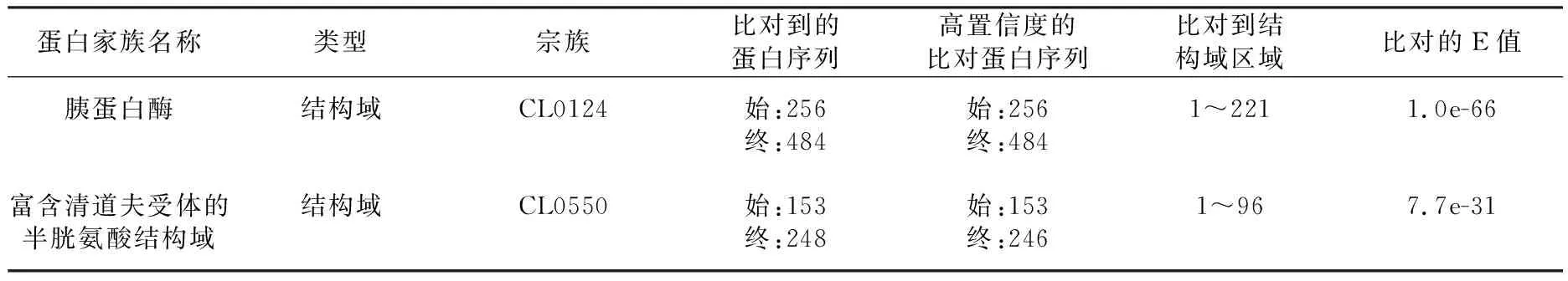
表3 Pfam预测TMPRSS2结构域信息
2.11 TMPRSS2三级结构预测分析
利用SWISS-MODEL服务器在线预测TMPRSS2三级结构,如图7所示。GMQE是一种将目标与模板对齐并进行模板搜索方法属性的质量评估方法,所得分数区间为0~1,可反映模型预测准确性以及覆盖范围,分数越高表明契合度越高;QMEAN是一种表示模型得分与相似大小实验结构所期望得分的配合度,分数在-4.0及以下表明模型质量较低。预测得知TMPRSS2蛋白酶第146~491位的氨基酸序列能与编号为5ce.1.1.A(SMTL ID)的模型同源建模,模型GMQE得分为0.53,QMEAN得分为-1.43,且其所匹配预测的寡聚态(Oligomeric state)为Monomer。如模型Ramachandran,如图8所示,绝大部分残基都位于可接受区域,表明所建三维模型较合理。同时采用CPHmodels服务器在线为TMPRSS2三级结构寻找同源模体,选取得分为200以上的模型,如表4所示,结果与SWISS-MODEL预测相符。
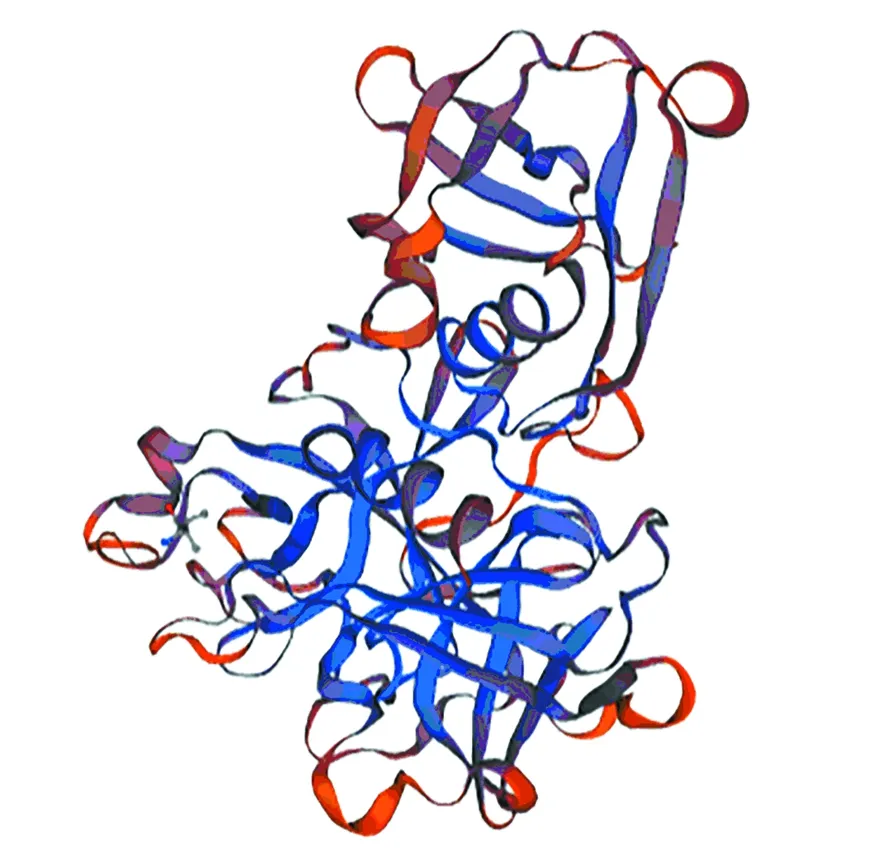
图7 SWISS-MODEL同源建模TMPRSS2三级结构Fig.7 Tertiary structure prediction of TMPRSS2 via SWISS-MODEL

图8 SWISS-MODEL同源建模TMPRSS2模型Ramachandran图Fig.8 Ramachandran Plots of TMPRSS2 via SWISS-MODEL
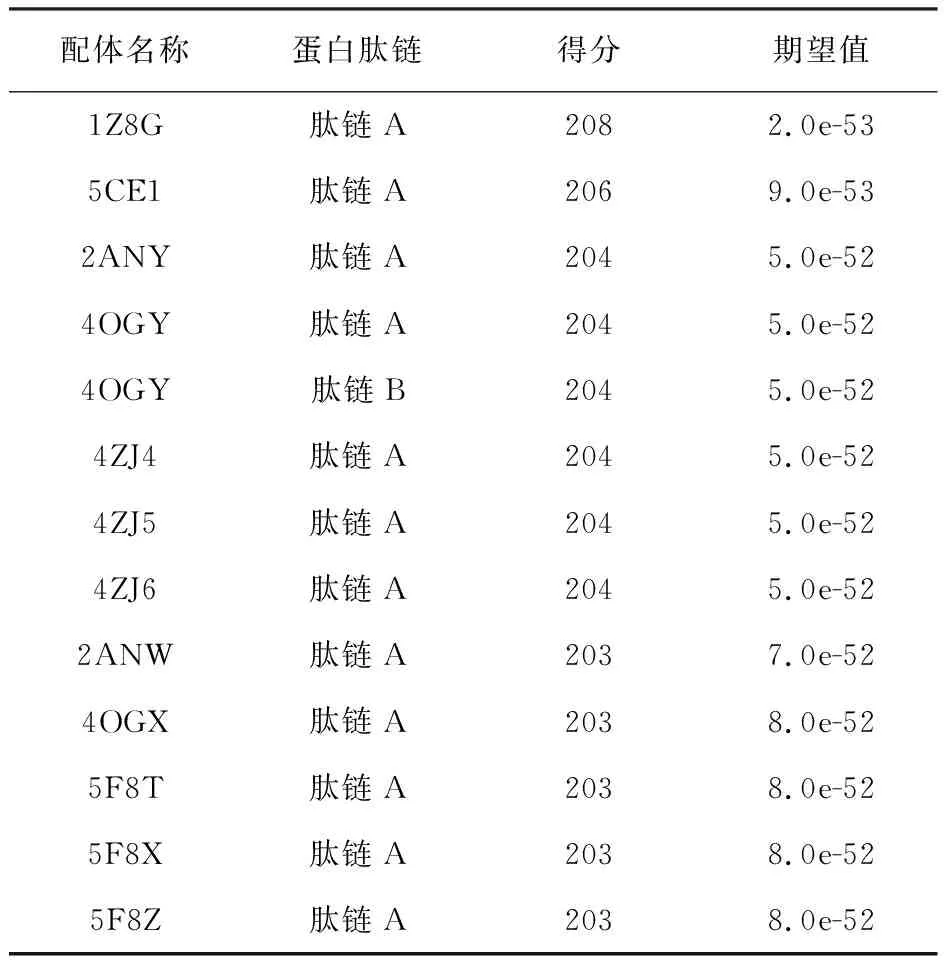
表4 CPHModels预测TMPRSS2同源模体
2.12 TMPRSS2蛋白酶B细胞表位、T细胞表位预测
利用IEBD服务器在线预测TMPRSS2蛋白酶B细胞表位,结果如图9、表5所示。以0.5为阈值可预测到表5所示的13个B细胞抗原表位,总体结果平均得分为0.506,最高得分为0.684,最低得分为0.260;利用IEBD服务器同时预测TMPRSS2蛋白酶T细胞表位,结果如表6所示。从肽-MHC I分子预测结果来看,以0.95为阈值可预测到表6所示的12个T细胞表位,其结果最高得分为0.981 967,最低得分为0.950 353。

图9 IEBD预测TMPRSS2 B细胞表位Fig.9 B cell epitopes analysis results of TMPRSS2 via IEBD

表5 IEBD预测TMPRSS2 B细胞表位信息

表6 IEBD预测TMPRSS2 T细胞表位信息
3 讨 论
SARS-CoV-2进入宿主的第一步为由病毒包膜糖蛋白控制的病毒粒子与宿主表面受体结合并与细胞膜融合的过程。研究表明,宿主蛋白酶激活病毒是膜融合的先决条件,蛋白酶的选择决定了细胞定位膜融合发生的位置,且病毒与ACE2受体的结合可以触发病毒粒子被宿主细胞内小体摄取,从而TMPRSS2得以在细胞表面或靠近细胞表面处切割病毒的S蛋白进而发挥协助病毒感染作用[17]。
本研究结果显示,由492个氨基酸组成的TMPRSS2蛋白酶被编码成功后则锚定于质膜上,可以通过Arg255和Ile256两位点间的自我催化转化成其本身的形式。本研究预测结果表明,TMPRSS2组成的氨基酸中丝氨酸占比程度最高为8.9%,而TMPRSS2蛋白酶催化结构域包含由His296、Asp345、Ser441三个氨基酸残基组成的催化三联体,正对应糜蛋白酶原的His57、Asp102、Ser195发挥其催化作用;TMPRSS2理论等电点为8.12,表明其为碱性蛋白,这与其带负电荷的氨基酸残基数小于带正电荷的氨基酸残基数预测结果是相契合的;TMPRSS2蛋白质性质较不稳定,为亲水蛋白,可用于离子交换色谱分离和纯化实验,为其体外实验探究奠定理论基础[18-19]。
蛋白质跨膜结构区可以将膜蛋白固定在包膜上,从TMPRSS2跨膜结构区预测结果来看其具有一定数量的跨膜区,提示可能作为膜受体或膜离子通道起作用;磷酸化是指肽链中的丝氨酸、苏氨酸、酪氨酸残基的侧链羟基被修饰成酸式磷酸酯多肽的过程,糖基化是指在糖基转移酶作用下将糖类转移至蛋白质和蛋白质上特殊的氨基酸残基形成糖苷键的过程,两者均是蛋白质修饰的重要环节。其中,磷酸化与转录调节、信号转导等生物学过程密切相关,糖基化可以使不同蛋白质拥有不同的标记,从而改变多肽的构象,增加蛋白质的稳定性,提示通过此二类预测修饰位点的干预可能会对抑制病毒进入宿主起作用[20-21]。
占据TMPRSS2二级结构比例最高的是无规则卷曲结构,其对于TMPRSS2的整体构象和活性有着极其重要的作用,是构成酶活性部位和特异功能部位的重要结构。从TMPRSS2结构域预测结果来看,胰蛋白酶与富含半胱氨酸结构域的清道夫受体是两个具有意义的功能结构域,两者可以由人类基因编码出具有两者酶活性的蛋白质。前者包含一个由组氨酸、天冬氨酸、丝氨酸组成的催化三联体,其中丝氨酸和组氨酸具有相等的质子份额从而增加了活性位点丝氨酸的亲核性,在蛋白水解过程中促进了其对酰胺碳的攻击;后者是一个富含清道夫受体的半胱氨酸结构域家族,主要存在于胰蛋白酶样跨膜丝氨酸蛋白酶Spinesin上游的脊椎动物序列上[22]。在寻找药物的实验设计中,晶体蛋白学的切入点即为测定蛋白质-配体复合物的晶体结构。如果配体是相对较小的分子,通常可以通过将不含有配体的蛋白质浸泡在含有配体的母液中来获得复合物的晶体结构。从TMPRSS2三级结构预测结构来看,1X50K为其一个非聚合物的配体,分子量为334.42,原子数为47,芳香键数为16。从此配体寻找蛋白质-配体作用点或许能成为治疗新冠肺炎的靶点;且从同源建模结果也可以得知1~255位氨基酸区域为TMPRSS2的非催化位点区,256~492位氨基酸区域为TMPRSS2的催化位点区;位于第296、345、441的三个位点为TMPRSS2三级结构预测活性区域;位于第160、254、329、449、491的五个位点为TMPRSS2三级结构的预测自然变异点。这些预测结果或为揭示TMPRSS2三级结构奠定了一定的理论基础。研究表明,从来自感染SARS-CoV和MERS-CoV的男性肺组织细胞系A549的雄激素依赖性可以得知雄激素反应元件参与了TMPRSS2的表达。基于此项发现以及TMPRSS2的三级结构特点,Devan等开发出一种可以与5′-WGWWCW-3′区域相互作用的聚酰胺化合物来抑制TMPRSS2的表达,聚酰胺化合物可以与TMPRSS2启动子的ARE结合,从抑制RNA转录的角度实现对TMPRSS2表达的抑制[23],因而针对TMPRSS2的聚酰胺化合物也有可能是防治SARS-CoV-2感染的治疗靶点。
B细胞表位是结合免疫球蛋白或抗体的抗原部位,B细胞在识别这些表位时可以构成抗原中暴露的溶剂区域且易接近B细胞受体,一般分为线性表位与构象表位。T细胞表位由Ⅰ类和Ⅱ类MHC分子呈现,由蛋白质降解后的多肽形成位于抗原分子内部的表位成分,在经抗原递呈细胞加工后分别被两个不同的T细胞亚群CD8和CD4 T细胞识别。从本研究预测结果来看,B细胞抗原表位平均分在阈值之上,且具有一定数量的抗原表位数,表明TMPRSS2抗原性较好。抗原表位通常位于转角与无规则卷曲结构处,呈较松散、扭曲并盘旋展示在蛋白表面和膜外区域,为B细胞表位形成提供依据。作为较理想的免疫原,抗原分子中应包含目的抗原的B细胞表位和T细胞表位,近年来更是新兴起一种重要疫苗即重组表位疫苗,因而结合本研究提出的上述B细胞表位与T细胞表位预测结果或许能为开发具有较强免疫原性的高效多价疫苗提供理论基础[24-25]。
TMPRSS2是SARS-CoV-2入侵宿主机制中的关键环节,研究报道TMPRSS2的抑制剂可抑制TMPRSS2蛋白酶活性从而抑制冠状病毒中S蛋白与肺宿主包膜融合, TMPRSS2的抑制剂可能是抗SARS-CoV-2药物研发的重要方向[26]。目前大多数研究偏向于某一个具体方面来研究TMPRSS2对新型冠状病毒的作用机制,而本研究结合生物信息学方法较全面地分析了TMPRSS2结构与功能特征,以期为丰富TMPRSS2认知以及抗SARS-CoV-2药物的相关研究提供参考。