草酸青霉线性五肽生物合成的比较转录组分析
2021-06-24宋瑾怡薛艳红刘士平
刘 超,宋瑾怡,薛艳红,熊 泽,刘士平
(三峡大学 生物与制药学院,湖北 宜昌443002)
草酸青霉(P.oxalicum)是一种广泛分布于土壤、根际、食物及动植物体内等环境中的丝状真菌[1],可防治番茄枯萎病、油菜菌核病、棉花炭疽病、柑橘绿霉病等20余种植物真菌性病害[2-3]。草酸青霉在溶磷、分解纤维素和生物修复等方面也发挥着重要作用[4-6]。2013年,我国测定第一个草酸青霉菌株(114-2)的全基因组序列,并完成基因注释:其基因组共约30 MB,预测有10 013个基因,编码9 979个蛋白质[7-9]。截止2020年4月,在GenBank数据库中共有6条草酸青霉的全基因组序列[7,10-11]。
前期从三峡河岸带地区筛选出两株草酸青霉SG-4和SJ-3,发现其对柑橘致腐菌指状青霉有强烈抑制作用[12]。通过活性追踪和系统分离鉴定,得知其抑菌物质是一种新型线性五肽(图1),结构是:O-Me-Baba-N-Me-Thr-Thr-N-Me-Val-Ser,其中Baba是β-氨基丁酸。进一步研究表明该肽活性稳定,对人体安全,是一种潜在抗生素和食品添加剂[12]。
自然界中寡肽的生物合成途径有两种,分别是核糖体途径和非核糖体途径[13-14]。前者指通过依赖核糖体的翻译途径合成寡肽,是一种初生代谢,在其基因组中一般存在目标寡肽的重复序列;后者是由一种不依赖于翻译途径的非核糖体肽合成酶(NRPS,non-ribosomal peptide synthetase)所催化,是一种次级代谢,它是寡肽合成的主要途径[15-16]。许多非核糖体肽是常见的抗生素,它们在医药、食品工业发挥了巨大作用[17-19]。根据NRPS的催化特点和寡肽的结构,开发出一系列基于NRPS基因序列来分析预测寡肽的软件,如fungiSMASH、NRPS predictor、Norine等,这些软件为寡肽生物合成途径研究提供了有利条件[20-22]。
为揭示新型五肽在草酸青霉中的合成机制,本研究测定了3株不同草酸青霉菌株的转录组序列,通过分析差异基因,从表达水平上确定候选合成基因簇,为剖析新型五肽合成机理提供了实验依据。

图1 草酸青霉中的新型线性五肽Fig.1 New linear pentapeptide in P.oxalicum
1 材料与方法
1.1 仪器与设备
BS-224s电子天平:德国赛多利斯公司产品;SW-CJ-2FD型双人实验操作台:苏州净化设备厂产品;XPX-9052 MBE数显培养箱:上海博讯实业有限公司医疗设备厂产品;ZQZY-85BN全自动控温摇床:上海知楚仪器有限公司产品;Master-s15和泰纯水仪:上海和泰仪器有限公司产品;MyCyclerTMThermal PCR仪:美国BIO-RAD公司产品;GDS-8000凝胶成像系统:美国UVP公司产品;Allegra 64R台式高速冷冻离心机:美国BECKMAN公司产品;DYCP-31DN水平电泳仪:北京市六一仪器厂产品;YM30F不锈钢智能型立式电热蒸汽消毒器:上海三申医疗器械有限公司产品;岛津高效液相色谱仪紫外-可见光检测器(SPD-16),C8液相柱(YMCTriart C8 250 mm×4.6 mm):岛津仪器苏州有限公司产品。
1.2 实验试剂
土豆:市售;葡萄糖:科密欧化学试剂有限公司产品;琼脂:金燕海洋生物股份有限公司产品;色谱级乙腈:Tedia公司产品;真菌RNA提取试剂盒:美国Omega Bio-Tek公司产品;GoldenstarTMRT6 cDNA Synthesis Kit Ver.2和T3 Super PCR Mix:均为北京擎科新业生物技术有限公司产品。
1.3 实验菌株及培养基
草酸青霉SG-4和SJ-3:来源于菌株均系三峡河岸带植物疏花水柏枝(Myricaria laxiflora)的内生真菌;114-2:来源于山东大学微生物技术国家重点实验室,由本实验室自行分离获得。以上菌株均保藏于4℃的PDA固体斜面培养基。PDA固体培养基:土豆200 g,葡萄糖20 g,琼脂20 g,水1 000 mL,pH值自然,121℃灭菌20 min。
1.4 发酵培养与RNA提取
将保存在PDA固体斜面的3株草酸青霉菌种活化培养4 d,采用无菌操作取1环孢子接种于装有200 mL的PDA液体培养基三角摇瓶中(总容积500 mL),于28℃、120 r/min条件下培养7 d,采用Omega试剂盒提取总RNA,提取方法按试剂盒的操作方法进行。
1.5 菌体质量测定和线性五肽相对丰度检测
将200 mL PDA液体中培养好的菌体抽滤,获取不同培养时间的菌丝和发酵液,菌丝烘干后称量。发酵液使用等体积无水乙醇混匀沉淀24 h,将上清液旋蒸冻干获得粗提物,使用纯水将粗提物配制成质量浓度为5 mg/mL的溶液。采用岛津高效液相色谱仪紫外-可见光检测器(SPD-16),YMCTriart C8 250 mm×4.6 mm、粒径5μm的C8色谱柱进行反向高效液相分析。流动相A为乙腈,流动相B为纯水,超声脱气后使用;检测波长为210 nm;进样体积为80μL;流量为1mL/min,柱箱温度为35℃,梯度洗脱:0~20 min,B体积分数由90%变为0%。记录5~6 min处五肽相对峰面积。
1.6 转录组测序
将质量检测合格的RNA样品使用BGISEQ-500,进行高通量RNA-Seq,具体过程如下:先用带OligodT的磁珠富集有polyA尾巴的mRNA,再用DNA探针杂交rRNA,利用RNaseH选择性消化DNA/RNA杂交链,再用DNaseI消化掉DNA探针,纯化后即得到所需RNA。然后将RNA片段化,用随机的N6引物进行反转录,进一步形成双链DNA。将双链DNA末端补平并进行5′端磷酸化,3′端形成突出一个“A”的粘末端,再连接一个3′端有凸出“T”的鼓泡状的接头。然后连接产物通过特异的引物进行PCR扩增和上机测序。
1.7 转录组数据分析
转录组数据以草酸青霉114-2为参考基因组进行有参分析,用过滤软件SOAPnuke进行统计,用Trimmomatic进行过滤,得到高质量数据(clean reads);使用HISAT软件和Bowtie2软件将clean reads分别比对到参考基因组和参考基因序列上。根据比对结果,利用RSEM软件定量分析,并以FPKM计算基因表达水平。差异表达分析重点在于找出样本之间差异表达的基因,并对这些基因进行深入挖掘分析。在分析中,差异表达基因默认定义为FDR≤0.001且倍数差异在1倍以上的基因。筛选获得的差异表达基因,根据GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genes and Genomes)注释进行结果分类,同时使用R软件中的phyper函数进行富集分析。
1.8 NRPS分析
用 线 上 工 具antiSMASH(https://fungismash.secondarymetabolites.org/#!/start?tdsourcetag=s_pcqq_aiomsg)分析草酸青霉114-2的基因组序列,统计草酸青霉次生代谢产物基因簇,根据线上产物预测功能及NRPS的A型结构域,结合转录组表达量分析NRPS基因簇。
1.9 RT-PCR验证
使用Oligo7软件设计引物,由生工生物工程(上海)股份有限公司合成,使用反转录试剂盒将所提RNA反转录成cDNA,添加引物进行RT-PCR验证,所用的引物序列见表1。

表1 实验中所用的引物序列Table 1 Primer sequences in the experiment
2 结果与分析
2.1 不同草酸青霉菌株的生产能力比较
将SG-4、SJ-3和114-2这3株草酸青霉菌在PDA液体培养基中培养7天,收集发酵液后进行高效液相色谱分析发现,菌株114-2不产线性五肽,相比之下,来自于三峡河岸带的SG-4和SJ-3线性五肽产量很高(图2(a)),同时对指状青霉显示出很强的抑制效应(图2(b)),其中菌株SJ-3的产量明显高于菌株SG-4(图2(a))。
为了明确线性五肽的合成特点,以菌株SG-4为对象,分析了菌体生长规律和合成五肽的时期。结果显示,菌体的生长从第6天开始进入对数生长期(图3(a)),而线性五肽在第4天开始累积,第8天达到最大值(图3(b)),符合次生代谢产物的特点。由于大多数寡肽由非核糖体肽合成酶(NRPS,non-ribosomal peptide synthetase)所催化,因此推测五肽极有可能由NRPS基因簇负责合成。

图2 不同草酸青霉的五肽产量及抑菌效果Fig.2 Pentapeptide productionand antifungal effect of P.oxalicum strains

图3 SG-4的生长曲线和五肽的合成时间Fig.3 Growth curve of SG-4 and synthesis time of pentapeptide
2.2 转录组测序质量分析
为明确线性五肽的生物合成机制,将上述3株草酸青霉在PDA培养基上生长6 d后提取其总RNA,质检合格后在武汉华大基因科技有限公司使用BGISEQ-500平台进行了转录组测序。由于114-2有完整的序列注释信息,故在测序时使用它为参照序列进行分析。
将测序的原始数据(raw reads)中低质量、接头污染以及未知碱基含量过高的数据过滤后,平均每个菌株产生了4 365万条高质量数据(clean reads),平均容量达6.55 GB,高质量数据比例接近97%。将所获得的clean reads使用HISAT进行参考基因组序列比对,平均比对率达到91.65%(表2)。上述结果表明,此次测序的质量有保证,结果可靠,而且3株草酸青霉的转录组序列均与参考基因组序列匹配程度高。
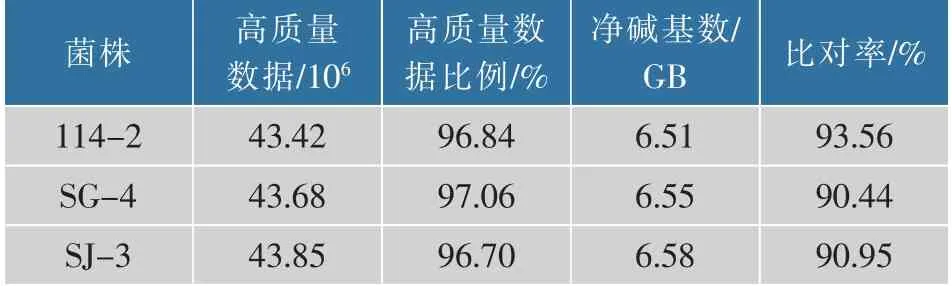
表2 转录组测序概况Table 2 A survey of transcriptome sequencing
2.3 差异表达基因分析
按照差异的倍数(|log2(Fold Change)|)和错误发现率(FDR)来筛选差异表达基因,分析中将同时满足|log2(Fold Change)|≥1且FDR≤0.001的转录本确定为差异表达基因[23]。3个转录组中的表达发生显著变化的基因数如表3所示。

表3 差异表达的基因数Table 3 Number of genes differentially expressed
上述结果表明,产五肽的菌株SG-4和SJ-3,其差异表达基因比例只有16.32%,明显比不产五肽的菌株114-2要少,这说明产五肽菌株的遗传背景有一定的共性。因此,在后期的比较中重点关注在SG-4和SJ-3中有相同变化趋势的基因,即114-2对SG-4及114-2对SJ-3中的2 239个基因(图4)。

图4 在114-2对SG-4和114-2对SJ-3中的差异表达基因Fig.4 Differentially expressed genes in 114-2 vs SG-4 and 114-2 vs.SJ-3
2.4 差异表达基因的功能注释
对于3株草酸青霉(114-2,SG-4和SJ-3)共同差异表达的基因分别采用GO(Gene Ontology)和KEGG(Kyoto Encyclopedia of Genesand Genomes)分类,富集分析了其可能的功能及所参与的细胞代谢过程。GO分类表明,上述1 823个基因主要参与生物调节、细胞成分组织或生物发生、细胞进程、代谢过程、大分子复合物、细胞膜、细胞器及催化活性等生物学过程中(图5(a))。而KEGG分类表明差异集中在糖代谢、脂代谢、氨基酸代谢、物质转运及信号传导过程(图5(b))。GO富集表明,差异基因主要在调节催化活性、膜结构与功能、氧化还原代谢等过程中发挥作用(图5(c)),KEGG富集表明,差异基因主要集中在抗生素合成、脂肪酸分解和氨基酸代谢等途径(图5(d))。

图5 差异基因表达分析Fig.5 Differential gene expression analyses
2.5 NRPS分析
由于草酸青霉所合成的线性五肽很有可能是由NRPS催化合成,因此利用线上分析工具antiSMASH[20],分析数据库内草酸青霉114-2的全基因组序列中可能的NRPS基因簇,发现共有33个可能的NRPS。将这些NRPS在有参转录组中对应后,分析其相应的表达量(表4)。分析结果表明,在33个NRPS中,共有16个NRPS的表达量与五肽合成的变化趋势一致(这16个NRPS在表4中用粗体给出),因此筛选范围进一步缩小。其中PDE_01071和PDE_01077属同一个基因簇,因此共筛选出15个基因簇。
为保证筛选结果的正确性,对部分候选基因进行了RT-PCR验证。结果表明,大多基因的表达均与测序结果一致,尤其是PDE_01071基因簇,该基因簇中有9个基因的表达在3株菌株中均存在明显差异,相对于不产五肽的草酸青霉114-2,均为上调基因,而且RT-PCR结果与测序表达结果完全一致(图6(a)),特别是结合了不同时间的表达情况,在生长4 d时该基因簇基本不表达(图6(b)),与五肽的生产时间相吻合,而且该基因簇中有多个甲基转移酶,说明该基因簇可能参与草酸青霉中线性五肽的合成。
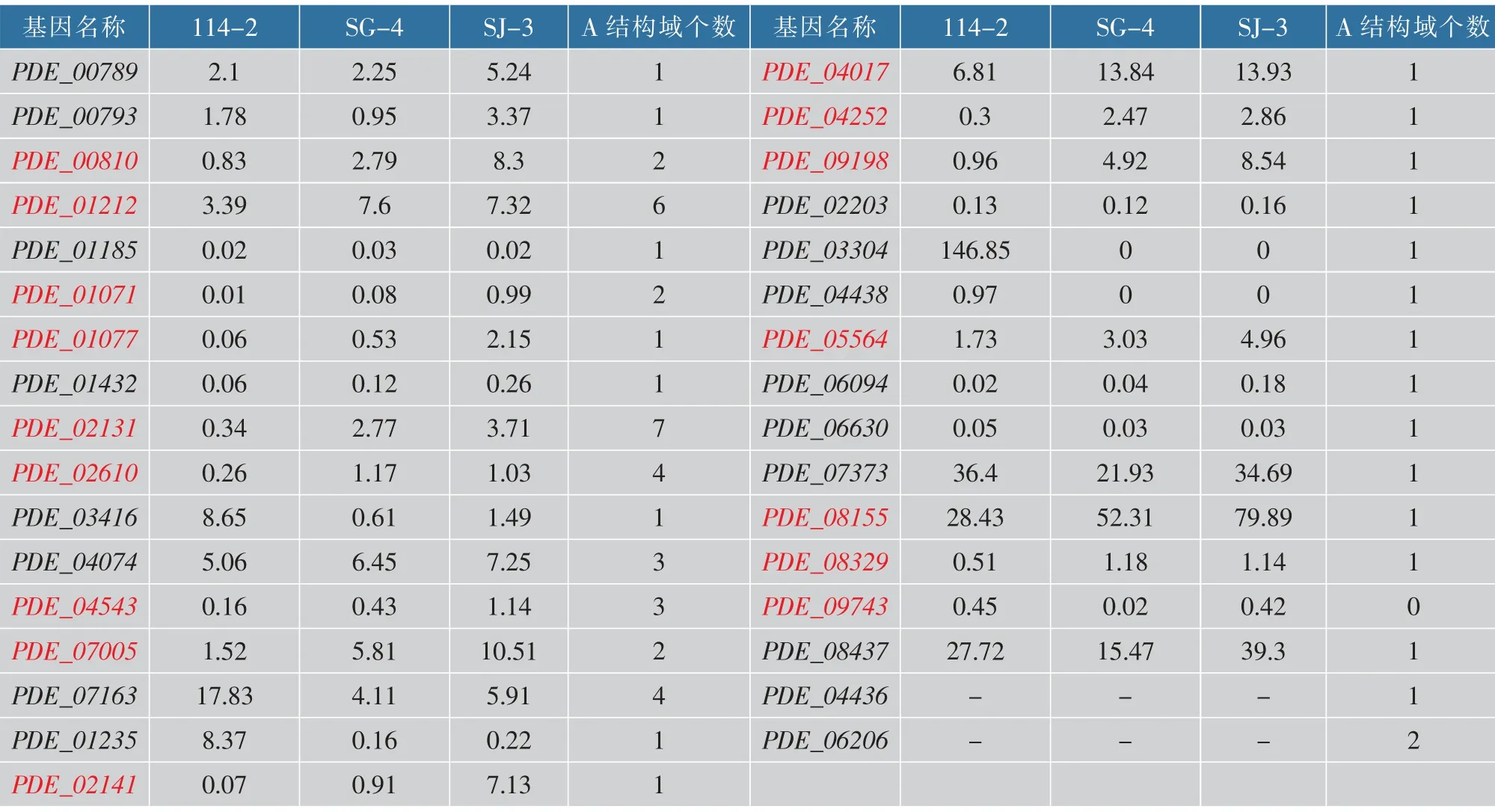
表4 草酸青霉中所有候选NRPS基因的表达量Table 4 Expression of all candidate NRPS genes in P.oxalicum

图6 RT-PCR验证表达结果Fig.6 RT-PCR to verify the expression
3 结语
本研究基于BGISEQ-500测序平台,分析比较了3株草酸青霉在培养7 d时的转录组测序数据,并基于草酸青霉114-2的全基因组进行了有参比对,同时对差异表达基因进行了分析;根据五肽的合成特点,筛选出一个最有可能负责草酸青霉中五肽合成的NRPS,即PDE_01071基因簇。值得一提的是,该结果与正在进行的不同生长时期的无参转录组测序结果相吻合(另文发表),所有这些尝试为明确其生物合成机制奠定了基础。后面的工作中,需对基因簇成员进行针对性诱变,在明确酶活性和基因功能的基础上,对其合成机理进行解析。另外,NRPS催化寡肽合成的过程和机理很复杂,目标产物是五肽,可是表3中候选NRPS的A结构域的数字很少有等于5的,这可能是由于在合成中采用了迭代模式。
