决明胰蛋白酶抑制剂的同源建模与定点突变
2021-06-23李超林周嘉裕
李超林,张 田,邹 萌,廖 海,周嘉裕
(西南交通大学 生命科学与工程学院,成都 610031)
Kunitz蛋白酶抑制剂(Kunitz protease inhibitor,KPI)是存在于植物中的一类活性肽,能够与胰蛋白酶等丝氨酸蛋白酶形成复合物,竞争性抑制靶酶的活性[1]。KPI在植物抵抗生物及非生物胁迫中发挥重要作用,具有抗虫、抗肿瘤、抗感染、抗病毒和抗真菌等多种生物学活性。牛蓓等[2]将麻疯树Kunitz型蛋白酶抑制剂基因转入烟草中,通过叶片饲养棉铃虫幼虫实验证明,转基因植株对棉铃虫具有一定的抗虫性;Fang等[3]从韩国大黑豆中纯化出Kunitz型胰蛋白酶抑制剂,能够抑制HIV-1反转录酶的活性,具有抑制HIV-1病毒增殖的潜能。
分析大豆胰蛋白酶抑制剂(Soybean trypsin inhibitor,STI)等KPI家族成员的三维结构晶体,发现它们具有典型的β-三叶草(β-trefoil)型三维结构。该类结构包含了一个由12个反向平行β-折叠组成的疏水核心,其中6个β-折叠形成底部的β-桶(β-Barrel),其余6个β-折叠形成疏水核心顶部的盖子(Lid)。该疏水核心的稳定性对于整体结构的维系发挥关键性作用。根据序列比对分析,发现疏水核心中存在较多的芳香族与脂肪族等疏水性氨基酸,推测它们通过疏水作用和氢键与邻近的氨基酸相互联系,参与核心结构的形成与稳定。由此,确定这些疏水性氨基酸并认识其作用能够有助于认识β-三叶草结构形成的分子机制,并对未来的结构改造具有指导作用。
决明(Cassiaobtusifolia)为豆科决明属植物。实验室在前期研究中,从决明种子中克隆得到决明胰蛋白酶抑制剂(Cassiaobtusifoliatrypsin inhibitor,CoTI,GenBank登录号AIU39184.1)基因的全长cDNA序列,并获得有活性的重组蛋白[4]。分析CoTI氨基酸序列,发现CoTI与其他KPI成员均含有1个保守的Trp114残基,随后利用同源建模的方法预测CoTI的三维结构及Trp114残基在三维结构中可能的作用,最后通过定点突变对推测进行验证,为CoTI的结构研究及可能的分子改造奠定理论基础。
1 材料与方法
1.1 材料
CoTI-pET28a重组大肠杆菌表达载体由本实验室构建并保存,大肠杆菌Rosseta(DE3)由本实验室保存;限制性内切酶、Prime star DNA聚合酶、T4 DNA连接酶均购自TaKaRa公司;BAPNA(N-Benzoyl-DL-arginine-p-nitroaniline)购自Sigma公司;质粒提取试剂盒Omega Plasmid Mini Kit I、胶回收试剂盒Gel Extraction Kit为OMEGA公司产品;定点突变系统Mut Express II Fast Mutagenesis Kit V2购自南京诺唯赞公司;异丙基硫代半乳糖苷(IPTG)购自MEKER公司;卡那霉素、氯霉素购自上海生工生物工程有限公司。
1.2 方法
1.2.1 序列比对及同源建模
从PDB数据库中选择同源建模的模板序列,其氨基酸序列与CoTI的一致性大于或等于30%,使用SWISS-MODEL对CoTI进行同源建模获得其三维结构。使用MEGA7.0进行多序列比对,使用PyMol对其模型进行可视化分析和处理。
1.2.2 模型验证
利用PROCHECK和Verify-3D程序对模型进行评估。Ramachandran plot用于阐述肽平面内两个二面角(φ与ψ)的比值,以表明氨基酸残基的允许和不允许的构象,通过PROCHECK 计算的 Psi/Phi Ramachandran图来评估模型的立体化学可靠性[5]。蛋白质侧链氨基酸残基的兼容性通过Verify-3D进行评价[6],至少80%的氨基酸残基得分≥0.2则通过验证。
1.2.3CoTI基因的定点突变
定点突变采用ClonExpress公司生产的Mut Express II Fast Mutagenesis Kit V2,基于一步法[7],利用CoTI-pET28重组载体为模板,根据W114所对应的核苷酸设计突变引物(表1)。

表1 突变引物及其序列
PCR反应体系总量为50 μL,在美国Applied Biosystems公司Veriti 96孔快速PCR 仪上进行。PCR反应条件:95 ℃预变性30 s;95 ℃变性15 s,63 ℃退火15 s,30个循环;72 ℃延伸50 s;最后72 ℃再延伸300 s。突变质粒DNA的片段回收、酶切、克隆、转化方法均参照文献[8]和试剂盒说明。最后突变体菌株提取质粒、酶切和PCR鉴定,送往成都擎科梓熙生物技术有限公司测序,验证突变的正确性。
1.2.4 突变体的表达、纯化及活性检测
(1)参照文献[8]:突变体菌株在含卡那霉素和氯霉素的液体LB培养基中37 ℃、200 r/min培养到A600值为0.6~0.8时,加入IPTG使其终浓度为1 mmol/L,过夜诱导表达(27 ℃,180 r/min)。诱导菌液使用超声破碎仪(总时长为30 min,超声3 s/间隙3 s,功率45 W)进行破碎,经Ni2+亲和柱纯化后得到纯化突变体。(2)突变体的活性检测参照文献[9]:采用BAPNA(N-benzoyl-DL-arginine-p-nitroaniline)为胰蛋白酶底物,活性检测实验重复3次。(3)菜青虫中肠胰蛋白酶提取方法参考文献[9]:取4~5龄虫固定于滴有石蜡的托盘,托盘置于冰上,截取菜青虫中肠及其内含物,用液氮研磨成粉末,加入150 mmol/L NaCl溶液1 mL,制成匀浆,于冰浴中抽提蛋白10 min后,使用12 000 r/min,4 ℃离心15 min,取上清液即为中肠酶液,BCA蛋白浓度测定试剂盒测定浓度,保存于-80 ℃备用。
1.2.5 突变体的荧光光谱检测
平衡CoTI和突变体蛋白液的终浓度为1.0 mmol/L,然后用日本日立公司F-7000光度计测定相应的吸光值。检测条件限定为:280 nm的激发波长,激发狭缝宽度为5 nm,发射狭缝宽度为10 nm,扫描速度为1 200 nm/min,在荧光分光光度计上记录CoTI和突变体蛋白在300~400 nm范围内的荧光发射光谱。
2 结果与分析
2.1 序列比对和同源建模
Blast结果显示:CoTI的氨基酸序列与大豆胰蛋白酶抑制剂STI(Soybean trypsin inhibitor,PDBid:1BA7)同源性最高,达到了35%的序列相似性(图1)。因此,选择STI作为同源建模的模板。二级结构预测结果表明:CoTI与大多数Kunitz类胰蛋白酶抑制剂一样,由12个β折叠组成(图1)。CoTI模型与STI的RMSD为1.33,Z Score为10.4,表明这两者具有相似的三维整体结构。
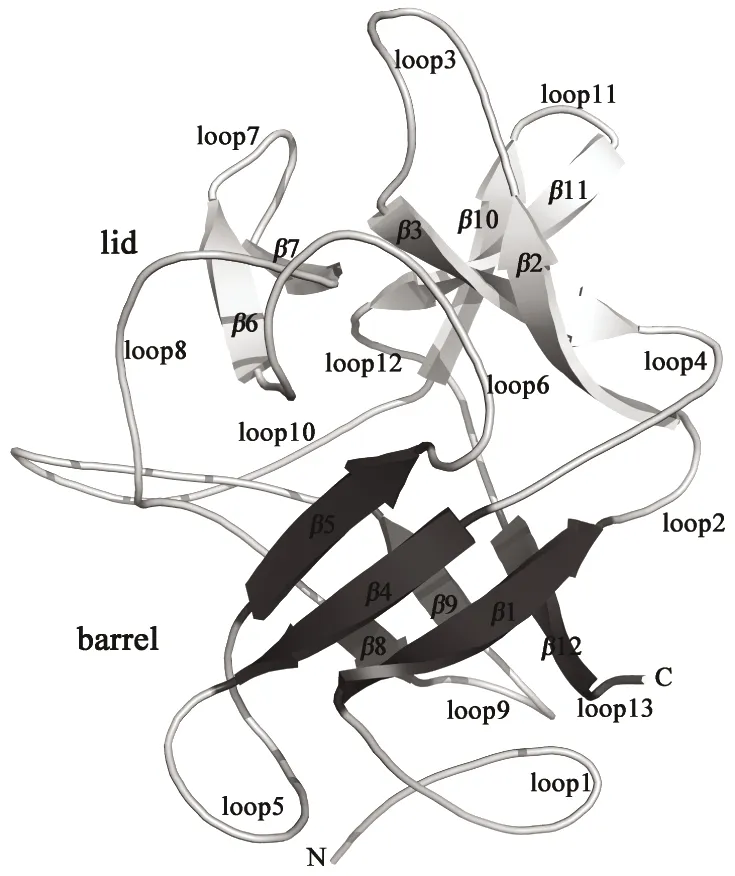
图1 CoTI同源建模结构
2.2 验证建模模型
使用PROCHECK和Verify-3D程序对CoTI模型进行评估。PROCHECK计算的Psi/Phi Ramachandran图结果表明CoTI模型的绝大多数φ、ψ二面角均处于合理范围内,其中允许区域占83.2%,最大允许区域占16.1%,不允许区域仅为0.7%,见图2(a)。Verify-3D 分析结果显示CoTI模型中有94.77%的侧链氨基酸残基得分在0.2以上,具体见图2(b)。

图2 拉氏图(a)和 CoTI模型的Verify 3D评价图(b)
2.3 CoTI蛋白模型分析
CoTI三维结构由12个反向平行β-折叠和13个loop环组成(图1),含有两个二硫键(Cys64-Cys108、Cys156-Cys164)。CoTI的抑制中心环位于β4与β5之间,活性基序为L84-V85-R86-P87-T88,在与靶蛋白酶反应时,该Loop可能插入到胰蛋白酶的底物口袋中,从而发挥抑制作用。CoTI形成β-三叶草结构,12个反向平行β-折叠中的6个形成底部的β-桶(β-Barrel),其余6个β-折叠形成疏水核心顶部的盖子(Lid)。Kunitz丝氨酸蛋白酶抑制剂β桶蛋白疏水核心的包装是家族特异性的。CoTI的Trp114残基位于桶盖连接处。通过与其他Kunitz丝氨酸蛋白酶抑制剂序列(ECTI[10]、STI[11]、DE-3[12]、DrTI[13]、TKI[14]、WCI[15]、BASI[16])比对可以看出,该残基在其他Kunitz蛋白酶抑制剂家族成员也高度保守(图3)。并且,将CoTI模型和其他Kunitz丝氨酸蛋白酶抑制剂的三维结构重叠比对,也可以看到Trp114在三维结构上高度重叠。
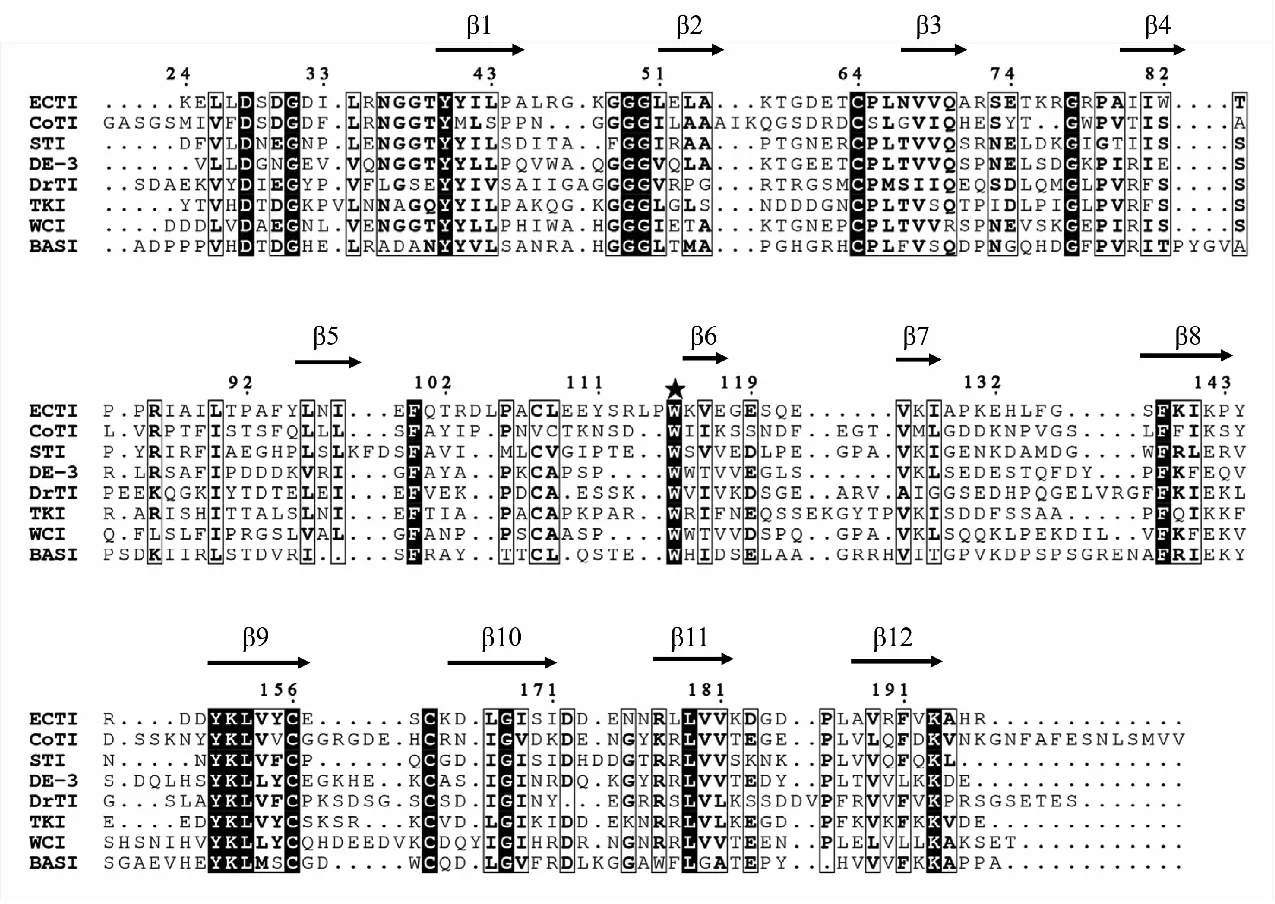
★为保守的W114
通过分析CoTI的W114发现,CoTI中的W114位于LID的β6折叠始端,与Leu98和Ser137形成氢键,其侧链伸向Barrel,与Leu42、Val68、Leu98、Phe100、Val126、Met127、Leu128、Phe139、Leu153和Val179形成疏水作用,从而参与三叶草结构的包装并决定结构的稳定性(图4)。

与W114形成疏水作用的氨基酸用灰色表示;与W114形成氢键的氨基酸用白色表示(Leu98与W114既形成氢键,又有疏水作用);黑色虚线代表氢键
2.4 突变体活性检测
经检测,菜青虫中肠蛋白酶液的比活力为2.286 IU/mg,表明菜青虫中肠中含有胰蛋白酶。图5表明:CoTI对牛胰蛋白酶抑制效果显著,为204.92 UI/mg,而CoTI(W114A)突变体的抑制活力为74.97 UI/mg,相较于野生型CoTI下降了63.4%;CoTI对菜青虫中肠胰蛋白酶也具有明显的抑制活性,为167.728 6 UI/mg。而CoTI(W114A)突变体抑制活性下降了62%,仅为63.92 UI/mg。
2.5 突变体荧光光谱检测
蛋白质中的天然荧光生色团为其芳香族氨基酸(Trp、Tyr、Phe等),其中由色氨酸残基发出的荧光占统治地位。通过对CoTI和突变体进行荧光光谱检测,由荧光光谱图(图6)分析可看出:CoTI(W114A)发生了轻微蓝移(342.8 nm蓝移至340.4 nm),荧光强度由318.2减弱到216.2,减弱了32%,可能原因是114位的色氨酸突变为丙氨酸后,减少了蛋白质中的天然荧光发色团,故荧光强度降低,且发生突变后,疏水腔内相互作用力减小,使得三维结构更加紧凑。

图6 CoTI与突变体的荧光光谱
3 讨论与结论
尽管KPI家族成员的氨基酸序列保守性不高,但却具有相似的三级结构,可能原因是β折叠上的若干疏水氨基酸之间形成的分子间相互作用。通过同源建模方法,获得CoTI的三维模型,发现其三维模型为典型的β-三叶草结构,其中6个形成β-桶,另外6个形成封闭桶上方的盖子,此结构为β-三叶草结构蛋白质的常见基序。分析CoTI疏水核心,发现Trp114在Kunitz家族中是高度保守的,其位于β-桶与盖子的连接点处,不仅与Leu98和Ser137形成氢键,而且疏水侧链伸向β桶内部,与邻近的氨基酸Leu42、Val68、Leu98、Phe100、Val126、Met127、Leu128、Phe139、Leu153和Val179产生疏水作用。W114通过这些相互作用,紧紧地将盖子与桶的顶层缝合在一起,从而参与β-三叶草整体结构的维系并保持结构的稳定性。
向缅等[8]曾做过CoTI(R86D)突变体的表达及活性检测,结果相较于野生型,突变体对胰蛋白酶抑制活性下降了93%,从而证实Arg86为CoTI抑制胰蛋白酶的关键残基,且在发挥抑制作用的过程中起决定性的作用。而本实验通过对CoTI中W114进行单定点突变后发现:无论是对牛胰蛋白酶还是菜青虫中肠胰蛋白酶的抑制活性,突变体较野生型都发生明显下降,但抑制活性下降并没有CoTI(R86D)大,可能原因是Arg86作为CoTI与胰蛋白酶作用的关键残基,对发挥抑制作用至关重要,而W114则主要是作为维持蛋白质三维结构的关键残基,间接的影响抑制作用的发挥。荧光光谱检测发现,W114作为疏水腔内关键的氨基酸,当发生突变后,会使CoTI的整体构象发生改变。我们推测:Trp114残基突变为Ala残基后,原有的氢键与疏水作用随即减弱,甚至消失,会使整体结构趋于不稳定,进而影响抑制中心的构象,导致W114A突变体与靶蛋白酶的结合变弱,从而导致抑制活性的下降。研究证实W114是CoTI疏水核心中的一个关键残基,其在CoTI蛋白的折叠形成过程以及发挥抑制活性中起到了重要的作用,该结果为CoTI的结构功能研究及相关的应用奠定了理论基础。
