基于GAN 异质网络表示学习的疾病关联预测算法
2021-06-18郭梦洁
郭梦洁,熊 贇
(复旦大学计算机科学技术学院上海市数据科学重点实验室,上海 201203)
0 概述
随着各种高通量生物技术的迅速发展,生物学领域产生了海量数据,使研究人员能够收集和研究大量数据,以更好地阐释复杂疾病的潜在生物学机制[1]。科研机构在生物医学数据的研究上取得了重要进展,但是由于利用海量的数据进行生物学实验需要耗费大量的时间和资源,大部分数据在最初的获取和分析后被搁置[2],因此,数据的生成和整合分析数据的能力之间的差距越来越大。
很多疾病关联数据可以表示成网络,其中节点代表生物实体,如疾病、基因等,节点间的边指代它们之间的关系。这些网络往往都包含多种类型的实体和关系,被称作异质网络[3]。疾病或其他生物实体在异质网络中是相似的,则它们有很大可能性存在关联。例如一种miRNA 在一种疾病起关键作用,则很有可能在相似疾病中起到相似的作用[2]。
为充分利用网络中的信息,研究人员采用网络表示学习算法[3],将网络映射到低维向量空间,同时保留原有的网络结构、节点内容等。近年来,兴起了异质网络表示学习算法的研究,一类是基于随机游走采样正负节点的训练,代表性的算法包括Metapath2vec[4]、HeteWalk[2],但它们都依赖合适的元路径[2],元路径的选择需要人工经验,另一类是将异质网络分解成子网络表示学习后进行信息融合,例如PTE[5]、AspEm[6],但在分解和融合过程中容易丢失网络中的重要信息。此外,上述算法都忽视了节点的数据分布,因此学习的向量表示缺乏鲁棒性。
本文提出一种基于生成式对抗网络(Generative Adversarial Network,GAN)[7]的异质网络表示学习算法DisGAN。该算法中的判别器和生成器设计通过异质网络中的关系区分不同关系链接的节点对,一对节点被认为是真实的必须满足基于网络拓扑结构的真实节点被正确的关系链接。DisGAN 算法考虑了网络中的关系以捕获丰富的异质信息,并通过对抗学习得到鲁棒的向量表示,同时为实现关联预测的目标并验证DisGAN 算法性能,本文整合6 个公开数据集构建一个生物异质网络,进行基因-疾病关联预测和miRNA-疾病关联预测。
1 问题定义
异质网络定义为G=(V,E),V 和E 分别代表节点集合和边集合。该网络也关联一个节点类型映射函数ϕ:V →A 和一个边类型映射函数φ:E →R,其中A 和R 分别代表节点类型和边类型集合且|A|+|R|>2[3]。
异质网络表示学习[3]的目标是学习一个映射函数,将网络中每个节点v∊V 映射到一个低维向量空间Rd,其中d≪|V|,尽可能保留网络原有信息。
生成式对抗网络[6]公式定义如下:
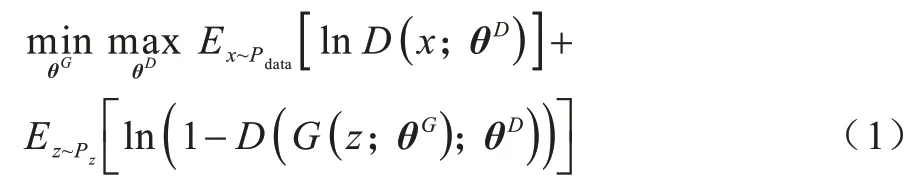
生成器G使用来自预定义分布Pz的噪声z,生成尽可能接近真实数据的伪样本,判别器D旨在区分来自Pdata的真实数据和生成器生成的伪数据,θG、θD表示参数。
2 DisGAN 算法
本节介绍DisGAN 算法,DisGAN 包括判别器Discriminator 和生成器Generator 两部分。网络中真实存在的节点对且通过正确的关系链接是正样本,其他均为负样本,判别器需要进行区分,而生成器需要生成和给定节点通过给定关系相连的伪节点。DisGAN 模型框架如图1 所示。

图1 DisGAN 模型框架Fig.1 Framework of DisGAN model
2.1 DisGAN 中的判别器和生成器
2.1.1 DisGAN 中的判别器
在异质网络中必须区分给定关系下的真实和虚假节点,因此判别器需要评估一对节点在给定关系下的链接性。给定异质网络G 中一个节点i∊V 和关系r∊R,参数为θD的判别器D给出一个样本j是否通过关系r和节点i相连的概率。样本j可以是真实节点,也可以是生成器生成的伪节点。
判别器D公式定义如下:
其 中,vi,vj∊Rd是节点i和j的d维表示向量,Mr∊Rd×d是关系r的关系矩阵,参数θD是判别器D学习的所有节点的表示向量和关系的表示矩阵。
如果样本j是通过关系r和节点i相连的真实节点,判别器给出的概率值应该较高,而对伪样本应该较低。通常,样本j与给定的i和r组成一个三元组
1)通过正确关系链接的真实节点
节点i和j是异质网络G 中的真实节点,并通过真实关系r连接,这样的三元组
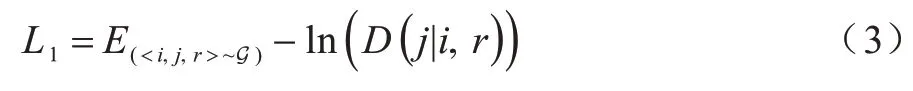
从网络G 中提取上述三元组,即
2)通过错误关系链接的真实节点
异质网络中的节点i和j通过一个错误的关系r′(r′≠r)链接。由于它们的链接性与给定关系r携带的期望语义信息不匹配,因此判别器希望将其判定为负样本:
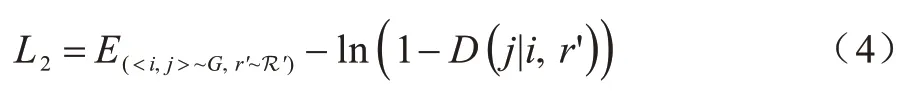
节点对(i,j)从网络G 提取,关系r′从R'=R -r获得。
3)通过正确关系链接的伪节点
给定异质网络中一个节点i和其关系r,然后通过生成器G(i,r)生成节点j'。该三元组
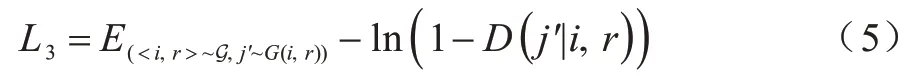
伪节点j'的表示向量是从生成器G学习到的分布中提取的,与生成器G的模型参数θG不同。
4)通过错误关系链接的伪节点
给定节点i和一个i中不存在的关系r*,输入生成器G(i,r*)生成一个伪样本j'。判别器的目标是将该三元组也判定为负:
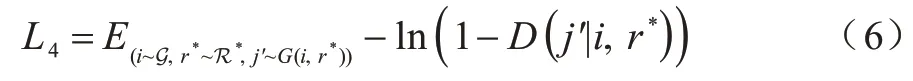
其中,R*代表网络G 的关系集合R 和节点i拥有的关系子集之间的差集。
整合上述4 个部分作为损失函数训练判别器:
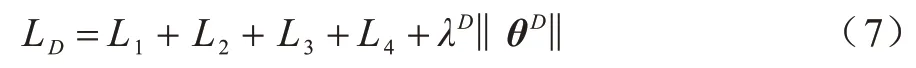
其中,λD‖θD‖是正则化项用来防止过拟合,通过最小化LD来优化判别器参数θD。
2.1.2 DisGAN 中的生成器
生成器同样考虑到网络的异质性,即给定来自异质网络G 的节点i∊V 和关系r∊R,参数为θG的生成器G希望生成尽可能和节点i通过关系r链接的节点。
生成器希望通过生成接近真实节点的伪样本来欺骗判别器,使判别器给伪样本赋予高分:
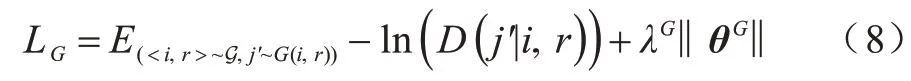
其中,λG‖θG‖是正则化项,通过最小化公式LG训练生成器。
2.2 DisGAN 模型训练与分析
DisGAN 模型使用迭代的数值计算方法[8]进行训练。首先初始化模型参数θG和θD,然后迭代训练判别器和生成器直到模型收敛。DisGAN 模型训练过程如算法1 所示。
算法1DisGAN 模型训练

DisGAN 模型生成器和判别器每次更新主要涉及节点向量和关系矩阵的更新,每轮迭代时间复杂度为O((nG+nD)˙ns˙|V|˙d2),其中,nG和nD是训练次数,ns是样本数目,|V|是节点数目,d是维度。本文将nG、nD、ns和d视为常数,所以DisGAN 每轮迭代的时间复杂度是O(|V|)。
DisGAN 模型的生成器是使用Leaky ReLU[9]激活函数的两层感知机,将最后一层输出当作伪节点无需softmax 计算采样伪节点,所以对于整个网络每轮迭代生成器采样伪节点的时间复杂度为O(|V|),|V|是网络中节点数目;而对每个节点每次softmax计算需要遍历网络中所有节点,因此每轮迭代时间复杂度为O(|V|2),计算代价非常高。
DisGAN 模型中判别器的参数θG是网络节点的表示向量和关系的表示矩阵,通过对抗学习优化模型参数。判别器和生成器在对抗学习过程中迭代训练:首先固定生成器G的参数θG,然后从网络中采样真实节点关系三元组,生成器G对每个给定的节点和关系生成ns个伪节点,最后通过最小化式(7)定义的损失函数优化参数θD从而训练判别器;固定判别器的参数θD,然后同样采样真实样本和伪样本,最后根据式(8)优化生成器参数θG以生成更好的伪节点。上述迭代过程直到模型收敛时停止。
DisGAN 相对于GAN[7]的改进主要在于将其扩展应用到网络表示学习:GAN 仅仅区分真伪节点无法捕获网络节点间的关系信息,而DisGAN 区分不同关系链接的节点对,从而捕获网络的结构和语义信息;GAN 中生成器输入为随机噪声,DisGAN 加上网络中的节点和关系,从而生成和真实节点更相似的伪节点进行训练提升模型表现。
3 实验结果与分析
3.1 实验数据集
本文实验所用数据集如下:
1)基因相互作用网络:从HPRD 数据库[10]中获得的39 240 条记录。
2)miRNA 相似性网络:从MISIM 数据库[11]中提取的56 289 条数据。
3)疾病相似性网络:从MimMiner[12]中提取 的3 162 016 条数据。
4)基因-疾病关联网络:从DisGeNET 数据库[13]中提取的19 714 条记录。
5)基因-miRNA 关联网 络:从miRTarBase 数据库[14]中提取的21 259 条记录。
6)miRNA-疾病关联网络:从文献[15]提供的数据集和miRNet[16]中提取的878 条数据。
通过共同节点链接上述6 个网络来构建一个生物异质网络。
3.2 对比算法
本文的实验对比算法主要包括:
1)HSSVM[17]:基于HeteSim 得分[18]衡量节点相关性,使用监督学习算法进行疾病关联预测。
2)GAN[7]:生成器输入从正态分布中采样的噪声生成伪节点,判别器区分网络节点和生成器产生的伪节点,将网络节点表示作为模型参数训练。
3)DeepWalk[19]:使用随机游走得到节点序列基于skip-gram[20]模型学习表示向量。
4)AspEm[6]:通过将异质网络分解成语义子图,分别学习每个子图中节点向量表示后进行拼接得到最终节点向量表示。
5)HeteWalk[2]:使用元路径和链接权重指导的随机游走并基于异质skip-gram 模型进行表示学习。
3.3 实验结果
本文分别进行基因-疾病关联和miRNA-疾病关联实验。每次实验将已知的关联数据随机划分为训练集和测试集,训练集所占比例(R)从50%变化到90%。在进行测试时,已知的关联作为正样本,随机选择相同数目且相同类型但没有关联的节点对作为负样本,通过算法得到节点表示向量的余弦相似度(归一化后的点积)得分作为预测值。不同算法在不同训练比例下的AUC 得分[21]如表1 和表2 所示。
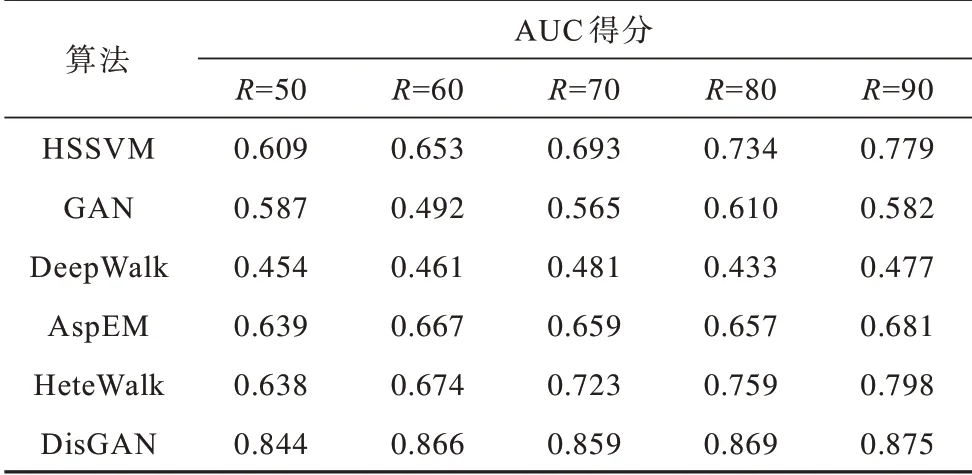
表1 基因-疾病关联预测实验的AUC 得分Table 1 AUC score of gene-disease association prediction experiment

表2 miRNA-疾病关联预测实验的AUC 得分Table 2 AUC score of miRNA-disease association prediction experiment
从表1 和表2 可以发现,DisGAN 算法在两个预测任务所有训练比例上的表现一直都超过所有对比算法。HSSVM 没有采用网络表示学习,只提取沿路径的两个节点之间可访问性的简单特征。GAN 尽管考虑了向量表示的鲁棒性,但是忽视了节点间的关系,没有捕获网络的拓扑结构和语义关系。DeepWalk 表现较差的主要原因是针对同质网络设计的网络表示学习算法,忽视了不同节点和链接类型。AspEm 在网络分解合并过程中可能会丢失一些重要信息。HeteWalk 尽管通过基于元路径的随机游走捕获到网络的异质信息,但是没有学习节点的数据分布,学习到的向量表示鲁棒性不高。在所有对比算法中,AspEm 和HeteWalk 表现较好,说明考虑网络异质性可以提升预测结果。
本文提出的DisGAN 模型超过了所有的对比算法,可以通过对抗学习节点的数据分布,得到更具鲁棒性的表示,能够较好地保留网络结构和异质语义信息。此外,DisGAN 模型在基因-疾病关联预测任务上的表现提升更明显,主要是由于异质网络中基因-疾病关联数据相对更多且数据可能更稀疏或存在噪声,因此需要更具鲁棒性的向量表示。
3.4 异质性分析
本节探究每个算法在处理异质性上的能力。实验中采用三折交叉验证,并去除3.1 节中部分数据集生成了另外两个只包含两种节点类型的子网络。从图2 和图3 可以发现,在只包含两种节点类型的子网络上进行关联预测的AUC 得分更低,整合3.1 节中所有网络数据,构建一个更加复杂的异质网络有明显的益处,尤其是在miRNA-疾病关联预测任务上。这主要是由于miRNA 和疾病之间的已知关联数据更稀少,因此单一网络无法保证预测的可靠性。基因相关的数据集可以帮助建立miRNA 和疾病之间的间接关联,这些关联很有可能被进行关联预测的算法捕获。整合多方面数据可以加深对复杂疾病的理解,结合间接关系信息,进一步提升预测结果。DisGAN 算法能够整合更多来源的异质网络数据。
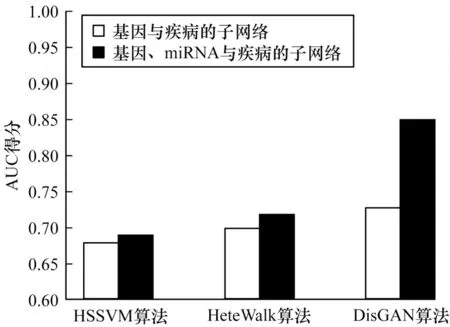
图2 基因-疾病关联预测中不同网络的AUC 得分Fig.2 AUC score on different networks in gene-disease association prediction

图3 miRNA-疾病关联预测中不同网络的AUC 得分Fig.3 AUC score on different networks in miRNA-disease association prediction
4 结束语
本文提出一种基于GAN 的异质网络表示学习算法DisGAN 进行疾病关联预测。DisGAN 中的判别器和生成器都考虑了网络中的关系捕获异质语义信息,通过对抗学习得到鲁棒的向量表示,并在构建的生物异质网络上进行基因-疾病关联预测和miRNA-疾病关联预测来衡量模型性能表现。实验结果证明了DisGAN算法的有效性和优越性。下一步将整合更多生物数据集来提升DisGAN 算法的预测性能。
